CREB结合蛋白(CREB-binding protein,CBP)于1993年被Chrivia等首次报道,因其能够特异性地结合磷酸化的环腺苷酸应答元件结合蛋白(cAMP-response element binding protein,CREB)而得名[1]。CBP作为一种重要的组蛋白乙酰基转移酶(Histone acetyltransferase, HAT)和转录辅助因子,通过对乙酰化以及转录进程的调节,在体内诸多生理过程中发挥着重要作用[2-4],包括细胞分化、细胞增殖、细胞凋亡以及细胞自噬。并通过调节细胞周期,参与多种肿瘤的恶性转化及转移[5]。
研究发现,CBP蛋白的功能与其结构中的多个保守区域密切相关[5],如转录适配器锌指结构域(Transcriptional adapter zinc finger domain,TAZ)、激酶诱导的CREB相互作用结构域(Kinase inducible domain of CREB interacting domain,KIX)、溴域(Bromodomain,BRD)、ZZ型锌指结构域(ZZ-type zinc finger domain,ZZ)和HAT催化结构域(HAT domain)。通过这些功能结构域的介导,CBP可与许多转录激活因子或转录因子相互作用,并乙酰化组蛋白,从而重塑染色质结构,参与基因转录的调控。越来越多的研究证据表明[6-8],CBP基因的突变以及HAT酶活性的异常与多种肿瘤疾病密切相关。然而,CBP参与肿瘤发生发展的作用机制尚不清晰。
生物信息学作为现今生命科学领域的有力工具,利用其相关方法分析蛋白质的信息使之助力相关研究的开展已经成为一种常态[9-10]。本研究基于生物信息学对CBP蛋白的理化性质、亲/疏水性、亚细胞定位、信号肽、跨膜区域、二级结构、三级结构进行分析并构建CBP蛋白相互作用网络与分子进化树,为CBP参与恶性肿瘤发生发展调控机制的深入研究奠定基础。
1 材料和方法 1.1 材料自UniProt网站下载人(Homo sapiens,Q92793)、黑猩猩(Pan troglodytes,K7DBC8)、恒河猴(Macaca mulatta,F6RRA8)、牛(Bos taurus,F1MD32)、马(Equus caballus,F6PZC1)、狗(Canis familiaris,F1PY87)、小型棕褐蝠(Myotis lucifugus,G1NVG2)、小鼠(Mus musculus,P45481)、大鼠(Rattus norvegicus,Q6JHU9)、爪蟾(Xenopus laevis,Q5U248)、斑马鱼(Danio rerio,A0A2R8Q4T2)、果蝇(Drosophila melanogaster,O01368)、秀丽隐杆线虫(Caenorhabditis elegans,P34545)的CBP蛋白序列。
1.2 方法运用生物信息学相关数据库及分析软件对人CBP蛋白有关信息进行分析与预测,各数据库及分析软件信息见表 1。
| 表 1 本文应用的数据库及分析软件 Table 1 Database and software for protein analysis |
采用ProtParam工具对人CBP蛋白的理化性质进行分析,结果显示人CBP蛋白共有2 442个氨基酸残基,总分子式为C11451H18225N3381O3590S145,分子质量为265 350.87,理论等电点为8.83。在构成CBP的氨基酸中,脯氨酸(Pro,276个)、谷氨酰胺(Gln,268个)以及丝氨酸(Ser,218个)的含量较高,并且带负电(Asp+Glu)和正电(Arg+Lys)氨基酸残基的总数分别是183和214。此外,其不稳定系数、脂肪系数和平均总亲水值分别为65.91、60.95和-0.690,提示人CBP为不稳定亲水蛋白质。
2.2 人CBP蛋白的同源性比较分析HAT结构域是CBP行使乙酰化催化功能的区域,该区域的保守氨基酸可能与其酶活性密切相关。CBP蛋白的HAT结构域氨基酸的多序列比对分析(见图 1)显示,人CBP蛋白与黑猩猩、恒河猴、牛、马、狗、小型棕褐蝠、小鼠、大鼠、爪蟾、斑马鱼、果蝇以及秀丽隐杆线虫中的同源蛋白相似度分别为99.96%、99.47%、94.48%、95.83%、93.38%、94.13%、95.26%、95.71%、74.31%、69.76%、68.83%、48.71%。HAT结构域的保守氨基酸集中于1 394位~1 400位、1 407位~1 415位、1 428位~1 440位、1 449~1 457位、1 470位~1 509位、1 537位~1 548位、1 626位~1 633位、1 659位~1 665位、1 677位~1 683位等区段。另外,CBP蛋白同源进化树分析结果(见图 2),亲缘关系与人类最为接近的是黑猩猩和恒河猴,其次为牛、马、狗等其它哺乳类动物,而果蝇、线虫与人类的亲缘关系较远。
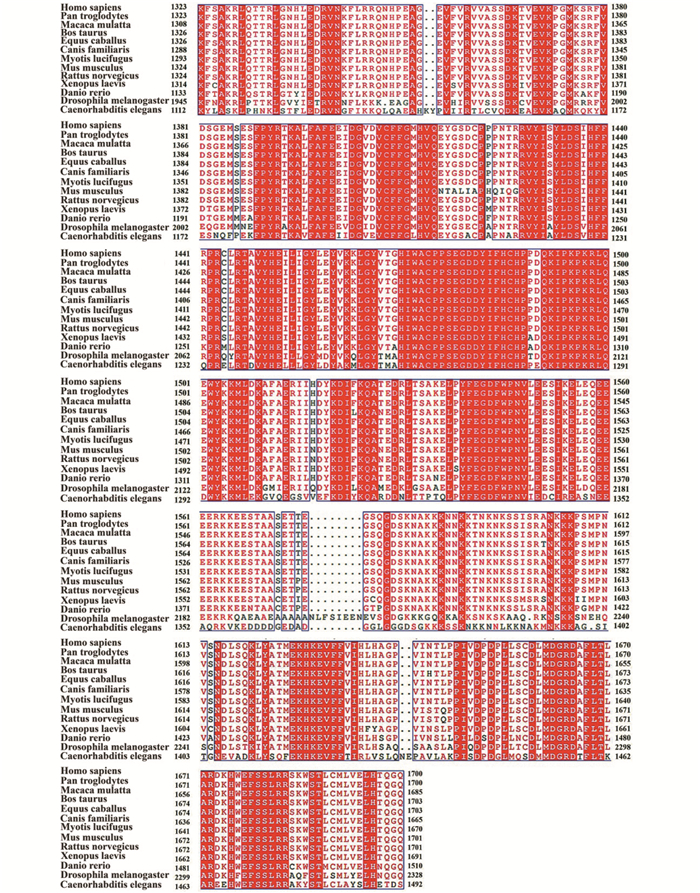
|
图 1 不同物种CBP蛋白HAT结构域多序列比对 Figure 1 Multiple sequence alignment of HAT domain of CBP in different species |
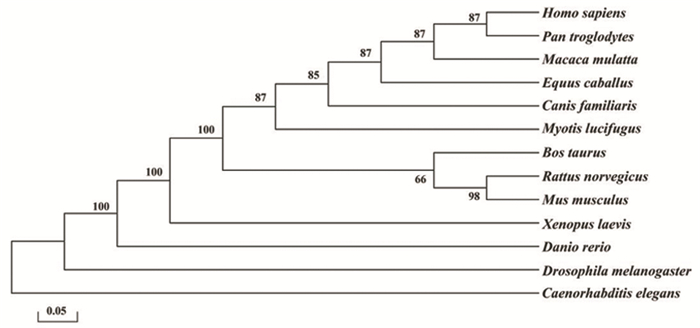
|
图 2 CBP蛋白HAT结构域同源进化树 Figure 2 Homology evolutionary tree of CBP based on HAT domain |
通过在线分析软件ProtScale的Hphob/Kyte & Doolittle算法分析人CBP蛋白的亲疏水性(见图 3)。人CBP蛋白中位于负值区的氨基酸占77.71%(1 980/2 432),其总分值为-1 944.087,亲水性最强的位点为1 561位、1 562位的谷氨酸以及1 563位的精氨酸,其分值为-3.7;位于正值区的氨基酸占21.8%(532/2 432),其总分值为254.966,疏水性最强的位点为1 635位的异亮氨酸,其分值为1.967。预测结果表明,人CBP蛋白的大部分氨基酸处于亲水区,故推测该蛋白为亲水蛋白质。
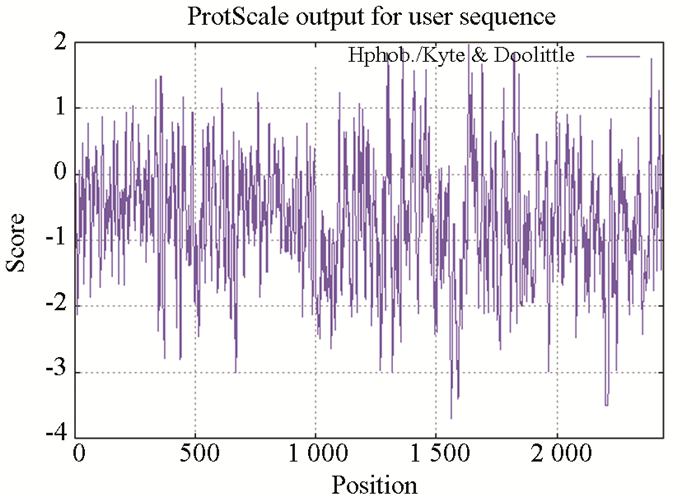
|
图 3 人CBP蛋白亲/疏水性分析 Figure 3 Analysis of CBP hydrophility/hydrophobicity in human |
通过在线分析软件PSORTⅡ对人CBP蛋白的亚细胞定位进行预测,结果显示人CBP蛋白定位于细胞核、细胞质、细胞膜上的概率分别为82.6%、8.7%、8.7%,表明人CBP蛋白存在于细胞核内的概率较大。
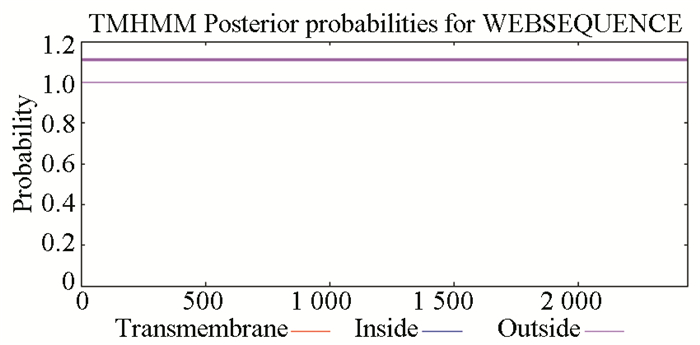
2.5 人CBP蛋白的信号肽及跨膜区域分析通过在线分析软件SignalP5.0[11]对人CBP蛋白的信号肽序列进行分析(见图 4),其中OTHER的数值为0.999 6,SP的数值为0.000 4,表明人CBP蛋白不存在信号肽序列,可以推测人CBP蛋白不是分泌蛋白。进一步利用在线分析软件TMHMM Server v.2.0对人CBP蛋白的跨膜结构进行分析(见图 5),表明人CBP蛋白不存在跨膜区域,提示人CBP蛋白可能为非跨膜蛋白质。
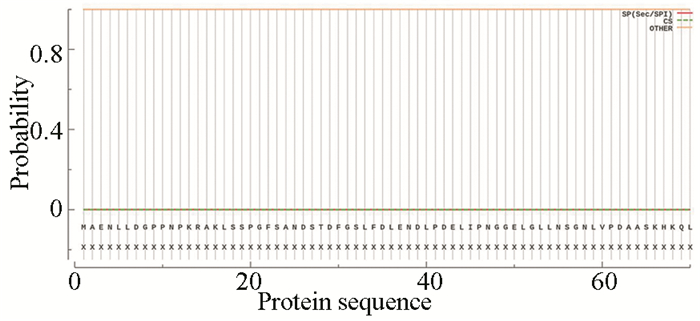
|
图 4 人CBP蛋白信号肽序列预测 Figure 4 Signal peptide prediction of CBP in human |
|
图 5 人CBP蛋白跨膜区域预测 Figure 5 Transmembrane structure prediction of CBP in human |
蛋白质序列二级结构预测软件SMOPA的分析结果(见图 6)显示,人CBP蛋白中α-螺旋、β-折叠、β-转角和无规卷曲所占比例分别为30.47%、9.95%、6.43%和53.15%,其中无规卷曲和α-螺旋是人CBP蛋白二级结构中数量较多的结构。
|
图 6 人CBP蛋白二级结构预测 Figure 6 Secondary structure prediction of CBP in human 注:蓝色,α-螺旋;紫色,无规卷曲;红色,β-折叠;绿色,β-转角. |
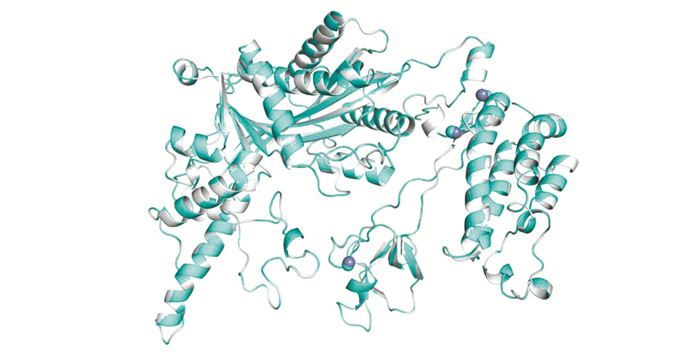
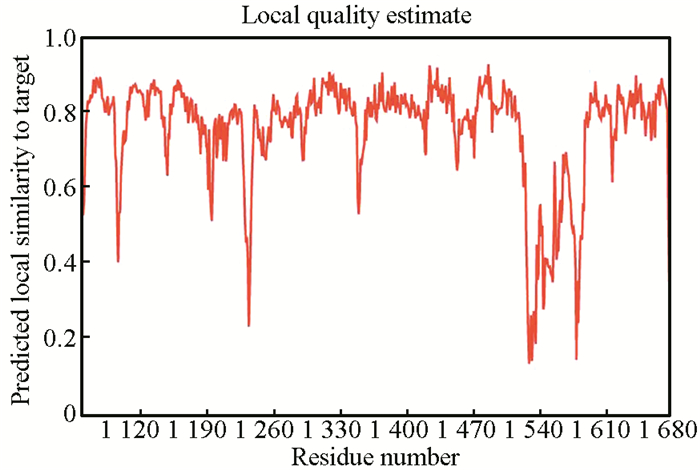
将人CBP蛋白氨基酸序列提交至SWISS-MODEL[12]中,选取序列一致性为87.05%,相似性为59%,包含整个HAT结构域的模板(Homo sapiens,PDB ID:6gyr)进行同源建模(见图 7)。根据相似波形图(见图 8)显示,大部分氨基酸残基得分大于0.6,表明该模型具有一定的可靠性。
|
图 7 人CBP蛋白优化前后的三级结构比较图 Figure 7 Comparison of tertiary structure of human CBP before and after optimization 注:白色为优化前结构;蓝色为优化后结构. |
|
图 8 人CBP蛋白三级结构与模板相似波形图 Figure 8 Waveform of similarity between tertiary structure of human CBP and template |
为提高酶活性中心关键氨基酸残基预测的准确性,采用WHAT-IF优化该蛋白三级结构[13],并利用拉式图分析网站The Structure Analysis and Verification Server对优化前后模型进行分析以评估该模型的可靠度。经优化后,落在拉氏图的红色以及黄色区域外的氨基酸残基由2%下降到了0.7%,而落在不合理区域氨基酸的数量明显减少,表明优化后模型的稳定性和可信度均有较大提高(见图 9)。

|
图 9 人CBP蛋白HAT结构域三级结构优化前后拉氏图 Figure 9 Ramachandran plot of tertiary structure of HAT domain of human CBP before and after optimization |
进一步结合序列比对结果(见图 1)分析该模型,发现HAT结构域保守序列共同组成了一个圆形的凹陷结构,推测该结构内的Tyr1433、Leu1434、Asp1435、Arg1664为酶活性中心的关键氨基酸位点。
2.8 人CBP相互作用蛋白质预测及其功能富集分析采用String 11.0蛋白质相互作用预测软件[14]分析与人CBP互作的蛋白网络结果(见图 10)所示,相互作用的蛋白质分别为CREB1、NCOA3、TP53、KAT2B、RELA/P65、SMAD3、MYB、PPARG、CTNNB1、HTT、STAT1。进一步采用Gene Ontology功能富集分析,预测结果(见图 11)显示,人CBP蛋白主要参与转录调控、细胞分化、组织发育、信号转导及细胞凋亡等生物学过程,具有与转录因子和染色质结合、转录辅激活因子和乙酰转移酶等生物学功能,而细胞组分分析显示为染色质、核质及染色体等。KEGG通路富集分析(见图 12)显示,人CBP蛋白主要涉及的通路,包括甲状腺激素和Wnt信号通路、细胞周期、及病毒感染和肿瘤等疾病。上述预测结果表明,人CBP蛋白能够通过与转录因子和转录辅激活因子在内的多种转录调节因子相结合,从而调控转录,参与细胞分化、细胞凋亡、组织发育及细胞周期等生物学过程。
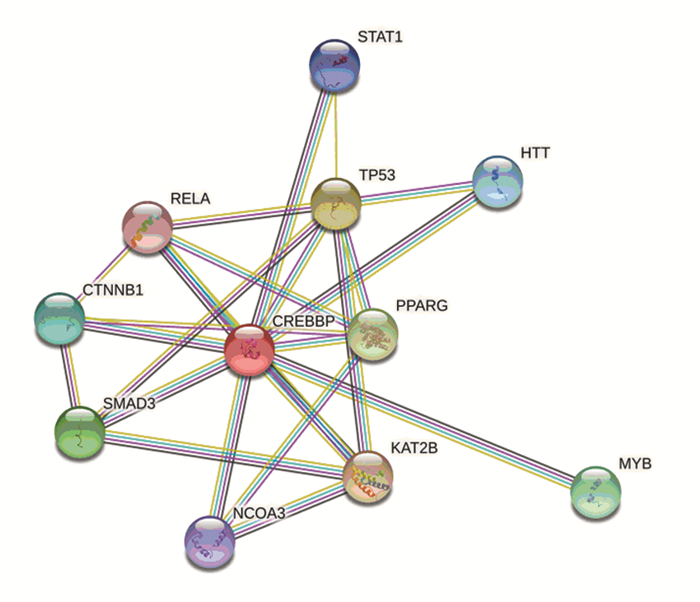
|
图 10 人CBP蛋白相互作用网络 Figure 10 Protein-protein interaction network of CBP in human |
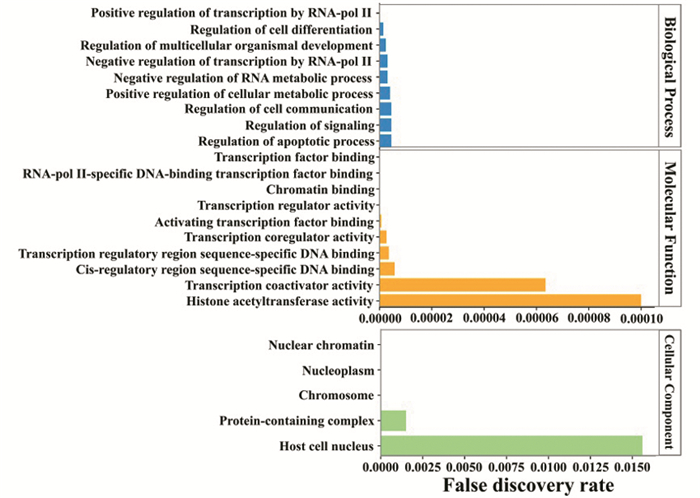
|
图 11 CBP蛋白GO功能富集分析 Figure 11 GO functional enrichment of CBP in human |
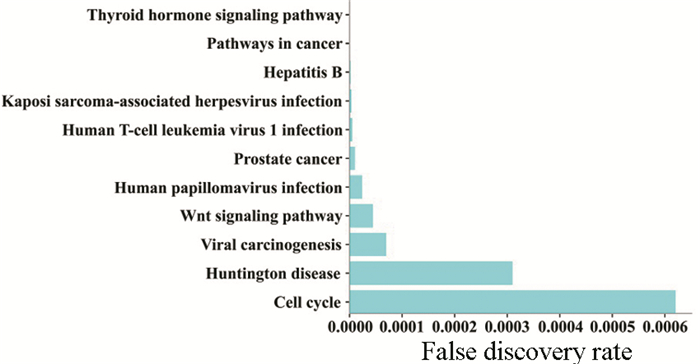
|
图 12 CBP蛋白KEGG通路富集分析 Figure 12 KEGG pathway enrichment analysis for human CBP |
CBP蛋白具有乙酰基转移酶的活性,能使组蛋白N-端赖氨酸残基乙酰化,从而改变染色质结构,参与转录调控,在细胞分化、增殖、凋亡和自噬等众多生理过程中发挥着重要作用。研究显示CBP主要通过以下三种方式调节转录:1)作为转录辅激活因子连接特异性转录因子和基本转录元件[15]。2)作为脚手架蛋白在转录时结合多种转录辅激活因子,形成转录辅激活因子复合物[6],以此促进转录辅激活因子-转录辅激活因子以及转录辅激活因子-DNA之间的相互作用。3)作为组蛋白/非组蛋白乙酰基转移酶分别乙酰化四种核心组蛋白以及包括转录因子在内的一系列非组蛋白[7, 16]。因此,在CBP的作用下,数量有限的转录激活因子以及转录辅因子就可以引起有效的应答反应。目前发现CBP基因的突变存在于众多恶性肿瘤患者当中,结合CBP的生理功能,人们推测CBP基因可能是一种肿瘤抑制基因[8]。然而,CBP参与恶性肿瘤发生发展的分子机制尚不清晰,仍有待深入研究。
本研究利用生物信息学方法[17]对人CBP蛋白结构和功能进行了分析和预测。亚细胞定位分析提示,人CBP蛋白是一种位于核内,无信号肽和跨膜区域的亲水蛋白质,这与大量研究报道其为细胞核蛋白质相一致[6-8, 15, 16, 18]。蛋白质二级结构分析显示,CBP蛋白以无规卷曲和α-螺旋居多。另外,CBP蛋白HAT结构域的氨基酸序列比对和三级结构分析表明,该结构域在进化上高度保守,并且其保守氨基酸形成一圆形凹陷结构,推测为底物结合区域,其中Tyr1433、Leu1434、Asp1435、Arg1664可能与酶活性密切相关。有研究报道[19],无规卷曲有利于CBP蛋白上的赖氨酸残基与HAT结构域结合,从而催化自乙酰化发生,增强自身的乙酰转移酶活性。而CBP蛋白结构中的溴域作为组蛋白乙酰化的“读码器”,对于CBP蛋白识别并结合乙酰化组蛋白,及调控靶基因转录功能的发挥都有着重要作用[20]。有研究表明[20-21],CBP蛋白的溴域含有大量的α-螺旋和无规卷曲,并进一步形成由柔性环连接的4个α-螺旋组成的左旋束结构,可促进CBP识别并结合乙酰化的组蛋白。
蛋白互作网络预测分析显示,人CBP蛋白可与CREB1、NCOA3、TP53、KAT2B、RELA/P65、SMAD3、MYB、PPARG、CTNNB1、HTT、STAT1等蛋白质发生相互作用。进一步分析发现,这些互作蛋白主要为转录因子或转录辅激活因子,这与CBP蛋白具有调控转录的生物学功能相吻合。另外,GO功能注释和KEGG通路富集分析提示,人CBP蛋白主要参与转录调控、细胞分化、组织发育、信号转导及细胞凋亡等生物学过程,并与肿瘤、神经退行性疾病和感染等疾病的发生发展密切相关。研究发现[20, 22],CBP蛋白可通过其HAT活性调控转录,参与细胞周期进展相关的过程,如细胞分化、细胞增殖、细胞凋亡、细胞周期以及DNA损伤修复,并与多种肿瘤疾病密切相关。然而,目前CBP的HAT结构域参与乙酰化调控的分子机制并不清楚,本研究关于CBP蛋白结构和功能的分析是基于生物信息学推测所得,后续还需要结合试验,对其相互作用的蛋白质及酶活性的关键氨基酸残基进行验证分析,从而阐释可能的分子机制。
4 结论通过对人CBP蛋白的理化性质、保守性、亚细胞定位、信号肽、跨膜区域、二级结构、三级结构以及相互作用蛋白质进行分析和预测,研究结果为深入探究人CBP蛋白在肿瘤恶性化进程中的作用机制提供了理论依据。
| [1] |
CHRIVIA J C, KWOK R P, LAMB N, et al. Phosphorylated CREB binds specifically to the nuclear protein CBP[J]. Nature, 1993, 365(6449): 855-859. DOI:10.1038/365855a0 (  0) 0) |
| [2] |
MEDRANO-FERNANDEZ A, DELGADO-GARCIA J M, DEL-BLANCO B, et al. The epigenetic factor CBP is required for the differentiation and function of medial ganglionic eminence-derived interneurons[J]. Molecular Neurobiology, 2019, 56(6): 4440-4454. DOI:10.1007/s12035-018-1382-4 (  0) 0) |
| [3] |
WONG C K, WADE-VALLANCE A K, LUCIANI D S, et al. The p300 and CBP transcriptional coactivators are required for β-cell and α-cell proliferation[J]. Diabetes, 2018, 67(3): 412-422. DOI:10.2337/db17-0237 (  0) 0) |
| [4] |
XU T J, LIU Y, LI P, et al. Insulin in combination with selenium inhibits HG/Pal-induced cardiomyocyte apoptosis by Cbl-b regulating p38MAPK/CBP/Ku70 pathway[J]. European Review for Medical and Pharmacological Sciences, 2016, 20(15): 3297-3303. (  0) 0) |
| [5] |
ZHANG X F, WEI B F, ZHANG X, et al. Current development of CBP/p300 inhibitors in the last decade[J]. European Journal of Medicinal Chemistry, 2021, 209: 112861. DOI:10.1016/j.ejmech.2020.112861 (  0) 0) |
| [6] |
KIM M S, YOO N J, LEE S H. Expressional and mutational analysis of CREBBP gene in gastric and colorectal cancers with microsatellite instability[J]. Pathology & Oncology Research, 2014, 20(1): 221-222. DOI:10.1007/s12253-013-9668-3 (  0) 0) |
| [7] |
PARK S, STANFIELD R L, MARTINEZ-YAMOUT M A, et al. Role of the CBP catalytic core in intramolecular SUMOylation and control of histone H3 acetylation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(27): e5335-e5342. DOI:10.1073/pnas.1703105114 (  0) 0) |
| [8] |
ZHANG J, VLASEVSKA S, WELLS V A, et al. The CREBBP acetyltransferase is a haploinsufficient tumor suppressor in B-cell lymphoma[J]. Cancer Discovery, 2017, 7(3): 322-337. DOI:10.1158/2159-8290.CD-16-1417 (  0) 0) |
| [9] |
张献, 彭涛, 张耀, 等. 氨基甲酸乙酯水解酶的家族生物信息学分析[J]. 生物信息学, 2022, 20(1): 64-74. ZHANG Xian, PENG Tao, ZHANG Yao, et al. Bioinformatics analysis of urethane hydrolase family[J]. Chinese Journal of Bioinformatics, 2022, 20(1): 64-74. DOI:10.12113/202011002 (  0) 0) |
| [10] |
黄劲, 王洁, 牛雪可, 等. 结核分枝杆菌pyrF基因的生物信息学分析[J]. 中国病原生物学杂志, 2019, 14(2): 125-130+136. HUANG Jin, WANG Jie, NIU Xueke, et al. Bioinformatics analysis of the pyrF gene encoding protein orotidine 5'-monophosphate decarboxylase in Mycobacterium tuberculosis[J]. Journal of Pathogen Biology, 2019, 14(2): 125-130+136. DOI:10.13350/j.cjpb.190201 (  0) 0) |
| [11] |
ALMAGRO J J, TSIRIGOS K D, SØNDERBY C K, et al. SignalP 5.0 improves signal peptide predictions using deep neural networks[J]. Nature Biotechnology, 2019, 37(4): 420-423. DOI:10.1038/s41587-019-0036-z (  0) 0) |
| [12] |
BIENERT S, WATERHOUSE A, DE BEER T A P, et al. The SWISS-MODEL Repository-new features and functionality[J]. Nucleic Acids Research, 2017, 45(D1): D313-d319. DOI:10.1093/nar/gkw1132 (  0) 0) |
| [13] |
LANGE J, BAAKMAN C, PISTORIUS A, et al. Facilities that make the PDB data collection more powerful[J]. Protein Science, 2020, 29(1): 330-344. DOI:10.1002/pro.3788 (  0) 0) |
| [14] |
SZKLARCZYK D, MORRIS J H, COOK H, et al. The STRING database in 2017: quality-controlled protein-protein association networks, made broadly accessible[J]. Nucleic Acids Research, 2017, 45: D362-D368. DOI:10.1093/nar/gkw937 (  0) 0) |
| [15] |
WANG L, ZHANG L, GONG X D, et al. PP-1β and PP-2Aα modulate cAMP response element-binding protein (CREB) functions in aging control and stress response through de-regulation of αB-crystallin gene and p300-p53 signaling axis[J]. Aging Cell, 2021, 20(9): e13458. DOI:10.1111/acel.13458 (  0) 0) |
| [16] |
WEINERT B T, NARITA T, SATPATHY S, et al. Time-resolved analysis reveals rapid dynamics and broad scope of the CBP/p300 acetylome[J]. Cell, 2018, 174(1): 231-244.e12. DOI:10.1016/j.cell.2018.04.033 (  0) 0) |
| [17] |
吴小霞, 许天委, 李国寅. 小鼠APOBEC3的结构与功能分析[J]. 生物信息学, 2021, 19(1): 66-74. WU Xiaoxia, XU Tianwei, LI Guoyin. Structural and functional analysis of mouse APOBEC3[J]. Chinese Journal of Bioinformatics, 2021, 19(1): 66-74. DOI:10.12113/202005006 (  0) 0) |
| [18] |
WADDELL A R, HUANG H, LIAO D, et al. CBP/p300: critical co-activators for nuclear steroid hormone receptors and emerging therapeutic targets in prostate and breast cancers[J]. Cancers (Basel), 2021, 13(12): 2872. DOI:10.3390/cancers13122872 (  0) 0) |
| [19] |
LASKO L M, JAKOB C G, EDALJI R P, et al. Discovery of a selective catalytic p300/CBP inhibitor that targets lineage-specific tumours[J]. Nature, 2017, 550(7674): 128-132. DOI:10.1038/nature24028 (  0) 0) |
| [20] |
LEE S Y, KIM J J, MILLER K M. Bromodomain proteins: protectors against endogenous DNA damage and facilitators of genome integrity[J]. Experiment and Molecular Medicine, 2021, 53(9): 1268-1277. DOI:10.1038/s12276-021-00673-0 (  0) 0) |
| [21] |
ZHANG F C, SUN Z Y, LIAO L P, et al. Discovery of novel CBP bromodomain inhibitors through TR-FRET-based high-throughput screening[J]. Acta Pharmacologica Sinica, 2020, 41(2): 286-292. DOI:10.1038/s41401-019-0256-2 (  0) 0) |
| [22] |
NARITA T, WEINERT B T, CHOUDHARY C. Functions and mechanisms of non-histone protein acetylation[J]. Nature Reviews Molecular Cell Biology, 2019, 20(3): 156-174. DOI:10.1038/s41580-018-0081-3 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


