环形RNA是一类特殊的呈封闭环状结构的RNA分子,其没有5’端帽子结构和3’端PolyA尾巴,由前体RNA(pre-mRNA)通过反向剪接形成。近来研究发现环形RNA也可以编码蛋白,且估计大约10%的环形RNA具有蛋白编码能力。尽管仅有少数编码蛋白的环形RNA被发现,但这些环形RNA编码的小肽在多个生物过程中发挥着重要的作用,且与疾病密切关联。目前尚有大量的环形RNA等待着人们去发现,因此本文对环形RNA编码蛋白的相关研究进行了综述,并对目前现有的可用于编码蛋白环形RNA识别的相关生物信息学工具和方法进行了总结。环形RNA通过编码蛋白这一机制,发挥了与疾病相关的一些作用。因此对于环形RNA编码蛋白的研究具有重要的意义。
1 环形RNA编码蛋白潜能的发现1976年人们首次在病毒中观察到环形RNA的存在[1]。1979年研究人员在电子显微镜下观察到真核细胞中的环形RNA[2]。随着二代测序技术的快速发展和生物信息学工具的开发,环形RNA被检测到广泛存在于真核生物中。在人类和小鼠脑组织分别检测到65 731和15 849个环形RNA [3]。2015年,Wang等人通过将内部核糖体进入位点(Internal Ribosome Entry Site, IRES)人工插入环形RNA的实验方法,发现这类人工构建的环形RNA可以翻译[4],这引起了研究者们的注意。
一直以来,环形RNA被认为不能编码蛋白质,是一类新的非编码RNA,通过竞争性结合microRNA调控基因表达。然而,人工构建的可编码蛋白环形RNA的出现使得人们开始怀疑是否存在内源性的可编码蛋白环形RNA。2015年Chen等人对人类环形RNA的编码能力进行了分析,发现相当多的环形RNA转录本具有蛋白质编码潜能,并通过质谱数据从中鉴定出21个编码蛋白的环形RNA。在2017年,Ivano等人通过northern blot、质谱技术等方法验证了环形RNA(circ-ZNF609)能够编码蛋白质,从而调控肌细胞增殖,并且验证了该环形RNA的非翻译区(Un-translated region, UTR)存在IRES结构[5]。Yang等人通过抗体检测、质谱结果发现了Circ-FBXW7能够编码与恶性胶质瘤发病机制相关的蛋白FBXW7-185aa[6]。Zhang等人验证了circ-SHPRH能够编码新型蛋白质SHPRH-146aa,该蛋白能够抑制神经胶质瘤的发生[7]。2018年Zhang等人还验证了circ-PINT能够编码新型蛋白PINT87aa,抑制多种癌基因转录延伸[8]。2019年Liang等人发现了circβ-catenin能够编码全新蛋白β-Catenin-370aa,调控Wnt/β连环蛋白信号通路[9]。Heesch等人在心脏组织中发现40种环形RNA能被翻译,其中6个在质谱检测中得到验证[10]。
综合以上已经发表的验证环形RNA编码蛋白的文章,验证的过程大致如下:预测环形RNA的开放阅读框(Open reading frame,ORF),包括了跨越接头位置(Junction site)的情况,具有开放阅读框的环形RNA则有编码蛋白质的潜在能力; 根据生物信息学的方法预测环形RNA中是否包含内部核糖体进入位点(Internal ribosome entry site,IRES)结构,如果有,进一步通过双顺反子实验验证IRES结构的活性; 如果预测的开放阅读框跨越反向剪接位点,预测其可能编码的蛋白质序列,通过质谱检测(MS)技术验证是否有环形RNA翻译形成的特定小肽片段,如果有则证实该环形RNA编码蛋白质。
2 环形RNA编码蛋白相关调控机制在基因组中,mRNA编码区的起始密码子必然在终止密码子之前。然而,对于环形RNA,因其闭环结构,其编码区的起始密码子在基因组中的位置可能在终止密码子之后,且编码区的长度可能大于环形RNA自身。此外,因闭环结构,环形RNA不含5’端帽子结构,因此无法依赖帽子结构招募核糖体起始翻译蛋白,而只能通过非帽依赖的内部翻译起始机制编码蛋白。IRES元件作为一段RNA内部序列,可直接招募核糖体结合,从RNA内部起始翻译蛋白,因此IRES元件可视为环形RNA编码蛋白的前提条件之一。m6A甲基化作为RNA中丰度最高的转录后修饰,其所在的短序列可作为IRES元件驱动环形RNA翻译蛋白,由此m6A甲基化也可视作环形RNA编码蛋白的标志。因此,以下将从编码区域的识别及翻译起始驱动方面介绍环形RNA编码蛋白的相关调控机制。
2.1 环形RNA中编码蛋白区域的识别机制环形RNA编码蛋白的先决条件是必须要有一定长度的开放阅读框(ORF)。开放阅读框是指从起始密码子(AUG)开始,结束于终止密码子(UAA, UAG, UGA)的一段连续碱基序列。由于密码子的读写起始位置不同,RNA序列可能按三种开放阅读框阅读和翻译。核糖体从起始密码子开始翻译,沿着RNA序列合成多肽链并不断延伸,遇到终止密码子翻译终止。然而,对于环形RNA这一呈现环状的特殊RNA,情况有所不同。不同于线性mRNA,环形RNA的开放阅读框可能跨越反向剪接位点(Junction site),开放阅读框可能绕环形RNA一圈或者两圈,长度甚至大于环形RNA本身。因此具有开放阅读框的环形RNA才可能编码蛋白质。
2.2 内部核糖体进入位点(IRES)介导的环形RNA内部翻译起始机制RNA的翻译起始可分为帽依赖翻译和非帽依赖翻译两种方式,其中帽依赖翻译主要依靠5’端的帽子结构招募起始因子复合物和核糖体亚基,在起始因子的辅助下,将RNA与40 S核糖体亚基结合,驱动翻译起始。而在非帽依赖翻译机制中,IRES介导的内部翻译起始占了很大一部分,其在反式作用因子的作用下直接招募40 S核糖体亚基与RNA结合,进而启动翻译过程。因此,尽管环形RNA是一个闭环结构,没有5’帽子结构,但环形RNA可以通过内部的IRES元件起始蛋白质翻译过程。
编码蛋白质的环形RNA内部大多都含有IRES元件,并且实验表明IRES确实驱动了环形RNA的翻译起始[5-9]。IRES实验验证的主要手段是通过双顺反子实验,通常使用荧光素酶质粒作为载体,在其5’UTR区插入待测序列,如果下游荧光素酶表达提升,则证明待测序列具有IRES活性。IRES元件不仅在5’非翻译区(5’Un-translated region, UTR)有分布,在CDS区及3’UTR区同样存在IRES元件[11]。并且,研究发现大约10%的人类mRNA的5’UTR区含有IRES元件。环形RNA大多来源于mRNA的外显子,因此有足够理由相信相当一部分的环形RNA含有IRES元件。一般来说,具有IRES元件结构的环形RNA,我们更相信其具有编码蛋白质的能力,因为IRES元件能够招募核糖体亚基与其结合从而启动翻译。
2.3 m6A(N6)甲基化修饰驱动的环形RNA翻译机制N6甲基化修饰促进环形RNA的翻译起始。N6甲基化修饰,即腺苷酸6号N发生甲基化修饰事件,又称m6A。m6A是真核细胞中最广泛的一种RNA甲基化修饰[12-13]。该修饰最可能出现的共有基序(Consensus motif)是“RRm6ACH”,其中R是A或G,H是A, C或U[14-15]。m6A在3’非编码区(UTR)通过与YTHDF1蛋白结合,提高翻译效率[16]。然而,在5’UTR区,m6A通过YTHDF2相关作用机制,促进非帽依赖翻译起始[17-18]。YTHDF3还能与核糖体蛋白相互作用促进mRNA的翻译[19]。
线性mRNA由核糖体扫描起始翻译,然而环形RNA的翻译起始机制完全不同。真核生物常规蛋白翻译起始由eIF4复合物开始,其中eIF4E结合5’帽子结构,eIF4G提供翻译起始复合物组装所需支架,募集核糖体后起始翻译过程。研究人员通过一系列实验表明eIF4G2与eIF3A结合位点与m6A修饰位点重合较高[20]。Yang等人通过circRNA-m6A-seq(m6A抗体免疫共沉淀反应深度测序)的实验手段证实内源性环形RNA中含有大量的m6A修饰位点,经过序列特征分析表明,m6A修饰经常出现在eIF4G2结合位点上游,说明了两者可能存在协同调控翻译活动的作用。基于m6A抗体测序组和全部环形RNA数量推理分析, 大约有13%环形RNA存在m6A修饰事件。因此,具有m6A修饰的环形RNA更有可能具有翻译能力,能够编码蛋白质。
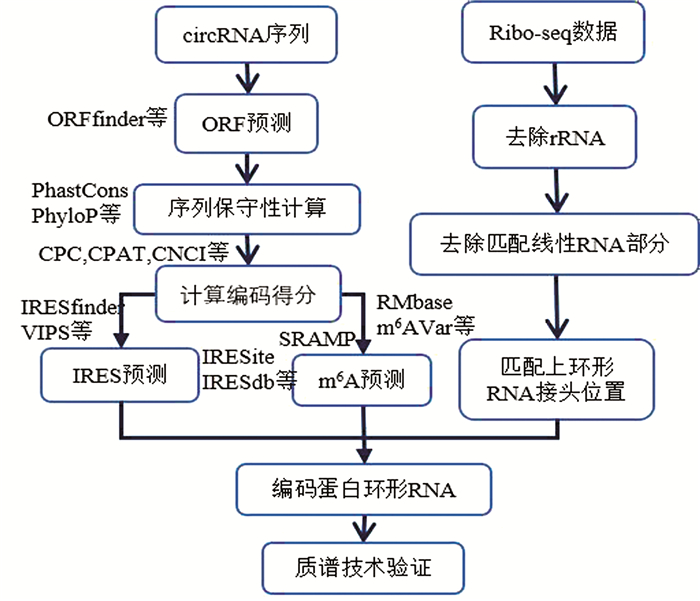
3 环形RNA编码蛋白的相关生物信息学预测工具 3.1 编码蛋白环形RNA的预测流程预测编码蛋白环形RNA的流程大致如下:(1)首先预测环形RNA的开放阅读框,具有开放阅读框的环形RNA则有编码蛋白质的潜在能力;(2)对开放阅读框的序列保守性进行计算;(3)通过一些现有工具计算编码得分;(4)根据生物信息学的方法和工具预测环形RNA中是否包含IRES结构;(5)接着进行m6A修饰的预测;(6)结合ribo-seq数据,过滤rRNA读段,去除匹配上线性RNA的部分,若环形RNA接头部分匹配上ribo-seq数据,更有理由相信环形RNA进行了翻译;(7)如果预测的开放阅读框跨越反向剪接位点,预测其可能编码的氨基酸序列,通过质谱检测(MS)技术验证是否有环形RNA翻译形成的特定小肽片段,如果有则证实该环形RNA确实能够编码小肽。流程图见图 1。
|
图 1 编码蛋白环形RNA预测流程图 Figure 1 Flow chart pipeline for predicting protein-coding circRNAs |
ORF预测软件主要有ORFfinder, ORF Investigator, ORF Predictor和ORFik。ORFfinder是一个图形分析工具,可以查找用户输入序列中大于一定长度的所有开放阅读框,或者在已有数据库中存在的序列,并通过BLAST服务器在数据库中检索氨基酸序列。ORF Investigator是基于perl语言编写的程序,能够有效地找到相应氨基酸序列的ORF并将它们转换成它们的单字母氨基酸代码,并在序列中提供它们的位置,还能在序列间进行全局比对,检测单核苷酸多态性。ORF Predictor使用两种不同ORF定义的组合,它搜索从起始密码子开始到终止密码子结束的延伸。作为另外的标准,它在5'非翻译区(UTR)中搜索终止密码子。ORFik是Bioconductor中的R包,用于寻找开放阅读框架并使用新一代测序技术来证明ORF的合理性。然而,环形RNA呈闭合环状结构,开放阅读框能够跨越接头位置,绕环一周以上,所以这些工具都不太适合环形RNA开放阅读框的预测,需要自编程序实现。
3.3 IRES预测工具及相关数据库目前预测IRES元件的工具主要有IRSS[21]、VIPS[22], IRESpred[23]和IRESfinder[24]。其中,IRSS和VIPS通过与已知IRES的二级结构进行相似度比对,得出待测序列为IRES元件的置信度。IRESpred通过支持向量机模型,构建了病毒和细胞IRES元件的35种特征,其中27种特征基于待测序列5'UTR区与小亚基核糖体蛋白结合的可能性,其他特征基于UTR区的序列和结构特征。IRESfinder通过文献验证[11]的583个IRES元件进行机器学习训练,经过10次交叉验证,ROC曲线分析的AUC值达到了0.825。其中,VIPS与已知病毒IRES二级结构进行比对,但当时已知病毒IRES只有4个,且运行时间较长,IRESfinder基于序列特征预测存在IRES元件的可能性,较适用于环形RNA中IRES的预测。
目前收录IRES元件的数据库主要有IRESdb[25], IRESite[26]和Rfam[27]。IRESdb构建于2002年,提供了30个来自病毒的IRES和50个来自真核细胞IRES相关mRNA信息。IRESite构建于2005年,数据库收录了125个IRES序列信息,来自43个病毒和70个真核mRNA。Rfam收集了IRES_RhPV, IRES_cyp24a1两个族类的IRES,提供了来源病毒和参考文献的相关信息。上述IRES数据库收录信息都比较久远,目前已验证的IRES元件已远超上述几个数据库。
3.4 m6A预测工具及相关数据库现有基于序列预测m6A修饰位点的软件主要有SRAMP(Sequence-based RNA adenosine methylation site predictor)[28]。SRAMP联合三种随机森林分类器(基于位置分类器、基于K最邻近算法分类器、基于核苷酸对分类器)给出综合打分。输入可以是基因组序列或是核心DNA序列(cDNA),分别对应两种模式。SRAMP在交叉验证和独立验证方面都具有优势,训练集正样本来自两篇验证哺乳动物单核苷酸分辨率的m6A位点的文章[29-30],负样本来自相同基序(DRACH)在同个数据集中的随机选取,因为m6A修饰并不是随机的[31]。SRAMP还做成了网页服务器的形式提供给用户使用。对于环形RNA中m6A修饰位点的预测,基于序列预测的工具SRAMP能够胜任。
目前收录m6A修饰位点的数据库主要有RMbase[32]和m6Avar[33]。RMbase通过m6A-CLIP的实验技术,收集了来自12个不同物种大约1 373 000个m6A修饰位点信息。m6Avar通过7组miCLIP,2组PA-m6A-Seq实验,244个MeRIP-Seq实验以及工具预测的渠道收集了三类m6A修饰位点数据,共414 241个m6A相关变异位点,基因类型包括了lincRNA, miRNA, piRNA等。
3.5 转录本蛋白编码预测工具目前常用转录本编码蛋白预测工具主要有CPC[34], CPAT[35]和CNCI [36]。工具主要分为两类,基于序列比对(Alignment-based)和不需要基于序列比对(Alignment-free)。其中CPC基于序列比对,可以识别保守性较好的蛋白编码基因,CPAT和CNCI不需要序列比对,主要用于物种间保守性较差的转录本。
2007年,Kong等人开发了评估转录本编码蛋白潜能的工具CPC[34]。CPC基于支持向量机分类器,通过提取具有重要生物学意义的六种序列特征。将输入序列分为编码序列或非编码序列并给出对应得分。训练集上通过十倍交叉验证,在大量数据集上展示出CPC具有很高的准确度(95.77%)。CPC提取的序列特征前三项关于预测的开放阅读框(ORF),由framefinder计算所得(包括The Log-odds score, Coverage of the predicted orf, Integrity of the predicted orf)。后三项特征通过假定ORF编码的蛋白与UniProt数据库经过blast比对结果所得(包括Number of hits, hit score, frame score)。CPC训练集正样本来自EMBL的121 914个编码区(CDS)序列,负样本来自Rfam和RNADB共34 766个非编码序列。
不同于CPC的是,CPAT不需要基于序列比对(Alignment-free),而是通过编码和非编码转录本的序列特征来进行区分[35]。CPAT运用逻辑回归分类器,基于四种序列特征来区分编码与非编码转录本,分别是:(1)开放阅读框长度(Open reading frame size);(2)开放阅读框覆盖度(Open reading frame coverage);(3)Fickett统计,基于碱基组成和密码子分布(Fickett TESTCODE statistic);(4)六聚体频率(Hexamer usage bias)。以上四种特征,都能较好区分编码与非编码转录本。正样本来自RefSeq数据库的10 000个编码蛋白转录本,负样本来自GENCODE数据库的10 000个随机选取的非编码RNA。通过十次交叉验证AUC曲线达到0.992 7。
而CNCI基于碱基三联子的构成来区分编码与非编码转录本,其利用人类和小鼠转录本构建支持向量机模型,用于对脊椎动物进行分类[36]。训练集正样本来自RefSeq数据库,负样本来自GENCODE。测试集数据物种包含了小鼠等脊椎动物和植物。对于人类编码和非编码转录本,经过十次交叉验证所得准确率达到97.3%。
针对环形RNA编码蛋白的预测,需要先将环形RNA序列预处理,保证ORF的完整性,避免跨越接头位置的ORF被分割,才能将环形RNA序列输入上述三种转录本编码蛋白预测工具进行分析。
3.6 编码蛋白环形RNA预测工具及相关数据库随着二代测序技术的快速发展,大量的环形RNA被发现,构建一个编码蛋白的环形RNA的数据库非常有必要。2016年Chen等人构建了首个人类环形RNA数据库circRNAdb,并对环形RNA的蛋白质编码潜能进行了分析[37]。研究者主要通过开放阅读框预测,IRES元件预测,以及蛋白质谱数据比对等几个方面,从32 914个人类环形RNA数据中,筛选出6 608个具有编码蛋白潜能的环形RNA,其中21个得到了质谱数据的验证。Yang等人和Zhang等人通过circRNAdb提供的参考信息,实验验证了Circ-FBXW7和circ-SHPRH能够编码蛋白质,ORF与数据库中预测的信息一致,IRES的验证也与数据库中的信息有很大重叠。由此可见,circRNAdb对于验证环形RNA编码蛋白质具有很大的参考意义。
目前环形RNA编码蛋白潜能预测工具主要有CircPro和CircCode。2017年,Meng等人开发了首个基于RNA-seq及Ribo-seq数据识别编码蛋白环形RNA的工具CircPro[38]。研究者首先使用转录组测序数据(RNA-seq)作为输入,结合.GTF基因注释文件,基因组文件,调用环形RNA检测工具CIRI2预测测序数据中的环形RNA[39]。其次,提取CIRI2所得结果的环形RNA序列,并经过拼接后调用CPC(Coding potential calculator)预测环形RNA编码能力得分。最后,使用翻译组测序数据(Ribo-seq)作为输入,寻找比对不上线性RNA的reads,将其与环形RNA反向剪接位点(Junction sites)的reads做比对,若能比对上,则能为该环形RNA的翻译潜能提供支持。CircPro总共会输出4个文件,其主要内容分别为:(a)预测的环形RNA序列;(b)每个circRNA的编码潜能得分(CPC预测);(c)每个circRNA的RNA-seq reads支持数和Ribo-seq reads支持数;(d)编码蛋白质的circRNAs。
2019年,Sun等人开发了环形RNA翻译的预测软件CircCode,这是一种基于机器学习的方法[40]。工作流程如下:首先应用Ribo-seq测序数据,保留比对不上基因组的reads,将其映射到环形RNA的接头位置,若能映射上则保留作为可翻译的候选环形RNA(该过程与CircPro最后一步类似)。接着通过机器学习工具BASiNET预测跨越街头部分的ribo-seq reads是否可以翻译,确定可以翻译的环形RNA。最后预测环形RNA的ORF及其可能编码的多肽。
CircPro与CircCode中基于ribo-seq数据分析的方法相似,有较高可信度,不同之处在于CircCode基于机器学习再预测这些比对上反向剪接位点的ribo-seq reads是否可翻译,而CircPro将比对上的reads都作为环形RNA可编码蛋白的一个证据。此外,CircCode使用FragGeneScan预测环形RNA开放阅读框,而CircPro通过CPC预测环形RNA编码蛋白潜能。
4 总结与展望一直以来,环形RNA被划分为非编码RNA。然而,近来研究发现,相当一部分的环形RNA具有编码蛋白质的潜能。目前,由于编码蛋白环形RNA的特征尚不明确,相关生物信息学预测及分析方法极为欠缺,严重阻碍了真核生物环形RNA编码蛋白的相关研究。现有RNA编码潜能的预测工具大都是基于线性RNA(mRNA和lncRNA)开发而成,而环形RNA中与mRNA的重叠部分,及其非线性的环状结构,都严重降低了现有工具对环形RNA编码潜能的预测能力。
环形RNA内的IRES及m6A修饰位点已被证实可介导其非帽依赖翻译起始过程,因此IRES及m6A修饰位点识别将有助于提高编码蛋白环形RNA的识别能力。此外,随着越来越多的编码蛋白环形RNA被发现,以及环形RNA编码蛋白机制的深入研究,相信会有更多更有效的编码蛋白环形RNA相关生物信息学工具及数据库出现,反过来进一步促进编码蛋白环形RNA的发现及对其编码起始机制的深入研究。
| [1] |
SANGER H L, KLOTZ G, RIESNER D, et al. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J]. Proceedings of the National Academy of Sciences, 1976, 73(11): 3852-3856. DOI:10.1073/pnas.73.11.3852 (  0) 0) |
| [2] |
HSU M T, MIGUEL C P. Electron microscopic evidence for the circular form of RNA in the cytoplasm of eukaryotic cells[J]. Nature, 1979, 280(5720): 339-340. DOI:10.1038/280339a0 (  0) 0) |
| [3] |
RYBAK-WOLF A, STOTTMEISTER C, GLAŽAR P, et al. Circular RNAs in the mammalian brain are highly abundant, conserved, and dynamically expressed[J]. Molecular Cell, 2015, 58(5): 870-885. DOI:10.1016/j.molcel.2015.03.027 (  0) 0) |
| [4] |
WANG Y, WANG Z. Efficient backsplicing produces translatable circular mRNAs[J]. Rna, 2015, 21(2): 172-179. DOI:10.1261/rna.048272.114 (  0) 0) |
| [5] |
LEGNINI I, DI T G, ROSSI F, et al. Circ-ZNF609 is a circular RNA that can be translated and functions in myogenesis[J]. Molecular Cell, 2017, 66(1): 22-37.e9. DOI:10.1016/j.molcel.2017.02.017 (  0) 0) |
| [6] |
YANG Y, GAO X, ZHANG M, et al. Novel role of FBXW7 circular RNA in repressing glioma tumorigenesis[J]. JNCI: Journal of the National Cancer Institute, 2017, 110(3): 304-315. DOI:10.1093/jnci/djy116 (  0) 0) |
| [7] |
ZHANG M, HUANG N, YANG X, et al. A novel protein encoded by the circular form of the SHPRH gene suppresses glioma tumorigenesis[J]. Oncogene, 2018, 37(13): 1805. DOI:10.1038/s41388-017-0019-9 (  0) 0) |
| [8] |
ZHANG M, ZHAO K, XU X, et al. A peptide encoded by circular form of LINC-PINT suppresses oncogenic transcriptional elongation in glioblastoma[J]. Nature Communications, 2018, 9(1): 4475. DOI:10.1038/s41467-018-06862-2 (  0) 0) |
| [9] |
LIANG W C, WONG C W, LIANG P P, et al. Translation of the circular RNA circβ-catenin promotes liver cancer cell growth through activation of the Wnt pathway[J]. Genome Biology, 2019, 20(1): 84. DOI:10.1186/s13059-019-1685-4 (  0) 0) |
| [10] |
VAN HEESCH S, WITTE F, SCHNEIDER-LUNITZ V, et al. The translational landscape of the human heart[J]. Cell, 2019, 178(1): 242-260. DOI:10.1016/j.cell.2019.05.010 (  0) 0) |
| [11] |
WEINGARTEN-GABBAY S, ELIAS-KIRMA S, NIR R, et al. Systematic discovery of cap-independent translation sequences in human and viral genomes[J]. Science, 2016, 351(6270): aad4939. DOI:10.1126/science.aad4939 (  0) 0) |
| [12] |
LI S, MASON C E. The pivotal regulatory landscape of RNA modifications[J]. Annual Review of Genomics and Human Genetics, 2014, 15: 127-150. DOI:10.1146/annurev-genom-090413-025405 (  0) 0) |
| [13] |
WEI C M, GERSHOWITZ A, MOSS B. Methylated nucleotides block 5' terminus of HeLa cell messenger RNA[J]. Cell, 1975, 4(4): 379-386. DOI:10.1016/0092-8674(75)90158-0 (  0) 0) |
| [14] |
CSEPANY T, LIN A, BALDICK C J, et al. Sequence specificity of mRNA N6-adenosine methyltransferase[J]. Journal of Biological Chemistry, 1990, 265(33): 20117-20122. DOI:10.1093/nar/18.19.5735 (  0) 0) |
| [15] |
HARPER J E, MICELI S M, ROBERTS R J, et al. Sequence specificity of the human mRNA N6-adenosine methylase in vitro[J]. Nucleic Acids Research, 1990, 18(19): 5735-5741. DOI:10.1093/nar/18.19.5735 (  0) 0) |
| [16] |
WANG X, ZHAO B S, ROUNDTREE I A, et al. N6-methyladenosine modulates messenger RNA translation efficiency[J]. Cell, 2015, 161(6): 1388-1399. DOI:10.1016/j.cell.2015.05.014 (  0) 0) |
| [17] |
ZHOU J, WAN J, GAO X, et al. Dynamic m6A mRNA methylation directs translational control of heat shock response[J]. Nature, 2015, 526(7574): 591. DOI:10.1038/nature15377 (  0) 0) |
| [18] |
MEYER K D, PATIL D P, ZHOU J, et al. 5' UTR m6A promotes cap-independent translation[J]. Cell, 2015, 163(4): 999-1010. DOI:10.1016/j.cell.2015.10.012 (  0) 0) |
| [19] |
LI A, CHEN Y S, PING X L, et al. Cytoplasmic m6A reader YTHDF3 promotes mRNA translation[J]. Cell Research, 2017, 27(3): 444. DOI:10.1038/cr.2017.10 (  0) 0) |
| [20] |
YANG Y, FAN X, MAO M, et al. Extensive translation of circular RNAs driven by N6-methyladenosine[J]. Cell Research, 2017, 27(5): 626. DOI:10.1038/cr.2017.31 (  0) 0) |
| [21] |
WU T Y, HSIEH C C, HONG J J, et al. IRSS: A web-based tool for automatic layout and analysis of IRES secondary structure prediction and searching system in silico[J]. BMC Bioinformatics, 2009, 10(1): 160. DOI:10.1186/1471-2105-10-160 (  0) 0) |
| [22] |
HONG J J, WU T Y, CHANG T Y, et al. Viral IRES prediction system-A web server for prediction of the IRES secondary structure in silico[J]. PLoS One, 2013, 8(11): e79288. DOI:10.1371/journal.pone.0079288 (  0) 0) |
| [23] |
KOLEKAR P, PATASKAR A, KULKARNI-KALE U, et al. IRESPred: Web server for prediction of cellular and viral internal ribosome entry site (IRES)[J]. Scientific Reports, 2016, 6: 27436. DOI:10.1038/srep27436 (  0) 0) |
| [24] |
ZHAO J, WU J, XU T, et al. IRESfinder: Identifying RNA internal ribosome entry site in eukaryotic cell using framed k-mer features[J]. Journal of Genetics and Genomics= Yi Chuan Xue Bao, 2018, 45(7): 403. DOI:10.1016/j.jgg.2018.07.006 (  0) 0) |
| [25] |
BONNAL S, BOUTONNET C, PRADO-LOURENÇO L, et al. IRESdb: The internal ribosome entry site database[J]. Nucleic Acids Research, 2003, 31(1): 427-428. DOI:10.1093/nar/gkg003 (  0) 0) |
| [26] |
MOKREJŠ M, MAŠEK T, VOPÁLENSKý V, et al. IRESite—A tool for the examination of viral and cellular internal ribosome entry sites[J]. Nucleic Acids Research, 2009, 38(suppl_1): D131-D136. DOI:10.1093/nar/gkp981 (  0) 0) |
| [27] |
KALVARI I, ARGASINSKA J, QUINONES-OLVERA N, et al. Rfam 13.0: Shifting to a genome-centric resource for non-coding RNA families[J]. Nucleic Acids Research, 2017, 46(D1): D335-D342. DOI:10.1093/nar/gkx1038 (  0) 0) |
| [28] |
ZHOU Y, ZENG P, LI Y H, et al. SRAMP: Prediction of mammalian N6-methyladenosine (m6A) sites based on sequence-derived features[J]. Nucleic Acids Research, 2016, 44(10): e91-e91. DOI:10.1093/nar/gkw104 (  0) 0) |
| [29] |
LINDER B, GROZHIK A V, OLARERIN-GEORGE A O, et al. Single-nucleotide-resolution mapping of m6A and m6 Am throughout the transcriptome[J]. Nature Methods, 2015, 12(8): 767. DOI:10.1038/nmeth.3453 (  0) 0) |
| [30] |
KE S, ALEMU E A, MERTENS C, et al. A majority of m6A residues are in the last exons, allowing the potential for 3' UTR regulation[J]. Genes & Development, 2015, 29(19): 2037-2053. DOI:10.1101/gad.269415.115 (  0) 0) |
| [31] |
MEYER K D, JAFFREY S R. The dynamic epitranscriptome: N6-methyladenosine and gene expression control[J]. Nature Reviews Molecular Cell Biology, 2014, 15(5): 313. DOI:10.1038/nrm3785 (  0) 0) |
| [32] |
SUN W J, LI J H, LIU S, et al. RMBase: A resource for decoding the landscape of RNA modifications from high-throughput sequencing data[J]. Nucleic Acids Research, 2015, 44(D1): D259-D265. DOI:10.1093/nar/gkv1036 (  0) 0) |
| [33] |
ZHENG Y, NIE P, PENG D, et al. m6A Var: A database of functional variants involved in m6A modification[J]. Nucleic Acids Research, 2017, 46(D1): D139-D145. DOI:10.1093/nar/gkx895 (  0) 0) |
| [34] |
KONG L, ZHANG Y, YE Z Q, et al. CPC: Assess the protein-coding potential of transcripts using sequence features and support vector machine[J]. Nucleic Acids Research, 2007, 35(suppl_2): W345-W349. DOI:10.1093/nar/gkm391 (  0) 0) |
| [35] |
WANG L, PARK H J, DASARI S, et al. CPAT: Coding-potential assessment tool using an alignment-free logistic regression model[J]. Nucleic Acids Research, 2013, 41(6): e74-e74. DOI:10.1093/nar/gkt006 (  0) 0) |
| [36] |
SUN L, LUO H, BU D, et al. Utilizing sequence intrinsic composition to classify protein-coding and long non-coding transcripts[J]. Nucleic Acids Research, 2013, 41(17): e166-e166. DOI:10.1093/nar/gkt646 (  0) 0) |
| [37] |
CHEN X, HAN P, ZHOU T, et al. circRNADb: A comprehensive database for human circular RNAs with protein-coding annotations[J]. Scientific Reports, 2016, 6: 34985. DOI:10.1038/srep34985 (  0) 0) |
| [38] |
MENG X, CHEN Q, ZHANG P, et al. CircPro: An integrated tool for the identification of circRNAs with protein-coding potential[J]. Bioinformatics, 2017, 33(20): 3314-3316. DOI:10.1093/bioinformatics/btx446 (  0) 0) |
| [39] |
GAO Y, WANG J, ZHAO F. CIRI: An efficient and unbiased algorithm for de novo circular RNA identification[J]. Genome Biology, 2015, 16(1): 4. DOI:10.1186/s13059-014-0571-3 (  0) 0) |
| [40] |
SUN Peisen, LI Guanglin. CircCode:A powerful tool for identifying circRNA coding ability[J]. Frontiers in Genetics, 2019, 10: 981. DOI:10.3389/fgene.2019.00981 (  0) 0) |
 2020, Vol. 18
2020, Vol. 18


