乙肝病毒属于DNA病毒,具有嗜肝性,该病毒感染会演变成肝炎甚至发展成肝硬化,相关疾病给患者及其家庭带来了沉重的负担。乙肝病毒是一个相当小的病毒,成环状、部分为双链DNA。乙肝病毒编码的几种蛋白中S蛋白是病毒的包膜蛋白,与病毒进入细胞有关,吸引了众多学者关注[1-2]。乙肝病毒S蛋白由226个氨基酸组成,属于疏水蛋白质[3]。乙肝病毒S蛋白是乙肝病毒亚病毒颗粒的主要组成部分,它的数量远比乙肝病毒颗粒多。以往研究者曾用生物信息学方法逐一对乙肝病毒基因区段的结构和功能的关系进行分析,不仅证明了乙肝病毒中部分功能区域的性质,而且发现了包膜蛋白上潜在的新功能域[4]。乙肝病毒大蛋白由S、PreS2和PreS1基因编码的HBsAg、PreS2和PreS1共同构成,已有部分研究证实乙肝病毒S大蛋白能够激活基因转录过程,促进肝癌发生,然而乙肝病毒S大蛋白在肝癌形成过程中所发挥的作用依然有很多问题有待研究。现有的乙肝免疫球蛋白都是针对乙肝病毒S蛋白的免疫球蛋白。和通过血液提纯的免疫球蛋白途径相比,单克隆抗体靶点清晰,灵敏度更高,特异性更强,同时,抗体的特异性结合能力和中和病毒的能力是评价中和抗体的关键指标。对乙肝病毒S蛋白进行深入研究有利于乙肝病毒的预防、诊断以及表位疫苗的研制[5]。本论文利用生物信息学方法分析乙肝病毒S蛋白的序列特征和抗原表位,预测该蛋白的抗原性,为进一步研究S蛋白结构与生物学功能提供参考依据。
1 材料及方法 1.1 材料登陆NCBI网站,从GenBank(NIH遗传序列数据库)数据库中获取乙肝病毒S蛋白氨基酸序列(检索号:ATY75544.1)。
1.2 方法利用https://web.expasy.org/protparam/网站统计与计算乙肝病毒S蛋白序列组成;利用https://web.expasy.org/protparam/网站分析乙肝病毒S蛋白的亲水性(疏水性);利用https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma. html网站分析乙肝病毒S蛋白的二级结构;利用http://www.cbs.dtu.dk/services/SignalP/网站分析乙肝病毒S蛋白的信号肽特征;利用http://www.cbs.dtu.dk/services/TMHMM/网站分析乙肝病毒S蛋白的跨膜区;利用http://tools.immuneepitope.org/bcell网站预测乙肝病毒S蛋白最佳抗原表位形成位置;利用http://www.cbs.dtu.dk/services/NetPhos-2.0/网站分析乙肝病毒S蛋白的磷酸化位点[6]。并将上述各预测的数值化结果进行重新绘图并分析。
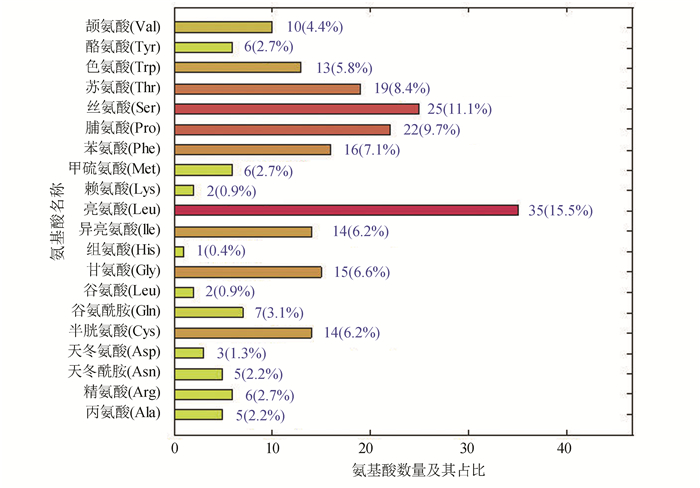
2 结果与分析 2.1 乙肝病毒S蛋白序列的基本组成成分乙肝病毒S蛋白由226个氨基酸组成,分子质量单位(Mr)为25 387.14。如图 1所示,图中纵坐标表示不同的氨基酸,226个氨基酸中亮氨酸(Leu)、丝氨酸(Ser)、脯氨酸(Pro)、苏氨酸(Thr)含量较高,分别占氨基酸总数的35%、25%、22%、19%。脂肪系数是99.60。理论等电点是8.21,原子总数是3 569。带负电荷残基的Asp(天冬氨酸)和Glu(谷氨酸)的数目是27,带正电荷残基的Arg(精氨酸)和Lys(赖氨酸)的总数目是18。N端的蛋白的为Met,S蛋白的不稳定系数是59.66,预测S蛋白属于不稳定蛋白。乙肝病毒S蛋白的半衰期在体外的哺乳动物网状细胞内是30 h,在酵母体内大于20 h,在大肠杆菌内大于10 h。
|
图 1 S蛋白中各个氨基酸含量 Figure 1 Content of each amino acid in S protein |
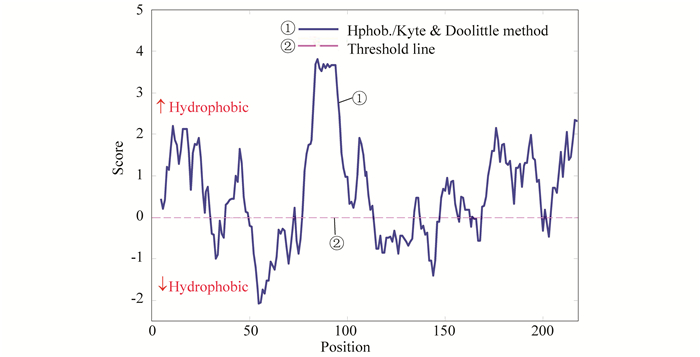
乙肝病毒S蛋白的总平均亲水性为0.649,可能是疏水蛋白质。该蛋白质的组成成分氨基酸的侧链大部分或者全部由碳原子和氢原子组成,不太可能与水分子形成氢键。图 2疏水性的测试结果集中反映了不同氨基酸位置的亲疏水性分布情况,阈值线为0,阈值线以上部分的氨基酸残基表现为疏水性,阈值线以下部分的氨基酸残基表现为亲水性。可以看出在(130-170)和(190-210)位置为高疏水值区域,附近可能存在潜在跨膜区。
|
图 2 S蛋白疏水性预测结果 Figure 2 Hydrophobicity prediction results for S protein |
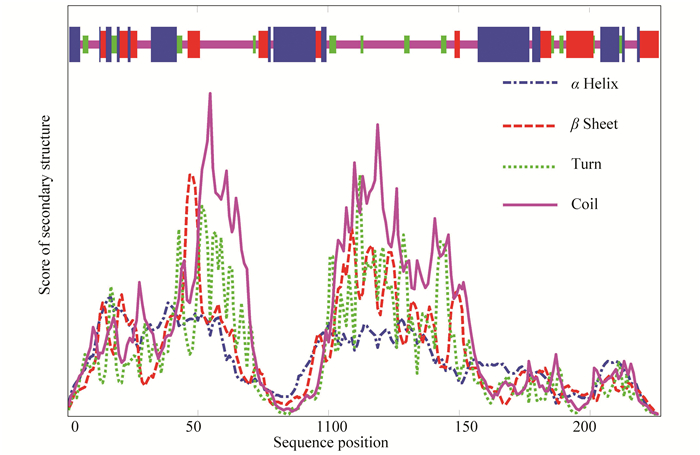
蛋白质一级结构中的氨基酸的组成和排列顺序决定蛋白质的空间结构,空间结构决定蛋白质的生物学功能。二级结构对其功能的影响较大,蛋白质进行二级结构预测是确定抗原表位,即确定抗原抗体结合部位的一种辅助手段[35]。乙肝病毒的S蛋白序列的二级结构见图 3,其中蓝色表示α螺旋,有72处氨基酸为α螺旋约占二级结构总数的31.86%;绿色表示β转角,有17处氨基酸为β转角约占二级结构总数的7.52%;红色表示β折叠,有44处氨基酸为β折叠约占二级结构总数的19.47%;紫色表示无规则卷曲,有93处氨基酸为无规则卷曲约占二级结构总数的41.15%。由β转角和无规则卷曲组成的区域占48.67%,表明在乙肝病毒S蛋白的这些区域中,抗原表位容易产生[7]。(彩图见电子版:http://swxxx.alljournals.cn/ch/index.aspx.2019年第2期
|
图 3 S蛋白二级结构 Figure 3 Secondary structure of S protein |
信号肽的本质是一段氨基酸序列,一般位于多肽链的末端,在蛋白质跨膜转运过程中起作用。如图 4所示,S经常被用于预测信号肽的剪切位点[8],该预测结果中S分值最大为0.606,大于信号肽阈值0.5,S分值与阈值线的交点处的氨基酸位点为5,说明乙肝病毒S蛋白存在信号肽。且信号肽长度为5个氨基酸残基,该蛋白的剪切部位在5-6氨基酸处。5氨基酸之后S分值较小这部分蛋白为成熟蛋白。

|
图 4 S蛋白信号肽预测结果 Figure 4 Signal peptide prediction results for S protein |
跨膜区是指蛋白质氨基酸序列中跨越细胞膜的区域。如图 5所示,蓝色表示细胞膜内,绿色表示细胞膜外,红色部分表示跨膜区。乙肝病毒S蛋白存在4个跨膜区位置分别是7-29,80-98,170-192,202-224。(彩图见电子版:http://swxxx.alljournals.cn/ch/index.aspx.2019年第2期)。

|
图 5 乙肝病毒S蛋白跨膜区预测结果 Figure 5 Transmembrane region prediction results of S protein |
如图 6所示,红色表示丝氨酸,绿色表示苏氨酸,蓝色表示酪氨酸。乙肝病毒S蛋白存在7个苏氨酸磷酸化位点,22个丝氨酸磷酸化位点,1个酪氨酸磷酸化位点,表明乙肝病毒S蛋白有30个潜在的磷酸化位点。(彩图见电子版:http://swxxx.alljournals.cn/ch/index.aspx.2019年第2期)。

|
图 6 乙肝病毒S蛋白磷酸化位点 Figure 6 Phosphorylation site of S protein in hepatitis B virus |
对乙肝病毒S蛋白进行可及性分析,可及性分析集中体现了乙肝病毒S蛋白抗原中氨基酸残基的分布情况。可及性越大说明相应的氨基酸残基更容易与抗体结合。如图 7所示,阈值线以上均为S蛋白抗原中氨基酸残基可被溶剂分子接触的部分,得分较高的氨基酸区域有10个分别为:0-3、24-34、48-62、66-69、96-99、109-118、122-132、139-143、165-168、198-204。

|
图 7 乙肝病毒S蛋白可及性 Figure 7 Accessibility of S protein in hepatitis B virus |
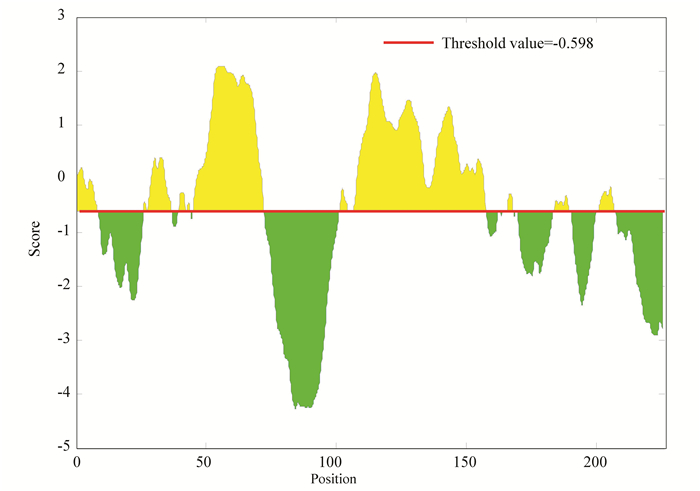
对乙肝病毒S蛋白进行线性表位分析,线性表位预测对于多种免疫研究是必不可少的,选择有效的线性表位有利于顺利进行抗体制备。如图 8所示,黄色部分即阈值线以上部分的线性表位对应的氨基酸位点较为适合作为抗原抗体结合部位,得分高的阈值线以上区域有7个分别为:0-8、25-36、46-69、100-155、165-168、182-188、200-206。(彩图见电子版:http://swxxx.alljournals.cn/ch/index.aspx.2019年第2期)。
|
图 8 乙肝病毒S蛋白线性表位 Figure 8 Linear epitope of S protein in hepatitis B virus |
对乙肝病毒S蛋白进行β转角结果预测,β转角一般处于蛋白质表面位置,为凸出结构,经常发生扭曲结构较为松散,更容易与抗体结合,由于β转角中的氨基酸残基结构和生物学特性的特殊性,使得β转角存在区域易形成抗原表位。本论文的抗原表位预测结果中阈值为1。如图 9所示,得分高的区域即阈值线以上的绿色区域有6个分别为:0-6、27-34、37-39、47-70、102-153、195-204。

|
图 9 S蛋白β转角预测结果 Figure 9 β angle prediction results for S protein |
对乙肝病毒S蛋白进行抗原性与分子量、化学组成、立体结构有关。抗原性越大,对应的氨基酸越稳定,越容易形成抗原位点。图 10为对乙肝病毒S蛋白进行抗原性结果预测的结果,得分较高的绿色阈值线以上区域有10个分别为:9-19、43-49、63-69、76-99、103-108、132-138、146-151、172-184、187-191、200-224。综合分析乙肝病毒S蛋白的可及性、线性表位、β转角、柔性、抗原性得到的潜在抗原表位有2个区域,分别为:48-49、66-69。

|
图 10 乙肝病毒S蛋白抗原性 Figure 10 Antigenicity of S protein in hepatitis B virus |
抗原表位是指位于抗原分子中的一种特殊的化学基团,对抗原特异性起决定性作用,抗原通过抗原表位完成免疫效应。表位疫苗是一种新型疫苗,在艾滋病等病毒上已经取得了初步研究进展。表位疫苗是指通过基因工程的手段,体外培养或人工合成微生物的表位,将得到的表位作为一种疫苗使用[9]。与传统疫苗相比,表位疫苗更加稳定安全[10]。为了提高抗原表位预测的准确性,基于蛋白质的二级结构预测结果,在此基础上进一步对乙肝病毒S蛋白的可及性、线性表位、β转角、抗原性进行预测分析,选出利用不同性质分析结果中相同区域,作为最佳抗原表位位置,综合分析得到的潜在抗原表位有2个区域,分别为:48-49、66-69。
乙型肝炎由乙型肝炎病毒引发,是一种全球性疾病,全世界乙型肝炎的慢性感染者约有4亿人。因为乙型肝炎危害极大,流传广,因此各国科学家对乙肝病毒的防治方法给予了高度的重视[11]。乙肝病毒顽强的抵抗力和潜伏性是乙肝病毒难以治愈的主要原因。蛋白的结构预测是生物学研究的热点之一[12]。自1965年乙肝病毒被发现就一直是一项难以攻克的难题,在现今的医疗水平上乙肝病毒几乎可以说是一种可以被控制,但不能被彻底杀死的病毒。乙肝病毒S蛋白在人体内的不仅会对肝脏造成伤害,还会通过影响男性患者的精子存活率以及受精能力导致生育能力下降[13]。关于乙肝病毒S蛋白生物信息学分析结果,是基于蛋白质的结构特点、数据库数据以及计算机分析得到的。这些预测结果对深入研究乙肝病毒、表位疫苗的研制具有指导意义,但还不能完全照搬,必须设计相应的实验进行验证,才能真正应用于乙肝病毒的防治过程。
李晋涛等人预测了一种抑制蛋白的表面特性和二级结构,采取多参数分析法进行预测,如对可塑性和亲水性、免疫原性和理化性质等方面进行分析,发现该抑制蛋白存在多个潜在的抗原表位[14]。黄继华采用多种方法检测乙肝病毒S蛋白对人精子凋亡和受精能力的影响,结果证明乙肝病毒S蛋白在精子的凋亡过程中发挥作用,因此精子的线粒体的部分功能甚至于受精能力都会受到不同程度的影响[15]。丁悦等人的系列研究表明乙肝病毒的主要构成成分乙肝病毒S蛋白能够引起精子线粒体膜电位、精子活力、受精能力显著下降,并导致精子大量死亡。同时,乙肝病毒S蛋白还可引起人精子产生氧化应激影响细胞膜的完整性,并进一步导致细胞凋亡而影响精子的功能[16]。本研究对乙肝病毒S蛋白运用生物信息学知识进行序列特征分析,成功预测到S蛋白的基本理化性质、结构和功能特征以及潜在的抗原表位所在位置,这些数据在一定程度上将作为对乙肝病毒以及乙肝病毒S蛋白深入研究和表位疫苗研制、免疫诊断的理论基础。
4 结论乙肝病毒存在逆转录过程并且具有极强的潜伏性。应用生物信息学分析网站进行乙肝病毒S蛋白的序列特征分析,并在分析的基础上研究乙肝病毒S蛋白的生物学特性。研究结果显示:(1)乙肝病毒S蛋白是不稳定蛋白,很难与水分子形成氢键属于疏水蛋白质,二级结构中α螺旋与无规则卷曲占大多数,同时乙肝病毒S蛋白存在信号肽、跨膜区以及潜在的磷酸化位点。(2)结合乙肝病毒S蛋白的序列可及性、线性表位、β转角、柔性、抗原性的预测结果,可以找到最合适的氨基酸位点进行抗原抗体结合,形成抗原表位,综合分析得到的潜在抗原表位有2个区域,分别为:48-49、66-69。研究结果可以作为表位疫苗的研制依据,有利于进一步研究乙肝病毒S蛋白的抗原性,对乙肝病毒S蛋白的研究进展有促进作用。
| [1] |
LI S, YANG Y, DING X, et al. LHBs can elevate the expression of MDR1 through HIF-1α in patients with CHB infection: A comparative proteomic study[J]. Oncotarget, 2017, 8(3): 4549-4562. DOI:10.18632/oncotarget.13941 (  0) 0) |
| [2] |
HASSEMER M, FINKERNAGEL M, PEIFFER K H, et al. Comparative characterization of hepatitis B virus surface antigen derived from different hepatitis B virus genotypes[J]. Virology, 2017, 502: 1-12. DOI:10.1016/j.virol.2016.12.003 (  0) 0) |
| [3] |
陈喆, 朱悦, 李亦学, 等. 人乙型肝炎病毒包膜蛋白多态性的生物信息学分析及其意义[J]. 复旦学报(医学版), 2006(06): 711-717. CHEN Zhe, ZHU Yue, LI Yixue, et al. Large-scale polymorphism analysis of hepatitis B virus envelope protein by bioinformatics[J]. Fudan University Journal of Medical Sciences, 2006(06): 711-717. DOI:10.3969/j.issn.1672-8467.2006.06.001 (  0) 0) |
| [4] |
STIRK H J, THORNTON J M, HOWARD C R. A topological model for hepatitis B surface antigen[J]. Intervirology, 1992, 33(3): 148-149. DOI:10.1159/000150244 (  0) 0) |
| [5] |
王葳, 何永刚, 浦永兰, 等. 乙型肝炎病毒Ⅹ蛋白的优势氨基酸序列与热点突变位点的生物信息学分析[J]. 微生物与感染, 2016, 11(06): 338-346. WANG Wei, HE Yonggang, PU Yonglan, et al. Bioinformatic determination of dominant amino acid sequence and mutation hotspots in hepatitis B virus X protein[J]. Journal of Microbes and Infections, 2016, 11(06): 338-346. DOI:10.3969/j.issn.1673-6184.2016.06.004 (  0) 0) |
| [6] |
刘畅, 刘安. 生物信息学分析RIPK4的分子结构与功能[J]. 生物信息学, 2018, 16(2): 105-112. LIU Chang, LIU An. Bioinformatics analysis of the structure and function of RIPK4[J]. Chinese Journal of Bioinformatics, 2018, 16(2): 105-112. DOI:10.3969/j.issn.1672-5565.201708006 (  0) 0) |
| [7] |
李广兴, 丛培君, 潘龙, 等. 鸡堆型艾美尔球虫3-1E抗原表位的生物信息学预测[J]. 东北农业大学学报, 2015, 46(05): 38-43. LI Guangxing, CONG Peijun, PAN Long, et al. Bioinformatics prediction on 3-1E antigen epitopes of Eimeria acervulina[J]. Journal of Northeast Agricultural University, 2015, 46(05): 38-43. DOI:10.19720/j.cnki.issn.1005-9369.2015.05.006 (  0) 0) |
| [8] |
熊伟, 杨勇琴, 张海洋, 等. 人线粒体转录终止因子1(hMTERF1)蛋白的生物信息学分析[J]. 生物信息学, 2015, 13(01): 23-30. XIONG Wei, YANG Yongqin, ZHANG Haiyang, et al. Bioinformatic analysis of human mitochondrial transcription termination factor 1(hMTERF1)[J]. Chinese Journal of Bioinformatics, 2015, 13(01): 23-30. DOI:10.13241/j.cnki.pmb.2016.05.038 (  0) 0) |
| [9] |
SOMPURAM S R, BASTAS G, VANIL K. Accurate identification of paraprotein antigen targets by epitope reconstruction[J]. Blood, 2008, 111(1): 302-308. DOI:10.1182/blood-2007-05-090654 (  0) 0) |
| [10] |
李玉娇, 王晶, 赵慧, 等. 细粒棘球绦虫Eg95抗原表位的生物信息学预测[J]. 中国人兽共患病学报, 2011, 27(10): 893-900. LI Yujiao, WANG Jing, ZHAO Hui, et al. Bioinformatics prediction on Eg95 antigen epitopes of Echinococcus granulosus[J]. Chinese Journal of Zoonoses, 2011, 27(10): 893-900. DOI:10.3969/j.issn.1002-2694.2011.10.008 (  0) 0) |
| [11] |
康祥锦, 丁悦, 杨洁, 等. 乙肝病毒S蛋白对人精子氧化应激的影响[J]. 癌变·畸变·突变, 2015, 27(03): 202-206. KANG Xiangjin, DING Yue, YANG Jie, et al. Effects of hepatitis B virus S protein exposure on human sperm oxidative stress[J]. Carcinogenesis, Teratogenesis & Mutagenesis, 2015, 27(03): 202-206. DOI:10.3969/j.issn.1004-616x.2015.03.009 (  0) 0) |
| [12] |
张静静, 付玉荣, 伊正君. 结核分枝杆菌RV2629基因编码蛋白的生物信息学分析[J]. 中国病原生物学杂志, 2017, 12(08): 736. ZHANG Jingjing, FU Yurong, YI Zhengjun. Bioinformatic analysis of the protein coded for by the Rv2629 gene of Mycobacterium tuberculosis[J]. Journal of Pathogen Biology, 2017, 12(08): 736. DOI:10.13350/j.cjpb.170808 (  0) 0) |
| [13] |
POLAT K Y, BALIK A A, CELEBI F. Hepatic alveolar echinococcosis: Clinical report from an endemic region[J]. Canadian Journal of Surgery, 2002, 45(6): 415-419. DOI:10.1016/S0305-4179(02)00189-4 (  0) 0) |
| [14] |
李晋涛, 陈正琼, 梁志清, 等. Eppin抗原的二级结构分析B细胞表位预测[J]. 第三军医大学学报, 2008(24): 2254-2257. LI Jintao, CHEN Zhengqiong, LIANG Zhiqing, et al. Prediction of B cell epitope for Eppin antigen[J]. Acta Academiae Medicinae Militaris Tertiae, 2008(24): 2254-2257. DOI:10.3321/j.issn:1000-5404.2008.24.002 (  0) 0) |
| [15] |
黄继华.乙型肝炎病毒S蛋白诱导人精子凋亡及功能异常[D].汕头: 汕头大学医学院, 2013. HUANG Jihua. Hepatitis B virus S protein induces sperm apoptosis and dysfunction[D]. Shantou: Shantou University Medical College, 2013. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=D380499 (  0) 0) |
| [16] |
丁悦, 杨洁, 杜红姿, 等. 乙肝病毒S蛋白暴露时间对人精子胞膜完整性及功能的影响[J]. 中国临床药理学与治疗学, 2015, 20(12): 1321-1327. DING Yue, YANG Jie, DU Hongzi, et al. Effects of hepatitis B virus S protein exposuring time on human sperm membrane integrity and function[J]. Chinese Journal of Clinical Pharmacology and Therapeutics, 2015, 20(12): 1321-1327. (  0) 0) |
 2019, Vol. 17
2019, Vol. 17

