2. 山西医科大学 基础医学院生物化学与分子生物学教研室,太原 030001;
3. 长治医学院 附属和济医院医务科,山西 长治 046000
2. Department of Biochemistry andMolecular Biology, Shanxi Medical University, Taiyuan 030001, China;
3. Medical Department, Heji Hospital Affiliated to Changzhi Medical College, Changzhi 046000, China
RIPK4最早通过酵母双杂交实验发现,是能够与蛋白激酶C(PKC)β1和PKCδ相互作用的一种蛋白质[1]。早期证据表明,RIPK4能够调控表皮细胞分化,RIPK4缺陷小鼠体内表皮细胞分化异常,围产期即会死亡[2]。人体内RIPK4突变可导致常染色体隐性Bartsocas-Papas综合征,该病患者面部、皮肤和四肢均有严重缺陷[3]。
尽管已知RIPK4在表皮分化过程中扮演重要角色,目前关于RIPK4在表皮分化、肿瘤发生和侵袭转移过程中的调控机制报道较少。Huang等[4]发现RIPK4通过磷酸化Dishevelled蛋白调控Wnt信号通路,在异体移植瘤模型中,RIPK4过量表达促进卵巢癌发生发展。此外,在皮肤癌和结直肠癌中,RIPK4均过量表达,推测RIPK4为致癌基因。然而在肝癌和舌鳞状细胞癌中,发现RIPK4低表达,其表达水平与细胞分化程度呈正相关,推测RIPK4为抑癌基因[5-6]。
RIPK4在肿瘤细胞侵袭转移过程中也发挥作用,此过程涉及上皮间质转化(EMT)的异常活化,受到Wnt/β-catenin等多种信号通路调控。RIPK4能够调控293T细胞中Wnt/β-catenin信号通路,增加β-catenin的转录活性[4]。在宫颈癌细胞中,抑制RIPK4表达能够显著抑制EMT途径关键调控分子(Vimentin,MMP2和Fibronectin)的表达,表明RIPK4能够促进宫颈癌细胞的侵袭转移[7]。然而,在Tca-8113舌癌细胞中,RIPK4抑制其侵袭转移[6]。以上矛盾的研究结果预示RIPK4在不同肿瘤中有不同的致癌机制,也表明了RIPK4作用的复杂性,其分子功能和具体调控机制亟待深入研究。
此外,RIPK4在细胞凋亡过程中也发挥作用。人舌癌细胞Tca-8113中过量表达RIPK4可调控顺铂诱导的细胞凋亡[6]。RIPK4能够激活NF-κB和JNK,在细胞凋亡、坏死和炎症过程中也能被caspase切割从而被激活[8-9]。
本文使用生物信息学分析方法,研究RIPK4蛋白的理化性质和结构功能,可为进一步研究该蛋白的分子功能及作用机制提供思路。
1 材料与方法 1.1 材料RIPK4蛋白的序列信息来自于NCBI的GenBank数据库(https://www.ncbi.nlm.nih.gov/),以“RIPK4+物种名”为关键词,搜索得到人RIPK4蛋白的氨基酸序列信息。
1.2 方法 1.2.1 RIPK4理化性质分析使用ExPASy数据分析系统中的ProtParam(http://web.expasy.org/protparam/)工具,对RIPK4的分子量、分子式、酸碱性和稳定性等理化性质进行分析。使用Protscale工具(http://web.expasy.org/protscale/)对RIPK4的亲疏水性进行分析。使用SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)分析RIPK4有无切割位点和信号肽,TMHMM 2.0工具(http://www.cbs.dtu.dk/services/TMHMM/)分析RIPK4的跨膜区域。
1.2.2 RIPK4组织表达特异性和亚细胞定位预测使用NCBI的UniGene数据库中EST结果对RIPK4在正常组织和癌组织中的表达情况进行分析。使用PSORTII(https://psort.hgc.jp/)进行RIPK4的亚细胞定位预测。
1.2.3 RIPK4二级结构和高级结构分析使用SOPMA工具(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)预测RIPK4的二级结构及各成分所占比例。使用NCBI的Conserved Domain数据库分析RIPK4的结构域。使用SWISS-MODEL建模服务器(https://swissmodel.expasy.org/)预测三维结构。
1.2.4 相互作用蛋白预测使用STRING数据库(http://string-db.org/),设置为高置信度0.7,不限制数量,构建与RIPK4相互作用的蛋白网络。
1.2.5 RIPK4系统进化树构建在NCBI中,通过BLASTp搜索得到RIPK4在不同物种中的同源蛋白质序列。使用Clustal2.1软件进行RIPK4同源蛋白间的多重序列比对。使用MEGA6软件,Neighbor-joining方法,设置Boot-strap分析重复数为1 000,构建分子进化树。
2 结果与分析通过对RIPK4蛋白的一级结构、二级结构、高级结构、组织表达特异性、亚细胞定位、蛋白质相互作用网络及序列同源性进行分析,旨在对RIPK4的分子结构与功能进行全面分析。
2.1 RIPK4基因的编码产物分析RIPK4基因的染色体定位为21q22.3,含有8个外显子,编码的P57078蛋白为RIPK4的共识编码序列,为832个氨基酸组成的多肽。转录产物经可变剪切后编码的另一种蛋白NP_065690.2与P57078相比,第278位到325位氨基酸缺失,为784个氨基酸组成的多肽。本文的分析对象是含有832个氨基酸的RIPK4蛋白。
2.2 RIPK4的理化性质分析ExPASy数据库中的ProtParam在线工具预测,RIPK4蛋白分子式为C4043H6486N1172O1200S28,分子量是91 610.79 Da,其中亮氨酸含量最高,占整个序列的13.3%。RIPK4蛋白的理论等电点为6.69,整个序列中带负电荷的酸性氨基酸(Asp和Glu)共有101个,带正电荷的碱性氨基酸(Arg和Lys)共有95个,因此RIPK4属于酸性蛋白质。RIPK4在哺乳动物网织红细胞内半衰期为30 h,不稳定系数为42.09,属于不稳定蛋白质。脂肪系数为95.07,总的平均亲水性为-0.237。
ExPASy数据库中的ProtScale在线工具预测,RIPK4亲水性最强的位点是第340位的天冬氨酸,分值为-2.522;疏水性最强的位点是第213位的异亮氨酸,分值为2.722。由图 1可知,RIPK4序列中亲水区域多于疏水区域,属于亲水蛋白质。

|
图 1 RIPK4蛋白的亲疏水性分析 Figure 1 Hydrophilic-hydrophobic analysis of RIPK4 |
SignalP 4.0预测RIPK4蛋白无信号肽序列(见图 2)。原始剪切位点C值最高点位于第23位氨基酸,分值为0.118;被结合的剪切点Y值最高点位于第12位氨基酸,分值为0.178;信号肽S值最高点在第4位氨基酸,分值为0.370;1~11位氨基酸序列的平均信号肽分值为0.275,不足以形成经典的信号肽区域。
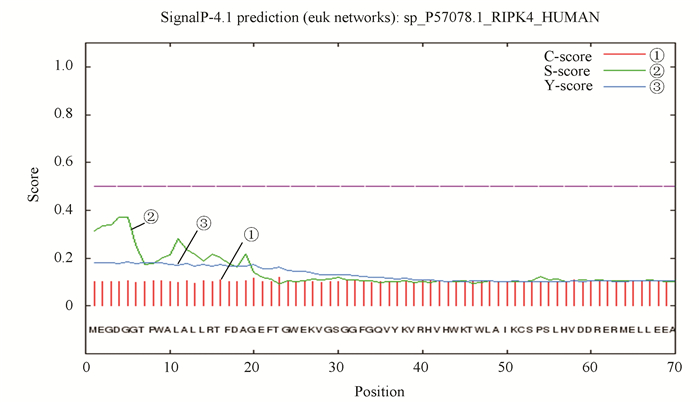
|
图 2 RIPK4蛋白信号肽分析 Figure 2 Signal peptides analysis of RIPK4 |
TMHMM 2.0预测RIPK4蛋白无跨膜结构域(见图 3)。RIPK4蛋白位于膜外(粉色细线)的概率几乎为100 %,位于膜内(蓝色细线)和跨膜区域(红色细线)的概率几乎为0。粉色粗线代表多肽链中跨膜区域所在位置,因RIPK4蛋白没有跨膜区域,所以在粗线上不显示相应标记。
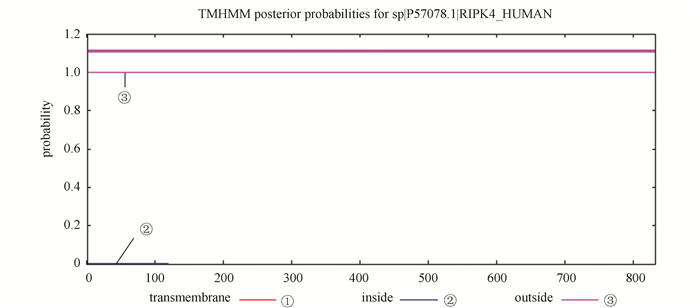
|
图 3 RIPK4蛋白跨膜结构分析 Figure 3 Trans-membrane domain analysis of RIPK4 |
NCBI的UniGene数据库中EST结果显示,RIPK4在以下正常组织中均有表达,拷贝数分别是:嘴347,气管193,甲状腺97,肾脏61,乳腺46,胃41,肺32,肝脏14。此外,在癌组织中也有表达,拷贝数分别是:头颈部肿瘤201,乳腺癌53,肝癌20,肾癌14,肺癌9,结直肠癌8。
PSORTII预测,RIPK4定位于细胞质的可能性最大(60.9 %),其次可能定位于细胞核(17.4 %),细胞骨架(17.4 %)和过氧化物酶体(4.3 %)。
2.4 RIPK4的空间结构分析SOPMA分析RIPK4的二级结构可知(见图 4),α-螺旋(图中蓝色)占38.94 %,无规卷曲(图中橙色)占31.85 %,延伸链(图中红色)占17.67 %,β-转角(图中绿色)占11.54 %。
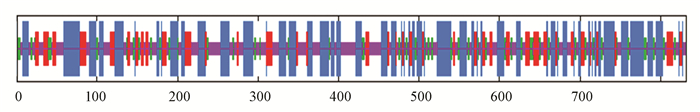
|
图 4 RIPK4的二级结构分析 Figure 4 RIPK4 secondary structure analysis |
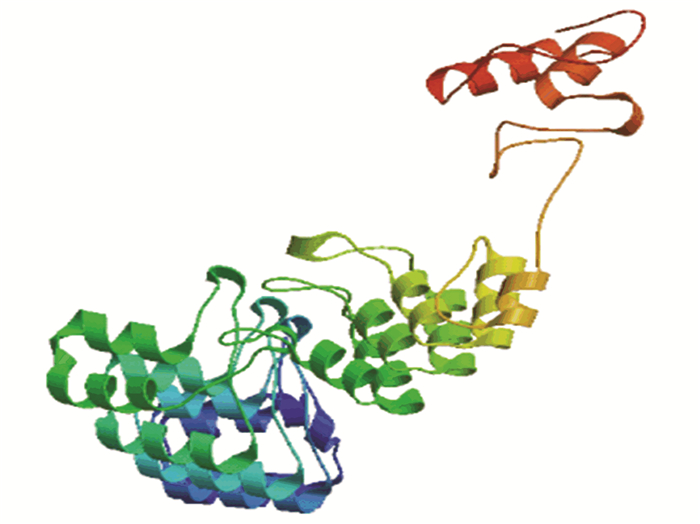
NCBI的Conserved Domain数据库预测RIPK4蛋白属于PKc_like超家族和锚蛋白重复序列(ankyrin repeats, ANK)超家族(见图 5)。PKc_like超家族中的STKc结构域能够催化磷酸基团从ATP转移到蛋白底物的丝氨酸/苏氨酸残基上,从而使蛋白底物磷酸化(见图 6)。ANK结构域常与其他结构域同时存在,能够调控蛋白之间的相互作用。使用SWISS-MODEL建模服务器预测RIPK4蛋白三维结构(见图 6),GMQE为0.28,QMEAN为-0.78,序列与模板相似度为42.51 %,覆盖率为40 %,该模型结构合理。
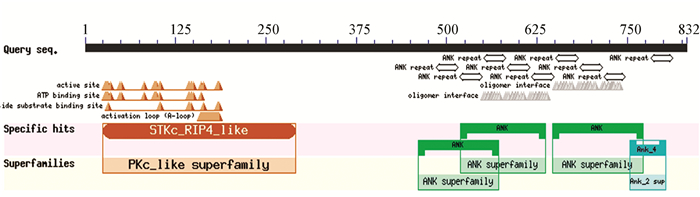
|
图 5 RIPK4功能结构域分析 Figure 5 Functional domain analysis of RIPK4 |
|
图 6 RIPK4三维结构预测 Figure 6 Prediction of RIPK4 Three dimensional structure |
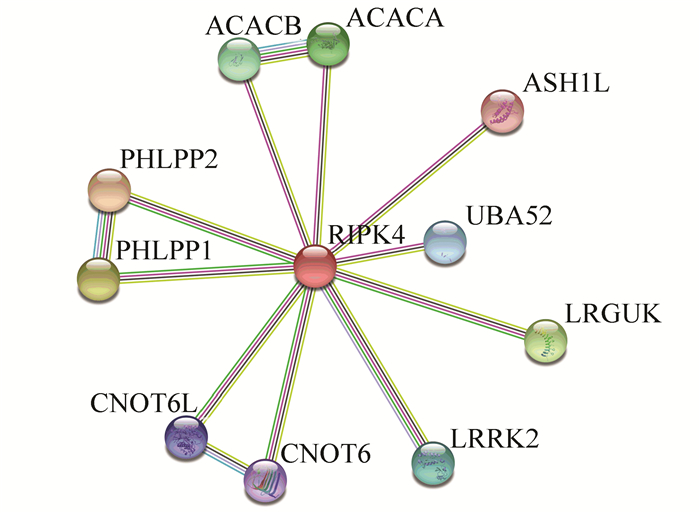
使用STRING数据库预测与RIPK4相互作用的蛋白质,设置为高置信度0.7,不限制数量,构建RIPK4蛋白相互作用网络(见图 7)。与RIPK4相互作用的蛋白质主要包括PHLPP1(PH domain and leucine rich repeat protein phosphatase 1),PHLPP2(PH domain and leucine rich repeat protein phosphatase 2),LRGUK(Leucine-rich repeats and guanylate kinase domain containing),乙酰辅酶A羧化酶α(acetyl-CoA carboxylase alpha,ACACA),乙酰辅酶A羧化酶β(acetyl-CoA carboxylase beta,ACACB),ASH1L(Ash1 (absent, small, or homeotic)-like (Drosophila)),UBA52(Ubiquitin A-52 residue ribosomal protein fusion product 1),LRRK2(Leucine-rich repeat kinase 2),CNOT6L(CCR4-NOT transcription complex, subunit 6-like),CNOT6(CCR4-NOT transcription complex, subunit 6)。
|
图 7 RIPK4蛋白相互作用网络 Figure 7 Protein-protein interaction network for RIPK4 |
对以上互作蛋白的基因进行GO分析和KEGG通路分析。GO分析结果表明,互作基因富集于细胞核和CCR4-NOT复合体(见表 1),参与乙酰辅酶A催化酶活性、离子结合和聚(A)-特异性核糖核酸酶活性等2个分子功能(见表 2),涉及含磷酸基团复合物的代谢、乙酰辅酶A代谢、能量代谢和调节细胞质中mRNA加工等生物过程(见表 3)。KEGG通路分析结果表明,互作基因显著富集于脂肪酸生物合成、脂肪酸代谢、丙酮酸代谢和RNA降解等通路中(见表 4)。由此推测,RIPK4与PHLPP1、PHLPP2、ACACA、ACACB、CNOT6L和CNOT6之间的关系值得进一步探究。
| 表 1 GO细胞组成分类结果 Table 1 GO cellular component classification results |
| 表 2 GO分子功能分类结果 Table 2 GO molecular function classification results |
| 表 3 GO生物学过程分类结果 Table 3 GO biological process classification results |
| 表 4 KEGG pathway路径列表 Table 4 KEGG pathway lists |
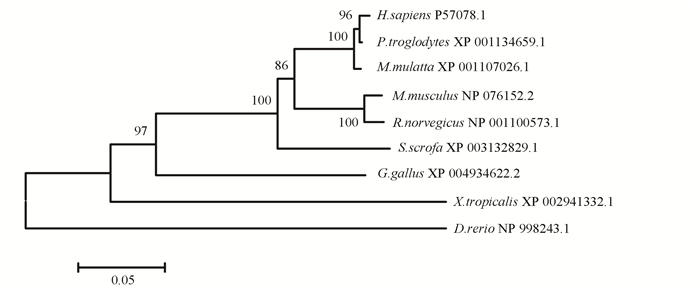
对不同物种RIPK4蛋白进行多重序列比对,并构建分子进化树(见图 8)。人RIPK4蛋白的氨基酸序列与黑猩猩、猕猴、小鼠、大鼠、野猪、鸡、非洲爪蟾和斑马鱼的相似性分别为93.41%、92.57%、85.25%、85.13%、83.39%、70.99%、60%和53.67%,与物种进化程度相一致,表明RIPK4蛋白在进化过程中具有保守的分子功能。
|
图 8 RIPK4蛋白的分子进化树 Figure 8 The phylogenetic tree of RIPK4 |
通过生物信息学方法,分析得到RIPK4是酸性不稳定的亲水蛋白,无信号肽和跨膜区域,定位于细胞质的可能性最大。RIPK4的主要二级结构是α-螺旋,属于PKc_like和ANK超家族,具有利于蛋白间相互作用的结构信息。
由RIPK4互作蛋白的基因GO分析和KEGG通路分析结果可知,互作基因主要参与乙酰辅酶A代谢和能量代谢等生物过程,显著富集于脂肪酸生物合成、脂肪酸代谢和丙酮酸代谢等通路中。因此,在RIPK4互作蛋白中,ACACA和ACACB引起关注。癌细胞中存在Warburg效应,即便在有氧条件下,癌细胞也更偏爱使用糖酵解途径进行葡萄糖代谢产生能量。目前研究发现,很多靶向Warburg效应的治疗方法中,癌细胞代谢可能依赖脂肪酸β-氧化途径,而不是糖酵解途径。AMPK(AMP-activated protein kinase)通过磷酸化乙酰辅酶A羧化酶(acetyl-CoA carboxylase,ACC),抑制细胞中脂肪酸合成,促进其发生β-氧化,从而维持胁迫条件下细胞中的能量稳态[10-11]。ACACA调控长链脂肪酸生物合成的限速反应[12]。作为导管原位癌的一个潜在诊断标志物,ACACB是一个复杂的多功能酶系统,能够调控脂肪酸氧化的催化速率。由此推测,RIPK4与ACACA和ACACB之间的关系及参与的脂代谢过程具有一定的研究价值,有助于阐明RIPK4的作用机制和生理功能。
此外,由GO分析结果可知,互作基因主要涉及含磷酸基团复合物的代谢等生物过程,参与催化活性等分子功能。因此,RIPK4与PHLPP1和PHLPP2之间的关系引起关注,预示RIPK4存在未知分子功能和作用机制。PHLPP1和PHLPP2在多种癌症中起到抑癌作用,能够抑制PI3K/Akt信号通路,使Akt的C末端473位Ser残基和PKC等其他AGC激酶的疏水基团发生去磷酸化,Akt和PKC的酶活性受到抑制,进一步抑制细胞增殖[13-14]。PHLPP1和PHLPP2也能够激活Mst1,促进细胞凋亡[15]。此外,PHLPP通过抑制Akt活性,促进溶酶体依赖性的整合素β1和β4降解,抑制EMT途径,进一步对细胞迁移起到负调控作用[16]。RIPK4与PHLPP1和PHLPP2之间的关系尚未见报道,本文分析得出它们之间存在相互作用,有望成为阐明RIPK4作用机制的突破口。
进一步,由GO分析和KEGG通路分析结果可知,互作基因参与聚(A)-特异性核糖核酸酶活性等4个分子功能,涉及调节细胞质中mRNA加工等生物过程,显著富集于RNA降解等通路中。因此,RIPK4与CNOT6L和CNOT6之间的关系值得继续探究。CNOT6L和CNOT6是CCR4-NOT复合体的亚基,该复合体是一个重要的去腺苷化酶,能降解mRNA的poly(A)尾,从而使mRNA发生降解,抑制翻译过程[17]。目前尚未有关于RIPK4与CNOT6L和CNOT6之间关系的报道,探究它们之间的相互作用,有助于深入认识RIPK4的作用机制。
4 结论对RIPK4的分子结构和理化性质进行生物信息学分析,为进一步研究其生物学功能及致癌机制提供一定的参考。结论如下:
1) RIPK4的理论分子量为91 610.79 Da,理论等电点为6.69,属于酸性蛋白质。RIPK4总平均亲水性为-0.237,不稳定系数为42.09,属于亲水蛋白质。
2) RIPK4的二级结构主要为α-螺旋,属于PKc_like超家族和ANK超家族,无信号肽和跨膜结构域。
3) RIPK4组织表达的特异性不强,最有可能定位于细胞质。
4) 根据系统进化树推测,人RIPK4蛋白在进化过程中高度保守。
5) 根据GO分析和KEGG通路分析结果推测,RIPK4主要涉及含磷酸基团复合物的代谢和能量代谢等生物学过程,参与催化活性和离子结合等分子功能。
| [1] |
CHEN L, HAIDER K, PONDA M, et al. Protein kinase C-associated kinase (PKK), a novel membrane-associated, ankyrin repeat-containing protein kinase[J]. Journal of Biological Chemistry, 2001, 276(24): 21737-21744. DOI:10.1074/jbc.M008069200 (  0) 0) |
| [2] |
HOLLAND P, WILLIS C, KANALY S, et al. RIP4 is an ankyrin repeat-containing kinase essential for keratinocyte differentiation[J]. Current Biology, 2002, 12(16): 1424-1428. DOI:10.1016/S0960-9822(02)01075-8 (  0) 0) |
| [3] |
KALAY E, SEZGIN O, CHELLAPPA V, et al. Mutations in RIPK4 cause the autosomal-recessive form of popliteal pterygium syndrome[J]. American Journal of Human Genetics, 2012, 90(1): 76-85. DOI:10.1016/j.ajhg.2011.11.014 (  0) 0) |
| [4] |
HUANG X, MCGANN J C, LIU B Y, et al. Phosphorylation of dishevelled by protein kinase RIPK4 regulates Wnt signaling[J]. Science, 2013, 339(6126): 1441-1445. DOI:10.1126/science.1232253 (  0) 0) |
| [5] |
HEIM D, CORNILS K, SCHULZE K, et al. Retroviral insertional mutagenesis in telomerase-immortalized hepatocytes identifies RIPK4 as novel tumor suppressor in human hepatocarcinogenesis[J]. Oncogene, 2015, 34(3): 364-372. DOI:10.1038/onc.2013.551 (  0) 0) |
| [6] |
WANG X, ZHU W, ZHOU Y, et al. RIPK4 is downregulated in poorly differentiated tongue cancer and is associated with migration/invasion and cisplatin-induced apoptosis[J]. International Journal of Biological Markers, 2014, 29(2): e150-e159. DOI:10.5301/jbm.5000073 (  0) 0) |
| [7] |
LIU D Q, LI F F, ZHANG J B, et al. Increased RIPK4 expression is associated with progression and poor prognosis in cervical squamous cell carcinoma patients[J]. Scientific Reports, 2015, 5: 11955. DOI:10.1038/srep11955 (  0) 0) |
| [8] |
MEYLAN E, MARTINON F, THOME M, et al. RIP4 (DIK/PKK), a novel member of the RIP kinase family, activates NF-kappa B and is processed during apoptosis[J]. Embo Journal, 2002, 3(12): 1201-1208. DOI:10.1093/embo-reports/kvf236 (  0) 0) |
| [9] |
MEYLAN E, TSCHOPP J. The RIP kinases: crucial integrators of cellular stress[J]. Trends in Biochemical Sciences, 2005, 30(3): 151-159. DOI:10.1016/j.tibs.2005.01.003 (  0) 0) |
| [10] |
CURRIE E, SCHULZE A, ZECHNER R, et al. Cellular fatty acid metabolism and cancer[J]. Cell Metabolism, 2013, 18(2): 153-161. DOI:10.1016/j.cmet.2013.05.017 (  0) 0) |
| [11] |
HARDIE D G, ROSS F A, HAWLEY S A. AMPK: a nutrient and energy sensor that maintains energy homeostasis[J]. Nature Reviews Molecular Cell Biology, 2012, 13(4): 251-262. DOI:10.1038/nrm3311 (  0) 0) |
| [12] |
EUSEBI P G, GONZÁLEZ-PRENDES R, QUINTANILLA R, et al. A genome-wide association analysis for carcass traits in a commercial Duroc pig population[J]. Animal Genetics, 2017, 48(4): 466-469. DOI:10.1111/age.12545 (  0) 0) |
| [13] |
GAO T, FURNARI F, NEWTON A C. PHLPP: a phosphatase that directly dephosphorylates Akt, promotes apoptosis, and suppresses tumor growth[J]. Molecular Cell, 2005, 18(1): 13-24. DOI:10.1016/j.molcel.2005.03.008 (  0) 0) |
| [14] |
GAO T, BROGNARD J, NEWTON A C. The phosphatase PHLPP controls the cellular levels of protein kinase C[J]. Journal of Biological Chemistry, 2008, 283(10): 6300-6311. DOI:10.1074/jbc.M707319200 (  0) 0) |
| [15] |
GRZECHNIK A T, NEWTON A C. PHLPPing through history: a decade in the life of PHLPP phosphatases[J]. Biochemical Society Transactions, 2016, 44(6): 1675-1682. DOI:10.1042/BST20160170 (  0) 0) |
| [16] |
SMITH A J, WEN Y A, STEVENS P D, et al. PHLPP negatively regulates cell motility through inhibition of Akt activity and integrin expression in pancreatic cancer cells[J]. Oncotarget, 2016, 7(7): 7801-7815. DOI:10.18632/oncotarget.6848 (  0) 0) |
| [17] |
MITTAL S, ASLAM A, DOIDGE R, et al. The Ccr4a (CNOT6) and Ccr4b (CNOT6L) deadenylase subunits of the human Ccr4-Not complex contribute to the prevention of cell death and senescence[J]. Molecular Biology of the Cell, 2011, 22(6): 748-758. DOI:10.1091/mbc.E10-11-0898 (  0) 0) |
 2018, Vol. 16
2018, Vol. 16

