由Science杂志数据库搜索查询,该刊最早于1911年33卷有两篇文章出现“合成生物学”一词。由Scirus搜索引擎搜索,合成生物学一词最早出现于1911年7月8日著名医学刊物《柳叶刀》发表的一篇书评中。后来虽然断断续续出现过多次,但在1980年第一次以“基因外科术:合成生物学的开始”为题出现在德文刊物的一篇长篇论文[1]。随着人类基因组计划的完成,2000年以后,合成生物学一词在学术刊物及互联网上逐渐大量出现。对于近几年合成生物学的突然变热,不同的人对其有不同的解释。著名科普刊物 The Scientist为此专门采访了一些合成生物学领域的参与者[2],其中加州大学伯克利分校(UCB)的化学工程教授Keasling说:合成生物学正在用“生物学”进行工程化,就像用“物理学”进行“电子工程”,用“化学”进行“化学工程”一样。目前合成生物学与传统的重组DNA技术之间的界限仍然是模糊的。从根本上说,合成生物学正在利用获得的生物“零件”进行下一层次的工作——对细胞进行实际的工程化。是利用我们所确信的一些标准“零件”构造新生物系统的工程。“合成生物学组织”网站上公布的合成生物学的定义包括两条路线:(1)新的生物零件、组件和系统的设计与建造;(2)对现有的、天然的生物系统的重新设计[3]。
合成生物学建立在“标准生物组件(BioBrick)”的基础上[4-5],所谓的标准生物构件就是一些具有生物学意义的DNA分子。合成生物学就是在活细胞中使用这些可互换的标准生物组件重新组合构造新的生物系统,并加以操纵来实现某种特定的生理功能。随着生物学的工程化和现代分子生物学的迅猛发展,这些所谓的“BioBrick”是以海量的形式出现的,对这些标准生物组件信息的组织、存储和操控必然依赖现代的数据库技术。本文就是通过使用现代数据库技术从常用的质粒表达载体中提取“生物组件”信息,将这些载体拆解成“零件”,提取信息加以组织、存储,然后期望使用这些零件构建新的载体。
1 常用质粒载体数据库gene_bank数据库是信息系统的核心,在信息社会中占据着举足轻重的地位。数据库技术主要研究如何科学地组织、存储和管理数据库中的数据。简单地说,数据库是存储、管理数据的容器:严格地说,数据库是“按照某种数据结构对数据进行组织,存储和管理的容器”[6]。合成生物学信息的组织、存储、管理以及操控就是依赖于现代的数据库技术。
Gene_bank数据库是源于常用质粒载体的数据库,这些质粒可以用来传染12种不同的宿主细胞(包括大肠杆菌、酿酒酵母、植物细胞、哺乳动物细胞、昆虫细胞等),这一信息在数据库中已予以存储。这是一个“生物组件”数据库,我们可以使用它来构造设计新的载体。每一个质粒载体的genbank文档中的FEATURES区域包含具有生物学意义的序列,可以用作开发标准生物组件(BioBrick)[7]。
通过Perl语言编写程序,可以提取FEATURES区域的信息。提取的信息包括features名,所有的注释信息即note,重要的是提取各个features的序列信息,需要按照各features的起止号码,根据ORIGIN区域的序列信息提取各个features的准确序列。将提取的信息输入MySQL数据库。输入时产生两个表,其中plasmids表包括了每个质粒的总体信息(包括完整的genbank文档)。Features表中包含了从质粒genbank文档FEATURES区域提取的信息,其中FEATURES名被定义为feature_qualifier,而第一个注释即note在数据库中被定义为feature_name (FEATURES名),第二个note被定义为description即FEATURES的描述,表中还包含各FEATURES的起止号码和相对应的准确序列信息。genebank_feature表对各个feature_qualifier的含义进行了描述。snapgene_qualifier表和genocad_qualifier表是对各个feature_qualifier在两种流行的合成生物学软件Snapgene[8]和GenoCAD[8]中的含义的描述,表结构与genebank_feature的表结构基本一致。Gene_bank这一关系型数据库中最重要的是features表,这个表里包含就是从质粒载体“拆解”下来的合成生物学“零件”信息,可以使用这些信息来开发BioBrick。该数据库源于常用质粒载体,与标准生物组件(BioBrick)数据库的组织与结构有所不同[5-6],其中最大的不同在于它源于成熟商业化的质粒可用来开发新的商用载体,而标准生物组件数据库主要支持iGEM(International Genetically Engineered Machine)竞赛。
2 gene_bank数据库的SQL查询利用MySQL查询gene_bank数据库的操作如下。
2.1 打开MySQLWindows+R——>cmd(进入DOS)——>mysql -u root -p——>password
2.2 显示库表show databases;
use gene_bank;
show tables;
经过查询可知,gene_bank数据库中共有5张表,分别是features,genebank_features,genocad_qualifier,plasmids,snapgene_qualifier。
2.3 查询表结构 2.3.1 Desc plasmidsPlasmid(质粒)表中共有7个字段,如图 1所示。其中plasmid_id即质粒号;plasmid_name即质粒名;definition是对质粒的基本描述;sequence是质粒的序列信息;complete_genbank_text区存储完整的质粒genbank文档;is_circular表示如果该质粒是环形闭合的该区域值为1否则为0;origin表示质粒来源的数据集。
2.3.2 Desc featuresFeatures(特性)表中共有10个字段,如图 2所示。其中feature_id即features号;feature_name即features名称;description是对该features的基本描述;feature_qualifier表示该features是属于哪一类;complement表示该features是否是反向互补序列,如果是该区域值取1否则取0;start表示该features在它所属质粒中序列的起始号;end表示该features在质粒中序列的终止号;sequence表示该features的序列信息;plasmid_id表示该features所属质粒的号;flag区域表示该features的序列是否含有除a,g,c,t之外的特殊字符,如果含有则予以标出。
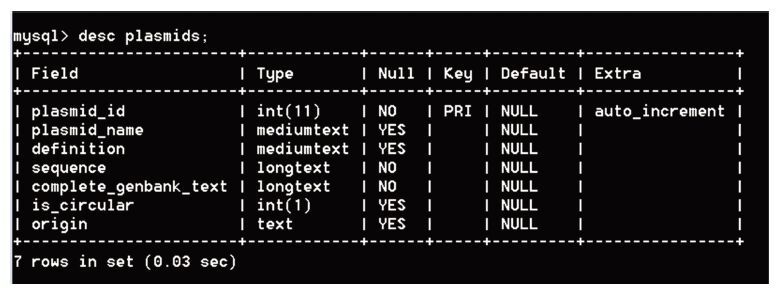
|
图 1 表plasmids的查询结果 Figure 1 The result of querying table plasmids |

|
图 2 表features 的查询结果 Figure 2 The result of querying table features |
Genebank_features表中共有3个字段,如图 3所示。其中qualifier_id表示feature_qualifier的号码;feature_qualifier就是各个feature_qualifier的名称;description是对各个feature_qualifier含义的解释。
2.4 查询feature表中的总记录Select count(*) as totalItem from features; 17 760 结果features表中总共有17 760条记录
Select count(*) from features where sequence is NULL; 0
Select count(distinct sequence) as totalsequence from features; 2 137sequence字段没有NULL值,完全不同的sequence只有2 137个,因此有大量sequence是冗余的,冗余的序列信息被标识并保留。

|
图 3 表genebank_features 的查询结果 Figure 3 The result of querying table genebank_features |
Select feature_name,sequence,description,feature_qualifier,count(feature_id) from features group by feature_name,sequence,description,feature_qualifier having count(feature_id)>1 order by count(feature_id) desc;
通过这个语句,可以查询到feature_name、feature_qualifier、description、sequence四者均相同的features有哪些,通过查询可知AmpR promoter,AmpR,ori,T7 promoter是使用最多的四种features(分别是781次、516次、454次、452次)。这一查询的意义在于知晓哪些序列使用最为普遍频繁,为下一步开发BioBrick做准备。
Select feature_name,sequence,description,feature_qualifier,count(feature_id) from features group by feature_name,sequence,description,feature_qualifier having feature_name='MCS' order by count(feature_id) desc;
通过这个语句,可以查询到当features是MCS(多克隆位点)时,所用序列的统计次数,可以得到使用次数最多的序列,并且feature_qualifier是misc_feature,对于这些序列用BioEdit做了分析,可以显示其中详细的多克隆位点。这一查询的意义在于知晓哪些多克隆位点使用最为普遍频繁,可以用来提取较为有效的多克隆位点构建克隆或表达载体。
3 gene_bank数据库的意义关于标准生物构件数据库,最著名的莫过于麻省理工学院(Massachusetts Institute of Technology,MIT)倡导的Standard Biological Parts[9]。但是之前还少有基于成熟并常用的克隆、表达载体的数据库[10]。Gene_bank数据库就是源于常用质粒载体的数据库,我们可以使用它构造新的载体。
Gene_bank数据库源于成熟常用的商业化质粒载体,可以用来开发用作BioBrick。
Gene_bank数据库便于合成生物学家查询合成生物学研究所需要的数据,了解各个组件的具体信息,组合成新的生物系统。
4 前景与展望合成生物学将催生下一次生物技术革命。目前,科学家们已经不局限于非常辛苦地进行基因剪接,而是开始构建遗传密码,以期利用合成的遗传因子构建新的生物体。合成生物学在未来几年有望取得迅速进展。据估计,合成生物学在很多领域将具有极好的应用前景,这些领域包括更有效的疫苗的生产、新药和药物的改进、以生物学为基础的制造、可再生能源利用、生产可持续能源、环境污染的生物治理、可以检测有毒化学物质的生物传感器。本文通过从常用的质粒载体中获取序列信息,将完整的质粒序列拆成“零件”构建成数据库,提供给合成生物学家使用。以期从这些零件中提取元素构建新的表达载体。
| [1] |
HOBOM B. Gene surgery:on the threshold of synthetic biology[J]. Medizinische Klinik, 1980, 75 (24) : 834 –841.
( 0) 0)
|
| [2] |
LUCENTINI, L. Just what is synthetic biology[J]. The Scientist, 2006, 20 (1) : 36 .
( 0) 0)
|
| [3] |
赵学明, 王庆昭. 合成生物学: 学科基础、研究进展与前景展望[J]. 前沿科学, 2007 (3) : 56 –66.
ZHAO Xueming, WANG Qingzhao. Synthetic biology: fundamentals, advances and prospect[J]. Frontier Science, 2007 (3) : 56 –66.
( 0) 0)
|
| [4] |
SHETTY R P, ENDY D. Engineering BioBrick vectors from BioBrick parts[J]. Journal of Biological Engineering, 2008, 2 (1) : 5 .
( 0) 0)
|
| [5] |
孔祥盛. MySQL核心技术与最佳实践(第一版). 北京: 人民邮电出版社, 2012 .
KONG Xiangsheng. MySQL core technology &best practice(1st ed.). Beijing: Posts &Telecom Press, 2012 .
( 0) 0)
|
| [6] |
ADAMES N R, WILSON M L, FANG G, et al. GenoLIB:A database of standard biogical parts derived from a library of common plasmid features[J]. Nucleic Acids Research, 2015, 43 (10) : 4823 .
( 0) 0)
|
| [7] |
COOLING M T, ROUILLY V, MISIRLI G, et al. Standard virtual biological parts: a repository of modular modeling components for synthetic biology[J]. Bioinformatics, 2010, 26 (7) : 925 –931.
( 0) 0)
|
| [8] |
CZAR M J, CAI Y, PECCOUD J. Writing DNA with GenoCAD[J]. Nucleic Acids Research, 2009, 37 (Web Server issue) : W40 –W47.
( 0) 0)
|
| [9] |
SMOLKE C D. Building outside of the box: iGEM and the BioBricks Foundation[J]. Nature Biotechnology, 2009, 27 (12) : 1099 –1102.
( 0) 0)
|
| [10] |
CAI Y, WILSON M L, PECCOUD J. GenoCAD for iGEM: a grammatical approach to the design of standard-compliant constructs[J]. Nucleic Acids Research, 2010, 38 (8) : 2637 –2644.
( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14

