转座子是染色体上可移位和自主复制的基本单位,其本质是具有不同结构和换位机制的移动序列[1]。转座子从原位点上单独复制或断裂下来,环化后插入到另一个位点,就像从基因组的一个位置“跳跃”到另一个位置,此基因切割整合的过程称为转座[2]。转座子依据转座机制的不同,可以分为:反转录转座子(I类转座子)和DNA转座子(II类转座子)[3]。反转录转座子通过自身编码的逆转录酶使用“copy-and-paste”机制实现转座,DNA转座子则通过自身编码的转座酶使用“cut-and-paste”转座机制[4]。
转座子被发现于20世纪50年代,一直被视为“垃圾序列”、假基因[5],然而近几十年研究已经证明,转座子在基因组进化、基因结构、染色体重组及基因活性的调节等很多领域都有着不可忽视的作用[6]。例如,转座子插入UTR和CDS区域可抑制或提前终止基因表达[7]、插入基因内部影响启动子功能实施[8]、或成为某些表观调控的靶位点[9]。而且,转座子可以在转录后水平调控基因的表达,有研究表明带有多聚腺苷酸信号的转座子基因插入3’端非编码区导致转录的终止[10],带有microRNA的结合位点的转座子插入到基因转录产物中导致基因表达沉默等[11]。
真核生物基因组测序计划揭示了较大基因组中转座子序列令人震惊的丰富度,几乎存在于所有生物体基因组,并在真核生物基因组中占据重要组成部分,尤其是脊椎动物,如人类基因组由总数3.2×109 bp的DNA序列构成,其中约50%为各种转座子,但大多在进化过程中失活。目前转座子在转基因上的应用研究报道较多[12-13],但对转座子在宿主中的功能了解较少,有研究表明转座子可能与衰老、肿瘤和大脑功能有关[14-16],众多学者也推测转座子可能在胚胎发育中发挥重要作用,但尚缺乏足够的实验证据,目前对各类转座子在早期胚胎中的表达特性和功能知之甚少。本文在生物信息学分析基础上,选择9个具有代表性DNA转座子和RNA转座子成员,这些转座子结构完整,可能具有转座活性,利用qPCR技术研究其在早期胚胎和成年组织中的时空表达,全面系统揭示其表达特性,为理解和认识其功能及后续的深入研究提供参考。
1 材料与方法 1.1 实验材料荧光定量PCR试剂盒FastFire qPCR PreMix(SYBR Green)、反转录试剂盒FastQuant RT Kit (With gDNase)购自天根生化有限公司;组织总RNA提取试剂TRIzol购自Invitrogen;其他试剂为国产分析纯级。斑马鱼(danio rerio)为Tuebingen品系,购自国家斑马鱼资源中心,饲养条件为每天光照14 h,黑暗10 h,温度为27~28 ℃。
1.2 方法 1.2.1 活性转座子数据来源本文所用的9种转座子来源于前期对斑马鱼转座子注释结果[17],主要通过MGEScan-nonLTR获得L1和L2序列,通过LTRHarvest获得ERV序列,Repbase数据中Tc1转座酶序列进行TBLASTN获得Tc1转座子超家族。其中Tc1-a、Tc1-b、Tc1-c、Tc1-d、Tc1-e为DNA转座子中Tc1/Mariner超家族成员,而ERV-1和ERV-2是逆转录病毒转座子,L1-323和L2-21为非长末端重复序列逆转录转座子(non-LTR),ERV-1、ERV-2、L1-323和L2-21均为RNA转座子。
1.2.2 胚胎收集选用6~10月龄Tuebingen品系的成年斑马鱼,于收卵前一天晚上,将雌鱼和雄鱼按1:2的比例置于繁殖盒,并用隔板隔开。第2天拿开隔板,公母鱼追逐产卵,从产卵开始计时,约10~15 min后收卵,置于50 mm培养皿,倒入E3培养液,放入28 ℃恒温培养箱中孵化,分别收集8个发育阶段胚胎,包括0.75(卵裂期)、2.00 (卵裂期/64-细胞)、3.00(囊胚期)、6.00(原肠期)、12.00(体节期/5-体节)、15.00(14-体节)、24.00(咽囊期)、48.00 h(孵化期)。收集各发育阶段胚胎适量,同一时间段至少收集3个平行样,置于1.5 ml的无核酸酶的离心管中,液氮速冻后,再放入-70 ℃保存备用。
1.2.3 成鱼组织器官收集用大头针分别固定斑马鱼的头部和尾部,用解剖剪去除从泄殖腔到鳃盖下缘一侧,暴露内脏器官,收集心脏、肝脏、睾丸、卵巢、肌肉、大脑等主要脏器,每种脏器至少收集3个平行样,置于1.5 ml的无核酸酶的离心管中,液氮速冻后,再放入-70 ℃保存备用。
1.2.4 RNA提取用Trizol法提取胚胎和组织总RNA,具体方法参照试剂使用说明。将获得的总RNA跑琼脂糖凝胶电泳检测RNA质量,并用微量核酸浓度测定仪测定RNA体积分数和纯度,质量合格者-70 ℃冻存备用。
1.2.5 逆转录合成cDNA逆转录反应按照试剂盒说明书,主要包括两步:先用DNase处理总RNA,去除污染的基因组,再用逆转录酶合成用于荧光定量实验的cDNA第一链。逆转录体系包括1 μg总RNA,2 μL 5×gDNA Buffer,2 μL 10×Fast RT Buffer,1 μL RT Enzyme Mix,2 μL FQ-RT Primer Mix,RNase-Free ddH2O补足至20 μL。先42 ℃孵育15 min,再95 ℃孵育3 min,-20 ℃保存备用。
1.2.6 实时荧光定量聚合酶链反应根据斑马鱼9个转座子编码框序列,用Primer5设计9对qPCR引物,并以斑马鱼延伸因子EF-1作为内参基因,引物由英潍捷基(上海)贸易有限公司合成,具体序列见表 1。qPCR反应体系包括:cDNA第一链1μL,2×SuperReal PreMix Plus10 μL,正反向引物(10 μmol/L)各0.6 μl,RNase-Free H2O补足至20 μL。使用Roche荧光定量PCR仪进行,先95 ℃预变性15 min,再进行40个循环:95 ℃10 s,60 ℃20 s,72 ℃20 s,最后72 ℃10 min。每个发育阶段或每个组织器官设3个平行样本,每个样本设3个复孔。
| 表 1 目的基因和内参基因引物序列 Table 1 Primer sequences of targeted gene and reference gene |
采用2-ΔΔCt方法对qPCR结果进行数据分析,试验数据以均数±标准误(mean±SE)表示,使用SPSS 22.0统计学软件进行数据的统计学处理和分析,表达差异用单因素方差分析,P<0.05为差异显著。
2 结果与分析 2.1 转座子在斑马鱼胚胎不同发育阶段表达水平通过qPCR法检测了9种转座子在斑马鱼胚胎7个发育阶段相对表达水平,结果如图 1、2所示,DNA转座子从6.00 h阶段开始表达,呈现先下降再上升或逐渐上升的表达趋势。反转录转座子启动表达时间不一致,其中ERV-1和L2-21在3.00 h阶段,L1-323在6.00 h,ERV-2在15.00 h阶段。而且3个反转录转座子在24.00 h时期表达水平显著上升。
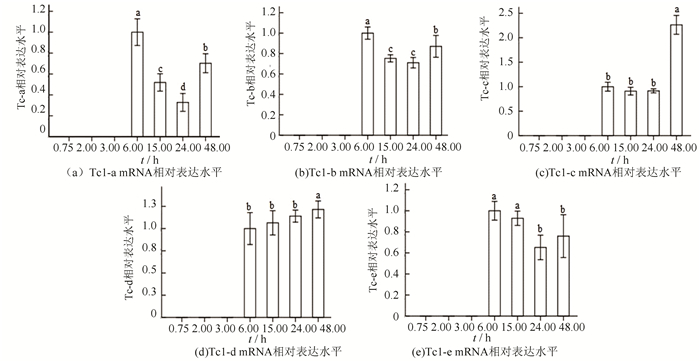
|
图 1 DNA转座子在斑马鱼胚胎发育节点表达水平检测 Figure 1 Expression level of DNA transposons at development nodes of zebrafish 注:不同字母表示差异显著(P<0.05) |
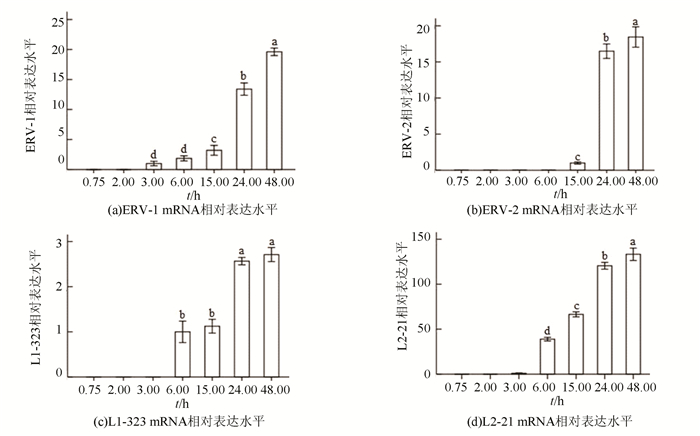
|
图 2 反转录转座子在斑马鱼胚胎发育节点表达水平检测 Figure 2 Expression level of retotransposons at development nodes of zebrafish 注:不同字母表示差异显著(P<0.05) |
提取成年斑马鱼心脏、大脑、肝脏、肌肉、睾丸、卵巢等组织总RNA,反转录成cDNA,以此cDNA为模板进行荧光定量PCR,检测各组织转座子表达水平。结果如图 3、4所示,DNA和RNA转座子在大脑和心脏的表达水平显著高于其他组织;卵巢和肝脏的转座子表达水平高于肌肉和睾丸;睾丸组织转座子表达水平最低。
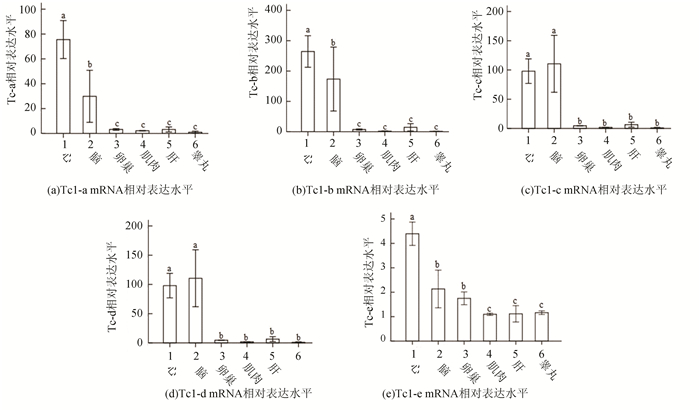
|
图 3 斑马鱼不同组织DNA转座子表达水平比较 Figure 3 Expression level of DNA transposons in different tissues of zebrafish 注:不同字母表示差异显著(P<0.05) |
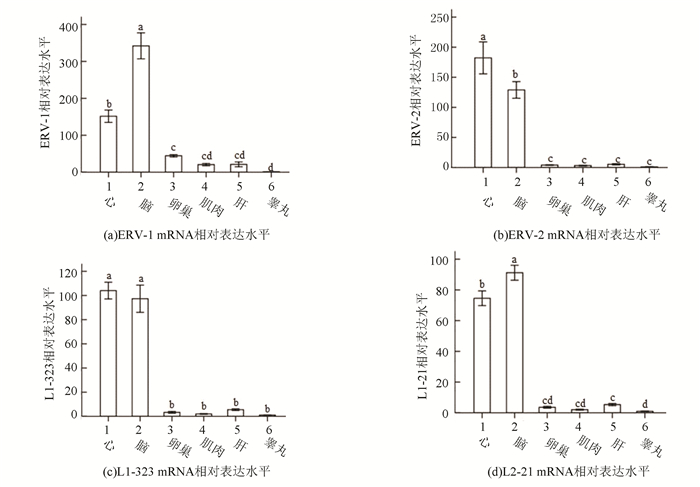
|
图 4 斑马鱼不同组织反转录转座子表达水平比较 Figure 4 Expression level of retrotransposons in different tissues of zebrafish 注:不同字母表示差异显著(P<0.05) |
转座子在动物基因组中占很大比例,占斑马鱼基因组54.96%。当转座元件入侵一个新的基因组后,增加了拷贝数,同时也积累了突变,最终导致失活的拷贝。由于转座子可能对基因组产生不利影响,宿主采用各种机制沉默活性转座子,如siRNAs和piRNAs,后者通常来源于转座子元件,可在转录和转录后水平阻断转座子表达[18-19]。转座子一直被认为是“垃圾”DNA,没有明确的功能。但近年来研究表明,转座子可被调控和定位表达。例如,在果蝇和小鼠胚胎检测到反转录转座子家族表达[20-21]。在热带爪蟾早期发育阶段,来自Tc1家族的一个DNA转座子在背部区域特异表达,这个区域最终形成神经系统[22],提示这些转座子可能在早期胚胎发育阶段具有生物功能。本文前期通过生物信息学分析,获得斑马鱼9个疑似活性转座子,qPCR检测结果表明,在斑马鱼早期发育阶段及成鱼各主要脏器,无论是DNA转座子还是反转录转座子均有表达活性,并具有时空特异性,在胚胎2 h及之前时期,9个转座子均无表达,3 h及之后几个阶段,转座子均出现表达活性,且不同转座子活性差异较大。转座子在成年鱼主要脏器也有表达活性,不同组织间差异显著。上述结果提示转座子的表达元件可能与胚胎发育和组织功能调控有关。
转座子发生转录的功能之一可能是产生非编码RNAs,如非洲爪蟾Tc1-2_Xt[22]。本文中9种转座子均发生转录,其中斑马鱼Tc1-a、Tc1-b、Tc1-c、Tc1-d、Tc1-e和非洲爪蟾的Tc1-2_Xt均为DNA转座子中Tc1/Mariner超家族成员,均在早期胚胎中表达,进一步证实这些Tc1/Mariner超家族成员可能参与早期胚胎发育调控。另外,本文实验结果还表明大脑和心脏中的表达水平显著高于其他4个组织,提示转座子在大脑和心脏组织中可能起重要生物学功能,Upton等[23]研究中也发现L1可能与脑细胞功能有关,在部分脑细胞表达和转座活性明显高于一切周围其他细胞,进一步证实L1可能在脑功能调控中发挥作用。本文发现大多转座子在睾丸组织中表达水平低,可能有利于保持遗传的稳定性。本文9种转座子均具有完整的转座酶编码序列,虽然本实验不能确定能否表达出活性转座酶,但在此基础上,本文将克隆并构建转座酶表达载体,进一步验证是否具有转座活性。
研究结果表明,在斑马鱼早期胚胎发育阶段,DNA转座子和RNA反转录转座子表达水平和表达趋势呈现出明显的差异。提示两类转座子发挥生物功能的机制可能不同。部分反转录转座子表达启动时间早于DNA转座子,但两者在胚胎2 h前均检测不出表达活性,说明早期胚胎母源mRNAs中无转座子或比例很低,转座子的表达可能对合子发育具有调控作用。
4 结论1) 9种转座子在斑马鱼胚胎早期不同阶段具有转录活性,其中5个DNA转座子从6 h阶段开始表达,总体呈现先下降再上升表达趋势;4个反转录转座子表达起始时间存在不一致性,最早为3 h,总体呈现持续上升表达趋势。
2) 9种转座子在成年鱼大脑、心脏、卵巢、肝脏、肌肉和睾丸均有转录活性,其中心脏和大脑的表达水平显著高于其他4种组织,为研究转座子功能提供重要参考。
| [1] |
NORIHIRO O. Transfer RNA-like structure of the human Alu family: implications of its generation mechanism and possible functions[J]. Journal of Molecular Evolution, 1990, 31(6): 500-510. DOI:10.1007/BF02102077 (  0) 0) |
| [2] |
MISKEY C, IZSVÁK Z, KAWAKAMI K, et al. DNA transposons in vertebrate functional genomics[J]. Cellular and Molecular Life Sciences, 2005, 62(6): 629-641. DOI:10.1007/s00018-004-4232-7 (  0) 0) |
| [3] |
FINNEGAN D J. Eukaryotic transposable elements and genome evolution[J]. Trends in Genetics, 1989, 5(4): 103-107. DOI:10.1016/0168-9525(89)90039-5 (  0) 0) |
| [4] |
LISCH D. How important are transposons for plant evolution?[J]. Nature Reviews Genetics, 2013, 14(1): 49-61. DOI:10.1038/nrg3374 (  0) 0) |
| [5] |
DOOLITTLE W F, SAPIENZA C. Selfish genes, the phenotype paradigm and genome evolution[J]. Nature, 1980, 284(5757): 601-603. DOI:10.1038/284601a0 (  0) 0) |
| [6] |
MANDAOKAR A, KUMAR V D, AMWAY M, et al. Microarray and differential display identify genes involved in jasmonate-dependent anther development[J]. Plant Molecular Biology, 2003, 52(4): 775-786. DOI:10.1023/A:1025045217859 (  0) 0) |
| [7] |
HIROCHIKA H. Contribution of the TosI7 retrotransposon to rice functional genomics[J]. Current Opinion in Plant Biology, 2001, 4(2): 118-122. DOI:10.1016/S1369-5266(00)00146-1 (  0) 0) |
| [8] |
MACIA A, BLANCO-JIMENEZ E, GARCÍA-PÉREZ J L. Retrotransposons in pluripotent cells: Impact and new roles in cellular plasticity[J]. Biochimica et Biophysica Acta (BBA) -Gene Regulatory Mechanisms, 2015, 1849(4): 417-426. DOI:10.1016/j.bbagrm.2014.07.007 (  0) 0) |
| [9] |
SLOTKIN R K, MARTIENSSEN R. Transposable elements and the epigenetic regulation of the genome[J]. Nature Reviews Genetics, 2007, 8(4): 272-285. DOI:10.1038/nrg2072 (  0) 0) |
| [10] |
MARINO-RAMIREZ L, LEWIS K C, LANDSMAN D, et al. Transposable elements donate lineage-specific regulatory sequences to host genomes[J]. Cytogenetic and Genome Research, 2005, 110: 333-341. DOI:10.1159/000084965 (  0) 0) |
| [11] |
SMALHEISER N R, TORVIK V I. Alu elements within human mRNAs are probable microRNA targets[J]. Trends in Genetics, 2006, 22(10): 532-536. DOI:10.1016/j.tig.2006.08.007 (  0) 0) |
| [12] |
IVICS Z, GARRELS W, MÁTÉS L, et al. Germline transgenesis in pigs by cytoplasmic microinjection of Sleeping Beauty transposons[J]. Nat Protoc, 2014, 9(4): 810-827. DOI:10.1038/nprot.2014.010 (  0) 0) |
| [13] |
IVICS Z, HIRIPI L, HOFFMANN O I, et al. Germline transgenesis in rabbits by pronuclear microinjection of Sleeping Beauty transposons[J]. Nat Protoc, 2014, 9(4): 794-809. DOI:10.1038/nprot.2014.009 (  0) 0) |
| [14] |
BAILLIE J K, BARNETT M W, UPTON K R, et al. Somatic retrotransposition alters the genetic landscape of the human brain[J]. Nature, 2011, 479(7374): 534-537. DOI:10.1038/nature10531 (  0) 0) |
| [15] |
GOODIER J L. Retrotransposition in tumors and brains[J]. Mobile DNA, 2014, 5(1): 11. DOI:10.1186/1759-8753-5-11 (  0) 0) |
| [16] |
St LAURENT Ⅲ G, HAMMELL N, McCAFFREY T A. A LINE-1 component to human aging: do LINE elements exact a longevity cost for evolutionary advantage?[J]. Mechanisms of Ageing and Development, 2010, 131(5): 299-305. DOI:10.1016/j.mad.2010.03.008 (  0) 0) |
| [17] |
GAO Bo, SHEN Dan, XUE Songlei, et al. The contribution of transposable elements to size variations between four teleost genomes[J]. Mobile DNA, 2016, 7(1): 4. DOI:10.1186/s13100-016-0059-7 (  0) 0) |
| [18] |
MALONE C D, HANNON G J. Small RNAs as guardians of the genome[J]. Cell, 2009, 136(4): 656-668. DOI:10.1016/j.cell.2009.01.045 (  0) 0) |
| [19] |
GIRARD A, HANNON G J. Conserved themes in small-RNA-mediated transposon control[J]. Trends in Cell Biology, 2008, 18(3): 136-148. DOI:10.1016/j.tcb.2008.01.004 (  0) 0) |
| [20] |
PARKHURST S M, CORCES V G. Developmental expression of Drosophila melanogaster retrovirus-like transposable elements[J]. The EMBO Journal, 1987, 6(2): 419-424. DOI:10.1002/j.1460-2075.1987.tb04771.x (  0) 0) |
| [21] |
PEASTON A E, EVSIKOV A V, GRABER J H, et al. Retrotransposons regulate host genes in mouse oocytes and preimplantation embryos[J]. Developmental Cell, 2004, 7(4): 597-606. DOI:10.1016/j.devcel.2004.09.004 (  0) 0) |
| [22] |
GREENE J M, OTANI H, GOOD P J, et al. A novel family of retrotransposon-like elements in Xenopus laevis with a transcript inducible by two growth factors[J]. Nucleic Acids Research, 1993, 21(10): 2375-2381. DOI:10.1093/nar/21.10.2375 (  0) 0) |
| [23] |
UPTON K R, GERHARDT D J, JESUADIAN J S, et al. Ubiquitous L1 mosaicism in hippocampal neurons[J]. Cell, 2015, 161(2): 228-239. DOI:10.1016/j.cell.2015.03.026 (  0) 0) |
 2017, Vol. 15
2017, Vol. 15


