尽管二代基因组测序(next-generation sequencing)技术日渐流行,Sanger测序依旧是SNP识别和分析的金标准。一般对Sanger测序结果的分析多依赖于Windows平台的SeqMan (DNASTAR Inc.)等软件进行。然而这类软件大多在测序数据与参考序列拼接后依靠肉眼观察来识别和记录测序结果中的SNP (single nucleotide polymorphism)位点,效率低下且容易发生错误。更为重要的是,当对多个个体进行序列测定时,这类软件无法完成对群体数据的自动比较、管理和输出,给研究人员带来了一定的不便。
Phred/Phrap/Consed/Polyphred是华盛顿大学(University of Washington)开发的基于类Unix平台(包括Unix、Linux、BSD、Solaris、Mac OS X等)的软件包[1-8],在群体测序数据的管理和SNP自动识别、标记与输出等方面具有强大的功能。然而,由于其安装、设置和使用较为复杂,在国内较少使用。鉴于此,本文对该软件包的组成、功能、使用和特点等进行了总结和介绍。
1 软件包主要组分该软件包由多个可执行文件和Perl脚本组成,主要组分及其功能见表 1。这些组分中,Phred[1-2]、Phrap、Consed[3-5]、Polyphred[6-8]是作为软件包核心的可执行程序,其余均为调用这些可执行程序完成特定功能的perl脚本。
| 表 1 软件包主要组分和功能 Table 1 The main component and function of the software package |
使用流程如图 1所示,具体说明如下。
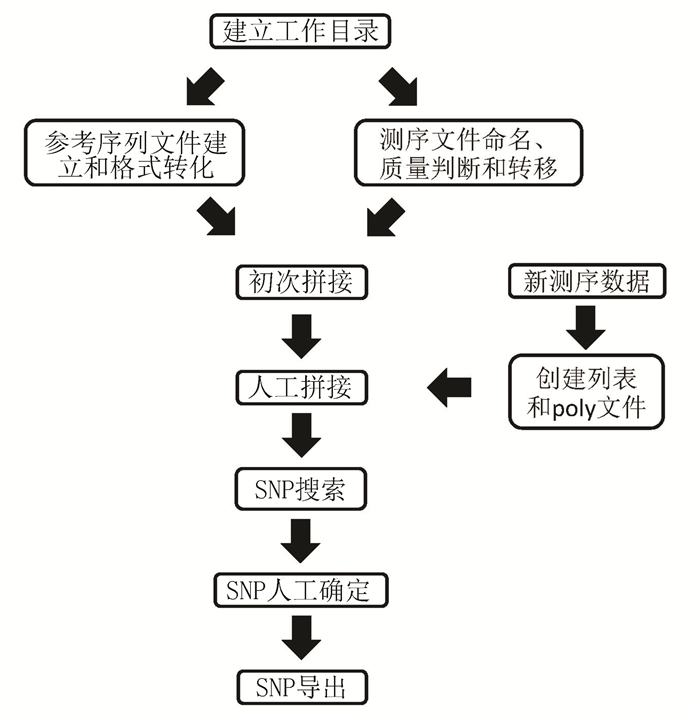
|
图 1 软件包的使用流程 Figure 1 The working flow of the software package |
在任意目录内,运行setup_std_dir.pl脚本,即可自动在该目录下建立工作目录,各个文件夹的名称和功能见表 2。
| 表 2 各个工作目录功能 Table 2 The function of each working directory |
人工生成fasta格式的参考序列并置于fasta_dir目录中,使用fasta2Phd.perl脚本转化为phd格式,并将其移入phd_dir目录。
2.3 测序数据文件命名、质量判断和转移将所有测序文件按照“基因名_个体名_引物名”的格式进行重命名,并保持大小写和长度一致,以方便后续SNP输出;将测序文件置于newchromats目录中,运行moveChromats脚本,质量较好的数据移入chromat_dir文件夹,低质量数据移入bad_chromats文件夹。
2.4 初次拼接进入edit_dir目录,使用phredPhrap脚本,对chromat_dir目录中的测序数据和phd_dir目录中的参考序列的进行初次拼接。
2.5 人工拼接由于算法的限制,初次拼接的结果往往为多个临近序列的集合(contig)。在这种情况下,在edit_dir目录中打开Consed界面,利用各个集合中可能重合的部分进行比对,将其拼接成一个完成的集合。具体方法见《使用说明》。
2.6 SNP搜索在edit_dir目录中运行Polyphred程序对每条序列进行等位基因确定,并对拼接的单个集合进行序列间比对,以确定潜在SNP位点。这些潜在的SNP位点,可在Consed界面中以高亮显示。
2.7 SNP人工确定在edit_dir目录中打开Consed界面,对于Polyphred搜索结果进行人工检视、修订和确认,以确定各条序列在该位置的基因型和SNP的真实性。这是整个数据处理过程中耗时最多的部分,具体方法见《使用说明》。
2.8 SNP导出生成包含需要导出个体名的文本文件。利用polyphredref2pb.pl脚本,指定其在测序文件名中的起始位置和长度,导出prettybase格式的群体变异文件,用于后续制图、计算或分析,具体方法见《使用说明》。
2.9 新数据加入对于新加入的测序数据,经测序数据文件命名、质量判断和转移步骤后,使用2fof.pl脚本,对chromat_dir目录中的测序数据和指定ace文件进行比对,生成新测序文件的列表(fof格式),并使用creatpoly.pl脚本对该列表中的新测序文件生成相关poly文件。在Consed界面中,将新测序数据加入集合,并重复人工拼接、SNP搜索、SNP人工确定、SNP导出步骤,对新增数据进行分析。
3 软件包特点 3.1 基于类Unix平台、命令行式操作方式该软件包主要可执行程序均只有类Unix操作系统版本,无Windows版本。除人工拼接、SNP搜索部分在Consed界面中以图形化方式操作外,其他主要工作均以命令的方式进行。这给初学者,尤其是不熟悉类Unix操作系统的学者,带来了一定的困难和麻烦。但是,由于需要掌握的命令不多,且类Unix操作系统多具有命令补齐和自动记录的功能,经一段时间练习后,大多使用者均可熟练使用该软件包。
3.2 标签式管理方式常见Windows平台的SeqMan等软件在遇到测序数据基因型判断失误、新发现SNP位点时,只能通过人工记录来保存数据分析过程和结果,极其不便且效率低下。与此不同的是,本软件包在序列处理过程中,Phred生成的质量评估结果、Polyphred生成的基因型判断、Consed界面中基因型的人工确认等各种处理分析结果,均以文本标签的形式,自动保存于ace或ace.out文件中。这些标签,由Consed读取,在界面中以不同颜色显示于序列相应位置上。所有标签,均可以手动在Consed界面中删除,或使用特定命令直接删除。在Polyphred自动生成的基因型与人工判断不一致时,优先输出人工标签。这种管理方式,在不改变原始测序文件的基础上,保存了数据处理过程的所有细节,同时方便了结果的输出。
3.3 数据管理、SNP数据输出的自动化常见Seqman等软件,在SNP识别、数据管理和输出方面,只能依赖操作者手动进行,效率低下且容易发生错误。本软件包在数据管理等方面具有较高程度的自动化,主要表现在:1)相同个体、引物多次测序数据的自动整理。在研究过程中,由于各种原因,有时需对同一个体应用同一引物进行多次测序,对于这种情况,软件会自动在测序文件名后加入“.1”、“.2”等后缀,分别代表第1、2次测序结果,并在后续分析中整合判断位点基因型;2)多态位点和质量的自动判断。对于测序结果,软件包会对每个个体进行比对,任何一个个体出现突变的位点,均视为潜在多态性位点,用特殊颜色标记以待人工检视;对于测序数据的低质量部分,会自动使用黄色背景表示,从分析中剔除;3)数据输出自动化。对于群体的测序数据,可输出每个个体的基因型,用于后续分析。
4 需要说明的问题 4.1 序列删除当需要去除特定序列时,可使用操作系统自带的文本编辑工具(如vi、Emacs、gedit等),建立一个含有需要去除测序文件名列表的文本文件,以回车键分隔(与下述fof后缀文本文件格式相同),并保存于edit_dir文件夹中。打开Consed界面,点击“Remove reads”,选择生成的文本文件,即可从集合中去除指定的测序数据。进而从chromat_dir文件夹中删除这些测序文件,以避免其在下一次加入新测序数据时被软件自动加入测序集合。
4.2 质量判断失误一般而言,该软件包对测序质量的判断较为准确。但当测序后纯化处理不当时,测序数据前端经常出现一个巨大的、横跨多个碱基的非特异峰形(俗称“酒精峰”),从而导致其下的碱基无法识别、后续的碱基峰图高度相对较低,因此Phred软件经常将其错误地标为低质量数据,而无法用于分析。对于这种情况,可在Consed中手动将其标为高质量数据,进入后续分析流程。
4.3 SNP自动识别的失效根据使用经验,Polyphred的SNP自动搜索算法阈值较低,因此有一定的假阳性(即实际并非多态性位点、而被软件标识)率,而假阴性(即实际为多态性位点但软件未能标识)发生的概率极低。本文使用该软件包分析了长达75.5 kb的测序数据、多达至800个SNP位点[9],仅发现了2个软件未识别的多态性位点,表明其假阴性率约在0.25%。假阴性的出现,多源于杂合子个体中两个等位基因的峰型高度差异过大、且稀有等位基因在群体中频率过低(导致无个体携带纯合稀有等位基因)。对于可能出现的假阴性,目前尚无良好、快速的解决办法,只能通过肉眼观察和比较测序胶图予以确定。
4.4 插入缺失(indel)的确定Polyphred软件具有自动识别indel的功能,但根据本文经验,其错误率较高、且无法识别indel的两个具体等位基因,因此建议对indel进行人工识别和标注(软件只允许标注为++、+-、--,不能标注具体序列)。对于两个等位基因都有纯合子个体的情况,可通过比对直接获得具体基因型。对于未发现稀有等位基因纯合子的情况,可通过分析杂合子个体序列胶图获得基因型信息。如图 2上部所示,从白色箭头所指的位置开始的15个碱基,每个位置的基因型为(A/T)(A/G)(A/T)(T/G)(T/T)(T/G)(T/T)(A/G)(T/T)(A/T)(A/T)(A/G)(T/T)(A/A)(A/G)。已知其中参考等位基因序列为TGTGTTTATAAGTAG(图 2下部),则另一等位基因序列为AAATTGTGTTTATAA,即为一个AAAT的插入。

|
图 2 Consed中杂合和纯合的indel示例 Figure 2 The example for homozygous and heterozygous indel in Consed *白色箭头代表indel起始的位置。 |
整个软件包处理过程中生成多种格式文件,大多为文本格式,主要文件格式说明如下。
phd:Phred对每个测序胶图文件均生成一个对应的同名phd格式文件,存放该测序胶图的碱基、质量、位置等信息[10]。
poly:Phred对每个测序胶图文件均生成一个对应的同名poly格式文件,存放该测序胶图的碱基、位置、峰面积等信息,用于后续突变检测。
ace:Consed读取、保存文件格式,保存有该contig中所有序列名称、碱基等信息。
wrk:Consed生成的临时文件,用于存储在Consed软件上的工作过程。当Consed意外停止运行时,可从wrk文件恢复之前的未保存修改。
ace.out: Polyphred输出格式文件,包含Polyphred运行时间、参数、结果,以及每条测序胶图在特定位点的基因型等信息。
prettybase:群体遗传学中常用的基因型数据文件格式,可用polyphredref2pb.pl脚本从ace.out文件中导出。一般由4列组成,第1列为位置,第2列为个体名,第3、4列为两个等位基因的基因型,其中A/C/T/G、+/-、N分别代表 4种碱基、indel、未确定碱基,X代表该个体不同测序数据存在矛盾,应人工检视以消除。
fof:2fof.pl脚本生成的新测序文件的列表,每行为一个新的测序文件名,以回车键分隔。
5 下载方法研究者可通过电子邮件向华盛顿大学各个软件负责人免费申请该软件包的非商业许可,联系方式见如下网页:http://www.phrap.org/index.html(Phred/Phrap/Consed)和http://droog.gs.washington.edu/polyphred/(Polyphred)。经包括本文通信作者在内的多名研究者安装测试表明,该软件包的Linux版本,可在Redhat 8.0、Ubuntu 8.04/12.04/14.04等不同GNU/Linux版本中正常运行和使用,表明该软件包具有较好的可移植性。
为方便研究者使用,本文将该软件包安装于Ubuntu 12.04操作系统中,下载链接http://pan.baidu.com/s/1slanyAL。研究者只需下载并导入VMware虚拟机,即可直接使用。详细使用说明见该压缩包中《使用说明》文件。
| [1] |
WING B, HILLIER L, WENDL M C, et al. Base-calling of automated sequencer traces using phred. Ⅰ. Accuracy assessment[J]. Genome Research, 1998, 8(3): 175-185. DOI:10.1101/gr.8.3.175 (  0) 0) |
| [2] |
EWING B, GREEN P. Basecalling of automated sequencer traces using phred. Ⅱ. Error probabilities[J]. Genome Research, 1998, 8(3): 186-194. DOI:10.1101/gr.8.3.186 (  0) 0) |
| [3] |
GORDON D. Viewing and Editing Assembled Sequences Using Consed[M]// BAXEVANIS A D, DAVISON D B, eds. In Current Protocols in Bioinformatics. New York: John Wiley & Co., 2004, 11.2.1-11.2.43. DOI: 10.1002/0471250953.bi1102s02.
(  0) 0) |
| [4] |
GORDON D, DESMARAIS C, GREEN P. Automated finishing with Autofinish[J]. Genome Research, 2001, 11(4): 614-625. DOI:10.1101/gr.171401 (  0) 0) |
| [5] |
GORDON D, ABAJIAN C, GREEN P. Consed: a graphical tool for sequence finishing[J]. Genome Research, 1998, 8(3): 195-202. DOI:10.1101/gr.8.3.195 (  0) 0) |
| [6] |
NICKERSON D A, TOBE V O, TAYLOR S L. Polyphred: automating the detection and genotyping of single nucleotide substitutions using fluorescence-based resequencing[J]. Nucleic Acids Research, 1997, 25(14): 2745-2751. DOI:10.1093/nar/25.14.2745 (  0) 0) |
| [7] |
STEPHENS M, SLOAN J S, ROBERTSON P D, et al. Automating sequence-based detection and genotyping of SNPs from diploid samples[J]. Nature Genetics, 2006, 38(3): 375-381. DOI:10.1038/ng1746 (  0) 0) |
| [8] |
BHANGALE T R, STEPHENS M, NICKERSON D A. Automating resequencing-based detection of insertion-deletion polymorphisms[J]. Nature Genetics, 2006, 38(12): 1457-1462. DOI:10.1038/ng1925 (  0) 0) |
| [9] |
SUN C, SOUTHARD C, HUO D, et al. SNP discovery, expression, and cis-regulation variation in the UGT2B family[J]. The Pharmacogenomics Journal, 2012, 12(4): 287-296. DOI:10.1038/tpj.2011.2 (  0) 0) |
| [10] |
王俊. 常用生物数据分析软件[M]. 北京: 科学出版社, 2008, 13-61. WANG Jun. Common analysis software for biological data[M]. Beijing: Science Press, 2008, 13-61. (  0) 0) |
 2017, Vol. 15
2017, Vol. 15


