2. 河南省-科技部共建细胞分化调控国家重点实验室培育基地(河南师范大学),河南 新乡 453007;
3. 漯河医学高等专科学校,河南 漯河 462002
2. State Key Laboratory Cultivation Base for Cell Differentiation Regulation and Henan Bioengineering Key Laboratory(Henan Normal University), Xinxiang 453007, Henan, China;
3. Luohe Medical College, Luohe 462002, Henan, China
在正常情况下,大部分肝细胞处于G0期,很少分裂,但是当受到某些机械、病毒、药物等刺激时,G0期细胞可以进入G1期,启动细胞周期。大鼠进行2/3肝切除后,剩余肝脏仍可代偿性的长到原来大小,并恢复其功能,这个过程称为肝再生(liver regeneration,LR)。很多激素、细胞因子、蛋白、信号通路参与肝再生过程,迄今为止,对肝再生过程还没有非常明确的了解,尤其是启动和终止阶段[1-3]。
溶酶体是广泛存在于真核细胞中对细胞功能有重要作用的细胞器,自噬(autophagy)是溶酶体介导的降解受损或冗余细胞成分为小分子物质的过程,降解后的物质可以被重新利用。这些功能主要是溶酶体膜蛋白和内腔中的酸性水解酶类来执行的。迄今为止,发现至少50种溶酶体酸性水解酶[4],这些水解酶直接行使催化功能,但溶酶体膜蛋白的作用也极其重要,比如,维持溶酶体内酸性环境,选择性的运输需要被降解的物质,介导溶酶体膜和其他膜的融合等[5]。自噬参与了机体很多重要的生理过程,如细胞发育、分化、衰老、死亡等[6-7]。越来越多的证据表明,自噬和人类的一些疾病和肿瘤发生有很大关系[8]。所以,自噬在细胞和机体的生命中扮演着重要角色。
关于肝再生与自噬的研究已有不少报道[9-13],研究发现,正常小鼠70%肝切除后,发生积极自噬而免于细胞老化,并通过维持健康的线粒体形态刺激线粒体代谢,通过氧化磷酸化为肝再生提供能量[14]。还有研究发现,肝再生早期,线粒体通透性没有明显改变,而24 h之后内膜通透性降低,氧化磷酸化偶联增强,效率提高[15]。为了解大鼠2/3肝切除肝再生与自噬的关系,自噬相关信号通路及相关蛋白在肝再生不同时间点的变化,并阐明其机制,本研究借助同位素标记相对和绝对定量(isobaric tags for relative and absolute quantitation,iTRAQ)结合质谱(mass spectrometry,MS)方法检测了大鼠肝再生2、6、12、24、30和36 h等6个时间点自噬相关蛋白的表达变化,并分析了他们的表达模式和相互作用及其与肝再生的关系。根据实验结果本文提出溶酶体和线粒体可能有共存的形式,且从分子水平阐释其存在的可能机理。除了自噬,真核细胞还有一种降解蛋白质的系统,现已有研究发现两种蛋白降解体系之间有着某种联系[16-19],但是机理有待研究。本文首次在蛋白水平分析了大鼠肝再生自噬与泛素介导的蛋白酶体途径之间可能的联系。对自噬和肝再生的研究对阐明肝再生的过程有重要的意义,并且可以对肝病的治疗提供新的思路和方法。
1 材料和方法 1.1 蛋白质组检测与数据分析实验Sprague-Dawle(SD)成年健康雄性大鼠由河南师范大学实验动物中心提供,体重210~250 g。在控制温度(20±3℃),相对湿度(60±10%),光照时间(8:00-20:00)条件下,实验大鼠可以自由饮水,摄食。取上述大鼠78只分为13组,每组6只,其中6组假手术(operation control, OC),6组肝切除(partial hepatectomy, PH),1组正常对照normal control group (NC)。参照实验室以前的方法构建大鼠2/3肝切除模型。简单来说,乙醚吸入法麻醉大鼠,腹部消毒,无菌条件下从剑突下方约1~2 cm处沿腹中线向上开腹至剑突,同时压迫大鼠两侧肋骨,将占全肝重68%的肝左,中叶挤出腹腔外,结扎蒂部,沿结扎线外侧切除肝叶,缝合切口,撒上磺胺以防感染。于固定时间取上述0、2、6、12、24、30、36 h的PH大鼠,乙醚吸入法麻醉,75%酒精消毒腹部皮肤,“十字”打开腹腔,取肝右叶。混合每组6只大鼠的肝组织置于液氮保存备用。SO组不进行肝切除,其他同PH组。
蛋白提取及iTRAQ8标同位素标记和肽段分离,方法详见文献[20]。概括说,组织样品加入500 μL SDT裂解液,匀浆后,进行超声和沸水浴裂解,离心取上清,BCA方法蛋白质定量后,-80℃冰箱保存。FASP酶解,OD280肽段定量。各组样品肽段分别取约80 μg, 按照AB公司试剂盒:iTRAQ Reagent-8plex Multiplex Kit(AB SCIEX)说明书进行标记。将标记后的所有肽段混合。样品采用AKTA Purifier 100 (GE Healthcare)液相系统进行分离。每份样品经毛细管高效液相色谱分离后用Q-Exactive质谱仪(Thermo Finnigan)进行质谱分析。具体为:分析时长为60 min,检测方式为正离子;母离子扫描范围为300~1 800 m/z;一级质谱分辨率为70 000 at m/z 200;AGC target为3e6;一级Maximum IT为10 ms;Number of scan ranges为1,Dynamic exclusion为40.0 s。多肽和多肽的碎片的质量电荷比按照下列方法采集:每次全扫描(full scan)后采集10个碎片图谱(MS2 scan)为MS2 Activation Type为HCD;Isolation window为2 m/z;二级质谱分辨率为17 500 at m/z 200;Microscans为1;二级Maximum IT为60 ms;Normalized collision energy为30eV;Underfill ratio为0.1 %。质谱分析原始数据为RAW文件,查库时将RAW文件通过Proteome Discoverer提交至Mascot服务器,选择已经建立好的数据库,然后进行数据库搜索。采用Proteome Discoverer1.4软件对肽段报告离子峰强度值进行定量分析。以混合样品通道标签为内参,各组蛋白质的iTRAQ比值均采取各通道标签与内参的比值(ratio值)。当ratio值≥对照2倍时,认为蛋白发生有意义表达上调,当ratio值≤对照0.5倍,认为蛋白发生有意义表达下调。
以“autophagy”为关键词在NCBI、GENEONTOLOGY、CAGP,GENMAPP、KEGG、QIAGEN和RGD等网站进行搜索,确认大鼠自噬相关基因和信号通路,有异议的进一步查阅文献进行确认。经查询确定mTOR,AMPK,PI3K/AKT,p53和Hedgehog等5条信号通路与自噬相关[21-25],并把自噬分为溶酶体、自噬、自噬调节和线粒体自噬4个生理过程。
1.2 统计与作图Cluster3.0和Treeview进行热图分析。具体为,将表达谱数据整理成txt文本文件,导入Cluster3.0软件,对gene和array进行聚类,选择Average linkage并生成cdt文件。把cdt文件导入Treeview软件生成层次聚类图。将iTRAQ检测的自噬及调节它们的信号通路的肝再生相关蛋白表达数据上传至IPA软件内置的Ingenuity © Knowledge Base数据库进行Heatmap分析[26]。
2 结果与分析iTRAQ检测到大鼠肝再生自噬相关蛋白共64种,其中,mTOR,AMPK,PI3K/AKT,p53和Hedgehog共5条信号通路与溶酶体、自噬、自噬调节和线粒体自噬等生理活动相关蛋白分别为4、12、35、18、6、6、1、4、3种。PSMD2、PSMA1、GFEF等蛋白上调显著,IL6、GAA、PARL等下调显著。对64种自噬相关蛋白聚类发现,第1聚类主要是6个时间点变化不是很明显,仅12 h有轻微下调的蛋白。第2聚类主要是几乎全部上调蛋白,且有些蛋白上调显著。第3聚类主要是30 h变化差异的蛋白。第4聚类主要是下调的蛋白。第5聚类主要是30和36 h表达下调,和前4个时间点不一致的蛋白如图 1所示。
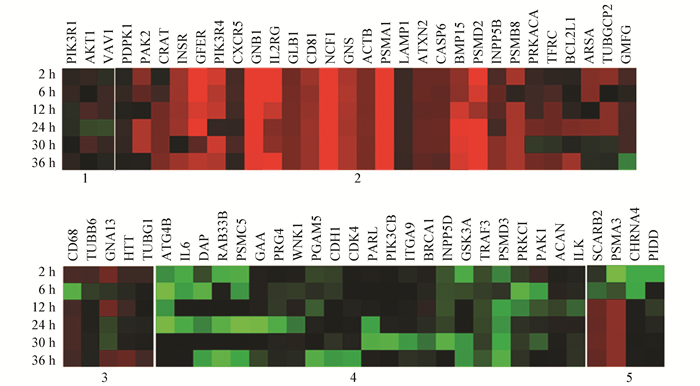
|
图 1 大鼠肝再生自噬相关蛋白聚类分析 Figure 1 The clustering analysis of proteins associated with autophagy in LR |
根据文献,本文把检测的6个时间点划分为两个阶段[27]。0~6 h为肝再生启动阶段,6~36 h为进展阶段。各蛋白表达变化及所参与的信号通路及各信号通路之间的联系如图 2所示。
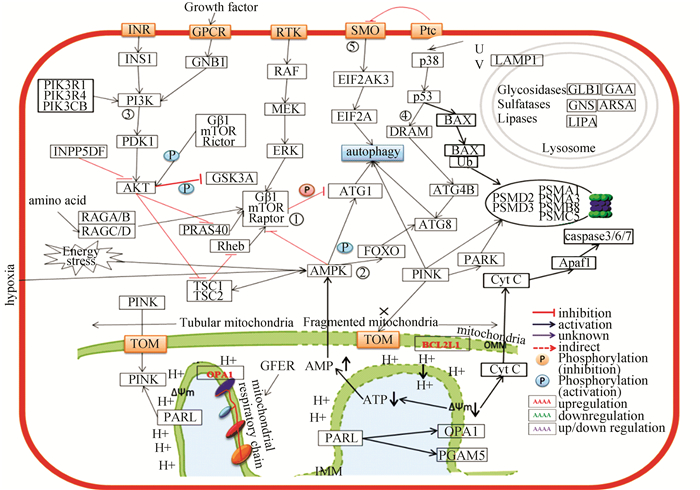
|
图 2 自噬相关蛋白之间的联系 Figure 2 The relationship of the proteins associated with autophagy |
肝切除后的低ATP状态,激活AMPK信号通路。而AMPK信号通路通过两条途径对自噬产生作用。一是直接磷酸化抑制raptor从而解除其对ATG1的抑制,诱导自噬。二是直接磷酸化ATG1,诱导自噬。另外,AMPK信号通路可以通过FOXO途径影响ATG8,影响自噬。AKT通过对TSC1/2的抑制作用,解除后者对Rheb的抑制,从而激活mTORC1复合体,最终抑制自噬的发生。Hedgehog信号通路和肝再生有着非常重要的关系,但是具体机制仍不明确,其可能通过EIF2AK3和EIF2A影响自噬,而本文未检测到这两种蛋白的变化。另外,GFER明显表达增多,和26S蛋白酶体相关的蛋白表达异常。所检测到的溶酶体内的酶,除GAA外,其余几乎都上调,溶酶体膜蛋白LAMP1,LAMP2皆表达增强。线粒体自噬相关蛋白OPA1表达明显上调,而OPA1是维持线粒体内膜完整性和电位差的重要的蛋白,这可以推测线粒体自噬没有大量发生。
根据图 3IPA结果可见,和自噬相关的5条信号通路中,有4条信号发生了有意义变化,其中,2条活性增强,即AMPK信号通路在大鼠肝再生的2~36 h信号转导活性增强,PI3K/Akt信号通路在大鼠肝再生的12、30 h信号转导活性增强。1条活性减弱,即p53信号通路在大鼠肝再生的2、6、24、30、36 h信号转导活性减弱。1条在不同时间点的变化不同,即mTOR信号通路在大鼠肝再生的2、6、30、36 h信号转导活性增强,在12 h信号转导活性减弱。
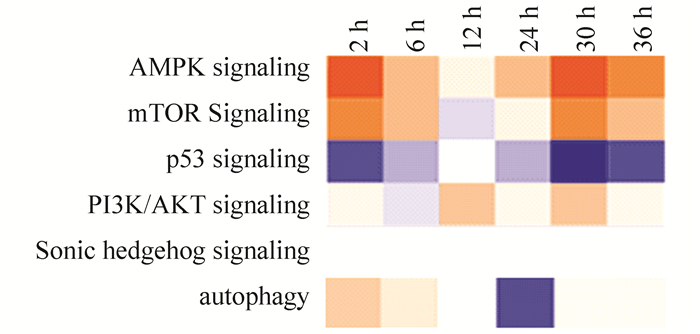
|
图 3 自噬及相关信号通路相关蛋白表达变化预示的自噬及信号转导活性 Figure 3 The comparison of canonical pathway and signaling pathways associated with autophagy 注:橙色为自噬反应活动增强,蓝色为自噬反应活动减弱。 |
在正常条件下,自噬的发生处于基础水平,但是在饥饿、低氧、胞内压力、高温、生长因子或激素缺乏等条件下,自噬急剧上升,以应对胞内ATP和构件分子的缺乏[28-29]。对细胞来说,维持自噬的基础水平非常重要,因为破损或衰老的细胞器,折叠错误的蛋白质在体内的聚集都会产生严重的后果,所以通过自噬的消除作用,提供能量的同时,还可以重新利用一些消化后的产物,维持细胞稳定[8]。大鼠2/3肝切除后,ATP含量迅速降低。而剩余肝脏在短时间内就可以重建,这个过程受到非常复杂的调控,各种激素,信号分子等参与其中,几乎所有的生化代谢过程都受到影响。
IPA分析结果可以发现,和自噬相关的信号通路除p53减弱外,而其余信号通路几乎多不同程度的被激活,自噬也反应活动增强。p53可能通过两个途径对自噬发挥作用,一个是核内调控转录因子,一个是调控线粒体[30-34],而以往所认知的p53和自噬的关系究竟是不是通过调控下游因子DRAM现在也受到一些挑战[35],这都有待进一步研究。Hedgehog信号通路作为最早发现的和肝再生相关的信号通路,其具体机制至今不是很清楚。它和自噬有着一定的联系,但具体的中介连接分子也不很确定[21, 36-38]。
线粒体自噬是自噬一种特殊形式,可以选择性降解功能障碍线粒体。自噬不足和过度都会导致细胞损伤和死亡。OPA1(optic atrophy 1)是核基因编码的线粒体蛋白,在维持内膜嵴的完整,呼吸链的完整等方面起着非常作用,而这直接和氧化磷酸化产生ATP相关[39-40]。OPA1在肝再生的6个时间点都发生明显上调,尤其是进展阶段。高表达的OPA1对线粒体的融合很重要,这也可以推测线粒体在肝再生中维持着良好的形态和功能,并没有形成大量片段化,而线粒体片段化对自噬是必需的,这应该和肝再生阶段大量的能量需求有着很重要的关系。在正常线粒体膜电位下,PARL(presenilin-associated rhomboid-like)会切割PINK1(PTEN induced putative kinase 1),一种线粒体丝氨酸/苏氨酸蛋白激酶,但在线粒体膜电位降低时,PARL则会切割PGAM5 (phosphoglycerate mutase family member 5),也就是说PARL会根据线粒体的状态选择不同的切割对象,因此它是一种比较独特的可以感应细胞压力的驻线粒体磷酸酶[41]。本文发现,PGAM5在2、24、36 h下调,其余时间点未发生变化。这可以推测肝切除后线粒体并未发生明显损伤,膜电位正常。
和肝再生与线粒体生成都有关系的肝再生增强因子ALR(augmenter of liver regeneration)在肝再生的整个阶段都高表达[42-43],尤其是进展阶段。而其表达降低阻碍线粒体生成,也会诱发细胞凋亡[44]。大鼠2/3肝切除后的6 h内ATP一直处于下降状态[14],而肝再生需要大量ATP供应[45],作为机体能量的发电站,线粒体功能直接影响ATP的供应。溶酶体跨膜蛋白LAMP1(Lysosomal-associated membrane protein 1)在肝再生的所有时间点均上调,这对溶酶体膜的完整,pH的维持和正常代谢起到非常重要的作用的作用。另外溶酶体中参与肝素等代谢的GNS(glucosamine (N-acetyl)-6-sulfatase), GLB1(galactosidase) CD81(cluster of differentiation 81),INPP5B(inositol polyphosphate-5-phosphatase B),CRAT(carnitine acetyltransferase),GLB1(Galactosidase, beta 1),ACTB(beta-actin)在肝再生整个阶段都明显上调。溶酶体内水解糖原的GAA(glucosidase)在2、12 h下调其余时间点没变化。GAA缺乏可能会因为溶酶体和细胞质之间形成渗透压梯度而导致溶酶体肿胀。而GAA基因敲除小鼠自噬泡增加,自噬功能改变。巧合的是,骨骼肌raptor(mTORC1复合体的重要亚基)基因敲除小鼠的表型与GAA非常相似[46],预示两者之间在自噬方面可能存在某些联系。溶酶体内氨基酸水平的改变导致自噬泡H+-ATP酶构象改变,mTORC1从细胞质到溶酶体迁移。有趣的是,mTORC1的上游激活剂Rheb也会在溶酶体膜上出现,并且受生长因子刺激而激活。在生长因子刺激的Rheb活性和氨基酸诱导的mTORC1的募集,共同导致mTORC1的充分活化。因此,考虑到GAA敲除小鼠溶酶体的肿胀,mTORC1的激活可能是对正常的溶酶体功能丧失的一种妥协。
综合上述分析,线粒体自噬在大鼠肝再生中起到非常重要的作用。大鼠肝切除后,合成活动旺盛,需要大量的能量供应。和能量相关的AMPK信号通路激活,以对抗这种应激状态。而和自噬直接相关的溶酶体膜蛋白和大量酸性水解酶表达上调,线粒体内膜相关蛋白表达增强,意味着线粒体功能并未受到大的影响,根据以上分析,本文提出一个设想,溶酶体和线粒体可以融合共存。也就是内体溶酶体和自噬体融合后,并未发生传统的线粒体完全被消化,而是像十几亿年前的原真核细胞吞噬原线粒体一样,线粒体内膜与溶酶体外膜共存,这无论从能量和节约体内物质成分,还是从快速提供能量上,都不失为一种合理的存在方式,示意图如图 4(a)所示,其共存机理如图 4(b)所示。线粒体内外膜间隙为酸性环境,而溶酶体内也是酸性环境,这就为它们的共存提供了可能。线粒体内膜电子传递链不断向膜间隙泵出电子,产生电位差。膜间隙的酸性环境对于酸性水解酶的催化作用必不可少,而水解后的成分转运出共存体,可以满足肝再生的需要,而H+向线粒体基质中的回流又可以产生ATP供机体需要。这样也就不难理解泛素-蛋白酶体的相关蛋白、溶酶体膜蛋白、线粒体膜蛋白等的不同寻常的表达变化。值的注意是GFER在其中究竟起了什么作用还不得知,但它的表达变化却暗示着其和肝再生的关系或许与此有关。
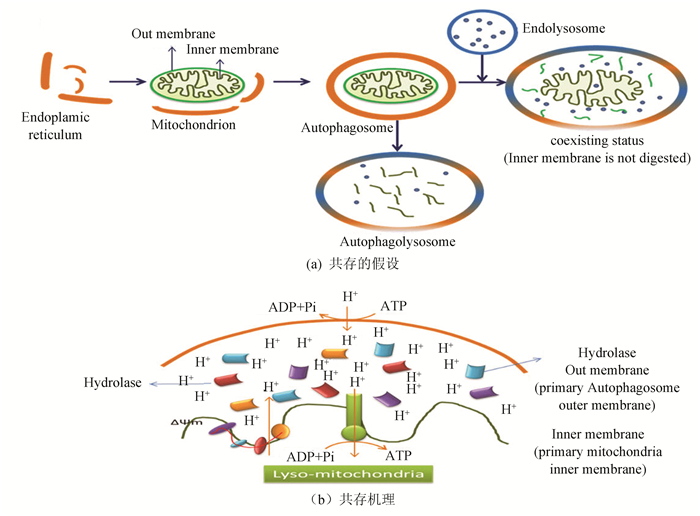
|
图 4 溶酶体和线粒体共存及可能性 Figure 4 The hypothesis of coexistance of lysosome and mitochondrion and the mechanism of coexistence of lysosome and mitochondria |
韩淑媛等[47]研究发现,肝切除后15 h左右,溶酶体数量和体积明显增加,许多细胞器衰退,唯独溶酶体数量增加并形成多种形式的自噬体,这表明细胞进入S期前,结构更新加速,溶酶体自噬增加,以清除这些衰退结构,这是细胞增殖的必要前提条件。缪明永等[15, 48]对肝再生中线粒体变化的长期研究发现,肝再生中线粒体通过降低线粒体内膜的通透性继而增强氧化磷酸化来适应再生中的能量需求,并且提出可以有两个方面增强线粒体功能,一是表达合成大量与氧化磷酸化有关的蛋白质提高ATP产能,但这会消耗很多ATP。二是直接调节线粒体内膜性质,呼吸链结构或内环境来提高氧化磷酸化的效率。显然,第2种比较合理。而线粒体溶酶体共存假说可以解释这个问题。早期,溶酶体外膜和线粒体外膜融合或把线粒体外膜降解,那么溶酶体本身含有的内含物和酶可以直接把物质降解成小分子跨越线粒体内膜进入线粒体,但是这个时候因为氧气下降,所以呼吸链功能不会太强,而溶酶体内部的酸性环境直接可以提供质子浓度梯度,从ATP合酶进入内膜,合成ATP,所以这个时候ATP不会下降太多。而后期24 h之后,随着功能的恢复,线粒体功能逐渐恢复,呼吸链恢复正常。已有实验证实,通过降低pH人为的加在线粒体膜质侧一个质子质量分数,即使缺少氧化型底物,也能合成ATP[49]。翟心慧等[50]研究发现,辅酶Q10在肝再生增值期有明显促进线粒体氧化磷酸化功能,且效果和剂量正相关,其中以pH后48 h最明显。依共存理论,肝再生初期,ATP生成溶酶体建立的质子梯度对ATP的生成起了重要的作用,那么前期添加CoQ10对氧化磷酸化的影响不显著而后期显著(后期线粒体内膜通透性恢复)就不难理解了。
泛素-蛋白酶体(the ubiquitin-proteasomes system,UPS)和自噬作为真核细胞两个最主要的蛋白降解系统,尤其本文发现UPS相关蛋白表达异常,很有必要分析下它和自噬及肝再生的相互联系。蛋白酶体降解得到的3~25个氨基酸残基的寡肽或多肽,最终要经由溶酶体降解[16-17]。研究发现,UPS抑制剂可以诱导自噬,这可能是作为UPS系统的一种补偿作用[16, 18]。如果UPS系统被抑制,短寿命和错误折叠蛋白质会增多,最终导致细胞毒性。持续的氨基酸缺乏导致蛋白质合成受阻,最终导致细胞凋亡。低氨基酸浓度会影响到mTOR信号通路,导致自噬活动增强。研究发现,大鼠2/3肝脏切除后,PSMA1,PSMD2,PSMB8在0~36 h明显上升甚至几百倍。PSMC5,PSMD3则在0~36 h一直明显下调。PSMA3则在0~24 h明显上升,30~36 h下调。这都显示26S蛋白酶体在大鼠2/3肝切除后发挥了重要作用。这一方面可能与受损肝脏产生大量受损蛋白需要清除有关,另一方面可能和肝切除后细胞增殖需要大量的氨基酸原料有关系,也可能参与溶酶体本身的构建。
4 结语大鼠肝切除后,能量的大量需求和组织损伤形成一种矛盾,而自噬可以在一定程度上解决这个问题。根据能量需求和自噬相关蛋白的表达变化,本文提出酶体和线粒体可以融合共存假说。无论从能量和节约体内物质成分,还是从快速提供能量上,都不失为一种合理的存在方式。如果本文提出的线粒体溶酶体共存假说成立,将对理解细胞功能和细胞器进化都有一定的启发作用。
| [1] |
ALISON M R, LIN W R. Diverse routes to liver regeneration[J]. The Journal of pathology, 2016, 238(3): 371-374. DOI:10.1002/path.4667 (  0) 0) |
| [2] |
MIYAOKA Y, MIYAJIMA A. To divide or not to divide: revisiting liver regeneration[J]. Cell Division, 2013, 8(1): 8. DOI:10.1186/1747-1028-8-8 (  0) 0) |
| [3] |
LAI H S, CHUNG Y C, CHEN W J, et al. Rat liver regeneration after partial hepatectomy: effects of insulin, glucagon and epidermal growth factor[J]. Journal of the Formosan Medical Association, 1992, 91(7): 685-690. DOI:10.1016/s0261-5614(13)60379-9 (  0) 0) |
| [4] |
SAFTIG P, KLUMPERMAN J. Lysosome biogenesis and lysosomal membrane proteins: trafficking meets function[J]. Nature reviews Molecular Cell Biology, 2009, 10(9): 623-635. DOI:10.1038/nrm2745 (  0) 0) |
| [5] |
GONZALEZ A, VALEIRAS M, SIDRANSKY E, et al. Lysosomal integral membrane protein-2: a new player in lysosome-related pathology[J]. Molecular Genetics and Metabolism, 2014, 111(2): 84-91. DOI:10.1016/j.ymgme.2013.12.005 (  0) 0) |
| [6] |
NIKOLETOPOULOU V, MARKAKI M, PALIKARAS K, et al. Crosstalk between apoptosis, necrosis and autophagy[J]. Biochimica et Biophysica Acta, 2013, 1833(12): 3448-3459. DOI:10.1016/j.bbamcr.2013.06.001 (  0) 0) |
| [7] |
CHEN Wenqi, SUN Yinyi, LIU Kangyong, et al. Autophagy: a double-edged sword for neuronal survival after cerebral ischemia[J]. Neural Regeneration Research, 2014, 9(12): 1210-1216. DOI:10.4103/1673-5374.135329 (  0) 0) |
| [8] |
崔丹蕊, 刘波, 刘伟. 细胞自噬与肿瘤发生关系的研究进展[J]. 中国科学:生命科学, 2015, 45(6): 593-603. CUI Danrui, LIU Bo, LIU Wei. The role of autophagy in tumorigenesis[J]. Scientia Sinica Vitae, 2015, 45(6): 593-603. DOI:10.1360/n052015-00107 (  0) 0) |
| [9] |
WANG Kewei. Autophagy and apoptosis in liver injury[J]. Cell Cycle, 2015, 14(11): 1631-1642. DOI:10.1080/15384101.2015.1038685 (  0) 0) |
| [10] |
王祺文, 常翠芳, 谷宁宁, 等. 自噬在肝再生中的作用[J]. 遗传, 2015, 37(11): 1116-1124. WANG Qiwen, CHANG Cuifang, GU Ningning, et al. Effect of autophagy on liver regeneration[J]. Hereditas(Beijing), 2015, 37(11): 1116-1124. DOI:10.16288/j.yczz.15-162 (  0) 0) |
| [11] |
VARGA M, SASS M, PAPP D, et al. Autophagy is required for zebrafish caudal fin regeneration[J]. Cell Death and Differentiation, 2014, 21(4): 547-556. DOI:10.1038/cdd.2013.175 (  0) 0) |
| [12] |
LIN C W, CHEN Y S, LIN C C, et al. Amiodarone as an autophagy promoter reduces liver injury and enhances liver regeneration and survival in mice after partial hepatectomy[J]. Scientific Reports, 2015, 5: 15807. DOI:10.1038/srep15807 (  0) 0) |
| [13] |
毛一雷, 陈蓉蓉, 杨华瑜, 等. 自噬现象在肝纤维化和肝切除时的表现形式[J]. 中国医学科学院学报, 2008, 30(4): 421-425. MAO Yilei, CHEN Rongrong, YANG Huayu, et al. Autophagy in fibrotic and postoperative remnant liver in rat[J]. Acta Academiae Medicinae Sinicae, 2008, 30(4): 421-425. DOI:10.3881/j.issn:1000-503x.2008.04.012 (  0) 0) |
| [14] |
TOSHIMA T, SHIRABE K, FUKUHARA T, et al. Suppression of autophagy during liver regeneration impairs energy charge and hepatocyte senescence in mice[J]. Hepatology, 2014, 60(1): 290-300. DOI:10.1002/hep.27140 (  0) 0) |
| [15] |
缪明永, 蔡在龙, 刘鹏飞, 等. 大鼠肝再生过程中线粒体氧化磷酸化的调控[J]. 中国病理生理杂志, 1999, 15(5): 398-401. MIAO Mingyong, CAI Zailong, LIU Pengfei, et al. Regulation of mitochondrial oxidation phosphorylation during rat regeneration after partial hepatectomy[J]. Chinese Journal of Pathophysiology, 1999, 15(5): 398-401. DOI:10.3321/j.issn:1000-4718.1999.05.005 (  0) 0) |
| [16] |
SANDRI M. Protein breakdown in muscle wasting: role of autophagy-lysosome and ubiquitin-proteasome[J]. The International Journal of Biochemistry & Cell Biology, 2013, 45(10): 2121-2129. DOI:10.1016/j.biocel.2013.04.023 (  0) 0) |
| [17] |
SCHREIBER A, PETER M. Substrate recognition in selective autophagy and the ubiquitin-proteasome system[J]. Biochimica et Biophysica Acta, 2014, 1843(1): 163-181. DOI:10.1016/j.bbamcr.2013.03.019 (  0) 0) |
| [18] |
WOJCIK S. Crosstalk between autophagy and proteasome protein degradation systems: possible implications for cancer therapy[J]. Folia Histochemica et Cytobiologica, 2013, 51(4): 249-264. DOI:10.5603/fhc.2013.0036 (  0) 0) |
| [19] |
SCHLOSSAREK S, FREY N, CARRIER L. Ubiquitin-proteasome system and hereditary cardiomyopathies[J]. Journal of Molecular and Cellular Cardiology, 2014, 71: 25-31. DOI:10.1016/j.yjmcc.2013.12.016 (  0) 0) |
| [20] |
GENG Xiaofang, WANG Gaiping, QIN Yanli, et al. iTRAQ-based quantitative proteomic analysis of the initiation of head regeneration in planarians[J]. PloS One, 2015, 10(7): e0132045. DOI:10.1371/journal.pone.0132045 (  0) 0) |
| [21] |
DU TOIT A. Cell signalling: hedgehog puts a damper on autophagy[J]. Nature Reviews Molecular Cell Biology, 2013, 14(1): 3. DOI:10.1038/nrm3487 (  0) 0) |
| [22] |
KIM J, KUNDU M, VIOLLET B, et al. AMPK and mTOR regulate autophagy through direct phosphorylation of Ulk1[J]. Nature Cell Biology, 2011, 13(2): 132-141. DOI:10.1038/ncb2152 (  0) 0) |
| [23] |
NAZIO F, STRAPPAZZON F, ANTONIOLI M, et al. mTOR inhibits autophagy by controlling ULK1 ubiquitylation, self-association and function through AMBRA1 and TRAF6[J]. Nature Cell Biology, 2013, 15(4): 406-416. DOI:10.1038/ncb2708 (  0) 0) |
| [24] |
NERI L M, CANI A, MARTELLI A M, et al. Targeting the PI3K/Akt/mTOR signaling pathway in B-precursor acute lymphoblastic leukemia and its therapeutic potential[J]. Leukemia, 2013, 28(4): 739-748. DOI:10.1038/leu.2013.226 (  0) 0) |
| [25] |
XU Xiuduan, WANG Hongli, LIU Shasha, et al. TP53-dependent autophagy links the ATR-CHEK1 axis activation to proinflammatory VEGFA production in human bronchial epithelial cells exposed to fine particulate matter (PM2.5)[J]. Autophagy, 2016, 12(10): 1832-1848. DOI:10.1080/15548627.2016.1204496 (  0) 0) |
| [26] |
高山, 欧剑虹, 肖凯. R语言与Bioconductor生物信息学应用[M]. 天津: 天津出版传媒集团, 2014.
(  0) 0) |
| [27] |
TAUB R. Liver regeneration: from myth to mechanism[J]. Nature Reviews Molecular Cell Biology, 2004, 5(10): 836-847. DOI:10.1038/nrm1489 (  0) 0) |
| [28] |
MELENDEZ A, NEUFELD T P. The cell biology of autophagy in metazoans: a developing story[J]. Development, 2008, 135(14): 2347-2360. DOI:10.1242/dev.016105 (  0) 0) |
| [29] |
KIEL J A K. Autophagy in unicellular eukaryotes[J]. Philosophical Transactions of the Royal Society of London Series B, Biological Sciences, 2010, 365(1541): 819-830. DOI:10.1098/rstb.2009.0237 (  0) 0) |
| [30] |
MRSCHTIK M, O'PREY J, LAO L Y, et al. DRAM-3 modulates autophagy and promotes cell survival in the absence of glucose[J]. Cell Death and Differentiation, 2015, 22(10): 1714-1726. DOI:10.1038/cdd.2015.26 (  0) 0) |
| [31] |
ZHANG Xingding, WANG Ye, WANG Yan, et al. p53 mediates mitochondria dysfunction-triggered autophagy activation and cell death in rat striatum[J]. Autophagy, 2009, 5(3): 339-350. DOI:10.4161/auto.5.3.8174 (  0) 0) |
| [32] |
O'PREY J, SKOMMER J, WILKINSON S, et al. Analysis of DRAM-related proteins reveals evolutionarily conserved and divergent roles in the control of autophagy[J]. Cell Cycle, 2009, 8(14): 2260-2265. DOI:10.4161/cc.8.14.9050 (  0) 0) |
| [33] |
MAIURI M C, MALIK S A, MORSELLI E, et al. Stimulation of autophagy by the p53 target gene Sestrin2[J]. Cell Cycle, 2009, 8(10): 1571-1576. DOI:10.4161/cc.8.10.8498 (  0) 0) |
| [34] |
CRIGHTON D, WILKINSON S, RYAN K M. DRAM links autophagy to p53 and programmed cell death[J]. Autophagy, 2007, 3(1): 72-74. DOI:10.4161/auto.3438 (  0) 0) |
| [35] |
CRIGHTON D, WILKINSON S, O'PREY J, et al. DRAM, a p53-induced modulator of autophagy, is critical for apoptosis[J]. Cell, 2006, 126(1): 121-134. DOI:10.1016/j.cell.2006.05.034 (  0) 0) |
| [36] |
JIMENEZ-SANCHEZ M, MENZIES F M, CHANG Y Y, et al. The Hedgehog signalling pathway regulates autophagy[J]. Nature Communications, 2012, 3: 1200. DOI:10.1038/ncomms2212 (  0) 0) |
| [37] |
LO RE A E, FERNANDEZ-BARRENA M G, ALMADA L L, et al. Novel AKT1-GLI3-VMP1 pathway mediates KRAS oncogene-induced autophagy in cancer cells[J]. The Journal of Biological Chemistry, 2012, 287(30): 25325-25334. DOI:10.1074/jbc.M112.370809 (  0) 0) |
| [38] |
WANG Ying, HAN Chang, LU Lu, et al. Hedgehog signaling pathway regulates autophagy in human hepatocellular carcinoma cells[J]. Hepatology, 2013, 58(3): 995-1010. DOI:10.1002/hep.26394 (  0) 0) |
| [39] |
SONG Zhiyin, GHOCHANI M, MCCAFFERY J M, et al. Mitofusins and OPA1 mediate sequential steps in mitochondrial membrane fusion[J]. Molecular Biology of the Cell, 2009, 20(15): 3525-3532. DOI:10.1091/mbc.E09-03-0252 (  0) 0) |
| [40] |
LANDES T, LEROY I, BERTHOLET A, et al. OPA1 (dys)functions[J]. Seminars in Cell & Developmental Biology, 2010, 21(6): 593-598. DOI:10.1016/j.semcdb.2009.12.012 (  0) 0) |
| [41] |
LU Wei, KARUPPAGOUNDER S S, SPRINGER D A, et al. Genetic deficiency of the mitochondrial protein PGAM5 causes a Parkinson's-like movement disorder[J]. Nature Communications, 2014, 5: 4930. DOI:10.1038/ncomms5930 (  0) 0) |
| [42] |
GANDHI C R. Augmenter of liver regeneration[J]. Fibrogenesis & Tissue Repair, 2012, 5(1): 10. DOI:10.1186/1755-1536-5-10 (  0) 0) |
| [43] |
ILOWSKI M, KLEESPIES A, DE TONI E N, et al. Augmenter of liver regeneration (ALR) protects human hepatocytes against apoptosis[J]. Biochemical and Biophysical Research Communications, 2011, 404(1): 148-152. DOI:10.1016/j.bbrc.2010.11.083 (  0) 0) |
| [44] |
FRANCAVILLA A, PESETTI B, BARONE M, et al. Transient GFER knockdown in vivo impairs liver regeneration after partial hepatectomy[J]. The International Journal of Biochemistry & Cell Biology, 2014, 53: 343-351. DOI:10.1016/j.biocel.2014.05.029 (  0) 0) |
| [45] |
HAN Lihong, DONG Lingyue, YU Hao, et al. Deceleration of liver regeneration by knockdown of augmenter of liver regeneration gene is associated with impairment of mitochondrial DNA synthesis in mice[J]. American Journal of Physiology Gastrointestinal and Liver Physiology, 2015, 309(2): G112-G122. DOI:10.1152/ajpgi.00435.2014 (  0) 0) |
| [46] |
SHEMESH A, WANG Y, YANG Y, et al. Suppression of mTORC1 activation in acid-alpha-glucosidase-deficient cells and mice is ameliorated by leucine supplementation[J]. American Journal of Physiology Regulatory, Integrative and Comparative Physiology, 2014, 307(10): R1251-R1259. DOI:10.1152/ajpregu.00212.2014 (  0) 0) |
| [47] |
韩淑媛, 黄志强, 刘永雄, 等. 大鼠肝部分切除后肝细胞再生时细胞形态的动态变化[J]. 军医进修学院学报, 1992, 4: 345. HAN Shuyuan, HUANG Zhiqiang, LIU Yongxiong, et al. The dynamic changes of cellular morphology during rat liver regeneration after partial Hepatectomy[J]. Academic Journal of Plan Postgraduate Medical School, 1992, 4: 345. (  0) 0) |
| [48] |
缪明永, 朱克军, 汪振诚, 等. 大鼠肝再生时线粒体通透性转换的变化[J]. 第二军医大学学报, 2004, 25(3): 292-294. MIAO Mingyong, ZHU Kejun, WANG Zhencheng, et al. Changes of mitochondrial permeability transition during rat liver regeneration[J]. Academic Journal of Second Military Medical University, 2004, 25(3): 292-294. DOI:10.3321/j.issn:0258-879x.2004.03.018 (  0) 0) |
| [49] |
RACKER E, STOECKENIUS W. Reconstitution of purple membrane vesicles catalyzing light-driven proton uptake and adenosine triphosphate formation[J]. The Journal of Biological Chemistry, 1974, 249(2): 662-663. (  0) 0) |
| [50] |
翟心慧, 来小海, 吴清华, 等. 辅酶Q10对大鼠再生肝细胞线粒体通透性转换的影响[J]. 解剖学报, 2013, 44(1): 44-48. ZHAI Xinhui, LAI Xiaohai, WU Qinghua, et al. Effects of coenzyme Q10 on mitochondrial permeability transition of regenerating hepatocytes in rats[J]. Acta Anatomica Sinica, 2013, 44(1): 44-48. DOI:10.3969/j.issn:0529-1356.2013.01.009 (  0) 0) |
 2017, Vol. 15
2017, Vol. 15


