自交不亲和(self-incompatibility)是广泛存在于显花植物中的一种生殖隔离现象,从而能够拒绝自交花粉的生长,使非自交花粉完成正常的授粉受精[1-2]。阐明自交不亲和性的分子机理在植物生殖学和培育无子品种等方面具有重要的理论和应用价值。目前研究表明,S-核酸酶(S-RNase)为花柱S-决定因子。花柱中的S-RNase进入花粉管,S-RNase的细胞毒素作用使自交花粉管中的RNA降解,导致蛋白质合成终止,花粉管生长受到抑制,产生自交不亲和现象[3-5]。在花粉决定因子方面,F-box基因可能在植物自交不亲和中发挥作用。Lai等[6]在金鱼草(Antirrhinum)中克隆了一个在花粉中特异性表达的F-box基因(AhSLF-S2)。随后,分别从茄科矮牵牛(Petunia inflate)中得到S-locus F-box基因PiSLF2[7],梅(Prunus mume)和扁桃(Prunus dulcis)中鉴定出特异性的S-locus F-box基因PmSLF和PdSFB[8-10]。
RING finger基因编码的蛋白是一个庞大的蛋白家族,其典型的结果特点是序列内包含环指结构域(RING finger domain)。目前的研究结果表明大部分的RING finger蛋白具有泛素连接酶E3(Ubiquitin-protein ligating enzyme)活性,是泛素调节的蛋白降解途径中识别底物的关键因子。RING finger蛋白通过单泛素化修饰和多聚泛素化修饰靶蛋白而广泛参与植物的胚胎发生、细胞周期调节、生长发育、激素信号转导、程序化死亡,以及逆境胁迫等过程[11]。泛素化降解途径为解释植物配子体自交不亲和机理的模型之一。RING finger蛋白在该途径中起关键作用[12-13]。金鱼草中发现的S位点F-box基因AhSLF-S2,在花粉中特异表达,能与Skp和Cullin-like蛋白结合形成SCF(Skpl-Cullin-F-box)复合物[6, 14]。SCF复合物中包括RING finger domain的蛋白Rbx/Roc/Hrt负责募集泛素化的E2(Ubquitin-conjugating enzyme)中间体[15-16]。单亚基的RING finger E3泛素蛋白连接酶ARC1,在结合底物之后经U-box结构域与同源的E2相互作用,将底物泛素化,再由26S蛋白酶体降解,导致自交不亲和反应的发生。
沙田柚属于芸香科配子体高度自交不亲和果树。目前,沙田柚自交不亲和的细胞学、形态学以及蛋白质化学等方面的研究取得一定进展:沙田柚属于配子体自交不亲和类型,花柱的1/2左右为花粉管生长抑制部位[17];分离和鉴定了沙田柚自交花柱[18]和花粉管中的特异蛋白[19];对花柱通道细胞中的特异蛋白和花粉管中特异蛋白产生部位以及分布位置进行了确认[20-21]。为了进一步探讨沙田柚自交不亲和的机理,本文对自交和异交花柱进行了转录组测序,通过对自交和异交花柱差异基因的比对,获得了沙田柚RING finger基因,并对该基因编码的蛋白质进行了生物化学特征分析,旨在为沙田柚自交不亲和分子机理的深入研究提供帮助。
1 试验材料与方法 1.1 试验材料沙田柚实验材料采自广西灵川县潮田乡大山口村果园10年生结果树。在盛花期对沙田柚样树进行人工自交(沙田柚×沙田柚)授粉和异交授粉(酸柚×沙田柚),分别收集自交和异交1~3 d的授粉花柱以及当天未开花的花柱,立即放入液氮中保存,并放入-80 ℃超低温冰箱备用。
1.2 试验方法 1.2.1 RNA的提取、建库和测序总RNA提取采用了改良的Trizol法[22],检测合格的RNA交由深圳华大基因科技服务有限公司进行建库和测序。建库和测序的程序和方法详见文献[23]。
1.2.2 序列分析和系统树构建使用DNAman、ORF Finder、TMPRED、Phyre2、NetPhos2.0等软件对序列以及蛋白质理化性质进行分析,使用DNAman软件对氨基酸序列RING-H2 finger蛋白的系统进化树进行构树。
1.2.3 基因表达量及差异统计使用RPKM法(Reads per kb per million reads),即每百万resd中来自某一基因每千碱基长度的resd数目计算基因表达量。如果一个基因存在多个转录本,则用该基因的最长转录本计算其测序覆盖度和表达量。根据基因的表达量(RPKM值)计算该基因在不同样本间差异表达倍数。FDR(false discovery rate)值越小,异倍数越大,表明表达差异越显著。本文确定FDR≤0.001且差异表达倍数不低于2倍(log 2Ratio≥1)的条件对自交和异交花柱之间的基因进行差异表达统计。自交/异交1 d花柱之间的差异表达基因为3 467个,自交/异交2花柱之间的差异基因为3 460个,自交/异交3 d花柱之间的差异表达基因数量为3 609个。分别将这些差异基因序列比对到NR、NT、Swiss-Port、COG等数据库中,得到差异基因编码蛋白的功能。其中Unigene20689_All定于为RING finger蛋白基因。
2 结果与分析 2.1 基因的生物信息学分析沙田柚RING finger蛋白(Unigene20689_All)的基因组序列全长为845 bp (GenBank登录号:KY885238)。通过生物信息学软件DNAman以及NCBI网站上的NCBI ORF Finder (http://www.ncbi.nlm.nih.gov/gorf/gorf.html)对序列进行分析,该序列的开放阅读框共有387 bp,编码128个氨基酸, 如图 1所示。
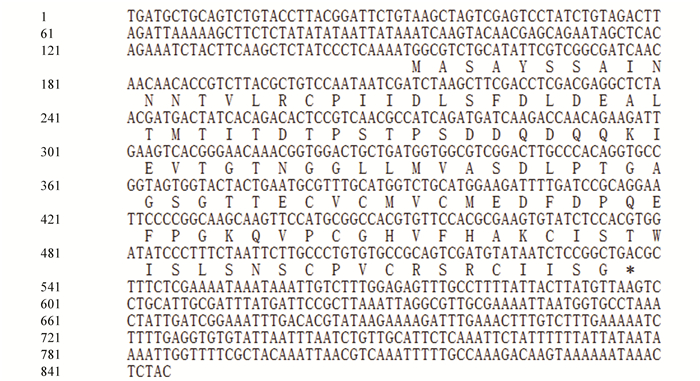
|
图 1 finger蛋白基因序列和推测的氨基酸序列 Figure 1 Nucleotide sequence of RING-H2 finger protein gene and the deduced amino sequence |
运用NCBI上的Conserved Domain Architecture Retrieval Tool (http://www.ncbi.nlm.nih.gov/gorf/gorf.html)因编码的氨基酸进行分析,该蛋白质具有一个与RING finger superfamily蛋白相同的保守结构域, 如图 2所示。

|
图 2 RING-H2 finger superfamily蛋白保守结构域 Figure 2 Conserved domains of porcine RING-H2 finger superfamily protein |
运用在线软件(http://web.expasy.org/protparam/)对该蛋白质进行分析该蛋白质的分子量为13.61 KDa,等电点pI为4.26,分子式为C577H919N155O196S14,不稳定系数为45.90,属于不稳定蛋白。其中,该蛋白质携带的负电荷氨基酸(Asp + Glu)总数为15,正电荷氨基酸(Arg + Lys)总数为6。蛋白质疏水性分析结果如图 3所示,从图 3中可以看出该基因编码的肽链中疏水性最大值约为2.96,最小值约为-3.65,初步鉴定该蛋白质为疏水蛋白。

|
图 3 RING-H2 finger蛋白疏水性分析 Figure 3 Hydrophobicity Analysing of RING-H2 finger protein |
运用TMHMM-2.0(http://www.cbs.dtu.dk/services/)对RING finger蛋白的跨膜区域进行预测。结果表明不属于跨膜蛋白,如图 4所示。
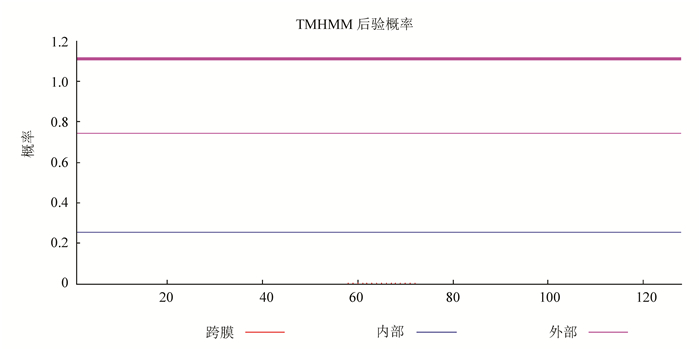
|
图 4 RING-H2 finger蛋白的跨膜区预测 Figure 4 Result of TMpred prediction on RING-H2 finger protein |
运用在线分析软件SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)对RING finger蛋白二级结构进行预测,预测结果如图 5所示。结果表明该蛋白的α-螺旋所占比例为10.94%,延伸链为34.38%,β-转角为7.03%,无规则卷曲为47.66%。

|
图 5 RING-H2 finger蛋白的二级结构预测 Figure 5 The secondary structure prediction of RING-H2 finger protein 注:h:α-螺旋;t:β-转角;c为无规则卷曲;e为延伸链. |
利用在线软件Phyre2[24]对RING finger蛋白氨基酸序列进行三级结构进行预测,结构如图 6所示。结果显示,以c5dOkI结构为模板,蛋白128个氨基酸序列中有64个氨基酸与模板达到99.7%的可信度。
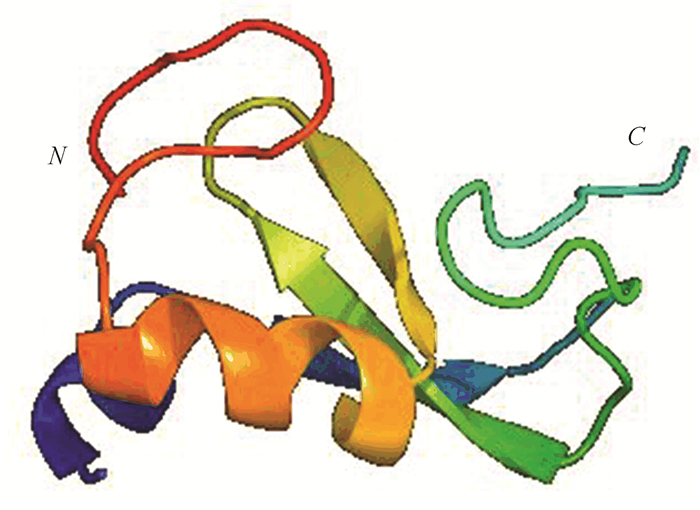
|
图 6 RING-H2 finger蛋白的三级结构 Figure 6 The tertiary structure of RING-H2 finger protein |
沙田柚Unigene20689_All序列与甜橙数据库(http://citrus.hzau.edu.cn/orange/)的RING-H2 finger蛋白(Cs4g15340.1)的cDNA相似度为98%。再从NCBI下载其他9种植物的RING-H2 finger基因编码的氨基酸序列,利用DNAMan构建系统发育树,结果表明沙田柚RING finger基因编码的蛋白质与芸香科的甜橙(Citrus sinensis)有很近的亲缘关系,属于同一进化分支,如图 7所示。经过同源性比较,在氨基酸的N端具有一个RING-H2 finger蛋白的保守结构域,推测沙田柚Unigene20689_All基因编码的蛋白质是RING-H2 finger蛋白。

|
图 7 10种植物RING-H2 finger蛋白的氨基酸序列比较 Figure 7 The comparison plant on amino acid sequence RING-H2 finger protein of 10 plant species 注:Unigene 20689(沙田柚, KY885228), Arabidopsis thaliana (拟南芥,NP_178507.1, ),Citrus sinensis (甜橙,Cs4g15340.1),Eutrema halophilum (山俞菜,AAM19707.1), Hevea brasiliensis (橡胶树,AAP46154.1),Medicago truncatula (蒺藜苜蓿,XP_003601512.1),Ricinus communis (蓖麻,XP_002533895.1),Solanum lycopersicum (番茄,XP_004251547.1),Solanum nigrum (龙葵,ADW66146.1), Zea mays (玉米,DAA40340.1). |
26S泛素蛋白酶体途径(ubiquitin-proteasome pathway)是已知真核生物中主要的蛋白质降解途径,主要由泛素(Ub)、泛素激活酶(E1)、泛素结合酶(E2)、泛素蛋白连接酶(E3)和26S蛋白酶体组成[25], 其中,泛素连接酶E3在底物的特异性选择降解过程中发挥关键作用。根据其结构和功能,目前已经发现的E3可以分为2大类:HECT结构域E3和RING E3。RING E3泛素连接酶具有一个RING finger域或一个与RING finger在结构上相关的U-box域,如SCF、ARC1等。
26S泛素蛋白酶体途径的生物学功能广泛,在植物的生长发育、抗病反应、激素反应、胁迫反应中起着重要的作用。此外,泛素/蛋白酶体途径与高等植物的有性生殖有关。Callis等[26]发现玉米花粉成熟时,其游离泛素和泛素缀合蛋白的含量都急剧下降,分别只有早期单核小孢子中的1/10和1/50。Kulikauskas等[27]发现一些禾本科植物花粉成熟时伴随着游离泛素水平的显著下降,而其他植物的花粉成熟时都具有高水平的游离泛素这一特征。Li等[28]发现泛素及泛素缀合蛋白在Nicotiana alata雌雄蕊中的分布具有时间、组织特异性,并认为泛素系统对高等植物生殖结构的分化过程起调节作用。同时,泛素/蛋白酶体途径在Actinidia deliciosa花粉萌发过程中起重要作用,并参与建立并维持Actinidia deliciosa花粉管的极性生长模式[29-30]。此外,26S泛素蛋白酶体途径还与植物自交不亲和相关。Qiao等[31]认为而在不亲和授粉中,花粉受体分子为SLF(S-locus-encoded F-box proteins),它含有一个F-box结构域SLF与S-RNase以等位基因特异性形式结合,这样就不会形成SCF复合体,S-RNase的核酸酶活性就会被保持下来,从而降解花粉管中的RNA,使花粉管停止生长,最终导致自交不亲和反应。
4 结论本文克隆的Unigene20689_All序列与甜橙的RING-H2 finger基因编码的氨基酸序列的相似度为98%,通过与其他含有RING-H2 finger结构域的植物进行同源性比对,其在C-端含有一个RING-H2 finger保守结构域,说明Unigene20689_All序列为RING-H2 finger基因。推测该基因可能在沙田柚泛素调节的蛋白质降解途径中扮演识别底物的角色,但在沙田柚自交不亲和反应中的具体功能还有待进一步研究。
| [1] |
De NETTANCOURT P D. Incompatibility and incongruity in wild and cultivated plants[M]. Berlin Heidelberg: Springer, 2001. DOI:10.1007/978-3-662-04502-2
(  0) 0) |
| [2] |
FRANKLIN-TONG V. Self-incompatibility in flowering Plants: evolution, diversity, and mechanisms[M]. BerlinHeidelberg: Springer, 2008. DOI:10.1007/978-3-662-04502-2
(  0) 0) |
| [3] |
HARING V, GRAY J E, MCCLURE B A, et al. Self-incompatibility: a self-recognition system in plants[J]. Science, 1990, 250(4983): 937-941. DOI:10.1126/science.2237440 (  0) 0) |
| [4] |
MCCLURE B A, FRANKLIN-TONG V. Gametophytic self-incompatibility: understanding the cellular mechanisms involved in "self " pollen tube inhibition[J]. Planta, 2006, 224(2): 233-245. DOI:10.1007/s00425-006-0284-2 (  0) 0) |
| [5] |
STONE J L. Molecular mechanisms underlying the breakdown of gametophytic self-incompatibility[J]. The Quarterly Review of Biology, 2002, 77(1): 17-32. DOI:10.1086/339200 (  0) 0) |
| [6] |
LAI Z, MA W S, HAN B, et al. An F-box gene linked to the self-incompatibility (S) locus of Antirrhinum is expressed specifically in pollen and tapetum[J]. Plant Molecular Biology, 2002, 50(1): 29-42. DOI:10.1023/A:1016050018779 (  0) 0) |
| [7] |
SIJACIC P, WANG Xi, SKIRPAN A L, et al. Identification of the pollen determinant of S-RNase-mediated self-incompatibility[J]. Nature, 2004, 429(6989): 302-305. DOI:10.1038/nature02523 (  0) 0) |
| [8] |
ENTANI T, IWANO M, SHIBA H, et al. Comparative analysis of the self-incompatibility (S-) locus region of Prunus mume: identification of a pollen-expressed F-box gene with allelic diversity[J]. Genes to Cells: Devoted to Molecular & Cellular Mechanisms, 2003, 8(3): 203-213. DOI:10.1046/j.1365-2443.2003.00626.x (  0) 0) |
| [9] |
YAMANE H, IKEDA K, USHIJIMA K, et al. A pollen-expressed gene for a novel protein with an F-box motif that is very tightly linked to a gene for S-RNase in two species of cherry, Prunus cerasus and P. avium[J]. Plant Cell Physiclogy, 2003, 44(7): 764-769. DOI:10.1093/pcp/pcg088 (  0) 0) |
| [10] |
IKEDA K, IGIC B, USHIJIMA K, et al. Primary structural features of the S haplotype-specific F-box protein, SFB, in Prunus[J]. Sexual Plant Reproduction, 2004, 16(5): 235-243. DOI:10.1007/s00497-003-0200-x (  0) 0) |
| [11] |
刘丽娟, 刘爱玲, 陈信波. 植物非生物逆境胁迫相关RING-H2 finger蛋白[J]. 生物技术通报, 2014(7): 1-7. LIU Lijuan, LIU Ailing, CHEN Xinbo. Abiotic stress-related RING-H2 finger proteins in plant[J]. Biotechnology Bulletin, 2014(7): 1-7. (  0) 0) |
| [12] |
LECKER S H, GOLDBERG A L, MITCH W E. Protein degradation by the ubiquitin-proteasome pathway in normal and disease states[J]. Journal of the American Society of Nephrology, 2006, 17(7): 1807-1819. DOI:10.1681/ASN.2006010083 (  0) 0) |
| [13] |
METZGER M B, HRISTOVA V A, WEISSMAN A M. HECT and RING-H2 finger families of E3 ubiquitin ligases at a glance[J]. Journal of Cell Science, 2012, 125(3): 531-537. DOI:10.1242/jcs.091777 (  0) 0) |
| [14] |
李彦泽. 拟南芥F-box基因AtPP2-B11的功能分析及苹果RING-H2 finger型泛素连接酶E3的家族分析[D]. 泰安: 山东农业大学, 2011. LI Yanze. Functional characterization of F-box-containing gene AtPP2-B11 and genome-wide analysis of the RING-H2 finger proteins in apple[D]. Taian: Shandong Agricultural University, 2011. http://d.wanfangdata.com.cn/Thesis/D143537 (  0) 0) |
| [15] |
LECHNER A, LEECH C A, ABRAHAM E J, et al. Nestin-positive progenitor cells derived from adult human pancreatic islets of Langerhans contain side population (SP) cells defined by expression of the ABCG2 (BCRP1) ATP-binding cassette transporter[J]. Biochemical and Biophysical Research Communications, 2002, 293(2): 670-674. DOI:10.1016/S0006-291X(02)00275-9 (  0) 0) |
| [16] |
RISSEEUW E P, DASKALCHUK T E, BANKS T W, et al. Protein interaction analysis of SCF ubiquitin E3 ligase subunits from Arabidopsis[J]. The Plant Journal: for Cell and Molecular Biology, 2003, 34(6): 753-767. DOI:10.1046/j.1365-313X.2003.01768.x (  0) 0) |
| [17] |
薛妙男, 陈腾土, 杨继华. 沙田柚自交和异交亲和性观察[J]. 园艺学报, 1995, 22(2): 127-132. XUE Miaonan, CHEN Tengtu, YANG Jihua. Observations on self and corss-compatibility in Shatinyu[J]. Acta Horticulturae Sinica, 1995, 22(2): 127-132. DOI:10.3321/j.issn:0513-353X.1995.02.006 (  0) 0) |
| [18] |
杨继华, 薛妙男, 陆景峰. 沙田柚自交、异交花柱蛋白质的比较分析[J]. 植物学通报, 1996, 13(专辑): 45. (  0) 0) |
| [19] |
秦新民, 李惠敏, 薛妙男, 等. 沙田柚自交、异交花粉管蛋白的双向电泳分析[J]. 广西植物, 2004, 24(6): 566-569. QIN Xinmin, LI Huimin, XUE Miaonan, et al. Two-dimensional electrophoresis of self and non-self pollinated pollen tube proteins of Shatinyu[J]. GUIHAIA, 2004, 24(6): 566-569. DOI:10.3969/j.issn.1000-3142.2004.06.018 (  0) 0) |
| [20] |
薛妙男, 李义平, 张杏辉, 等. 沙田柚自交花柱中识别蛋白的免疫金定位[J]. 园艺学报, 2001, 28(1): 59-61. XUE Miaonan, LI Yiping, ZHANG Xinghui, et al. Immuno-gold localization of recognition prorein in the self-pollination style of Shatinyu[J]. Acta Horticulturae Sinica, 2001, 28(1): 59-61. DOI:10.3321/j.issn:0513-353X.2001.01.012 (  0) 0) |
| [21] |
秦新民, 莫花浓, 石菁萍, 等. 沙田柚花粉管S1-RNase免疫胶体金定位研究[J]. 广西农业科学, 2009, 40(5): 483-485. QIN Xinmin, MO Huanong, SHI Jingping, et al. Immunogold localization of S1-RNase in pollen tube of Cirus grandis var. Shatinyu Hort[J]. Guangxi Agricultural Sciences, 2009, 40(5): 483-485. DOI:10.3969/j.issn.2095-1191.2009.05.007 (  0) 0) |
| [22] |
BARIOLA P A, HOWARD C J, TAYLOR C B, et al. The Arabidopsis thaliana ribonuclease gene RNA1 is tightly controlled in response to phosphate limitation[J]. The Plant Journal, 1994, 6(5): 673-685. DOI:10.1046/j.1365-313X.1994.6050673.x (  0) 0) |
| [23] |
秦新民, 张渝, 刘玉洁, 等. 沙田柚S-RNase基因的克隆及序列分析[J]. 广西师范大学学报(自然科学版), 2015, 33(1): 139-145. QIN Xinmin, ZHANG Yu, LIU Yujie, et al. Cloning and sequence analysis of S-RNase gene from Citrus grandis var.Shatinyu hort[J]. Journal of Guangxi Normal University: Natural Science Edition, 2015, 33(1): 139-145. DOI:10.16088/j.issn.1001-6600.2015.01.023 (  0) 0) |
| [24] |
KELLEY L A, MEZULIS S, YATES C M, et al. The Phyre2 web portal for protein modeling, prediction and analysis[J]. Nature Protocols, 2015, 10(6): 845-858. DOI:10.1038/nprot.2015.053 (  0) 0) |
| [25] |
SMALLE J, VIERSTRA R D. The ubiquitin 26S proteasome proteolytic pathway[J]. Annual Review of Plant Biology, 2004, 55(1): 555-590. DOI:10.1146/annurev.arplant.55.031903.141801 (  0) 0) |
| [26] |
CALLIS J, BEDINGER P. Developmentally regulated loss of ubiquitin and ubiquitinated proteins during pollen maturati on in maize[J]. Proceedings of the National Academy of Sciences, 1994, 91(13): 6074-6077. DOI:10.1073/pnas.91.13.6074 (  0) 0) |
| [27] |
KULKAUSKAS R, HOU A, MUSCHIETT J, et al. Comparisons of diverse plant species reveal that only grasses show drastically reduced levels of ubiquitin monomer in mature pollen[J]. Sexual Plant Reproduction, 1995, 8(6): 326-332. DOI:10.1007/BF00243199 (  0) 0) |
| [28] |
LI Y Q, SOUTHWORTH D, LINSKENS H F, et al. Localization of ubiquitin in anthers and pistils of Nicotiana[J]. Sexual Plant Reproduction, 1995, 8(3): 123-128. DOI:10.1007/BF00242254 (  0) 0) |
| [29] |
SPERANZA A, SCOCCIANTI V, CRINELLI R, et al. Inhibition of proteasome activity strongly affects kiwifruit pollen germination[J]. Plant Physiology, 2001, 126(3): 1150-1161. DOI:10.1104/pp.126.3.1150 (  0) 0) |
| [30] |
SCOCCIANTI V, OVIDI E, TADDEI A R, et al. Involvement of the ubiquitin/proteasome pathway in the organisation and polarised growth of kiwifruit pollen tubes[J]. Sexual Plant Reproduction, 2003, 16(3): 123-133. DOI:10.1007/s00497-003-0182-8 (  0) 0) |
| [31] |
QIAO H, WANG H, ZHAO L, et al. The F-box protein AhSLF-S2 physically interacts with S-RNases that may be inhibited by the ubiquitin/26S proteasome pathway of protein degradation during compatible pollination in Antirrhinum[J]. Plant Cell, 2004, 16(3): 582-595. DOI:10.1105/tpc.017673 (  0) 0) |
 2017, Vol. 15
2017, Vol. 15


