基因芯片以其高通量、微型化和自动化等优点逐渐成为基因表达检测、突变检测、基因组多态性分析和基因文库作图以及杂交测序等方面的重要工具,并已广泛应用于疾病诊断和治疗、药物筛选、农作物的优育优选、司法鉴定、食品卫生监督、环境检测、国防、航天等许多领域[1-3]。
探针设计和筛选是制备高质量基因芯片的关键步骤之一[4-5]。目前,已有不少探针设计软件被开发出来,它们针对不同的设计对象,显示出各自的优势和局限性[6]。与其他探针设计软件相比,ARB软件更适用于大量寡核苷酸(16S/23S)的探针设计、处理大量的序列、构建广泛的亲缘关系、处理16S/23S/18S rRNA序列等。ARB软件是用于设计系统发育芯片(PhyloChip)探针的主要软件,在世界范围内被广泛使用[7]。目前随着环境问题的增加,为了更好地解决环境问题,探索环境微生物群落的多样性和复杂性,解析在不同的生态系统下微生物群落的功能,出现了越来越多的基于ARB软件设计的系统发育芯片(PhyloChip) [8-10]。然而由于安装困难,相关中文介绍文章较少等原因,使得ARB软件在国内使用较少。
1 ARB软件简介迅速增加的rRNA基因序列数据和日益重要的rRNA基因识别系统,以及用于数据处理和分析的软件工具集成包的缺失,使得德国慕尼黑工业大学的Oliver Strunk, Wolfgang Ludwig, Oliver Gross, Boris Reichel, Norbert Stuckmann, Michael May, Björn Nonhoff, Michael Lenke, Toni Ginhart, Alexander Vilbig, Ralf Westram等[11]于1992年开发了一个免费的图形化的序列分析软件包ARB(拉丁语,树)。这个软件包的开发距今已有二十多年的历史,但还在不断的优化和改进。ARB起初功能比较简单,主要用于rRNA序列分析,而现在ARB的功能已经非常强大,可以广泛用于核酸和蛋白质序列分析。ARB软件作为一款具备图形界面、操作可视化的探针设计软件,可便于用户对数据实现人机交互式拓展处理。避免了在使用各式各样的软件时,由于处理顺序的不同,需频繁变换输入、输出文件格式的缺点,从而实现各种分子数据(核酸序列、蛋白序列)的综合分析。
2 ARB软件包的安装ARB需要在Linux系统下安装。如果在windows系统中安装,需要先装一个虚拟机软件(如VMWare、VirtualBox等),虚拟一台PC,然后安装Linux系统,然后再在这个Linux系统中安装ARB[12-13]。
首先需要根据Linux系统版本信息从http://download.arb-home.de/release/latest/下载合适的arb.tgz文件、arb_README.txt文件以及arb_install.sh文件,并放到一个文件夹中。打开Linux系统终端,进入文件所在目录,利用命令“sudo sh arb_install.sh”进行安装,会出现脚本提出的第1个问题“Enter full installation path: [/usr/arb]”,建议输入默认安装路径,即“/usr/arb”。之后会出现脚本提出的第2个问题“Where do you want to put your pt_server data”,并给出2个选项,“1.
ARB提供了小亚基核糖体RNA和大亚基核糖体RNA基因序列的数据库并定期更新,网址为http://download.arb-home.de/data/。从上述网站选择合适的数据库,进行下载。在“ARB INTRO”窗口选择数据库所在的位置,鼠标左键点击目标文件,并点击“OPEN SELECTED”打开数据库,如图 1所示。

|
图 1 ARB INTRO窗口 Figure 1 ARB INTRO window |
每个窗口右上角均有一个“HELP”按钮,按这个按钮显示“HELP WINDOW”窗口,窗口包含当前软件工具上的信息,并提供访问帮助文本和相关主题。
不同的序列数据集(对齐),例如分配到相同物种的不同基因序列或基因及基因产物(蛋白质)的序列可以被保持在一个ARB数据库。点击ARB主窗口“Tools”下面的按钮(第2行第3个按钮)即可弹出“SELECT AN ALIGNMENT”窗口,从列表中选择一个合适的序列类型数据库。之后任何操作将在当前选定的数据集(对齐)的序列数据进行。
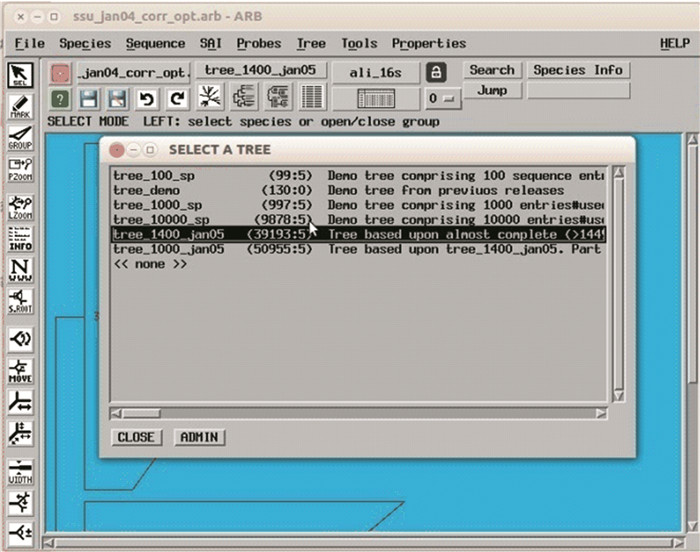
从“Tree”菜单中选择“Select...”或按“Probes”下面的按钮(第2排第2个按钮),会出现“SELECT A TREE”窗口。ARB_NT主窗口中的树可在“SELECT A TREE”窗口的列表中进行选择(例如选择tree_1400_jan05(39193:5))。当前窗口中的树的名称显示在按钮中如图 2所示。
|
图 2 ARB主窗口及SELECT A TREE窗口 Figure 2 ARB window and SELECT A TREE window |
辐射状树或树状显示模式可分别通过按





|
图 3 SEARCH and QUERY窗口 Figure 3 SEARCH and QUERY window |
在ARB主窗口点击“Probes”,从下拉列表中选择“PT_SERVER Admin”打开“PT_SERVER ADMIN”窗口,点击“Build server”,出现“QUESTION BOX”,选择“Do it”,出现“STATUS BOX”,几分钟后出现“MESSAGE BOX”说明已经生成基于本数据库的PT_server。鼠标左键选中新建的PT_server,点击“Start server”即可激活调用选中的PT_server进行探针设计如图 4所示。

|
图 4 PT_SERVER ADMIN窗口 Figure 4 PT_SERVER ADMIN window |
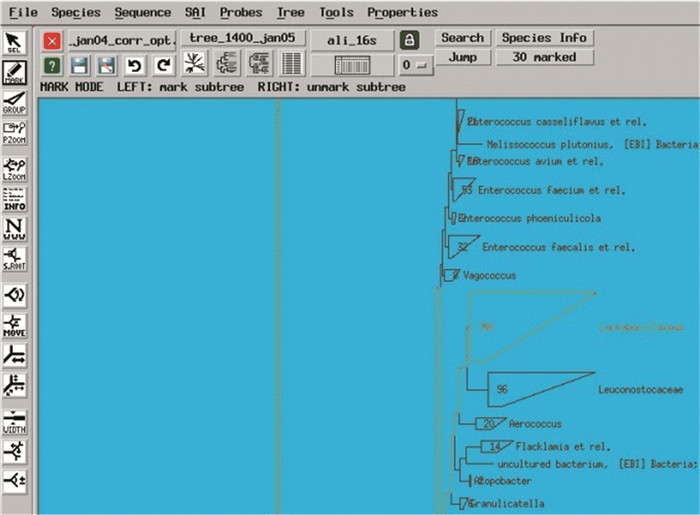
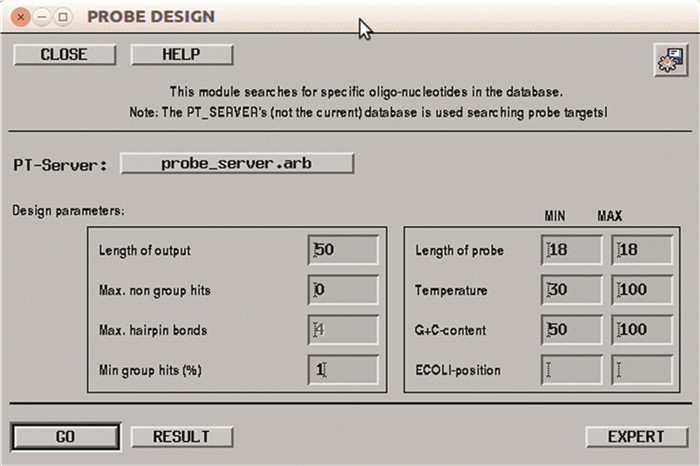
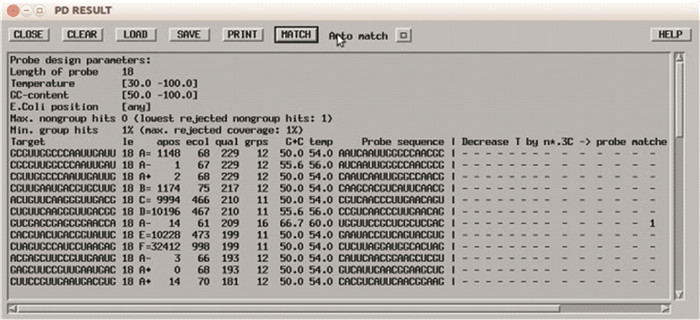
选定、标记目标物种或类群,例如设计Lactobacillaceae科的探针,利用“Mark”按钮选中Lactobacillaceae科的所有物种如图 5所示。在ARB主窗口点击“Probes”,从下拉列表中选择“Design Probes”打开“PROBE DESIGN”窗口如图 6所示。在“PT-Server”子窗口选择已经激活的PT_server。在“Design parameters”子窗口填写合适的参数,例如探针的数量(Length of output)、长度(Length of probe)、T(Temperature)、G+C含量(G+C content)、位置(ECOLI-position)、发卡结构数量(Max. hairpin bonds)(越小越好)、最大非目标物种数量(Max. non group hits)(越小越好)、最小目标种群覆盖度(Min group hits)(越大越好)等。参数确定之后,点击“GO”,出现“STATUS BOX”,等待几分钟后出现“PD RESULT”的窗口,窗口中将列出所有符合设定参数的探针及其详细信息如图 7所示。可通过点击“PD RESULT”窗口上方的“SAVE”,将所有探针信息进行保存。如果无探针出现,可重新调整最大非目标物种数量(Max. non group hits)和最小目标种群覆盖度(Min group hits)的参数。
|
图 5 物种选择 Figure 5 Species selection |
|
图 6 PROBE DESIGN窗口 Figure 6 PROBE DESIGN window |
|
图 7 PD RESULT窗口 Figure 7 PD RESULT window |
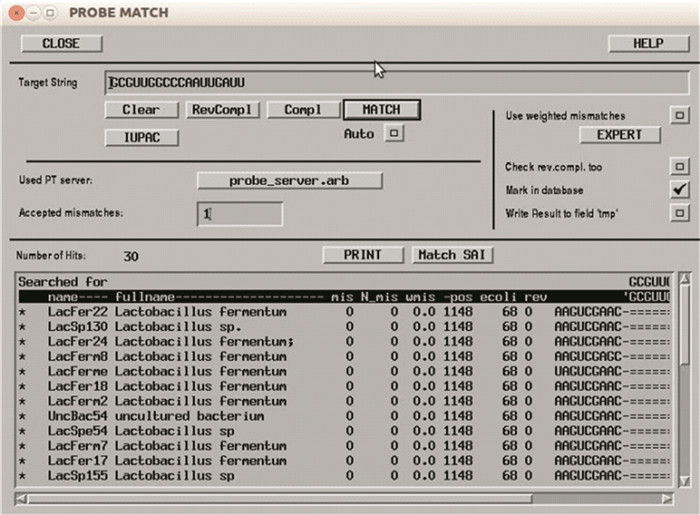
鼠标左键选定某个探针,点击“MATCH”,可打开“PROBE MATCH”窗口,确定目标序列后(可通过点击“RevCompl”、“Compl”等对序列进行反向互补、互补等操作),在“Accepted mismatches”子窗口选择合适的错配数,点击“MATCH”,相应的物种及相关信息将出现列表中如图 8所示。
|
图 8 PROBE MATCH窗口 Figure 8 PROBE MATCH window |
本文利用ARB软件及数据库针对海洋环境中丰度较高的放线菌门的10个科、α变形菌门的22个科、β-变形菌的9个科、γ变形菌门的31个科、拟杆菌门的13个科初步设计了约8 500个探针,通过“PROBE MATCH”从中初步筛选了850个特异性较好的探针。值得注意的是通过ARB软件设计的探针,还需要在SILVA、Greengenes database、BLAST、Ribosomal DataBase Project's Probe Match等网站进行特异性验证,甚至还需要通过实验验证,以最大限度的保证探针的特异性。本文将ARB软件设计的850个探针在BLAST网站进行了特异性验证,最后为每个科选择了2~3条特异性较好、T值相近的探针,研制成了用于海洋环境放线菌门[14]、α变形菌门[15]、β-变形菌[16]、γ变形菌门[17]、拟杆菌门[18]群落检测的基因芯片。通过纯菌、单克隆、混合克隆等的杂交实验验证,证明设计的探针具有良好的特异性。
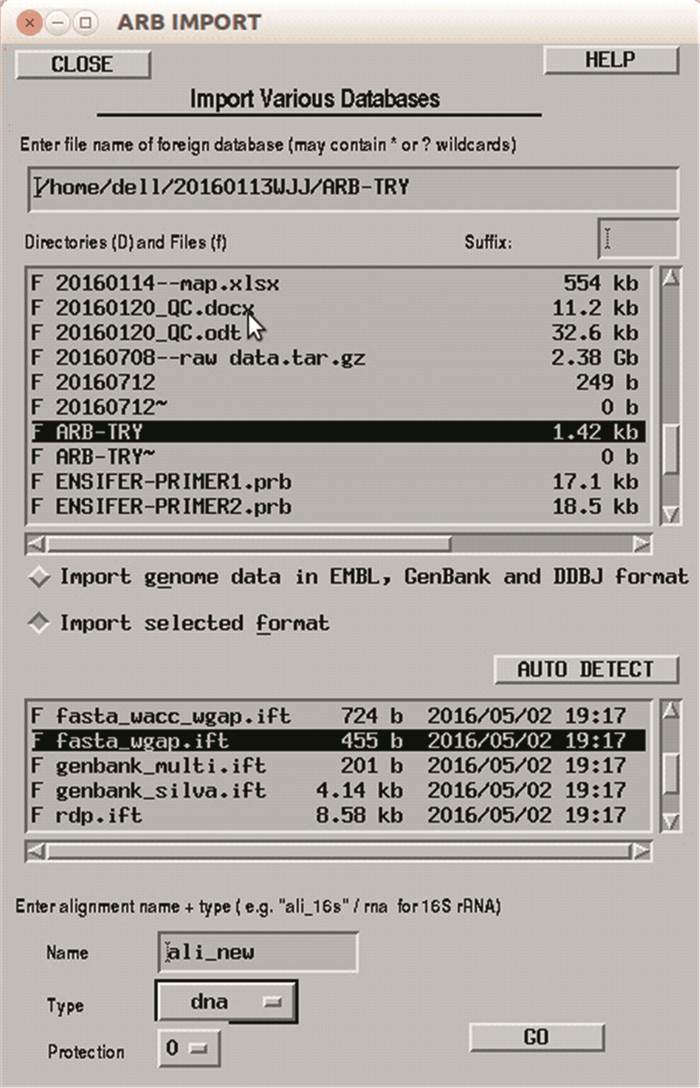
3.2 基于本地数据库的探针设计ARB软件同样支持使用本地序列数据进行探针设计。在“ARB INTRO”窗口点击“CREATE AND IMPORT”打开“ARB IMPORT”窗口。在“Directories(D)and Files(f)”子窗口中选中本地序列文件,点击“AUTO DETECT”或在其子窗口中选择相应的序列格式,在“Enter alignment name + type”子窗口中填写“Name(可默认)”,在“Type”下拉列表中选择相应的序列类型,点击“GO”出现“STATUS BOX”,等待几分钟后连续两个“QUESTION BOX”,选择默认的选项后,即可将序列导入如图 9所示。
|
图 9 ARB IMPORT窗口 Figure 9 ARB IMPORT window |
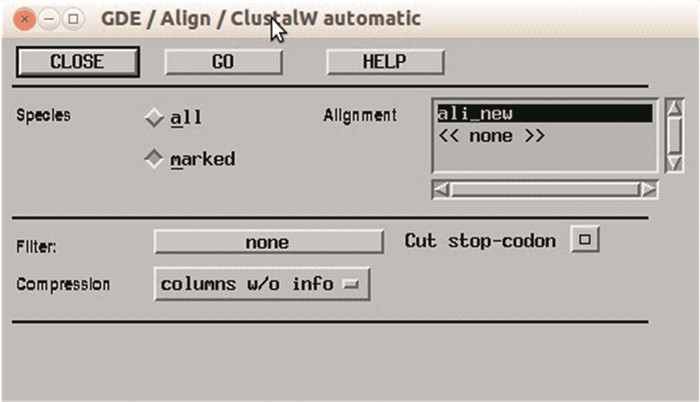
在ARB主窗口的“Sequence”下拉菜单中选择“Align Sequences”,根据序列特征选择合适的比对方法,如DNA序列可选择“ClustalW automatic”(亦可选择ClustalW DNA Alignment),打开“GDE/Align/ClustalW automatic”窗口,默认参数,点击“GO”出现“STATUS BOX”,几分钟后可完成序列比对,亦会再出现一个“QUESTION BOX”,选择“Overwrite species (all)”,将比对好的序列重新导入如图 10所示。
|
图 10 GDE/Align/ClustalW automatic窗口 Figure 10 GDE/Align/ClustalW automatic window |
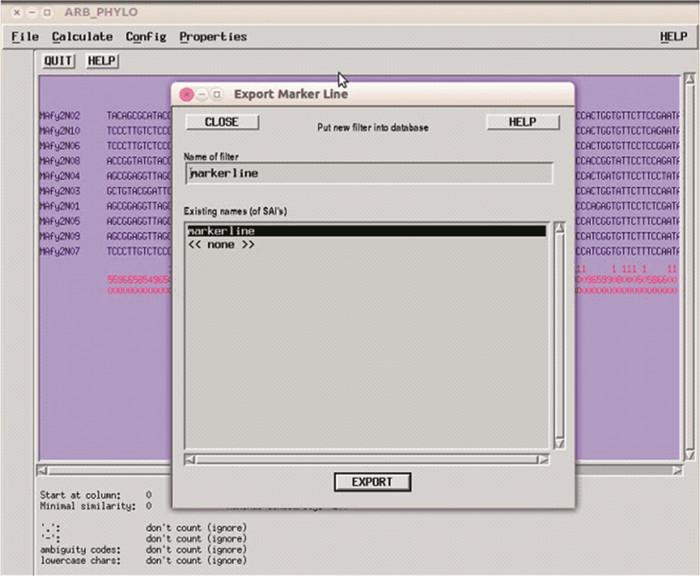
在ARB主窗口的“SAI”下拉菜单中选择“Create SAI using ……”,然后选择“Filter by base frequency”打开“ARB_PHYLO”窗口,点击“Calculate”下拉菜单中的“Column Filter”,然后点击“File”下拉菜单中的“Export Filter”打开“Export Marker Line”窗口,点击“EXPORT”即可在“Existing names”子窗口出现新生成的SAI如图 11所示。
|
图 11 ARB_PHYLO窗口和Export Marker Line窗口 Figure 11 ARB_PHYLO and Export Marker Line window |
在ARB主窗口的“Tree”下拉菜单中选择“Build tree from sequence data”,子菜单中列出了构建系统发育树的主要3种方法。距离矩阵法(distance matrix method)是根据每对物种之间的距离,其计算一般很直接,所生成的树的质量取决于距离尺度的质量。距离通常取决于遗传模型。距离矩阵法中的邻接法(neighbor-joining)是基于最小进化原理的一种算法,构建的树相对准确,假设少,计算速度快,只得一颗树。其将序列上的所有位点等同对待,且所分析的序列的进化距离不能太大。适用于进化距离不大,信息位点少的短序列。最大简约法(maximum parsimony method)较少涉及遗传假设,它通过寻求物种间最小的变更数来完成的。最大简约法是基于进化过程中碱基替代数目最少这一假说,其推测的树不是唯一的,变异大的序列可能会导致建树错误,适用于序列残基差别小,具有近似变异率,包含信息位点比较多的长序列。最大似然法(maximum likelihood method)的原理是将每个位点所有可能出现的残基替换概率进行累加,产生特定位点的似然值,对所有可能的系统发育树都计算似然函数,似然函数值最大的那棵树即最可能的系统发育树。在进化模型确定的情况下,最大似然法是与进化事实吻合最好的建树算法,但是其计算强度非常大,极为耗时[19-20]。可根据自己的数据选择合适的算法进行系统发育树构建。例如可选择“Distance matrix methods”,然后选择“Distance Matrix + ARB NJ”,打开“NEIGHBOUR JOINING”窗口,在“Which Species”下拉列表中选择“all”,在“Alignment”子窗口选择目标数据,在“Filter”下拉列表中选择新生成的SAI,在“Correction”下方点击“AUTODETECT”自动选择合适的计算方法,或者根据序列特征进行选择,如300-1 000/2 000的DNA序列适合选择“jukes-cantor”,大于1 000/2 000的DNA序列适合选择“felsenstein”,具体的选择标准可点击“HELP”按钮进行查看。然后点击“Calculate tree”或“Calculate bootstrap tree”即可成功构建系统发育树如图 12所示。在此系统发育树基础上,可按照公共数据库的探针设计的方法进行探针设计[14]。
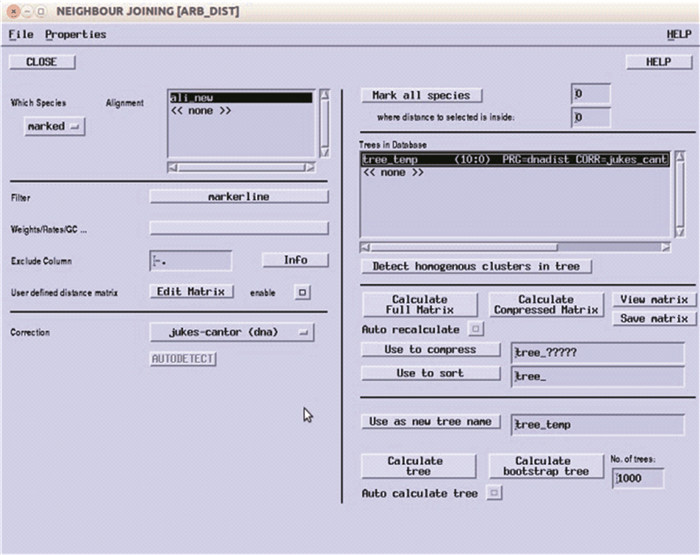
|
图 12 NEIGHBOUR JOINING窗口 Figure 12 NEIGHBOUR JOINING window |
近年来由于高通量测序技术的飞速发展,获得核酸序列的数量每年都在成倍增加。很多研究需要基于自己获得的本地数据进行探针设计,以增强探针的针对性和特异性。尤其是针对本地数据中一些不可培养、未命名种类的探针设计,很多公共数据库中并不包含相应的序列,因此基于本地数据库的探针设计在高通量技术蓬勃发展的今天显得尤为重要。本文亦基于本地数据库进行了大量的探针设计,尤其是针对不可培养、未命名的种类进行了大量的探针设计。然而由于不可培养、未命名的种类在BLAST中无法有效的进行验证,亦无纯菌和单克隆进行有效地实验验证,因此这些探针的特异性无法进一步进行评估。但是之前的经验表明基于ARB软件设计、验证的探针,在整个数据库中具有良好的特异性。因此只要将特定环境中的数据库健全,基于此库设计的所有种类的探针,在此环境中应用时均会具有良好的特异性。
4 ARB软件包的其他功能ARB软件的大致有以下一些功能:序列数据以系统发育树的形式在主窗口中显示,用鼠标点击或搜索工具可进行一系列查找及编辑操作;序列及附属数据可以不同格式导入和导出;可生成和输出各种版式的系统发育树;提供各种各样的算法构建系统发育树;ARB有一个功能强大的编辑器,具有查找、初级结构自动对齐、局部对齐优化、自动二级结构检查、显示二级结构等功能;根据用户定义的标准,应用不同的程序,可建立保护配置文件和列过滤器;ARB的PT server提供了强大的搜索功能,可以在整个数据库中快速查找最近的亲缘关系或特定的序列特征,在整个数据库中,这样的特征可被评估为类群特异性探针;ARB团队维护核糖体RNA处理数据库并选择进化保守基因;ARB也可处理和可视化注释的基因组序列数据[13, 20]。
| [1] |
HELLER T, KIRCHHEINER J, ARMSTRONG V W, et al. AmpliChip CYP450 GeneChip: a new gene chip that allows rapid and accurate CYP2D6 genotyping[J]. Therapeutic Drug Monitoring, 2006, 28(5): 673–677.
DOI:10.1097/01.ftd.0000246764.67129.2a( 0) 0)
|
| [2] |
WILSON W J, STROUT C L, DESANTIS T Z, et al. Sequence-specific identification of 18 pathogenic microorganisms using microarray technology[J]. Molecular and Cellular Probes, 2002, 16(2): 119–127.
DOI:10.1006/mcpr.2001.0397( 0) 0)
|
| [3] |
NELSON T, HOLMES S, ALEKSEYENKO A V, et al. PhyloChip microarray analysis reveals altered gastrointestinal microbial communities in a rat model of colonic hypersensitivity[J]. Neurogastroenterology and Motility, 2011, 23(2): 169–e42.
DOI:10.1111/j.1365-2982.2010.01637.x( 0) 0)
|
| [4] |
ZHOU Jizhong, THOMPSON D K. Challenges in applying microarrays to environmental studies[J]. Current Opinion in Biotechnology, 2002, 13(3): 204–207.
DOI:10.1016/S0958-1669(02)00319-1( 0) 0)
|
| [5] |
HE Zhili, WU Liyou, LI Xingyuan, et al. Empirical establishment of oligonucleotide probe design criteria[J]. Applied and Environmental Microbiology, 2005, 71(7): 3753–3760.
DOI:10.1128/AEM.71.7.3753-3760.2005( 0) 0)
|
| [6] |
陈熹, 吴灶全, 刘正春. 寡核苷酸芯片探针设计软件研究进展[J]. 生物医学工程学杂志, 2014, 31(1): 214–221.
CHEN Xi, WU Zaoquan, LIU Zhengchun. Research progress of probe design software of oligonucleotide microarrays[J]. Journal of Biomedical Engineering, 2014, 31(1): 214–221. DOI:10.7507/1001-5515.20140041(  0) 0)
|
| [7] |
LOY A, SCHULZ C, LVCKER S, et al. 16S rRNA gene-based oligonucleotide microarray for environmental monitoring of the betaproteobacterial order "Rhodocyclales"[J]. Applied and Environmental Microbiology, 2005, 71(3): 1373–1386.
DOI:10.1128/AEM.71.3.1373-1386.2005( 0) 0)
|
| [8] |
KYSELKOVA M, KOPECKY J, FELFOLDI T, et al. Development of a 16S rRNA gene-based prototype microarray for the detection of selected actinomycetes genera[J]. Antonie Van Leeuwenhoek International Journal of General and Molecular Microbiology, 2008, 94(3): 439–453.
DOI:10.1007/s10482-008-9261-z( 0) 0)
|
| [9] |
LILES M R, TURKMEN O, MANSKE B F, et al. A phylogenetic microarray targeting 16S rRNA genes from the bacterial division Acidobacteria reveals a lineage-specific distribution in a soil clay fraction[J]. Soil Biology and Biochemistry, 2010, 42(5): 739–747.
DOI:10.1016/j.soilbio.2010.01.007( 0) 0)
|
| [10] |
SANGUIN H, REMENANT B, DECHESNE A, et al. Potential of a 16S rRNA-based taxonomic microarray for analyzing the rhizosphere effects of maize on Agrobacterium spp. and bacterial communities[J]. Applied and Environmental Microbiology, 2006, 72(6): 4302–4312.
DOI:10.1128/AEM.02686-05( 0) 0)
|
| [11] |
LUDWIG W, STRUNK O, WESTRAM R, et al. ARB: a software environment for sequence data[J]. Nucleic Acids Research, 2004, 32(4): 1363–1371.
DOI:10.1093/nar/gkh293( 0) 0)
|
| [12] |
洪亮, 石志芳. 虚拟机安装Linux及网络设置[J]. 价值工程, 2012, 31(11): 156–157.
HONG Liang, SHI Zhifang. To install Linux on virtual machine and set network[J]. Value Engineering, 2012, 31(11): 156–157. DOI:10.3969/j.issn.1006-4311.2012.11.092(  0) 0)
|
| [13] |
万国良, 武守东. VirtualBox安装及使用[J]. 中国科技信息, 2011(15): 103.
WAN Guoliang, WU Shoudong. VirtualBox installation and use[J]. China Science and Technology Information, 2011(15): 103. DOI:10.3969/j.issn.1001-8972.2011.15.060(  0) 0)
|
| [14] |
王敬敬, 黄志勇, 梁亚杰, 等. 一种海洋环境放线菌群落检测基因芯片及应用: CN103866044B[P]. 2015-08-26.
WANG Jingjing, HUANG Zhiyong, LIANG Yajie, et al. A gene chip for marine environmental actinomycetes community and its application:CN103866044B[P].2015-08-26.
( 0) 0)
|
| [15] |
王敬敬, 黄志勇, 赵维, 等. 一种海洋环境α变形菌群落检测基因芯片及其应用: CN104862410A[P]. 2015-08-26.
WANG Jingjing, HUANG Zhiyong, ZHAO Wei, et al. A gene chip for marine environmental α-Proteobacteria community and its application:CN104862410A[P].2015-08-26.
( 0) 0)
|
| [16] |
赵维, 王敬敬, 黄志勇. 一种检测海洋环境中β-变形菌群落的基因芯片: CN106048077A[P]. 2016-10-26.
ZHAO Wei, WANG Jingjing, HUANG Zhiyong. A gene chip for marine environmental β-Proteobacteria community and its application:CN106048077A[P].2016-10-26.
( 0) 0)
|
| [17] |
赵维, 王敬敬, 黄志勇. 一种检测海洋环境中γ-变形菌群落的基因芯片: CN105483268A[P]. 2016-04-13.
ZHAO Wei, WANG Jingjing, HUANG Zhiyong. A gene chip for marine environmental γ-Proteobacteria community and its application:CN105483268A[P]. 2016-04-13.
( 0) 0)
|
| [18] |
赵维, 黄志勇, 王敬敬. 一种检测海洋环境拟杆菌群落的基因芯片及应用: CN105401221A[P]. 2016-03-16.
ZHAO Wei, HUANG Zhiyong, WANG Jingjing. A gene chip for marine environmental Bacteroidetes community and its application:CN105401221A[P]. 2016-03-16.
( 0) 0)
|
| [19] |
吕宝忠. 分子进化树的构建[J]. 动物学研究, 1993, 14(2): 186–193.
LV Baozhong. The construction of molecular evolution trees[J]. Zoological Research, 1993, 14(2): 186–193. (  0) 0)
|
| [20] |
李建伏, 郭茂祖. 系统发生树构建技术综述[J]. 电子学报, 2006, 34(11): 2047–2052.
LI Jianfu, GUO Maozu. A review of phylogenetic tree reconstruction technology[J]. Acta Electronica Sinica, 2006, 34(11): 2047–2052. DOI:10.3321/j.issn:0372-2112.2006.11.024(  0) 0)
|
 2017, Vol. 15
2017, Vol. 15


