2. 江苏恒瑞医药股份有限公司,江苏 连云港 222047
2. Jiangsu Hengrui Medicine Co., Ltd., Lianyungang Jiangsu 222047, China
蛋白质二级结构指定是研究蛋白质结构与功能的基础,二级结构支撑着蛋白质组织构架并且是产生蛋白质三维折叠模式的关键,二级结构的指定是蛋白质结构预测的前提条件并且为蛋白质比较和功能分析提供有效的方法。然而由原子坐标正确地指定蛋白质二级结构是一项重要且具有挑战的工作。虽然目前已经有超过20种蛋白质二级结构指定程序,但是不同程序指定结果之间差异较大。由于目前尚没有二级结构指定方法的综述文献,因此本文对已有的方法进行总结并且介绍不同方法使用的二级结构指定模式。
1 蛋白质二级结构介绍在结构生物学和生物化学中,蛋白质二级结构是指在蛋白质中有规则重复的构象。1951年,Linus Pauling和同事根据模型蛋白骨架氢键准确地预测出理想的螺旋和片层结构,同时指出,310-螺旋由于键角不合适而不能出现在蛋白质中[1],但是后来发现,310-螺旋残基在蛋白质中占有大概4%的比例[2]。随后,在1952年,Kaj Ulrik Linderstrm-Lang在Linus Pauling工作基础上,首次引入二级结构的概念,与此同时,蛋白质一级结构和三级结构的概念也被他一同引入[3]。Pauling预言α-螺旋和π-螺旋是通过蛋白质骨架的氢键:α-螺旋具有重复的(i,i+4)氢键而π-螺旋具有重复的(i,i+5)氢键[1]。本文中,(i,i+n)氢键模式是指i+n号氨基酸残基的N-H基和i号氨基酸残基的C=O基形成氢键。Pauling这篇文章在20世纪科研中被认为具有里程碑的意义:第一次在分子生物学中使用了模型并且获得了巨大的成功;α-螺旋和β-片层的发现是蛋白质研究的基石[4]。
α-螺旋和β-片层是蛋白质二级结构中最主要的元素,具有这两种二级结构的残基占蛋白质残基总数的一半[1, 5]。另外还有一些其他二级结构,例如除了α-螺旋还有其他几种螺旋:π-螺旋,310-螺旋,左手螺旋和PPII螺旋。π-螺旋经常出现在α-螺旋的末端或者α-螺旋的中间位置[6],而且π-螺旋和左手螺旋被发现和蛋白质功能关系密切[6-8];α-片层,在天然状态下虽然稀少,但是被认为是蛋白质折叠的重要中间媒介结构[9];转角和环,在蛋白质结构中扮演着连接规则元素的作用[10, 11];无规则卷曲其实并不是一种特定二级结构,它是在不能被指定为其他规则二级结构情况下的一种统称。
310-螺旋,α-螺旋和π-螺旋分别是形成连续的(i,i+3),(i,i+4),(i,i+5)氢键模式(见图 1a)[1]。而左手螺旋又分为左手310-螺旋和左手α-螺旋,其氢键模式和相应的右手螺旋一致。左手和右手的定义是采用拇指指向螺旋轴延伸的方向,残基的Cα原子的走向是右手的为右手螺旋,走向是左手则为左手螺旋。本文在没有特别说明的情况下,螺旋指的是右手螺旋。
β-片层是相邻肽链骨架原子的N─H基CO基之间形成连续的氢键[5],因为肽链的走向有两种:由N端到C端;由C端到N端。因此,根据这两条相邻肽链的走向,β-片层可分为平行(走向一致)和反平行(走向相反)两种(见图 1b)。在反平行的β-片层结构中,氢键是平行的,平行的氢键使反平行β-片层结构更加稳定,因此,反平行β-片层在蛋白质结构中含量要比平行β-片层多[12]。
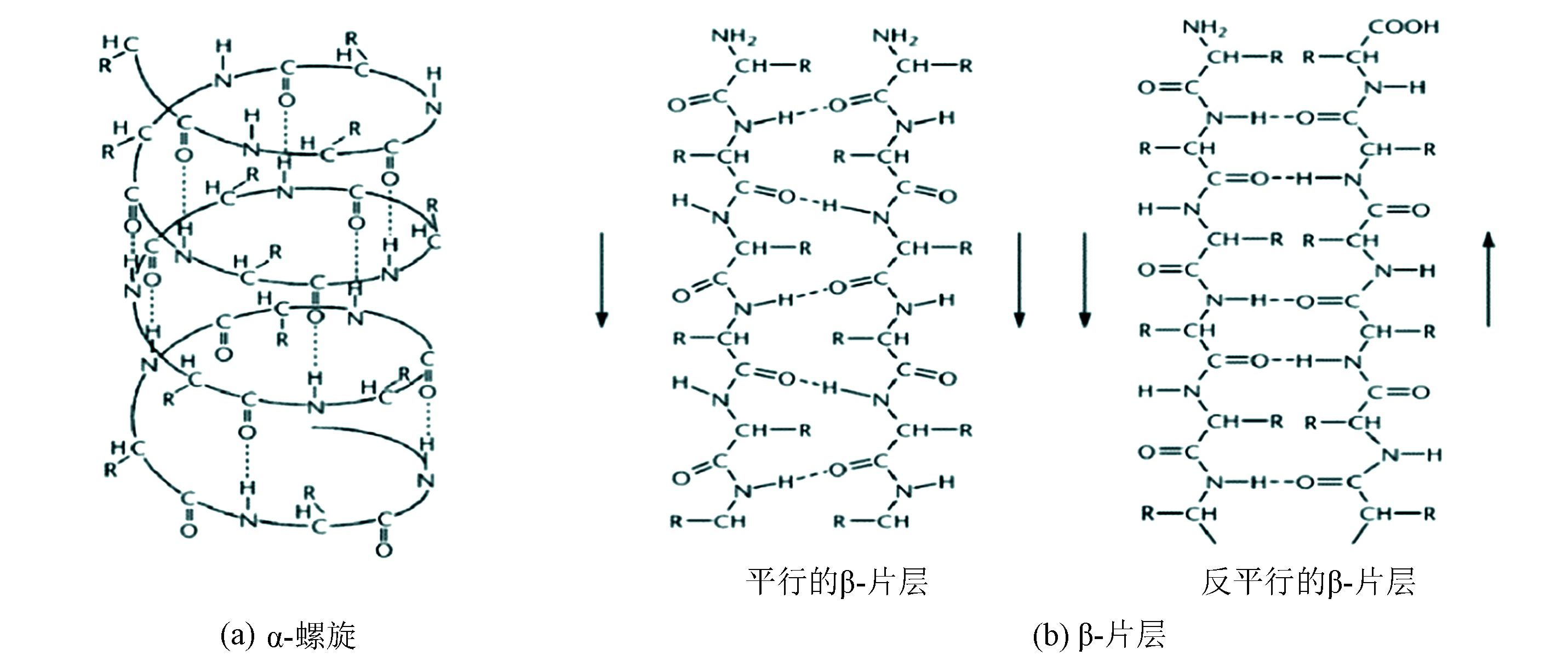
|
图 1 α-螺旋和β-片层氢键模式示意图 Figure 1 Hydrogen-bond patterns of α-helix and β-sheet |
蛋白质二级结构在结构生物学的诸多领域具有重要的作用,具体来说,在蛋白质结构的可视化[13]、蛋白质结构的比较与分类[14-16]、蛋白质的建模与结构预测[17]、蛋白质的结构检查[18]、蛋白质折叠[19],蛋白质动力学的结构变化[20, 21]以及蛋白-蛋白相互作用和蛋白质功能分析方面都有着广泛的应用[22]。在结构生物学中,很多研究是基于蛋白质二级结构正确指定基础上进行的。
2 蛋白质二级结构指定方法Pauling通过蛋白质骨架原子间氢键准确地预测出理想的α-螺旋、π-螺旋和β-片层模型,由于Pauling第一次将氢键引入二级结构使得后来蛋白质二级结构的定义也是基于氢键模式。实际上二级结构是结构生物学家通过肉眼对三维蛋白质晶体结构进行的一种指定,然而,这种指定具有主观和任意性,不同的结构生物学家会有不同的指定结果。为了解决这一问题,Levitt和Greer通过氢键和连续四个Cα原子的距离与二面角开发了第一个蛋白质二级结构指定程序[23]。随后,Kabsch和Sander在1983年开发了基于近似氢键能量的蛋白质二级结构自动指定方法:DSSP,DSSP现在依然是蛋白质二级结构指定领域中最流行的方法[24],并且,根据氨基酸序列进行蛋白质二级结构预测的结果一般是和DSSP指定结果进行比较来检验预测结果的好坏[25]。1995年Frishman和Argos开发了另一个基于氢键模式的二级结构指定程序STRIDE,STRIDE可以看作是DSSP的一个改进:同样是计算氢键能量,与DSSP不同的是,STRIDE氢键能量计算是基于经验的氢键公式而不是像DSSP那样使用静电能量近似氢键能量并且STRIDE将二级结构残基骨架二面角(φ/φ)限制在拉氏图 (Ramachandran plot) 的特定区域,具有相应的氢键模式但是(φ/φ)在拉氏图其他区域的残基将不被指定为相应的二级结构[26]。SECSTR是Fodje等人于2002年开发的,SECSTR的主要目标是识别蛋白质中π-螺旋的结构[27]。SECSTR发现DSSP和STRIDE由于氢键模式的优先性原因(i,i+4氢键模式优先于i,i+5氢键模式),经常会将π-螺旋错误地指定为α-螺旋。通过改变氢键模式的优先性,SECSTR指定的π-螺旋的含量是DSSP的10倍左右,而π-螺旋被发现经常和蛋白质的功能相联系,因此正确的识别π-螺旋对研究蛋白质结构功能关系十分重要。我们阅读了最新版本DSSP(version 2.21)的源代码,发现源代码中已经修改了氢键模式的优先性问题,将π-螺旋的(i,i+5)氢键模式定义为最优先[28]。PSSC (Protein Secondary Structure Characterization,2014年)使用DSSP的输出结果进行二级结构指定,它和DSSP的主要不同点在于:1)PSSC 使用八个字符编码一个残基的二级结构特征,这八个字符包含了指定二级结构所需要的信息;2)PSSC匹配相邻的β-链的要求是这两个β-链间至多存在一个残基的β-凸起,而DSSP可以容忍四个残基的β-凸起;3)DSSP将X-螺旋结构中具有Y-螺旋氢键模式(XY不同)的残基指定为转角,而PSSC将此残基指定为相应的Y-螺旋(X,Y为310,α,π[29]。
以上介绍的五个程序都是基于氢键模式,或者在氢键模式的基础上,通过原子间距离和角度的限制从而对蛋白质残基的二级结构进行指定。除了使用氢键模式外,很多蛋白质二级结构指定方法利用蛋白结构的几何特征,而基于几何的二级结构指定方法又可以分为两类:1)使用蛋白质局部的几何特征;2)将蛋白质骨架原子拟合到一条直线或者一系列直线上。
对于基于几何蛋白质二级结构指定方法的第一类,使用蛋白质局部的几何特征进行蛋白质二级结构指定的主要方法有(按照发表时间顺序进行介绍):
P-SEA(Protein Secondary Element Assignment,1997年):利用Cα原子的距离标准(i和i+2,i和i+3,i和i+4之间的距离)和角度标准(i,i+1,i+2,i+3四个Cα原子形成的二面角和i,i+1,i+2三个Cα原子形成的夹角)进行二级结构定;P-SEA只能指定出三类二级结构:螺旋,片层和不规则卷曲[30]。
Xtlsstr(1999年):蛋白质在远紫外区域的圆二色性是由骨架酰胺-酰胺相互作用决定的[31],Xtlsstr通过计算蛋白质骨架二面角和三个距离(其中两个是氢键距离)对二级结构进行指定,结果发现310-螺旋,π-螺旋和β-片层的指定结果与圆二色光谱中观测到的酰胺-酰胺相互作用结果相一致[32]。
VoTAP(VOronoï Tessellation Assignment Procedure,2004年)利用泰森多边形(VOronoï diagram)将蛋白质分为残基多面体,如果两个残基多面体共享一个面,那么认为这两个残基是存在相互作用的,根据这个共享接触面面积大小将这两个残基的相互作用强度分成三种:无作用,中等相互作用和很强的相互作用,这样就产生了残基之间作用强度的矩阵。通过对DSSP,STRIDE,P-SEA和DEFINE这四个程序的二级结构指定结果统计并结合残基间作用强度矩阵的分析VoTAP做出新的二级结构的指定。VoTAP的指定结果分成三类:螺旋,片层和不规则卷曲[33]。
KAKSI(KAKSI means “two” in Finnish,2005年)利用距离和角度的限制先对蛋白质中残基进行一次螺旋扫描,将符合螺旋距离和角度标准的残基指定为螺旋,之后再对剩余残基进行两次β-片层扫描:先找出符合β-单链距离和角度标准的残基,然后再将β-单链的残基配对为β-片层。KAKSI将蛋白二级结构分为三类:螺旋,片层和不规则卷曲[34]。
Beta-Spider(2005年)是专门指定β-片层的程序,Beta-Spider给出β-片层间两条链形成氢键残基的Cα原子距离和角度的限制,另外Beta-Spider给相邻两个β-链设定了堆积能(包括氢键和范德华力)的阈值,当同时满足几何条件和能量条件后,两个相邻β-链被匹配为β-片层结构。Beta-Spider指定的平行β-片层和反平行的β-片层含量分别比DSSP指定的多11%和6%[35]。
PALSSE(Predictive Assignment of Linear Secondary Structure Elements,2005年)刻画出Cα原子向量之间的转角和距离标准,先找到二级结构的核心区域,再由核心区域进行延伸拓展。PALSSE只指定两种二级结构:螺旋和片层,蛋白质80%的残基都被PALSSE归于两种结构。和其他程序不同的是,PALSSE发现两个二级结构之间会有重叠,因此PALSSE可以给一个残基指定两种二级结构[36]。
SABA(Secondary structure Assignment program Based on only Alpha carbons,2011年)定义了一个虚拟中心(两个连续残基Cα原子的中心),通过给出虚拟中心间的距离和四个虚拟中心形成的二面角标准进行蛋白质二级结构的指定。SABA可以指定出α-螺旋,310-螺旋,平行和反平行的β-片层结构[37]。
SST(2012年)是基于最小信息长度推断的贝叶斯方法来指定二级结构,它把蛋白质二级结构指定作为假设来解释蛋白质的坐标数据(Cα原子坐标),SST可以指定出螺旋的精细结构[38]。
DISICL(DIhedral-based Segment Identification and CLassification,2014年)仅使用蛋白质骨架二面角信息进行二级结构指定。DISICL首先将拉氏图分为19个区域,然后将连续两个残基的二面角分别配对到拉氏图相应区域中,根据配对的区域进行二级结构指定。DISICL将二级结构划分为18个小类,这18个小类可以合并为8个大类的二级结构[21]。
PCASSO(Protein C-Alpha Secondary Structure Output,2014年)提取出每个残基Cα原子和虚拟中心(与SABA虚拟中心定义一致)与其他残基(包括序列附近残基和相差超过6个序列的残基)的距离特征,每个残基产生258个距离属性。PCASSO利用随机森林从258个属性中随机选择16个属性计算最佳的分裂方式。PCASSO指定的二级结构分为三类:螺旋,片层和不规则卷曲,PCASSO和DSSP在残基水平有95%的指定是相同的[20]。
HELIX-F(HELIX Fitting,2015年)将螺旋指定问题分为两个子问题:最小化问题与约束满足问题。HELIX-F通过拟合算法搜索一系列空间螺旋曲线以最佳地拟合到蛋白质连续四个残基的Cα原子上,这部分解决的是第一个最小化问题。利用最佳拟合的螺旋曲线我们可以得到相应的螺旋参数,这些螺旋参数被我们用于蛋白质中螺旋的指定。结果显示,HELIX-F可以准确地指定310-螺旋,α-螺旋,π-螺旋,并且可以指定左手螺旋和PPII螺旋[39]。
SACF(Secondary structure Assignment using Cα Fragment,2016年)的核心思想是找到DSSP指定二级结构片段中的离群Cα片段并将其排除,对剩余片段进行几何聚类,聚类后每个簇的中心Cα片段作为模板,新的指定只需要和模板Cα片段进行比较即可。SACF与STRIDE相同之处在于都是通过几何特征排除离群的构象,但是我们将二级结构片段看做一个整体结构而不是像STRIDE那样关注残基局部几何特征:φ/φ,这么做的好处是使得SACF指定结果在整体Cα片段上更加一致[40]。
此外,基于局部几何特征的二级结构指定程序还有:PROSS(1999年)只是根据蛋白质骨架的二面角进行二级结构指定[41];SEGNO(2005年)根据Cα原子二面角和氢键距离以及角度来指定二级结构[42];P-CURVE(1989年)的二级结构指定基于对蛋白质曲率的数学分析,P-CURVE利用微分几何通过一系列肽平面的固定轴系统产生螺旋轴,并计算出一系列参数(螺旋半径,倾斜值,扭曲值和滚动值),再利用这些参数值进行蛋白质二级结构的指定[43]。
第二类基于几何的指定方法代表的程序有:
DEFINE(1988年)首先给出不同种类二级结构的标准距离矩阵,根据蛋白质残基的距离矩阵和标准距离矩阵的均方根(RMS)差异来指定二级结构片段第一个残基和末端残基。因为DEFINE只给出β-链的标准距离矩阵,因此DEFINE会指定出没有匹配为β-片层的β-链[44]。
STICK(2001年)可以看做是DEFINE的一个改进,由于蛋白质结构中存在弯曲和扭曲,将Cα原子拟合到一条直线上会产生较大偏差。STICK将残基Cα原子轨迹近似到一系列直线上,STICK通过每个残基在轴上上升的距离来描述二级结构,而不是用经典的螺旋和片层结构来描述。这么做的好处就是可以用线段编码结构从而进行蛋白质结构的比较[45]。
最后,有几种二级结构指定方法不属于上面的分类,这些方法或者是为了指定蛋白质中一些稀少结构(如PPII螺旋,转角等),或者利用其他程序的指定结果,具体的有:DSSP-PPII(2011年)利用DSSP的输出结果进行指定,DSSP-PPII主要为了指定蛋白质中的PPII螺旋[46];DSSPcont(CONTinuous DSSP Assignment,2003年)利用DSSP使用不同的氢键能量阈值对蛋白质指定多次,残基的最终指定结果为每次DSSP指定结果的加权平均值[47];PROMITIF(1996年)利用DSSP输出结果去指定和分析一些稀少结构,如β-转角,γ-转角,β-凸起,β-发夹,ψ-环等结构[48];SKSP(The consensus of STRIDE,KAKSI,SECSTR,and P-SEA,2008年)的蛋白质二级结构指定结果是四个方法(STRIDE,KAKSI,SECSTR和P-SEA)指定的平均[49]。
3 蛋白质二级结构指定结果的比较截止到2016年,已经有超过20种蛋白质二级结构指定程序发表在生物信息以及生物学领域的期刊上。由于不同程序指定的二级结构元素不同,例如,DSSP指定了八种二级结构元素(表 1)而KAKSI,PSEA等方法只是提供了三种二级结构元素(螺旋,片层,不规则卷曲),甚至,有的程序采取另外的标准来描述二级结构:STICK利用残基在轴上上升的距离来描述二级结构。为了进行比较,一般采取的策略是将不同的二级结构元素分成三个大类:螺旋,片层,不规则卷曲,这种策略被广泛使用[30, 34, 38]。1993年,Colloc等人利用154个蛋白质对DSSP,P-CURVE和DEFINE的指定结果做了比较,发现只有63%的残基被这三个程序指定结果是相同的[50]。相互比较时,DSSP和DEFINE,P-CURVE和DEFINE的符合率都是74%,而DSSP和P-CURVE的符合率是79%。2005年,Martin等人对不同二级结构指定方法在高分辨率X-射线蛋白质数据集上的指定结果进行了一次比较[34],参与比较的程序有DSSP,STRIDE,KAKSI,PSEA,SECSTR,XTLSSTR和PDB文件中的二级结构指定,发现:DSSP,STRIDE,PDB和SECSTR结果相近,符合率在87.4%到95.4%之间,其中DSSP和STRIDE的符合率最高(95.4%),原因是这两个程序都是基于氢键的;SECSTR和DSSP紧密相关,所以二者符合率达到了93.4%;XTLSSTR是这几个程序中和其他程序差异最大的:XTLSSTR指定结果和其它程序指定结果的符合率都低于81%;KAKSI、PSEA和其它程序表现出中等差异。另外根据二级结构指定程序相关文献,我们收集了一些指定结果之间的比较(见表 1)。值得注意的时,不同文献符合率的计算方法有所不同,而且,不同文献中所使用的测试集也是不同的,所以同样的两个方法在不同文献中符合率是有差异的。我们选择其中一篇文献的结果列于表中,表中的符合率数据只作为的参考。
| 表 1 不同二级结构指定程序的比较 Table 1 Comparison of different secondary structure assignment methods |
由表 1可以看出,除了SECSTR,PCASSO,SABA这三个程序,其他程序二级结构指定结果与DSSP的符合率都在85%以下,甚至有的低于75%(DISICL,DEFINE,STICK)。
曹晨等人通过比较10种蛋白质二级结构指定结果发现,在基于几何的蛋白质二级结构指定程序中,PCASSO与DSSP指定结果最为一致,其整体符合率达到了93.5;SACF,KAKSI,PROSS这三种方法指定结果与DSSP指定结果接近,从83.5%到84.7%;而DISICL,PALSSE与DSSP指定结果的符合率只有78.9%和72.9%。不同二级结构指定方法指定结果的差异主要是在二级结构的N端和C端,如果以DSSP指定结果作为标准:PCASSO和SACF倾向于缩短二级结构的两段,而SEGNO,KAKSI,P-SEA更有可能延伸二级结构的N端和C端[40]。
4 结论与展望准确且一致地指定蛋白质二级结构是一个重要的问题。目前最流行的二级结构指定方法是DSSP,但是DSSP方法存在以下几个问题:1)利用静电能量近似地代替氢键能量;2)氢原子坐标是近似得到的,与实际位置可能存在误差;3)DSSP将介电常数视为定值但是实际上介电常数在蛋白质表面和内部疏水环境差异很大;4)不同氢键模式之间会有交叉,这些问题会导致DSSP指定结果中出现几何上异常的二级结构。基于几何的指定方法特别是基于Cα原子坐标的指定方法可以利用最少的蛋白质原子信息对二级结构指定指定。蛋白质中一些稀少的二级结构(例如π-螺旋,PPII螺旋等)对于蛋白质功能研究具有重要的作用,因此二级结构指定方法不仅需要指定出大类的二级结构:螺旋、片层、卷曲,还需要对二级结构中的细微结构进行指定与分析。
| [1] |
PAULING L, COREY R B, BRANSON H R. The structure of proteins; two hydrogen-bonded helical configurations of the polypeptide chain[J]. Proceedings of National Academy of Sciences of the United States of America, 1951, 37(4): 205–211.
DOI:10.1073/pnas.37.4.205 ( 0) 0)
|
| [2] |
ANDERSEN C A. Protein structure and the diversity of hydrogen bonds[D].Anker Engelunds:The Technical University of Denmark, 2001.
( 0) 0)
|
| [3] |
LINDERSTRØM-LANG K. Proteins and enzymes. Palo Alto,California: Stanford University Press, 1952 .
( 0) 0)
|
| [4] |
DUNITZ J D. Pauling’s left-handed alpha-helix[J]. Angewandte Chemie-International Edition, 2001, 40(22): 4167–4173.
DOI:10.1002/1521-3773(20011119)40:22<>1.0.CO;2-D ( 0) 0)
|
| [5] |
PAULING L, COREY R B. Configurations of polypeptide chains with favored orientations around single bonds: two new pleated sheets[J]. Proceedings of National Academy of Sciences of the United States of America, 1951, 37(11): 729–740.
DOI:10.1073/pnas.37.11.729 ( 0) 0)
|
| [6] |
COOLEY R B, ARP D J, KARPLUS P A. Evolutionary origin of a secondary structure: pi-helices as cryptic but widespread insertional variations of alpha-helices that enhance protein functionality[J]. Journal of Molecular Biology, 2010, 404(2): 232–246.
DOI:10.1016/j.jmb.2010.09.034 ( 0) 0)
|
| [7] |
WEAVER T M. The pi-helix translates structure into function[J]. Protein Science, 2000, 9(1): 201–206.
( 0) 0)
|
| [8] |
NOVOTNY M, Kleywegt G J. A survey of left-handed helices in protein structures[J]. Journal of Molecular Biology, 2005, 347(2): 231–241.
DOI:10.1016/j.jmb.2005.01.037 ( 0) 0)
|
| [9] |
MILNER-WHITE E J, WATSON J D, QI G Y, et al. Amyloid formation may involve alpha-to beta sheet interconversion via peptide plane flipping[J]. Structure, 2006, 14(9): 1369–1376.
DOI:10.1016/j.str.2006.06.016 ( 0) 0)
|
| [10] |
RICHARDSON J S. The anatomy and taxonomy of protein structure[J]. Advances in Protein Chemistry, 1981(34): 167–339.
DOI:10.1016/S0065-3233(08)60520-3 ( 0) 0)
|
| [11] |
ROSE G D, GIERASCH L M, SMITH J A. Turns in peptides and proteins[J]. Advances in Protein Chemistry, 1985(37): 1–109.
( 0) 0)
|
| [12] |
PERCZEL A, GASPARI Z, CSIZMADIA I G. Structure and stability of beta-pleated sheets[J]. Journal of Computer Chemistry, 2005, 26(11): 1155–1168.
DOI:10.1002/(ISSN)1096-987X ( 0) 0)
|
| [13] |
RICHARDSON J S. Schematic drawings of protein structures[J]. Methods Enzymol, 1985(115): 359–380.
( 0) 0)
|
| [14] |
SALI A, BLUNDELL T L. Definition of general topological equivalence in protein structures. A procedure involving comparison of properties and relationships through simulated annealing and dynamic programming[J]. Journal of Molecular Biology, 1990, 212(2): 403–428.
DOI:10.1016/0022-2836(90)90134-8 ( 0) 0)
|
| [15] |
ORENGO C A, MICHIE A D, JONES S, et al. CATH--a hierarchic classification of protein domain structures[J]. Structure, 1997, 5(8): 1093–1108.
DOI:10.1016/S0969-2126(97)00260-8 ( 0) 0)
|
| [16] |
GIBRAT J F, MADEJ T, BRYANT S H. Surprising similarities in structure comparison[J]. Current Opinion in Structural Biology, 1996, 6(3): 377–385.
DOI:10.1016/S0959-440X(96)80058-3 ( 0) 0)
|
| [17] |
DROZDETSKIY A, COLE C, PROCTER J, et al. JPred4: a protein secondary structure prediction server[J]. Nucleic Acids Research, 2015, 43(W1): W389–94.
DOI:10.1093/nar/gkv332 ( 0) 0)
|
| [18] |
MORRIS A L, MACARTHUR M W, HUTCHINSON E G, et al. Stereochemical quality of protein structure coordinates[J]. Proteins, 1992, 12(4): 345–364.
DOI:10.1002/(ISSN)1097-0134 ( 0) 0)
|
| [19] |
KUWAJIMA K, YAMAYA H, MIWA S, et al. Rapid formation of secondary structure framework in protein folding studied by stopped-flow circular dichroism[J]. FEBS Letters, 1987, 221(1): 115–118.
DOI:10.1016/0014-5793(87)80363-0 ( 0) 0)
|
| [20] |
LAW S M, FRANK A T, BROOKS C L. PCASSO: a fast and efficient c alpha-based method for accurately assigning protein secondary structure elements[J]. Journal of Computational Chemistry, 2014, 35(24): 1757–1761.
DOI:10.1002/jcc.v35.24 ( 0) 0)
|
| [21] |
NAGY G, OOSTENBRINK C. Dihedral-based segment identification and classification of biopolymers I: proteins[J]. Journal of Chemical Information and Modeling, 2014, 54(1): 266–277.
DOI:10.1021/ci400541d ( 0) 0)
|
| [22] |
YU C Y, CHOU L C, CHANG D T. Predicting protein-protein interactions in unbalanced data using the primary structure of proteins[J]. BMC Bioinformatics, 2010(11): 167.
( 0) 0)
|
| [23] |
LEVITT M, GREER J. Automatic identification of secondary structure in globular proteins[J]. Journal of Molecular Biology, 1977, 114(2): 181–239.
DOI:10.1016/0022-2836(77)90207-8 ( 0) 0)
|
| [24] |
KABSCH W, SANDER C. Dictionary of protein secondary structure: pattern recognition of hydrogen-bonded and geometrical features[J]. Biopolymers, 1983, 22(12): 2577–2637.
DOI:10.1002/(ISSN)1097-0282 ( 0) 0)
|
| [25] |
FRISHMAN D, ARGOS P. Incorporation of non-local interactions in protein secondary structure prediction from the amino acid sequence[J]. Protein Engineering, 1996, 9(2): 133–142.
DOI:10.1093/protein/9.2.133 ( 0) 0)
|
| [26] |
FRISHMAN D, ARGOS P. Knowledge-based protein secondary structure assignment[J]. Proteins, 1995, 23(4): 566–579.
DOI:10.1002/(ISSN)1097-0134 ( 0) 0)
|
| [27] |
FODJE M N, Al-KARADAGHI S. Occurrence, conformational features and amino acid propensities for the pi-helix[J]. Protein Engineering, 2002, 15(5): 353–358.
DOI:10.1093/protein/15.5.353 ( 0) 0)
|
| [28] |
TOUW W G, BAAKMAN C, BLACK J, et al. A series of PDB-related databanks for everyday needs[J]. Nucleic Acids Research, 2015, 43(Database issue): D364–368.
( 0) 0)
|
| [29] |
ZACHARIAS J, KNAPP E W. Protein secondary structure classification revisited: processing dssp information with PSSC[J]. Journal of Chemical Information and Modeling, 2014, 54(7): 2166–2179.
DOI:10.1021/ci5000856 ( 0) 0)
|
| [30] |
LABESSE G, COLLOC’H N, POTHIER J, et al. P-SEA: a new efficient assignment of secondary structure from C alpha trace of proteins[J]. Computer Applications of Biosciences, 1997, 13(3): 291–295.
( 0) 0)
|
| [31] |
TETIN S Y, PRENDERGAST F G, VENYAMINOV S Y. Accuracy of protein secondary structure determination from circular dichroism spectra based on immunoglobulin examples[J]. Analytial Biochemistry, 2003, 321(2): 183–187.
DOI:10.1016/S0003-2697(03)00458-5 ( 0) 0)
|
| [32] |
KING S M, JOHNSON W C. Assigning secondary structure from protein coordinate data[J]. Proteins, 1999, 35(3): 313–320.
( 0) 0)
|
| [33] |
DUPUIS F, SADOC J F, MORNON J P. Protein secondary structure assignment through Voronoi tessellation[J]. Proteins-Structure Function and Bioinformatics, 2004, 55(3): 519–528.
DOI:10.1002/prot.10566 ( 0) 0)
|
| [34] |
MARTIN J, LETELLIER G, MARIN A, et al. Protein secondary structure assignment revisited: a detailed analysis of different assignment methods[J]. BMC Structural Biology, 2005(5): 17.
DOI:10.1186/1472-6807-5-17 ( 0) 0)
|
| [35] |
PARISIEN M, MAJOR F. A new catalog of protein beta-sheets[J]. Proteins-Structure Function and Bioinformatics, 2005, 61(3): 545–558.
DOI:10.1002/prot.20677 ( 0) 0)
|
| [36] |
MAJUMDAR I, KRISHNA S S, GRISHIN N V. PALSSE: a program to delineate linear secondary structural elements from protein structures[J]. BMC Bioinformatics, 2005(6): 202.
( 0) 0)
|
| [37] |
PARK S Y, YOO M J, SHIN J, et al. SABA (secondary structure assignment program based on only alpha carbons): a novel pseudo center geometrical criterion for accurate assignment of protein secondary structures[J]. BMB Reports, 2011, 44(2): 118–122.
DOI:10.5483/BMBRep.2011.44.2.118 ( 0) 0)
|
| [38] |
KONAGURTHU A S, LESK A M, ALLISON L. Minimum message length inference of secondary structure from protein coordinate data[J]. Bioinformatics, 2012, 28(12): i97–105.
DOI:10.1093/bioinformatics/bts223 ( 0) 0)
|
| [39] |
CAO C, XU S, WANG L. An algorithm for protein helix assignment using helix geometry[J]. Plos One, 2015, 10(7): e0129674.
DOI:10.1371/journal.pone.0129674 ( 0) 0)
|
| [40] |
CAO C, WANG G S, LIU A, et al. A new secondary structure assignment algorithm using Cα backbone fragments[J]. International Journal of Molecular Sciences, 2016, 17(3): 333.
DOI:10.3390/ijms17030333 ( 0) 0)
|
| [41] |
SRINIVASAN R, ROSE G D. A physical basis for protein secondary structure[J]. Proceedings of National Academy of Sciences of the United States of America, 1999, 96(25): 14258–1463.
DOI:10.1073/pnas.96.25.14258 ( 0) 0)
|
| [42] |
CUBELLIS M V, CAILLIEZ F, LOVELL S C. Secondary structure assignment that accurately reflects physical and evolutionary characteristics[J]. BMC Bioinformatics, 2005, 6(suppl4): s8.
DOI:10.1186/1471-2105-6-S4-S8 ( 0) 0)
|
| [43] |
SKLENAR H, ETCHEBEST C, LAVERY R. Describing protein structure: a general algorithm yielding complete helicoidal parameters and a unique overall axis[J]. Proteins, 1989, 6(1): 46–60.
DOI:10.1002/(ISSN)1097-0134 ( 0) 0)
|
| [44] |
RICHARDS F M, KUNDROT C E. Identification of structural motifs from protein coordinate data: secondary structure and first-level supersecondary structure[J]. Proteins, 1988, 3(2): 71–84.
DOI:10.1002/(ISSN)1097-0134 ( 0) 0)
|
| [45] |
TAYLOR W R. Defining linear segments in protein structure[J]. Journal of Molecular Biology, 2001, 310(5): 1135–1150.
DOI:10.1006/jmbi.2001.4817 ( 0) 0)
|
| [46] |
MANSIAUX Y, JOSEPH A P, GELLY J C, et al. Assignment of PolyProline II conformation and analysis of sequence-structure relationship[J]. Plos One, 2011, 6(3): e18401.
DOI:10.1371/journal.pone.0018401 ( 0) 0)
|
| [47] |
CARTER P, ANDERSEN C A F, ROST B. DSSPcont: continuous secondary structure assignments for proteins[J]. Nucleic Acids Research, 2003, 31(13): 3293–3295.
DOI:10.1093/nar/gkg626 ( 0) 0)
|
| [48] |
HUTCHINSON E G, THORNTON J M. PROMOTIF-a program to identify and analyze structural motifs in proteins[J]. Protein Science, 1996, 5(2): 212–220.
( 0) 0)
|
| [49] |
ZHANG W, DUNKER A K, ZHOU Y Q. Assessing secondary structure assignment of protein structures by using pairwise sequence-alignment benchmarks[J]. Proteins-Structure Function and Bioinformatics, 2008, 71(1): 61–67.
DOI:10.1002/(ISSN)1097-0134 ( 0) 0)
|
| [50] |
COLLOC’H N, ETCHEBEST C, THOREAU E, et al. Comparison of three algorithms for the assignment of secondary structure in proteins: the advantages of a consensus assignment[J]. Protein Engineering, 1993, 6(4): 377–382.
DOI:10.1093/protein/6.4.377 ( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14


