2. 广东省农业科学院农业生物基因研究中心,广州510640;
3. 西北农林科技大学生命科学学院,陕西 杨凌 712100
2. Agro-biological Gene Research Center, Guangdong Academy of Agricultural Sciences, Guangzhou 510640,China;
3. College of Life Sciences, Northwest A & F University, Yangling Shaanxi 712100,China
茉莉酸(Jasmonic acid,JA)是植物应对病虫害防御反应的关键激素,可以调控植物的生长发育、信号转导、基因表达以及应答外界刺激等[1]。当植物受到一系列生物或非生物刺激时,茉莉酸信号途径上的关键基因就会激活并表达[2]。在JA信号转导中需要多个基因的共同参与,而COI1/JAZ/MYC2则是其信号转导中最为核心的基因[3]。COI1(COR-insensitive 1)作为茉莉酸信号的受体蛋白,在其中发挥关键作用[4]。COI1(COR-insensitive1) 受体蛋白可以与SKP1(S-phase kinase-associated protein 1)、CUL1(Cullin1) 和RBX1(Ring-box1)组合并且形成SCFCOI1 泛素连接酶复合体,进而参与介导JA信号[5]。Xie等从拟南芥中分离到COI1 基因,并证明COI1是F-box 蛋白超家族成员,COI1蛋白功能缺失直接影响JA信号转导[6]。随后,Sheard等揭示了COI1与JAZ蛋白相互作用的感受机制,并且构建了COI1 蛋白的晶体结构:该结构与TIR1(生长素受体)结构相似。在其N 端有一个三螺旋的F-box 结构域,可以和SKP1蛋白结合形成SCFCOI1复合物,C 端为LRRs 结构域,可特异性识别底物[7]。
目前,尽管对拟南芥AtCOI1 基因进行生理生化等多方面的研究,然而,对COI基因家族在植物基因组中的进化规律仍不清楚。因此,本研究采用生物信息学方法,从藻类植物、苔藓植物、蕨类植物、裸子及单、双子叶植物等多谱系角度对COI基因家族进行比较基因组学研究,揭示植物COI基因家族的起源与进化规律。
1 材料与方法 1.1 COI家族同源基因的鉴定采用HMMsearch和BLAST两种检索方法对植物COI家族同源基因进行检索。(1)HMMsearch检索结果如下:首先,利用已报道AtCOI1 氨基酸序列进行BLASTp检索PLAZA数据库;其次,利用HMMER v3.0[8]软件包的hmmbuild程序建立COI蛋白家族保守结构域序列HMMprofile;最后,利用hmmsearch程序在默认参数下进行本地非冗余蛋白数据库的检索。(2)本地BLAST检索方法如下:以拟南芥的AtCOI1 氨基酸序列为靶序列,利用BioEdit v7.0软件中的BLASTp程序分别检索本地蛋白数据库(注:含有15种代表植物的非冗余蛋白序列,详见表 1);同时,在线检索NCBI数据库〔注:含绿藻类(Chlorophyta)、红藻类(Rhodophytes)、硅藻类(Bacillariophytes)、灰胞藻类(Glaucophytes)以及褐藻类(Phaeophytes)等低等藻类植物〕[9]。(3)利用SMART数据库[10]对HMMsearch和BLASTp检索所获得的COI同源候选蛋白进行结构域分析,并下载候选蛋白所对应基因序列和CDS序列进行基因结构分析[11]。
| 表 1 不同植物中 COI同源基因及相关信息 Table 1 The COI gene family in different plants and the related information |
采用在线EBI中 Clustalw2 (http://www.ebi.ac.uk/Tools/msa/clustalw2/)[12]在默认参数下对COI同源蛋白序列进行序列比对;然后,利用在线Gblock[13]对比对结果进行分析,并选择序列保守区段;最后,利用PhyML3.0软件,选择Jones-Taylor-Thornton (JTT)模型,bootstrap检验选用100次重复,Maximum Likelihood [14]构建COI蛋白家族系统发育树[15-17]。
1.3 COI家族基因结构、定位及区段重复分析利用基因结构系统(Gene Structure Display Server,GSDS)分析COI家族基因外显子-内含子的分布及相位[18]。利用PLAZA数据库WGMapping工具(http://bioinformatics.psb.ugent.be/plaza/genoe_mapping/index)对COI家族基因进行染色体定位。利用植物区段重复数据库(Plant Genome Duplication Database,PGDD)中Locus-Search在线工具(http://chibba.agtec.uga.edu/duplication/index/locus) 对COI家族基因进行区段重复分析[19]。
1.4 COI家族基因组织和诱导表达谱分析利用Bio-Array Resource for Plant Biology (BAR)(http://142.150.215.220/)数据库中的Rice eFP Browser[20]和 Arabidopsis eFP Browser[21]网页工具,对水稻和拟南芥COI家族基因进行分析,查找的不同发育时期以及诱导表达芯片数据[22]。
2 结果与分析 2.1 COI同源基因鉴定结果通过HMMsearch和BLAST检索结果发现,在所选择的7种陆生植物中一共发现了55个COIs同源基因。然而,在绿藻类(Chlorophytes)、红藻类(Rhodophytes)、 硅藻类(Bacillariophytes)、灰胞藻类(Glaucophytes)、及褐藻类(Phaeophytes)等基因组中均未发现COI同源基因。这表明,在低等的水生藻类植物基因组中,COI及其同源基因并未出现,见图 1。
此外,保守结构域分析表明(见图 2b),55个陆生植物COIs同源基因保守结构域可分为四种类型。第一种含有典型的F-box和LRR结构域(AtCOI2、 AtCOI3和AtCOI5);第二种类型仅含有典型的F-box结构域(AtCOI4);第三种类型仅含有典型的LRR结构域(AtCOI6);第四种类型既没有典型的F-box结构域,也没有典型的LRR结构域(AtCOI1和AtCOI7)。由此推测,COIs蛋白家族结构域分化可能会产生功能的分化。

|
图 1 植物COI家族基因在不同植物中同源基因的拷贝数 Figure 1 the number of COI homologue genes among different plant |
为了探究COI家族基因的进化规律,本研究利用检索的COI同源蛋白序列构建系统进化树。根据COI家族进化树的拓扑结构,可分为4个亚家族,命名为Subs.Ⅰ~Ⅳ(见图 2a)。序列比对结果发现,SmCOI7保守性较差,因此未包含于进化树。其中Sub.Ⅰ和Sub.Ⅲ两个亚家族包含了苔藓类、蕨类、裸子及单、双子叶植物的COIs;Sub. Ⅱ中也有苔藓类、蕨类、单子叶及双子叶植物的COIs;而Sub.Ⅳ只包含苔藓植物的COIs,并且在进化树的最下端。与此同时,苔藓植物的PpCOIs也分布于其它三个亚家族Subs. Ⅰ~Ⅲ。由此提示,植物COI家族基因可能最先起源于苔藓植物中,且在陆生植物中发生基因扩增后进而形成多个COI亚家族。
进一步分析发现,Sub.Ⅰ亚家族的AtCOI3即AtTIR1是生长素信号途径的受体蛋白,该亚家族所包含的COIs基因可能是参与生长素信号途径的候选基因。而Sub. Ⅲ亚家族中的AtCOI1是茉莉酸信号途径的受体蛋白,该亚家族中包含的COIs基因可能是参与茉莉酸途径的候选基因。由此表明,植物COI家族在陆生植物发生扩增后形成的多个亚家族已开始发生功能分化。
2.3 COI基因家族扩增与进化机制COI家族基因进行染色体定位结果发现(见图 3),在高粱、水稻、毛果杨和拟南芥染色体上均未发现串联重复现象。而利用PDGG数据库进行区段重复分析发现(见图 3),植物COI家族基因在进化过程中存在物种内区段重复,其中,在单子叶植物高粱基因组中有1对重复区段即SbCOI1/SbCOI2;而双子叶植物拟南芥和毛果杨基因组中共存在6对区段重复即AtCOI4/AtCOI5、AtCOI6/AtCOI7、PtCOI3/PtCOI4、PtCOI5/PtCOI6、PtCOI8/PtCOI9和PtCOI7/PtCOI10。
此外,在COI基因家族系统发育树中存在11对旁系同源基因,其中,苔藓类植物小立碗藓4对即PpCOI1/PpCOI2、PpCOI7/PpCOI8、PpCOI9/PpCOI11和PpCOI10/PpCOI12。蕨类植物江南卷柏1对即SmCOI3/SmCOI4,双子叶植物拟南芥和毛果杨共6对即AtCOI4/AtCOI5、AtCOI6/AtCOI7、PtCOI3/PtCOI4、PtCOI5/PtCOI6、PtCOI7/PtCOI10和PtCOI8/PtCOI9。旁系同源基因主要发生在物种内的基因复制,因此,该结果提示COI家族基因在各个物种内可能发生了特异性的扩增。
2.4 COI家族基因结构进化基因结构分析显示(见图 2c),植物COI家族基因结构表现多样性,主要体现在内含子的数目和长度上。从内含子数目上发现,COI家族基因的内含子数在2~5个之间,其中有31个COIs基因含有2个内含子,约占59.6%,15个COIs基因含有3个内含子,约占28.8%,4个COIs基因含有4个内含子,约占7.7%,2个COIs基因含有5个内含子,约占3.8% (见图 2c);从内含子长度上发现,该家族最短的内含子仅有47 bp(SmCOI7的第三个内含子),而最长的内含子有3236 bp(OsCOI9基因的第一个内含子)。内含子数目和长度的变化提示,COI家族基因在物种的进化过程中可能发生内含子的插入缺失。另外,鉴于裸子植物云杉(Picea sitchensis)基因组尚未完成测序,故PsCOIs基因的结构未能确定。
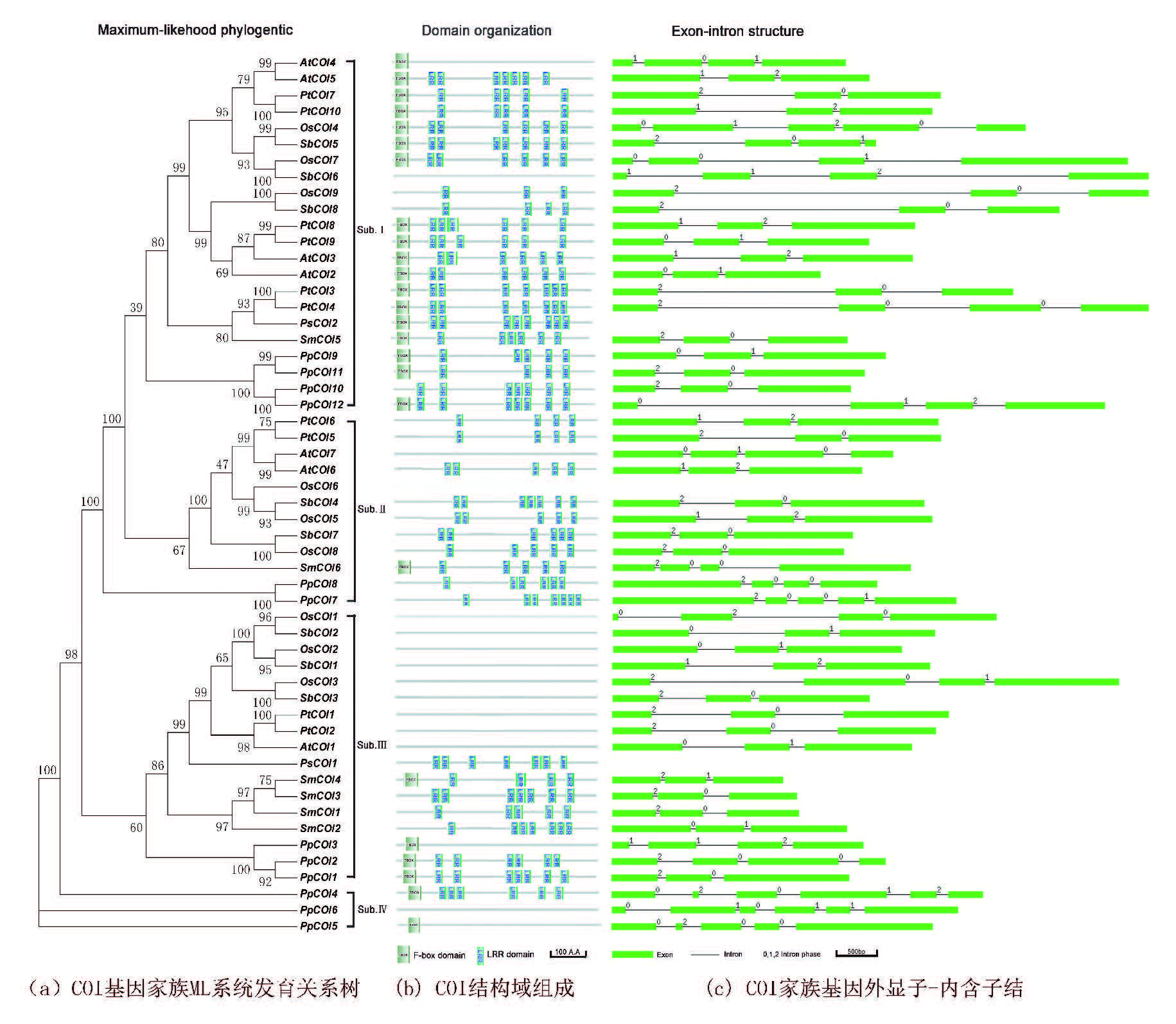
|
图 2 植物COI蛋白家族系统发育关系、保守结构域及基因结构* Figure 2 Phylogenetic relationship,domain organization and exon-intron structure of COI genes in plants 注:*图中绿方框代表外显子,线条代表内含子;数字0,1和2分别代表内含子的相位。 *彩图见电子版(http://swxxx.alljournals.cn/ch/index.aspx)(2016年第3期doi:10.3969/j.issn.1672-5565.2016.03.04 )。 |
进一步分析发现,苔藓类植物小立碗藓PpCOIs含有2~5个内含子,蕨类植物江南卷柏SmCOIs含有2~3个内含子,单子叶植物水稻OsCOIs和高粱SbCOIs含有2~4个内含子,双子叶植物拟南芥AtCOIs和毛果杨PtCOIs含有2~3个内含子。而COI家族基因中内含子的数目以2个或3个为主,约占88%。结合系统进化树发现,苔藓植物的PpCOI4和PpCOI6都含有5个内含子,且位于进化树的最下端。由此提示,COI基因家族在陆生植物发生扩增同时可能发生了内含子的丢失。
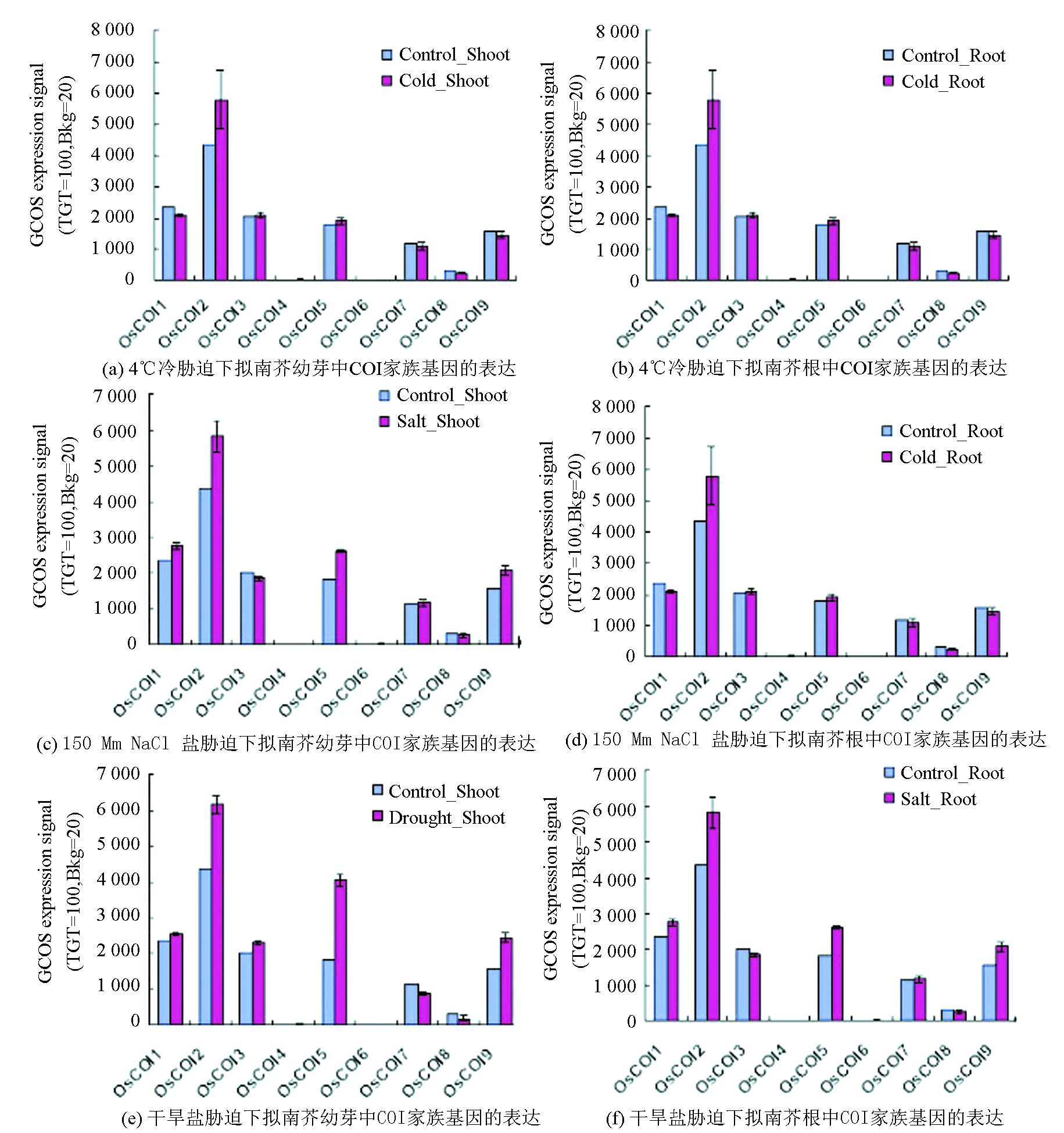
2.5 COI基因家族不同发育时期和诱导表达结果分析为了阐明COI基因家族在植物生长发育过程中可能存在的功能分化现象,我们对拟南芥和水稻基因芯片表达谱数据进行收集和分析。分析结果表明,拟南芥的7个AtCOIs在不同组织器官中均有表达,但是表达量存在差异(见图 4)。其中AtCOI2根和花发育12期花瓣中的表达量明显高于其他家族成员;AtCOI6在茎、叶和花发育12期心皮中的表达量也显著高于其他家族成员;AtCOI3在干种子的表达量最高。不同胁迫条件下,拟南芥AtCOIs家族成员的表达量也存在差异,具体表现为,在冷胁迫下(见图 5a~b),拟南芥幼芽中AtCOI7表达量有所上调,其他6个AtCOIs表达量均表现为下调;在拟南芥根中除AtCOI1表现为上调,其他6个AtCOIs表达量也表现为下调。在盐胁迫下(见图 5c~d),拟南芥幼芽中AtCOI2、AtCOI4和AtCOI5表达量明显下调,AtCOI3、AtCOI6和AtCOI7表现为上调;而在拟南芥根中除了AtCOI3表达量上调,其他6个6个AtCOIs表达量均出现下调。在干旱胁迫下(见图 5e~f),拟南芥COIs幼芽和根中表达规律一致,其中AtCOI1、AtCOI2和AtCOI4表达量明显下调,AtCOI3、AtCOI5、AtCOI6和AtCOI7表现为上调。

|
图 3 植物COI家族基因在染色体上的定位及物种内区段重复 Figure 3 Chromosomal locations and intra-specie segmental duplications of COI family genes in monocots and eudicots 注:虚线表示在物种内的COI同源基因重复区段。 |
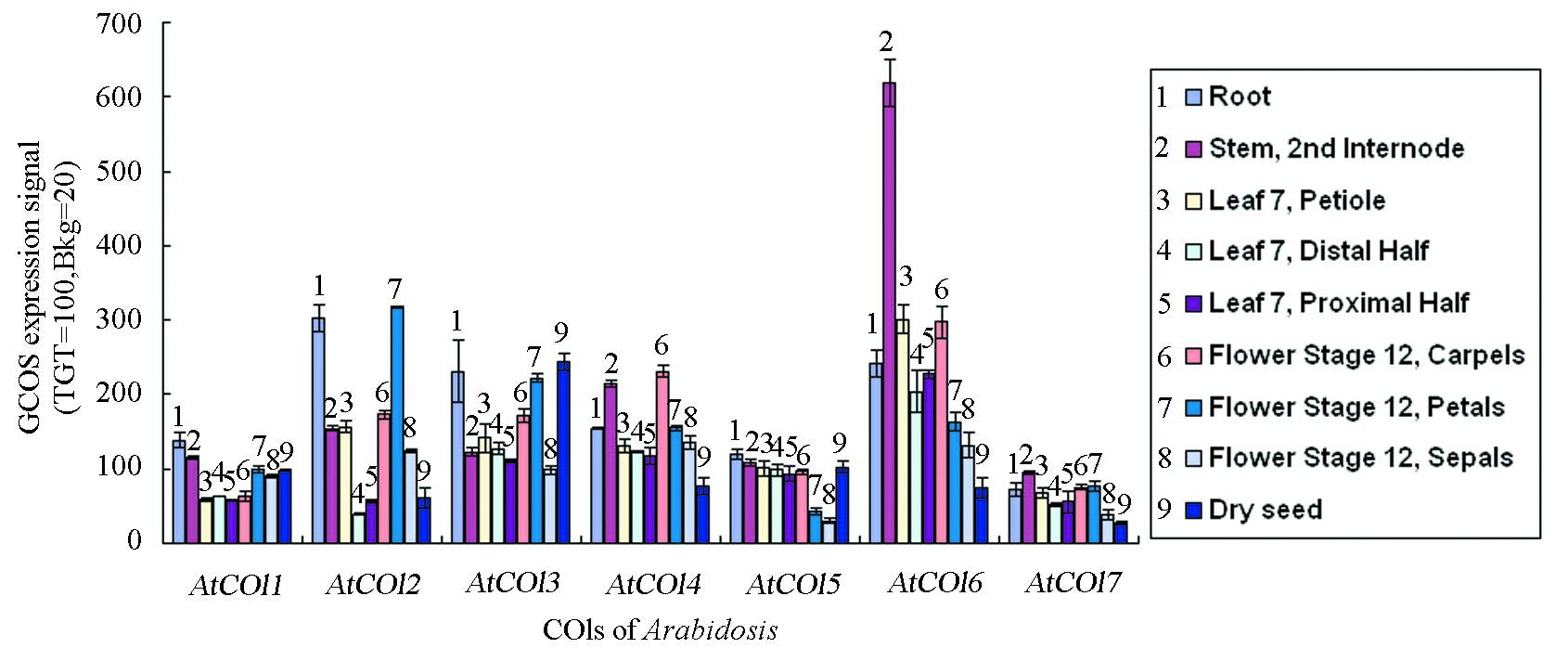
|
图 4 拟南芥COI家族基因在不同器官中的表达 Figure 4 The expression profiles COI family genes among different organs in Arabidosis 注:拟南芥的不同器官包括根、茎、第7叶(叶柄,叶近端半、叶远侧半)、花发育12期(萼片、花瓣、雄蕊)和种子。 |
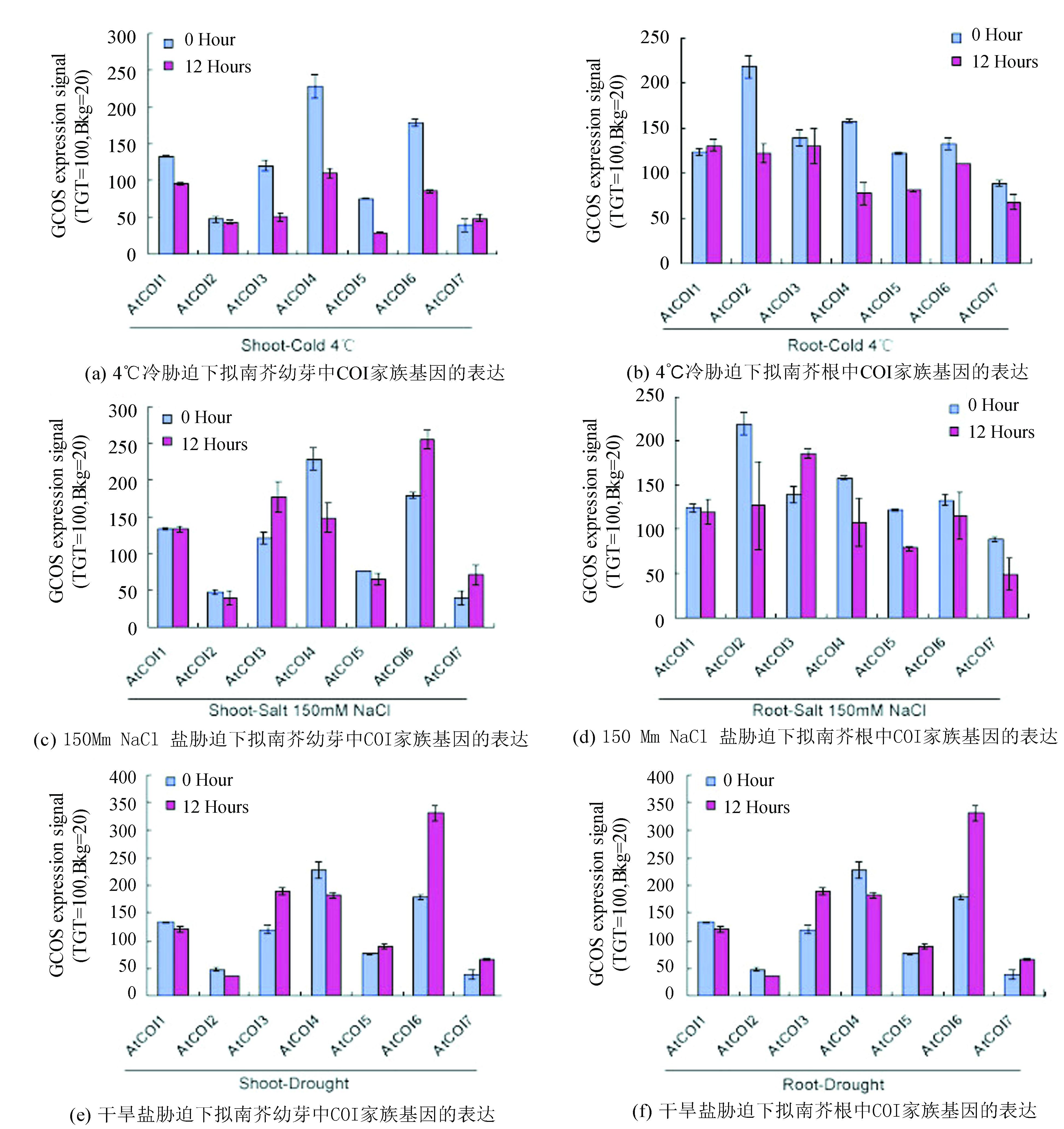
|
图 5 拟南芥 COI家族基因在不同胁迫下的表达 Figure 5 The expression profiles of COI family genes among different stress in Arabidosis |
与拟南芥相比,水稻的9个OsCOIs在所选的7个不同组织器官中均有不同程度的表达(见图 6)。其中OsCOI2在水稻叶、花以及种子的表达量显著高于其他8个OsCOIs,相反,OsCOI4和OsCOI6在所选的7个组织器官中表达量显著低于其他OsCO
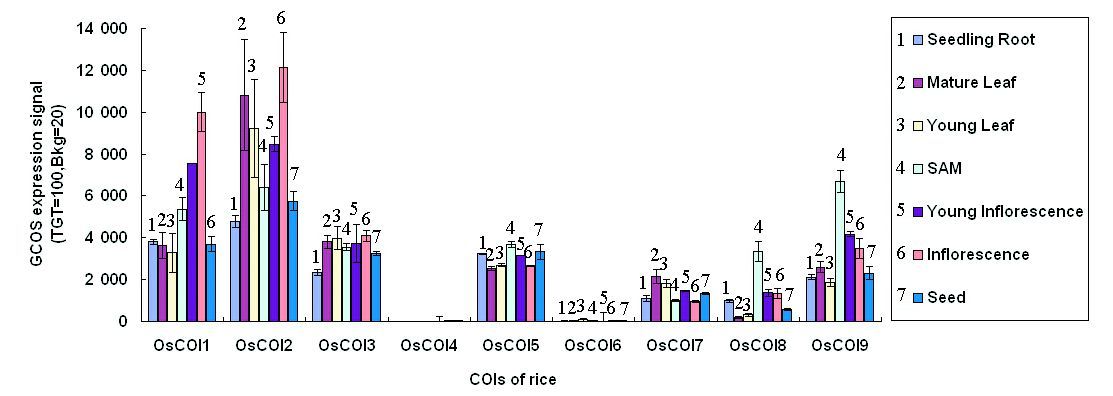
|
图 6 水稻COI家族基因在不同器官中的表达 Figure 6 The expression profiles COI family genes among different organs in rice 注:水稻不同器官包括根、叶(成熟叶、未成熟叶)、苗端分生组织、花和种子。 |
|
图 7 水稻COI家族基因在不同胁迫下的表达 Figure 7 The expression profiles of COI family genes among different stress in rice |
以上结果表明,COIs基因家族成员参与植物生长发育的多个时期,且在不同组织器官以及在不同的胁迫应答反应中发挥不同的作用。COI基因家族在拟南芥和水稻中表达出现了显著差异分化,有可能导致家族成员出现功能差异,从而增加了COI基因家族功能多样性和丰富度,但COI家族基因的表达也不是完全独立的,而是存在一定的规律,这说明他们之间的功能也存在一定的协作和相互作用。
3 讨论及结论通过HMMsearch和BLAST检索结果发现,在低等的藻类基因组中均未发现COI同源基因。然而,在所选的代表陆生植物中均发现了COI同源基因,且存在多个拷贝。已有研究表明,在低等水生藻类植物中有发现F-box家族成员,但是数量相对较少;而在陆生植物中发现大量的F-box家族成员,其中拟南芥和水稻中分别有692和779个[23]。结构域分析发现,茉莉酸受体COI1含有F-box-like,而 F-box-like(PF12937)结构域即分化的F-box(PF00646)结构域。由此推测,F-box基因家族在由水生到陆生植物的进化过程中发生大量基因扩增,同时,在陆生植物中形成了一类新的家族即COI基因家族。
在拟南芥中,AtCOI1是茉莉酸的受体,AtCOI3(TIR1)是生长素的受体[24],两者在序列和结构上均有较高的同源性,然而,在功能上已出现了分化现象。此外,从拟南芥中鉴定的AtCOIs家族成员中,除AtCOI1外,其他6个家族成员AtCOI2(AFB1) 、AtCOI3(TIR1)、AtCOI4(AFB3)、AtCOI5(AFB2)、AtCOI6(AFB5)和AtCOI7(AFB4)都是生长素信号的关键组分[14]。值得一提的是,系统发育树提示,AtCOI1和其他6个AtCOIs有明显的聚类分歧关系。具体表现为,Sub. Ⅲ亚家族中仅存在一个拟南芥基因即(AtCOI1)。同时,SMART结构域分析显示,该亚家族中COIs基因都没有典型的F-box和LRR 结构域(见图 2b)。但是利用Pfam (http://pfam.janelia.org/)对AtCOI1进行结构域分析显示,AtCOI1含有F-box-like和LRR_4结构域,但是均为insignificant即不典型,E值分别为0.009 7和0.069。由此推测,植物COI家族基因的主要功能是参与植物激素的信号调控,而在进化的过程中发生了功能的分化现象,既有参与生长素信号的调控,也有参与茉莉酸信号的调控。
同源基因在不同的组织器官中表达,可为该家族基因的生理、生化、基因功能分化研究提供重要的信息。通过分析拟南芥和水稻COIs基因表达数据发现,COIs基因在不同组织器官中都有表达,但在不同组织部位以及不同胁迫下的表达存在着差异。由此推测,植物COIs家族在由低等到高等植物的进化的过程中,为适应外界环境而分化出不同的家族成员,且不同家族成员之间存在明显的功能差异,进而增加了COI蛋白功能多样性和丰富度。
| [1] |
MOSBLECH A, THUROW C, GATZ C, et al. Jasmonic acid perception by COI1 involves inositol polyphosphates in Arabidopsis thaliana[J]. The Plant Journal, 2011, 65(6): 949–957.
DOI:10.1111/tpj.2011.65.issue-6 ( 0) 0)
|
| [2] |
FONSECA S, CHICO J M, SOLANO R. The jasmonate pathway: the ligand, the receptor and the core signalling module[J]. Current Opinion in Plant Biology, 2009, 12(5): 539–547.
DOI:10.1016/j.pbi.2009.07.013 ( 0) 0)
|
| [3] |
CHINI A, BOTER M, SOLANO R. Plant oxylipins: COI1/JAZs/MYC2 as the core jasmonic acid-signalling module[J]. The FEBS Journal, 2009, 276(17): 4682–4692.
DOI:10.1111/j.1742-4658.2009.07194.x ( 0) 0)
|
| [4] |
KATSIR L, SCHILMILLER A, STASWICH P E, et al. COI1 is a critical component of a receptor for jasmonate and the bacterial virulence factor coronatine[J]. Proceedings of National Academy of Sciences of the United States of America, 2008, 105(19): 7100–7105.
DOI:10.1073/pnas.0802332105 ( 0) 0)
|
| [5] |
ADAMS E, TURNER J. Illuminating COI1: a component of the Arabidopsis jasomonate receptor complex also interacts with ethylene signaling[J]. Plant Signaling & Behavior, 2010, 5(12): 1682–1684.
( 0) 0)
|
| [6] |
XIE D X, FEYS B F, JAMES S, et al. COI1: an Arabidopsis gene required for jasmonate-regulated defense and fertility[J]. Science, 1998, 280(5366): 1091–1094.
DOI:10.1126/science.280.5366.1091 ( 0) 0)
|
| [7] |
SHEARD L B, TAN X, MAO H, et al. Jasmonate perception by inositol-phosphate-potentiated COI1-JAZ co-receptor[J]. Nature, 2010, 468(7322): 400–405.
DOI:10.1038/nature09430 ( 0) 0)
|
| [8] |
WONG W C, YAP C K, EISENHABER B, et al. dissectHMMER: a HMMER-based score dissection framework that statistically evaluates fold-critical sequence segments for domain fold similarity[J]. Biology Direct, 2015(10): 39.
DOI:10.1186/s13062-015-0068-3 ( 0) 0)
|
| [9] |
段龙飞, 慕小倩, 李文燕. 茉莉酸信号途径中转录抑制因子JAZ蛋白家族的分子进化分析[J]. 植物学报, 2013, 48(6): 623–624.
DUAN Longfei, Mu Xiaoqian LI Wenyan. Molecular evolution of transcriptional repressor JAZ protein family in jasmonic acid signaling pathway[J]. Chinese Bulletin of Botany, 2013, 48(6): 623–624.
( 0) 0)
|
| [10] |
LETUNIC I, COPLEY R R, SCHMIDT S, et al. SMART 4.0: towards genomic data integration[J]. Nucleic Acids Research, 2004, 32(Database issue): D142–144.
( 0) 0)
|
| [11] |
段龙飞. 茉莉酸信号途径上关键基因家族COI/JAZ/MYC分子进化分析[M]. 杨凌:西北农林科技大学,2013.
DUAN Longfei.Molecular evolution analysis of the key gene families COI/JAZ/MYC in Jasmonic Acid Signaling Pathway[M]. Yangling:Northwest A&F University,2013.
http://cdmd.cnki.com.cn/article/cdmd-10712-1013345380.htm ( 0) 0)
|
| [12] |
LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. ClClustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947–2948.
DOI:10.1093/bioinformatics/btm404 ( 0) 0)
|
| [13] |
TALAVERA G, CASTRESANA J. Improvement of phylogenies after removing divergent and ambiguously aligned blocks from protein sequence alignments[J]. Systematic Biology, 2007, 56(4): 564–577.
DOI:10.1080/10635150701472164 ( 0) 0)
|
| [14] |
BASTER P, ROBERT S, KLEINE-VEHN J, et al. SCF(TIR1/AFB)-auxin signalling regulates PIN vacuolar trafficking and auxin fluxes during root gravitropism[J]. The EMBO Journal, 2013, 32(2): 260–274.
DOI:10.1038/emboj.2012.310 ( 0) 0)
|
| [15] |
LI Wenyan, WANG Xiang, LI Ri, et al. Genome-wide analysis of the NADK gene family in plants[J]. PLoS One, 2014, 9(6): e101051.
DOI:10.1371/Journal.pone.0101051 ( 0) 0)
|
| [16] |
LI W, LIU B, YU L, et al. Phylogenetic analysis, structural evolution and functional divergence of the 12-oxo-phytodienoate acid reductase gene family in plants[J]. BMC Evolutionary Biology, 2009(9): 90.
DOI:10.1186/1471-2148-9-90 ( 0) 0)
|
| [17] |
GUINDON S, DELSUC F, DUFAYARD J F, et al. Estimating maximum likelihood phylogenies with PhyML[J]. Methods in Molecular Biology, 2009(537): 113–137.
DOI:10.1007/978-1-59745-251-9_6 ( 0) 0)
|
| [18] |
Guo A Y, Zhu Q H, Chen X, et al. GSDS: a gene structure display server[J]. Hereditas, 2007, 29(8): 1023–1026.
( 0) 0)
|
| [19] |
LEE T H, TANG H, WANG X, et al. PGDD: a database of gene and genome duplication in plants[J]. Nucleic Acids Research, 2013, 41(Database issue): D1152–1158.
DOI:10.1093/nar/gks1104 ( 0) 0)
|
| [20] |
JAIN M, NIJHAWAN A, ARORA R, et al. F-box proteins in rice. Genome-wide analysis, classification, temporal and spatial gene expression during panicle and seed development, and regulation by light and abiotic stress[J]. Plant Physiology, 2007, 143(4): 1467–1483.
DOI:10.1104/pp.106.091900 ( 0) 0)
|
| [21] |
SCHMID M, DAVISON T S, HENZ S R, et al. A gene expression map of Arabidopsis thaliana development[J]. Nature Genetics, 2005, 37(5): 501–506.
DOI:10.1038/ng1543 ( 0) 0)
|
| [22] |
汤龙军, 朱璐, 王瑞丰, 等. 天冬氨酸蛋白酶在拟南芥和水稻中的分子进化?表达模式以及在花药发育中的功能分析[J]. 植物生理学报, 2015, 51(3): 323–336.
TANG Longjun, ZHU Lu, WANG Ruifeng, et al. Identification, evolutionary and expression profile analysis of the Aspartic Protease gene superfamily in Arabidopsis thaliana and rice[J]. Plant Physiology Journal, 2015, 51(3): 323–336.
DOI:10.13592/j.cnki.ppj.2014.0621 ( 0) 0)
|
| [23] |
XU G, MA H, NEI M, et al. , Evolution of F-box genes in plants: different modes of sequence divergence and their relationships with functional diversification[J]. Proceedings of National Academy of Sciences of the United States of America, 2009, 106(3): 835–840.
DOI:10.1073/pnas.0812043106 ( 0) 0)
|
| [24] |
QI J, LI J, HAN X, et al. , Jasmonic acid carboxyl methyltransferase regulates development and herbivory-induced defense response in rice[J]. Journal of Integrative Plant Biology, 2015, 58(6): 564–76.
DOI:10.1111/jipb.12436 ( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14


