蛋白质折叠规律研究是生命科学重大前沿课题,折叠分类是蛋白质折叠研究的基础。围绕蛋白质折叠类型进行系统化研究,将为蛋白质的功能分类和预测[1]提供依据,研究结果用于蛋白质空间结构预测,可缩小蛋白质三级结构预测的搜素范围,加快搜索的速度[2]。
蛋白质折叠类型是一种粗粒化的结构,反映了蛋白质核心结构的拓扑模式[3-6]。蛋白质折叠类型包括蛋白质分子空间结构的三个主要方面:二级结构单元、二级结构单元的相对排布位置以及蛋白质多肽链的整个路由关系(即肽链走向)[7]。蛋白质的空间结构十分复杂,但它的框架结构(折叠类型或拓扑结构或折叠子)却相对简单[8]。现在一般认为蛋白质的折叠类型是有限的,只有数百到数千种[9-10],许多同源性很差的蛋白质却存在相同的骨架结构——折叠子[11],进一步的研究也表明,蛋白质的折叠速率和折叠机制,在很大程度上是由天然状态的拓扑所决定的[12]。因此,对自然界存在的数百到数千种折叠类型进行系统研究,探索构建蛋白质折叠类型模板的方法,建立蛋白质折叠类型分类方法,为进一步识别研究奠定基础。
目前,SCOP[13]数据库是蛋白质结构分类数据库的典型代表,包括蛋白质结构类、折叠类型、超家族、家族等不同层次,其中蛋白质折叠类型,由专家凭经验指定,是手工分类的结果,伴随PDB数据库资料的迅速增长,专家人为指定存在的弊端日益突显。2013年,SCOPe[14]数据库建立,利用ASTRAL提供的几个有助于蛋白质结构分类的工具,在SCOP已有分类的基础上,对新的蛋白质结构样本进行自动管理并归类,但依然有很多蛋白质不能使用自动管理方法,需要通过手动选择来完成。最近7年,SCOP数据中折叠层所包含的折叠类型总数基本保持在1 393种左右,折叠类型总数基本稳定。如何利用并进一步挖掘SCOP人工分类结果,并在此基础上建立蛋白质折叠类型分类方法,实现蛋白质折叠类型的自动分类,是迫切需要解决的问题。
蛋白质折叠类型分类方法的建立,首先需要解决的是折叠类型对应的模板的选取问题。研究结果表明,模板的好坏直接影响了预测结果的好坏,即预测的结果倾向于模板的模型[15]。在折叠识别选择模板时,第一步选通过序列比对在结构数据库中寻找同源性高、结构上冗余小、分辨率高并且折叠核心清晰的天然蛋白质作为原始模板,这些模板具有相似的二级结构组成、数目和排列方式,第二步建立具体模板时,将目标序列与第一步的天然模板进行序列比对,是目标氨基酸残基和模板的残基匹配,并确定保守区和可变区,保留保守区中不连续的二级结构片段作为过程模板,之后对过程模板进行优化并构建侧链和环区得到最优模板。折叠类型分类的模板选择方法和蛋白质结构预测中折叠识别的模板选择的方法类似,折叠类型分类只是对已知结构的蛋白进行分类,在以往的蛋白质折叠类型分类方法[16-17]中,通常会选取一个天然蛋白质作为折叠类型模板,所选的天然蛋白质在结构上冗余少并且折叠核心清晰。但我们的研究发现:在一个以结构简单的天然样本作为模板的分类结果中,折叠类型内部部分样本的分类结果并不好,其原因是在一个蛋白质折叠类型内部,通常会包含多了家族和多个超家族,以结构简单的天然样本为模板,该模板具有所在家族的个性化结构特征,但不足以代表折叠类型所属全部超家族样本的共性特征,即普适性不够;另外,蛋白质折叠类型的模板应该围绕折叠核心的的规则二级结构片段(保守区域结构)来构建,这样天然模板折叠核心以外的其它结构(非保守结构loop区域)会干扰折叠分类的结果,因此,需要通过设计反映折叠类型特征的无结构冗余的多模板来解决上述问题。本文将利用前期我们给出了BRD-like折叠类型模板设计方法[18],设计生成该折叠类型模板,设计的模板具有普适性,能够用于蛋白质的分类,并用于本文的综合分类方法的建立。
基于模板的分类方法需要建立一个量化的评判方法。通常,蛋白质的折叠分类方法是将已知空间结构的待测蛋白和折叠类型的模板进行结构比对,以结构比对的量化打分函数来确定待测蛋白是否属于某一折叠类型。结构比对是蛋白质结构分类的基础,目前结构比对算法如CE[19]、DALI[20]、SSM[21]、TM-align[22]、MUSTANG[23]、GOSSIP[24]。CE是基于组合扩展的方法但发时间较早,DALI是在两蛋白质间寻找最佳的距离比对并生成距离矩阵得到Z-score,该方法忽略了结构比对后建模的准确性且很大程度上依赖于蛋白质的序列长度,MUSTANG是在DALI双结构比的基础上发展的一种多结构比对方法,对于空间折叠、残基接触模式有较强的识别能力,TM-align是一个基于TM-score结构比对程序,其比对速度是CE比对的4倍,是DALI的20倍[22]。同时,TM-align利用比对结果计算待测蛋白与模板的α-碳原子坐标距离生成打分函数,得到两个比对质量的评估参数RMSD和TM-score,若TM-score>0.5,待测蛋白质通常与模板属于同一折叠类型,即以TM-score阈值0.5作为折叠类型分类的基础,TM-score克服了打分值与蛋白质大小的幂率依赖[16],但是,TM-score是基于单模板比对的打分,仅利用TM-score来评判分类,无法克服单模板分类的弊端,并且以0.5作为TM-score阈值的分类结果并不理想。
利用多模板的TM-score结果,建立BRD-like折叠类型综合分类方法。依据多模板打分的综合分类方法的建立,利用多模板之间的互补性能够解决单模板在结构上的单一性问题,提高分类准确性,此外,多模板的综合分类方法将模板的分类阈值提高,从而进一步提高分类的正确性。该综合分类方法的建立,对其它蛋白质折叠类型综合分类方法的建立具有示范和借鉴作用,并为统一的蛋白质折叠类型综合分类方法的建立奠定基础。
1 材料和评估参数 1.1 材料 1.1.1 实验集和独立检验集Bromodomain(BRD)蛋白因其在基因转录过程中发挥重要的作用,并与肿瘤、神经紊乱、炎症、肥胖和心血管疾病发生相关[25]成为近年的研究热点。BRD家族在人体内能特异性识别蛋白中的乙酰化赖氨酸(KAc)[26],并具有辨别不同蛋白结合物的能力[27-29],是蛋白质交互模块中探索药物发现领域的代表。
实验集:SCOPe astral 2.03数据库序列相似度小于40%、分辨率高于0.25 nm的全部12 117样本。其中BRD-like折叠类型对应Bromodomain(BRD)蛋白,样本总数为52,记为Set-Ⅰ,图 1为BRD蛋白结构及其对应的拓扑结构模型,该折叠类型在SCOPe Astral 2.03数据库中其对应编号为a.29,包含15个超家族、20个家族。数据集中非BRD-like折叠类型的样本为12 065,记为SSet-Ⅱ。

|
图 1 BRD蛋白模型和拓扑结构模型 Figure 1 BRD protein model and topological structure model |
独立检验集:SCOPe astral 2.05中剔除SCOPe astral2.03所含样本,余下的2 260样本,记为Set-Ⅲ。Set-Ⅲ中,17个样本属于BRD-like折叠类型,2 243个样本属于非BRD-like折叠类型样本。
1.1.2 模板信息及模板数据库在前期工作中[18],我们利用Set-Ⅰ样本,通过多结构比对及数据分析,建立了折叠类型家族模板的设计方法,并结合家族模板的系统聚类图,提出了蛋白质折叠类型模板的设计方法。利用该方法对BRD-like折叠类型设计生成了4个模板,分别记为Model_1、Model_2、Model_3、Model_4,模板的文本信息见表 1,其对应的结构信息以Model-ID为文件名,保存在相应的PDB格式文件中,并形成模板数据库。Model_1的ID号为a. 29. 2. 0_2. 1,其中α代表结构类,即全α类,29代表SCOPe astral 2.03数据库中BRD-like折叠类型的编号,2. 0_2. 1代表形成该模板的2. 0和2. 1超家族和家族,其它模板ID编号类同。
| 表 1 BDR折叠类型蛋白质的模板信息 Table 1 Template information of BRD-like type |
打分函数TM-score[16](Template Model Score,模板建模打分)定义为:
| $TM - sore = \frac{1}{L}{\left[{\sum\limits_{i = 1}^{{L_{ali}}} {\frac{1}{{1 + d_i^2/d_0^2}}} } \right]_{\max }}$ |
其中L是模板蛋白的长度,Lali是模板蛋白与待测蛋白中等价残基的数量,di是模板蛋白与待测蛋白质中第i个等价残基之间的距离,d0[16]的定义是将TM-score标准化,使得打分值与蛋白质大小不存在幂率的关系。TM-score的取值范围为(0, 1],取值越大,表明待测蛋白与模板蛋白相似性越高。TM-score>0. 5,待测蛋白与模板蛋白属于同一折叠类型,否则为不同折叠类型[16]。
利用敏感性、特异性、Matthew相关系数三个指标对分类方法进行评估,参数定义如下:
| $\begin{array}{l} {\rm{敏感性:}}{S_n} = \frac{{{t_p}}}{{{t_p} + {f_n}}} \times 100\% \\ {\rm{特异性}}:{S_p} = \frac{{{t_n}}}{{{t_n} + {f_p}}} \times 100\% \end{array}$ |
相关系数:
| $MCC = \frac{{\left( {{t_p} \times {t_n}} \right)-\left( {{f_n} \times {f_n}} \right)}}{{\sqrt {\left( {{t_p} + {f_n}} \right) \times \left( {{t_n} + {f_p}} \right) \times \left( {{t_p} + {f_p}} \right) \times \left( {{t_n} + {f_n}} \right)} }}$ |
式中tp为真阳性个数,tn为真阴性个数,fp为假阳性个数,为fn假阴性个数。
2 分类方法与结果讨论 2.1 基于单模板的分类方法及结果讨论 2.1.1 TM-score计算及统计分析对Set-Ⅰ及Set-Ⅱ数据集中任意样本,分别与Model_1~Model_4进行TM-align比对,并计算TM-score,分别记为TM-score1~TM-score4,部分结果见表 2。
| 表 2 实验集中样本的TM-score Table 2 The TM-score of training set |
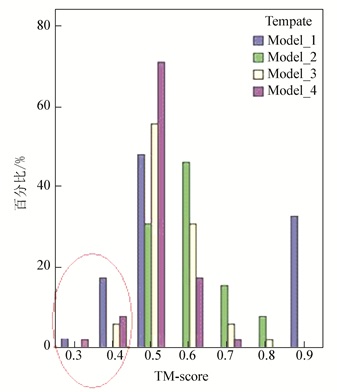
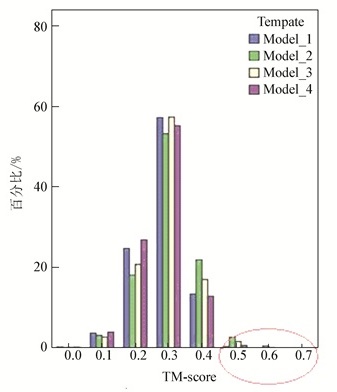
根据表 2的TM-score数据,分别对Set-Ⅰ、SSet-Ⅱ所属的TM-score数据,以模板为分组变量,进行描述性统计分析,Set-Ⅰ对应的TM-score的分组直方图见图 2,SSet-Ⅱ对应的TM-score的分组直方图见图 3。图 2和图 3中圈内的部分分别代表TM-score小于0.5和TM-score大于0.5;各个模板的统计指标见表 3,其中mean代表均值,confidence interval为均值95%的置信区间,max代表SSet-Ⅱ的TM-score最大值,min代表Set-Ⅰ的TM-score最小值。
|
图 2 实验集SSet-Ⅰ的TM-score直方图 Figure 2 The TM-score histogram of Set-Ⅰ |
|
图 3 实验集SSet-Ⅱ的TM-score直方图 Figure 3 The TM-score histogram of SSet-Ⅱ |
由表 3、图 2、图 3可知,Set-Ⅰ中,Model_1对应的TM-score最小值为0.37 nm,均值为0.66 nm;SSet-Ⅱ中,Model_1对应的TM-score最大值为0.61 nm,均值为0.33 nm。Set-Ⅰ和SSet-Ⅱ的TM-score均值相差较大,数值分布区间重叠部分较小,其它模板类同。说明设计模板的TM-score取值在所属折叠类型内部及非所属折叠类型内部具有良好的聚集性,而在两者之间具有离散性,这与张扬文章[16]中基于天然模板的TM-score分布是一致的,说明设计模板与天然模板具有相同的TM-score取值分布。
| 表 3 各个模板的统计指标 Table 3 The TM-score statistical index of templates |
由图 2不同模板对Set-Ⅰ的TM-score数值分布图可知:当TM-score数值在0.5~0.7时,四个模板能够识别本折叠类型的大多数样本,说明不同模板具有相同折叠类型的属性;当TM-score数值大于0.8时,只有Model_1能够识别的样本数较多,为30%左右,其它三个模板识别数在10%以下,Model_1的TM-score分布与其它模板不同,出现两级分化现象,Model_2与Model_3和Model_4的TM-score峰值位置也不同,说明模板间具有差异性。SSet-Ⅱ中,不同模板的TM-score分布基本一致,呈正态分布。
2.1.2 基于单模板的分类结果根据表 2的计算结果,将TM-score取值0.5作为分类阈值,当TM-score≥0.5时,待测蛋白与模板蛋白属于同一折叠类型,否则为不同折叠类型[16]。分别计算Model_1~Model_4的敏感性、特异性及Mattew相关系数,结果见表 4。表中S表示BRD-like折叠类型样本数量,S'表示打分在0.5以上的样本数量。
| 表 4 不同模板的敏感性、特异性以及MCC值 Table 4 Sensitivity, specificity and MCC of different template |
由表 4可知,4个模板的敏感性均在80%以上,特异性在95%以上,说明设计模板本身抓住了折叠类型的基本特征,具有相同的折叠类型属性,模板设计是合理的,但MCC值均未达到0.6,且敏感性高对应的特异性会低,即敏感性、特异性是一对矛盾体。
对于单模板分类,提高TM-score的阈值,特异性会提高,但敏感性会降低,降低TM-score的阈值,敏感性会提高,特异性又会降低,矛盾无法解决。
2.2 基于多模板的综合分类方法及结果讨论如何使MCC值得到提高,同时特异性、敏感性也保持较高水平?需要综合利用多模板打分,建立基于设计模板的综合分类方法。
2.2.1 模板的互补性分析为进一步检验模板之间的相似性和差异性,将任意两模板进行TM-align比对,获得模板之间的RMSD和TM-score,见表 5。
| 表 5 各个模板之间的RMSD和TM-score Table 5 The RMSD and TM-score between the templates |
可知,模板间两两比对后的RMSD都在0.4 nm以内,打分值都在0.5以上,说明各个模板具用相同折叠类型的属性,即模板间具有相似性。但模板间的TM-score均小于0.61,说明各个模板间存在差异性。
在Set-Ⅰ数据集内部,对表 2提供的TM-score1~TM-score4的4组数据,利用SPSS软件计算任意两组间Pearson相关系数,结果见表 6。
| 表 6 Pearson相关系数 Table 6 The Pearson Correlation |
表 6中,Pearson相关系数的绝对值均在0.5以下。Pearson相关系数小说明:相同样本不同模板打分值之间关联度比较小,不同模板的TM-score数组间不存在共线性问题,模板彼此相对独立;另外,Model_1打分TM-score1与Model_2~Model_4打分的TM-score2~TM-score4数组间为负相关,说明对相同样本,对应的打分值存在取值大小上的互补性。
2.2.2 双模板分类方法及结果讨论提高TM-score阈值,并采用双模板组合对实验集Set-Ⅰ和SSet-Ⅱ进行分类,并按照以下原则搜索可能的双模板阈值组合:能识别Set-Ⅰ中95%以上样本;每个模板的阈值大于0.5且能识别Set-Ⅰ中50%(识别数为26)以上样本。选取其中模板互补性良好的阈值组合,并对实验集样本进行分类,结果见表 7。
| 表 7 双模板组合的敏感性、特异性以及MCC值 Table 7 Sensitivity, specificity and MCC of two templates |
由表 7可知,采用双模板打分并且提高阈值以后,MCC值提高到0.63以上,分类结果的敏感性和特异性与单模板相应结果比也均有提高。说明利用模板间的互补性进行折叠类型分类,既提高了打分函数的阈值,也提高了敏感性、特异性及MCC值。
2.3 综合分类方法的建立对BRD-like折叠类型,设计生成了4个模板,综合利用四个模板的差异性及其在分类识别中的互补性,建立综合分类方法,提高分类方法的有效性。
四模板最佳阈值组合寻找方法:
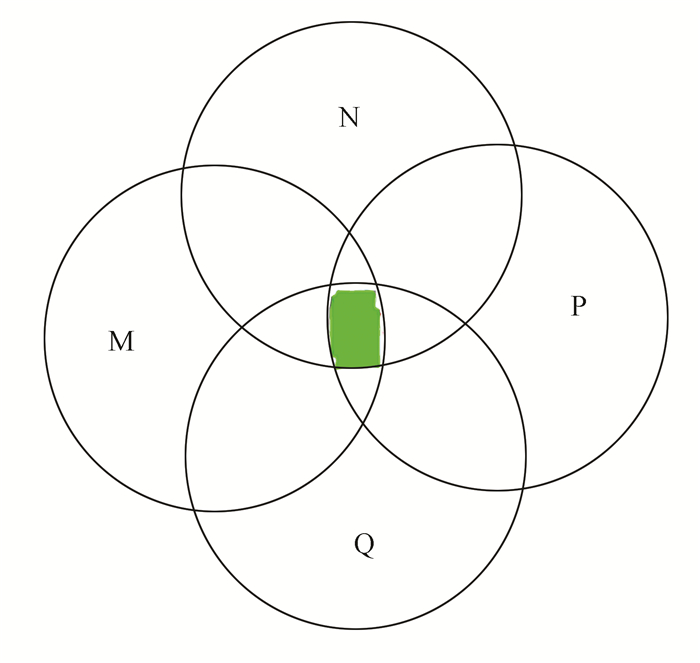
(1) 假设Model_1~Model_4模板的阈值分别为score1、score2、score3和score4,阈值以上能够识别Set-Ⅰ数据集样本个数分别为M、N、P、Q,见图 4。
|
图 4 四个模板对于Set-Ⅰ实验集中样本的识别个数 Figure 4 The dentification number of four templates for the Set-Ⅰ database |
(2) 根据集合的容斥原理,得到四模板综合打分的识别总数,M∪N∪P∪Q。根据四模板的阈值TM-score组合,得到四模板打分的最佳阈值的组合。
(3) 集合的容斥原理如下:
| $\begin{array}{c} M \cup N \cup P \cup Q = M + N + P + Q-M \cap N-M\\ \cap P-M \cap Q - N \cap P - N \cap \\ Q - P \cap Q + M \cap N \cap P + M\\ \cap N \cap Q + M \cap P \cap Q + N \cap \\ P \cap Q - M \cap N \cap P \cap Q \end{array}$ |
利用最佳阈值组合方式筛选本折叠类型的52个样本,得到正确识别50、51、52个样本的阈值组合为分别为244 244、302 907、205 600,占阈值组合的比例分别为3.3%、4.1%、2.8%。
从正确识别52个样本的205 600种阈值组合中,选取每个模板正确识别数在13以上且对应模板的阈值大于0.5的阈值组合,对Set-Ⅰ及SSet-Ⅱ进行分类,分类的敏感性均为100%,特异性在98.23%以上,但MCC值均低于0.62。且最佳阈值组合对表 2中TM-score取值依赖性强,阈值的普适性也比较差。
进一步对正确识别数为52的最佳阈值组合进行统计分析,我们发现:38.5%的组合可以简化为:ScoreMax≥0.60,且ScoreLarge≥0.55的阈值组合;31.9%的组合可以简化为:
| $\begin{array}{l} \overline {Scor{e_{{\rm{Highly-2}}}}} = \frac{1}{2}\left( {Scor{e_{{\rm{Large}}}} + Scor{e_{{\rm{Max}}}}} \right)\\ \overline {Scor{e_{Average}}} = \frac{1}{4}\left( {Scor{e_{{\rm{Min}}}} + Scor{e_{{\rm{Minor}}}} + Scor{e_{{\rm{Large}}}} + Scor{e_{{\rm{Max}}}}} \right) \end{array}$ |
分别代表待分类样本与四模板打分值TM-score1~TM-score4的由小到大排序。
基于上述分析,建立综合分类方法,对于任意待分类样本,满足以下阈值组合条件:
分类方法(1): ScoreMax≥0.60,且ScoreLarge≥0.55;
分类方法(2):
将分类方法对Set-Ⅰ和SSet-Ⅱ样本进行分类,分类结果见表 8。
由表 8可知:MCC值达到了0.7以上,特异性达到99.6%以上,敏感性也在92%以上,其真阳性个数差别在1之内,2种分类方法结果差别不大,但从综合指标MCC的结果看,方法2略好于方法1。
| 表 8 综合分类方法的自洽性检验 Table 8 Self check of the classification method |
对方法2结果中的4个假阴性样本进行分析发现,4个假阴性样本中d1v9va1和d2hgka1为核磁共振样本,d2hi7b1原子信息缺失较多的样本,d1w07a2为结构冗余较大样本,4个假阴性样本对应的
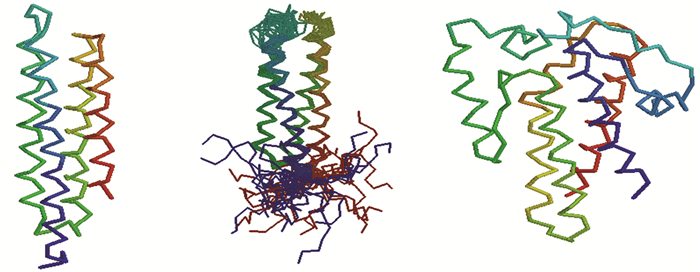
对方法2结果中的36个假阳性样本的分析发现:有3个样本—d1sj8a2、d1u89a1、d2xola_的拓扑结构与BRD-like折叠类型相同,见图 5,对应的SCOPe分类编号为a.216、a.216和a.184;其它33个假阳性样本中,8个样本为4螺旋结构但拓扑核心连接顺序不同,10个样本为5螺旋结构,15个样本为7螺旋以上结构,这些样本,当其所属折叠类型模板参与折叠类型分类时,可以通过竞争实现正确分类。
|
图 5 假阳性样本 Figure 5 Partial false positive samples |
综合分类方法用于独立性检验集Set-Ⅲ所属样本的的分类结果见表 9。
| 表 9 综合分类方法的独立性检验 Table 9 Independent test of the classification method |
对独立验集,两种分类方法的敏感性在88%以上,特异性在99.6%以上,MCC值在0.75以上,其中方法2的敏感性为94.12%,MCC值达到0.86,说明综合分类方法具有普适性,用于BRD-like折叠类型分类是可行的。对独立检验集,方法2的分类结果比方法1的分类结果好,与自洽性检验的结果吻合。综合自洽性检验及独立性检验的结果,确定方法2为基于多模板的BRD-like折叠类型的分类方法。
3 结论本文构建了BRD-like折叠类型模板数据库,利用基于单模板的方法进行分类,综合指标MCC值范围为0.34~0.59;利用基于双模板的方法进行分类,综合指标MCC值范围为0.63~0.64;利用基于多模板的综合分类方法,对实验集序列相似度小于40%的12 117个样本进行检验,检验结果的敏感性、特异性分别为0.923和0.997,MCC值为0.72。将基于多模板的综合分类方法对序列相似度小于40%的独立检验集的2 260个样本进行检验,结果为:敏感性、特异性分别为0.941和0.998、MCC值为0.86。结果表明:基于多模板的综合分类方法可用于蛋白质折叠类型分类,分类结果优于单模板分类结果。
| [1] |
VOLKAMER A, KUHN D, RIPPMANN F, et al. Predicting enzymatic function from global binding site descriptors[J]. Proteins Structure Function & Bioinformatics, 2013, 81(3): 479–489.
( 0) 0)
|
| [2] |
ISIK Z, YANIKOGLU B, SEZERMAN U.Protein structural class determination using support vector machines.[C]//Proceedings of the 19th International Symposium on Computer and Information Sciences. Kemer-Antalya, Turkey, 2004:82-89.
( 0) 0)
|
| [3] |
VALERIE D, ALAN F. The present view of the mechanism of protein folding[J]. Nature Reviews Molecular Cell Biology, 2003, 4(6): 497–502.
DOI:10.1038/nrm1126 ( 0) 0)
|
| [4] |
DAGGETT V, FERSHT A R. Is there a unifying mechanism for protein folding[J]. Trends in Biochemical Sciences, 2003, 28(1): 18–25.
DOI:10.1016/S0968-0004(02)00012-9 ( 0) 0)
|
| [5] |
ONUCHIC J N, WOLYNES P G. Theory of protein folding[J]. Current Opinion in Structural Biology, 2004, 14(1): 70–75.
DOI:10.1016/j.sbi.2004.01.009 ( 0) 0)
|
| [6] |
STEFANO G, GUYDOSH N R, FAAIZAH K, et al. Unifying features in protein-folding mechanisms[J]. Proceedings of the National Academy of Sciences, 2003, 100(23): 13286–13291.
DOI:10.1073/pnas.1835776100 ( 0) 0)
|
| [7] |
阎隆飞. 蛋白质分子结构. 北京: 清华大学出版社, 1999 .
YAN Longfei. Protein molecular structure. Beijing: Tsinghua University Press, 1999 .
( 0) 0)
|
| [8] |
LUO L F, LI X. Recognition and architecture of the framework structure of protein[J]. Proteins Structure Function & Bioinformatics, 2000, 39(1): 9–25.
( 0) 0)
|
| [9] |
CHOTHIA C. One thousand families for the molecular biologist[J]. Nature, 1992, 357: 543–544.
DOI:10.1038/357543a0 ( 0) 0)
|
| [10] |
WANG Z X. How many fold types of protein are there in nature?[J]. Proteins Structure Function & Bioinformatics, 1996, 26(2): 186–191.
( 0) 0)
|
| [11] |
BAKER D, SALI A. Protein structure prediction and structural genomics[J]. Science, 2001, 294(5540): 93–96.
DOI:10.1126/science.1065659 ( 0) 0)
|
| [12] |
BAKER D. A surprising simplicity to protein folding[J]. Nature, 2000, 405(6782): 39–42.
DOI:10.1038/35011000 ( 0) 0)
|
| [13] |
ANTONINA A, DAVE H, JOHN-MARC C, et al. Data growth and its impact on the SCOP database: new developments[J]. Cancer Research, 2006, 66(7): 3688–3698.
DOI:10.1158/0008-5472.CAN-05-4564 ( 0) 0)
|
| [14] |
FOX N K, BRENNER S E, CHANDONIA J M. SCOPe: Structural classification of proteins-extended, integrating SCOP and ASTRAL data and classification of new structures[J]. Nucleic Acids Research, 2014, 42(Database issue): D304–309.
( 0) 0)
|
| [15] |
KELLEY L A, MACCALLUM R M, STERNBERG M J. Enhanced genome annotation using structural profiles in the program 3D-PSSM[J]. Journal of Molecular Biology, 2000, 299(2): 499–520.
( 0) 0)
|
| [16] |
JINRUI X, YANG Z. How significant is a protein structure similarity with TM-score=0.5?[J]. Bioinformatics, 2010, 26(7): 889–895.
DOI:10.1093/bioinformatics/btq066 ( 0) 0)
|
| [17] |
马帅, 王勤, 李晓琴. α/β类蛋白质折叠类型的分类方法研究[J]. 生物信息学, 2014, 12(2): 123–132.
MA Shuai, WANG Qin, LI Xiaoqin. Research on the classification method of α/βprotein fold type[J]. Chinese Journal of Bioinformatics, 2014, 12(2): 123–132.
( 0) 0)
|
| [18] |
孔令强, 李晓琴. 基于特征片段信息的PH domain-like barrel蛋白质折叠类型分类分析[J]. 生物信息学, 2012, 10(2): 125–129.
KONG Lingqiang, LI Xiaoqin. A method of PH domin-like barrel protein fold classification based on characteristics fragments[J]. Chinese Journal of Bioinformatics, 2012, 10(2): 125–129.
( 0) 0)
|
| [19] |
SHINDYALOV I N, BOURNE P E. Protein structure alignment by incremental combinatorial extension (CE) of the optimal path[J]. Protein Engineering, 1998, 11(9): 739–747.
DOI:10.1093/protein/11.9.739 ( 0) 0)
|
| [20] |
HOLM L, PARK J. DaliLite workbench for protein structure comparison[J]. Bioinformatics, 2000, 16(6): 566–567.
DOI:10.1093/bioinformatics/16.6.566 ( 0) 0)
|
| [21] |
KRISSINEL E H K. Secondary-structure matching (SSM), a new tool for fast protein structure alignment in three dimensions. Acta Crystallogr[J]. Acta Crystallographica Section D: Biological, 2004, 60(12-1): 2256–2268.
( 0) 0)
|
| [22] |
ZHANG Yang, SKOLNICK J. TM-align: a protein structure alignment algorithm based on the TM-score[J]. Nucleic Acids Research, 2005, 33(7): 2302–2309.
DOI:10.1093/nar/gki524 ( 0) 0)
|
| [23] |
KONAGURTHU A S, WHISSTOCK J C, STUCKEY P J, et al. MUSTANG: A multiple structural alignment algorithm[J]. Proteins Structure Function & Bioinformatics, 2006, 64(3): 559–74.
( 0) 0)
|
| [24] |
KIFER I, NUSSINOV R, WOLFSON H J. GOSSIP: A method for fast and accurate global alignment of protein structure[J]. Bioinformatics, 2011, 27(7): 925–32.
DOI:10.1093/bioinformatics/btr044 ( 0) 0)
|
| [25] |
VIDLER L R, PANAGIS F, OLEG F, et al. Discovery of novel small-molecule inhibitors of BRD4 using structure-based virtual screening[J]. Journal of Medicinal Chemistry, 2013, 56(20): 8073–88.
DOI:10.1021/jm4011302 ( 0) 0)
|
| [26] |
FILIPPAKOPOULOS P, KNAPP S. The bromodomain interaction module[J]. Febs Letters, 2012, 586(17): 2692–2704.
DOI:10.1016/j.febslet.2012.04.045 ( 0) 0)
|
| [27] |
DHALLUIN C, CARLSON J E, ZENG L, et al. Structure and ligand of a histone acetyltransferase bromodomain[J]. Nature, 1999, 399(6735): 491–496.
DOI:10.1038/20974 ( 0) 0)
|
| [28] |
CONWAY S J. Bromodomains: are readers right for epigenetic therapy?[J]. Acs Medicinal Chemistry Letters, 2012, 3(9): 691–4.
DOI:10.1021/ml300221t ( 0) 0)
|
| [29] |
VOLLMUTH F, BLANKENFELDT W, GEYER M. Structures of the dual bromodomains of the P-TEFb-activating protein Brd4 at atomic resolution[J]. Journal of Biological Chemistry, 2009, 284(52): 36547–36556.
DOI:10.1074/jbc.M109.033712 ( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14


