可变剪接作为基因表达调控的重要机制之一,使得同一基因可以通过不同剪接方式产生不同的RNA异构体,进而扩大了蛋白质的多样性。研究表明基因的异常剪接与癌症的发生发展及预后密切相关。近来,lncRNA被发现在基因剪接过程中扮演着重要角色,可通过以下三种方式调控可变剪接[1]:①lncRNA与剪接因子直接或间接相互作用; ②lncRNA与pre-mRNA分子形成RNA-RNA双链; ③lncRNA影响染色质重塑。尽管目前已有部分lncRNA被揭示参与剪接调控过程,但由于lncRNA数量众多且相关分子机制复杂,lncRNA在剪接调控中的功能与作用机制仍有待进一步探索。特别是,与癌症异常剪接调控相关lncRNA的识别与分子机制研究,将为癌症诊断与治疗策略提供新的见解。
1 lncRNA概述lncRNA是长度超过200个核苷酸的非编码RNA,序列保守性较差[2]。根据其与mRNA在基因组上的相对位置关系,lncRNA主要可分为以下几种类型:正义型、反义型、内含子型、基因间型和增强子型[3]。随着高通量测序技术及计算平台的发展,在过去的十多年间人们发现并注释了大量的lncRNA。lncRNA从一开始的“转录噪声”转变成为具有重要生物学功能的调控分子,包括充当分子支架、与pre-mRNA或miRNA形成RNA-RNA双链体作为转录后调节因子, 转录后募集或抑制调节蛋白以及编码小肽或产生发挥生物学效应的小RNA分子[2]。
lncRNA可通过多种机制参与生物过程[4],其调控功能通常具有以下特征:①lncRNA的功能与其在细胞中的位置有关。细胞核内的lncRNA参与调节诸如转录、剪接和染色质重塑等生物过程,而细胞质中的lncRNA则负责调节mRNA运输、稳定性, 翻译以及翻译后修饰等生物过程; ②lncRNA的功能取决于其相对丰度; ③lncRNA的功能与和其相互作用的RNA或RNA结合蛋白密切相关。随着研究的不断深入,越来越多的证据表明lncRNA在癌症等人类复杂疾病的发生发展中起着关键作用,包括肝癌、胃癌、卵巢癌, 胰腺癌和结直肠癌等[5-9]。尽管已有许多lncRNA的功能被发现,但鉴于其数量众多且调控机制复杂,仍有大量lncRNA的功能及作用机制未知。
2 lncRNA调控可变剪接的分子机制可变剪接是指同一RNA前体生成不同RNA剪接异构体的过程,可变剪接类型主要包括外显子跳跃, 可变5'或3'剪接位点、互斥外显子和内含子保留[10]。据估计,92%~94%的人类基因都会经历可变剪接[11],表明可变剪接在生命活动中扮演着极其重要的角色。影响剪接调控的因素有很多,近年来随着对lncRNA研究的不断深入,研究人员发现lncRNA是剪接过程的重要参与者和调节者。
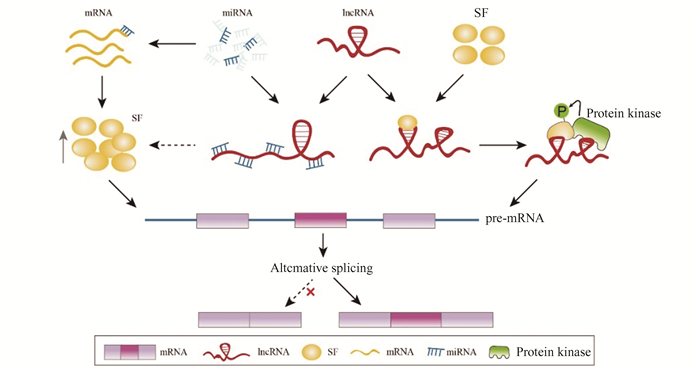
2.1 lncRNA与剪接因子直接或间接相互作用催化剪接过程的剪接体复合物包括100多种反式作用因子,其中hnRNP和SR蛋白属于公认的剪接因子(Splicing factor, SF)家族[12]。剪接因子对于剪接位点的识别以及剪接体的组装至关重要,而lncRNA可通过与剪接因子相互作用影响可变剪接(图 1)。一方面,lncRNA与剪接因子直接相互作用,例如MALAT1通过与SR剪接因子结合调节其表达水平和磷酸化,使SR剪接因子能够在正确的时间、地点和浓度下完成pre-mRNA的可变剪接[13]; 另一方面,lncRNA在转录或转录后水平上间接与剪接因子相互作用,例如ZNF561-AS1通过与miR-26a-3p和miR-128-5p相互作用来调节SRSF6的表达水平,而SRSF6作为SR剪接因子家族成员,其对pre-mRNA的剪接至关重要[14]。此外,剪接因子调节靶基因剪接通常由转录因子(Transcription factor, TF)触发,例如c-Myc通过上调PTB, hnRNPA1和hnRNPA2的转录进而调节丙酮酸激酶PKM2亚型的剪接[15]。lncRNA作为转录调控分子,可与转录因子相互作用,进而影响下游剪接因子对基因的可变剪接。
|
图 1 LncRNA与剪接因子互作 Figure 1 Interaction between lncRNA and splicing factor 注:一方面,lncRNA作为脚手架,通过介导蛋白激酶与剪接因子间的相互作用影响后者的磷酸化修饰水平,进而调节下游基因的可变剪接;另一方面,lncRNA可海绵吸附miRNA,进而降低miRNA与其靶基因间的结合,提高剪接因子的表达水平,从而影响下游基因的剪接过程. |
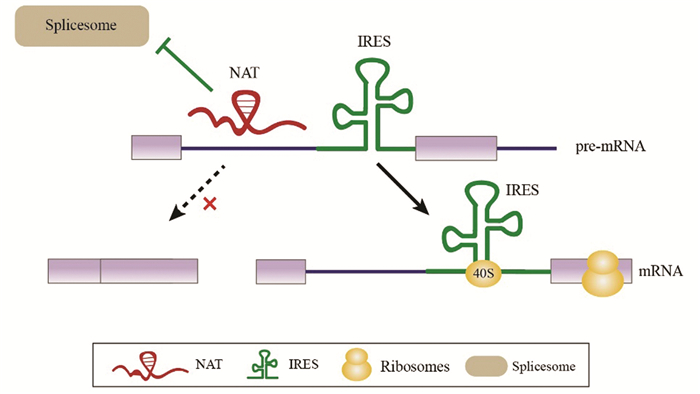
lncRNA可与pre-mRNA互补配对形成RNA-RNA双链体,进而掩盖pre-mRNA内部与剪接因子结合所必需的顺式作用元件,改变其剪接形式(图 2)。2008年Beltran M等[16]发现ZEB2的天然反义转录物(Natural antisense transcript, NAT)与ZEB2 5’-UTR的内含子区域互补形成RNA-RNA双链,从而防止了ZEB2 5’-UTR的剪接,进而增加了ZEB2蛋白的表达水平。此后,人们陆续发现许多反义lncRNA与pre-mRNA形成RNA-RNA双链,进而影响剪接包含或去除相关外显子,从而产生相应的致癌转录本。lncRNA Saf通过与Fas pre-mRNA碱基互补配对,形成特定的双链RNA中间体。进一步的研究发现,二者之间最强的相互作用发生在外显子5-6和外显子6-7连接处,从而导致剪接因子SPF45选择性剪接Fas外显子6[17]。LncRNA PLANE通过与NCOR2 pre-mRNA形成RNA-RNA双链,招募hnRNPM,从而抑制剪接产物NCOR2-202的生成,降低其蛋白质水平,促进癌细胞的增殖[18]。
|
图 2 pre-mRNA与其天然反义转录产物NAT形成RNA-RNA双链 Figure 2 pre-mRNA and its natural antisense transcript (NAT) form RNA-RNA double strand 注:RNA-RNA双链体的形成使得pre-mRNA的内含子在剪接过程中得以保留,此区域内的IRES元件可直接招募核糖体起始翻译过程. |
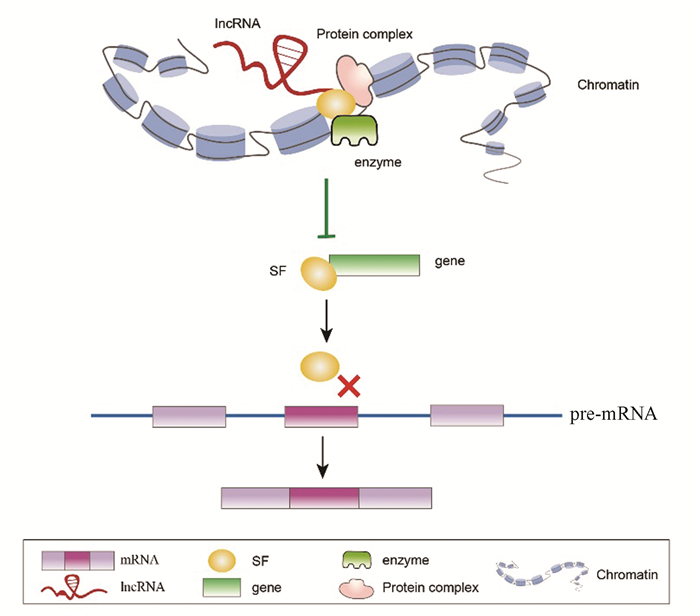
lncRNA可以与染色质相互作用,然后招募蛋白质复合物来重塑染色质状态,从而调节基因表达[19](图 3)。SWI/SNF复合物是重要的染色质重塑因子,其利用ATP水解的能量来改变染色质结构[20]。MALAT1将染色质重塑复合物SWI/SNF的催化亚基BRG1招募到促炎介质IL-6和CXCL8的启动子区域,从而促进NF-κB诱导这些炎症因子的表达[21]。Pandey RR等[22]发现,Kcnq1ot1与PRC2复合物相互作用,并将H3K9和H3K27甲基转移酶募集到作用部位,从而调节染色质水平。其次,研究表明,染色质重塑在可变剪接的调控中具有重要功能[23]。Sims RJ 3rd等[24]发现ATP依赖性染色质重塑蛋白和组蛋白伴侣CHD1通过与U2 snRNP的SF3a亚复合物的特异性相互作用将核心剪接体组分连接到H3K4me3。当CHD1和H3K4me3减少时,SF3a的募集和pre-mRNA剪接的效率均受到干扰[24]。
|
图 3 LncRNA介导的染色质重塑 Figure 3 LncRNA mediated chromatin remodeling 注:LncRNA将相关蛋白质复合物和酶募集到染色质,使其结构发生改变,进而抑制剪接因子与基因外显子的结合,使得该外显子在剪接过程中得以保留. |
卵巢癌是一种异质性肿瘤,其死亡率超过任何其他类型的女性生殖系统恶性肿瘤[25]。卵巢癌发生发展的分子机制甚为复杂,至今仍不明确。Gordon MA等[7]发现lncRNA MALAT1敲低会出现细胞增殖, 侵袭和失巢细胞增多的情况,并且抑制MALAT1还会导致剪接因子RBFOX2的表达减少,进而调控可变剪接朝着KIF1B促凋亡亚型KIF1B-β表达增多的方向发展。Ma Y等[26]发现PAXIP1-AS1通过miR-6744-5p上调剪接因子PCBP2的表达,从而加剧卵巢癌细胞的生长和迁移。Wu J等[27]的研究表明lncRNA CRNDE通过SRSF1/TIA1信号通路诱导卵巢癌顺铂耐药,但其具体调控机制仍需进一步研究。
3.2 结直肠癌结直肠癌是全球第三大常见癌症,也是第四大常见的癌症死亡原因[28]。为确定新的治疗靶点,探索其发生发展的分子机制是十分迫切的。Lan Z等[29]发现lncRNA SNHG6可以诱导hnRNPA1特异性剪接mRNA PKM,从而增加PKM2/PKM1的比例,这可能是增强结肠癌细胞中有氧糖酵解进而致癌的重要机制。Hu ZY等[30]研究表明,lncRNA MALAT1介导的SRPK1过表达会导致细胞核中SRSF1磷酸化水平升高,而核磷酸化-SRSF1水平的恢复可促进AKAP-9 pre-mRNA的可变剪接,提高AKAP-9蛋白的表达,进而增强结直肠癌细胞的增殖、迁移和侵袭能力。Kong J等[31]则发现lncRNA LINC01133可与剪接因子相互作用促进结直肠癌中EMT和转移的调节。
3.3 胃癌胃癌是全球第五大常见癌症,由于多数胃癌患者在确诊时已处于晚期,因此死亡率极高,位于癌症相关死亡的第三位[32]。Zhang F等[33]研究发现,lncRNA CRNDE可通过与剪接因子SRSF6相互作用降低其蛋白质稳定性,而SRSF6可调节PICALM的可变剪接使之产生外显子14跳跃的剪接异构体,进而触发亚型转换,促进PICALM长亚型的表达,从而减弱胃癌的化学耐药性。He Q等[34]发现LINC00924调控剪接因子hnRNPC在e14a处与Mnk2 pre-mRNA结合,使得Mnk2a亚型表达减少,进而调控p38 MAPK/PPARα信号通路,促进胃癌腹膜转移。Zhu W等[35]发现lncRNA SNHG8通过与hnRNPA1结合促进TROY的表达,而TROY已被证实会促进胃癌细胞的化学抗性。
3.4 肝癌肝细胞癌是最常见的原发性肝癌,被列为第六大常见肿瘤,并且是肝硬化患者死亡的主要原因[36]。Gonzalez I等[37]阐明了lncRNA AS-FGFR2通过诱导多梳抑制复合体2(PRC2)的SUZ12和EZH2与FGFR2基因座结合,进而募集H3K36去甲基化酶KDM2a,促进外显子IIIb包含。Amann T等[38]研究表明含有IIIb的mRNA FGFR2-IIIb会抑制肝细胞癌细胞的致瘤性。Lin YH等[39]首次证明lncRNA LINC01348在肝细胞癌中显著下调并与患者生存密切相关,其通过抑制剪接因子SF3B3介导的EZH2可变剪接来调节肝癌细胞的转移,表明其作为生物标志物的效用。另外,Malakar P等[40]报道了lncRNA MALAT1在肝细胞癌中上调进而诱导剪接因子SRSF1调节其剪接靶点导致抗凋亡亚型的产生。另一方面,lncRNA MALAT1通过调节S6K1的可变剪接激活mTOR途径,这可能在肝细胞癌发生中发挥着关键作用。
3.5 胰腺癌胰腺癌是全球癌症死亡的主要原因,随着全球人口老龄化,胰腺癌的发病率日益升高[41]。Li J等[8]研究发现lncRNA DKFZp434J0226的表达增加与胰腺导管腺癌的预后不良呈正相关,进一步的内源性RNA pull-down实验与质谱分析揭示了DKFZp434J0226与剪接因子SF3B6相互作用从而促进其磷酸化,进而影响MDM4基因的可变剪接,使得致癌转录本MDM4-S的表达升高。类似的,Meng LD等[42]研究表明LINC01232通过上调剪接因子HNRNPA2B1参与A-Raf的可变剪接,使得A-Raf全长亚型的mRNA水平增加和A-Raf短亚型的mRNA水平下调,并通过激活MAPK/ERK信号通路促进胰腺癌细胞转移。Zhang Y等[43]研究证实LINC00857通过剪接因子SRSF1调节CLDN12的可变剪接,增加其蛋白产物表达水平,进而促进胰腺癌细胞的上皮-间质转化。
3.6 其他癌症除以上癌症外,lncRNA在其他癌症的异常剪接调控中也扮演着重要角色。Xu S等[44]发现lncRNA EGOT通过RNA-蛋白质互作招募剪接因子hnRNPH1,促进ITPR1的选择性剪接并提升其表达水平,进而增加乳腺癌细胞自噬水平,提高了肿瘤对紫杉醇化疗的敏感性。Singh R等[45]在乳腺癌中发现BC200可招募剪接因子hnRNP A2/B1改变Bcl-x基因的剪接模式,使其蛋白产物由促凋亡亚型Bcl-xS转为抗凋亡亚型Bcl-xL,进而起到致癌作用。Duan Y等[46]发现lncRNA DGCR5在食管鳞状细胞癌中与SRSF1相结合继而调节mRNA Mcl-1的可变剪接,促使其抗凋亡亚型Mcl-1L的表达增加。Zhang Z等[47]对前列腺特异性lncRNA PCGEM1进行了研究,发现其可与包括hnRNP A1和U2AF65在内的剪接因子相互作用,调节AR基因的可变剪接模式从而产生不同的功能后果。
3.7 小结可变剪接异常往往在肿瘤发生发展中起着主导作用[48],这些异常剪接事件的发生会导致一些关键基因,诸如TP53[49], RB1 [50]和PTEN [51]等的失调,使其功能丧失。基因剪接异常会增加肿瘤细胞的异质性[52],对肿瘤细胞和微环境的代谢重编程、增殖、转移及耐药性均有显著影响,并以此驱动肿瘤的发生发展[[53]-[54]]。目前,剪接调控相关研究主要集中在剪接因子[55-57],lncRNA可与剪接因子的相互作用,进而已被认为是调控剪接事件的重要参与者[58-59]。特别是,lncRNA可作为miRNA的分子海绵,而后者可靶向调控剪接因子表达[60-62]。此外,lncRNA还可通过与pre-mRNA分子形成RNA-RNA双链或影响染色质重塑两种机制,调控可变剪接事件。鉴于其调控过程复杂且研究手段有限,目前仍需更多的研究来深入探索其分子机制。lncRNA在肿瘤异常剪接中的功能作用研究,或可为肿瘤的诊断、治疗及预防提供新的思路和研究方向,有助于深入理解肿瘤发生发展的分子机制。
4 生物信息学工具及数据库 4.1 生物信息学工具IncRNA调控可变剪接研究相关的生物信息学工具见表 1。
| 表 1 lncRNA调控可变剪接研究相关生信工具 Table 1 Bioinformatics tools for the study of alternative splicing regulated by lncRNA |
rMATS是目前最常用的针对RNA-seq数据的差异可变剪接分析工具,由Shen S等[63]于2014年开发。与先前其它工具[64-65]相比,rMATS是首个可处理生物学重复样本RNA-seq数据的工具。rMATS采用层次框架来同时考虑单个重复样本的采样不确定性和重复样本间的变异性对计算结果的影响。rMATS不仅可以处理非配对生物学重复样本RNA-seq数据,还可计算配对生物学重复样本RNA-seq数据的差异剪接事件。
SpliceSeq是Ryan MC等[66]开发的用于探索TCGA数据库中肿瘤组织可变剪接模式的标准工具,分析结果收录于TCGA SpliceSeq[67]数据库中。TCGA SpliceSeq包含来自33种不同类型的肿瘤样本(包括可用的相邻正常样本)上的剪接事件的剪接百分比(Percent spliced in, PSI),研究人员可以通过输入基因名来寻找所选肿瘤类型之中表现出最高剪接比的基因,或者探索肿瘤和相邻正常样本之间的剪接模式变化。TCGA SpliceSeq还提供剪接模式, reads计数及各种统计结果的直观图形。
LeafCutter是Li YI等[68]在2018年开发的从短读长RNA-seq数据中识别可变剪接事件的软件,其无需转录本注释信息。LeafCutter采用基于计数的统计模型,使用狄利克雷多项式-广义线性模型来检验整个样本集中的内含子使用变化,而不是分别在不同条件或基因型下检验内含子的差异切除。与其他方法不同,LeafCutter通过测序数据中拼接的读段来识别和量化可变剪接事件,可发现高复杂性的事件。此外,LeafCutter还可用于绘制剪接数量性状基因座(sQTL)。
SUPPA2是由Trincado JL等[69]开发并与LeafCutter同年发表的可变剪接分析工具,其主要基于转录本定量来识别可变剪接事件,具有计算快和存储要求低的优点。SUPPA2可根据转录本注释生成剪接事件,定量剪接事件或转录本的剪接水平(PSI),识别不同条件下的差异剪接事件,以及根据PSI值对剪接事件进行聚类。SUPPA2同样可以处理具有生物学重复的RNA-seq数据。
FLAIR是Tang AD等[70]开发的一套适用于纳米孔cDNA, native RNA及PacBio测序数据,分析差异可变剪接事件或剪接异构体的工具。FLAIR可选择与短读长测序数据一起运行,以提高长读长测序reads的剪接位置识别精度。FLAIR流程主要包括比对、校正、折叠、量化, 差异表达及差异剪接分析,使用多个比对步骤和剪接位点过滤方法提高结果置信度。
4.1.2 lncRNA-RNA互作分析TargetScan是Lewis BP等[71]于2003年开发的软件,用于识别miRNA的靶标,最后一次更新时间为2018年。TargetScan通过搜索RNA序列中与miRNA种子区域配对的保守8mer,7mer和6mer位点来预测miRNA-RNA相互作用,采用位点的累积加权分值对靶点可能性进行评估。对于lncRNA和miRNA的相互作用预测,用户仅需提供lncRNA的原始序列并下载miRNA种子序列文件,然后设置相应参数即可进行预测。
RNAhybrid是Krüger J等[72]在2006年发表的可简单、快速和灵活地预测miRNA靶标的工具。RNAhybrid提供在线版本,用户只需上传miRNA和候选靶序列即可进行预测,并且有多个参数可供选择。RNAhybrid的算法核心是RNA二级结构预测的变体。与其他二级结构预测软件相比,RNAhybrid的算法专门为RNA杂交而设计,计算速度显著提升。
LncTar是Li J等[73]在2015年开发的一种通过自由能最小化来预测lncRNA-RNA相互作用的软件。与其他工具相比,LncTar具有以下优势:首先,LncTar对RNA大小没有限制,可以处理当前RNA分子的所有长度。值得注意的是,LncTar提供了一种基于自由能的定量标准,可以自动确定两个RNA分子是否相互作用。其次,LncTar运行速度快,因此可用于lncRNA的RNA靶标的大规模鉴定。最后,LncTar具有很高的预测精度。
RIblast是Fukunaga T等[74]开发的一种基于种子区域扩展方法的RNA-RNA相互作用预测算法。其主要使用后缀阵列发现种子区域,随后根据RNA二级结构能量模型扩展种子区域。RIblast由数据库构建和RNA相互作用搜索两个步骤组成。虽然RIblast预测精度与现有方法相似,但其速度比现有方法快64倍以上,适用于大型lncRNA数据集,全面分析lncRNA-RNA相互作用。
RIsearch2是Alkan F等[75]在2017年开发的大规模RNA-RNA相互作用预测工具,能够快速定位查询序列和目标序列之间潜在相互作用位点。RIsearch2使用基于后缀数组的种子扩展框架,采用修正后的Smith-Waterman-Gotoh算法来识别RNA-RNA相互作用。另外,RIsearch2中还提出了一种siRNA脱靶识别方法,可预测脱靶转录本,同时还可计算给定siRNA的脱靶潜力。
miRWalk是Sticht C等[76]在2018年开发的综合性的miRNA靶基因数据库,收录有人类、小鼠、大鼠, 狗和牛等多个物种的miRNA靶基因信息。miRWalk收录有TargetScan, RNAhybrid, TarPmiR等多个miRNA靶标预测工具的结果,收录有包括5’-UTR, CDS和3’-UTR在内的完整转录序列上的miRNA靶位点信息。miRWalk可以从miRNA出发,检索其靶基因,也可以从基因出发,检索可靶向结合的miRNA。除可以表格形式导出检索结果,miRWalk还提供miRNA和靶基因之间调控网络可视化及基因集富集分析功能。
4.1.3 lncRNA-蛋白互作分析CatRAPID是Agostini F等[77]在2013年开发的用于大规模计算蛋白质-RNA相互作用的工具,并由Armaos A等[78]于2021年更新至第二版。CatRAPID可在八种模式生物的全转录组水平或全蛋白质组水平计算蛋白质-RNA相互作用倾向。用户可以根据已知蛋白质序列预测与其结合的RNA序列,根据已知RNA序列预测与其结合的蛋白质,或预测一对特定RNA与蛋白质之间的作用倾向。预测结果会以图及表格的形式展示,其中表格部分包含蛋白质与RNA间的交互倾向、RNA及蛋白质结合区域等共计十项内容。
LncADeep是Yang C等[79]开发的基于深度学习的lncRNA识别、lncRNA-蛋白质互作预测以及功能注释的生物信息学工具。作者基于lncRNA-蛋白质相互作用对的序列和结构特征,使用DNN构建深度学习模型,用于预测lncRNA的蛋白质靶标。此外,LncADeep还可根据KEGG, Reactome数据库中功能注释,从预测的相互作用蛋白推断lncRNA的生物学功能。
Capsule-LPI是Li Y等[80]开发的一种基于胶囊网络的lncRNA-蛋白质相互作用预测工具。Capsule-LPI集成了四组多模态特征,包括序列特征、基序信息、理化性质和二级结构特征。其多通道框架不仅可以考虑每个特征的预测趋势,还可以考虑其他交互信息和不同特征之间的关系,从而更好地集成和学习多个特征。与现有的lncRNA-蛋白质相互作用预测工具相比,Capsule-LPI集成了更多的特征信息,如物理化学特征和基序信息,并且预测精度也有所提升。
4.1.4 lncRNA-染色质互作分析ChIRP-seq是Chu C等[81]开发的可绘制全基因组范围内RNA结合位点的高通量测序技术。生物素或链霉亲和素探针的互补寡核苷酸与目标RNA杂交后,使用磁性链霉亲和素珠分离并纯化与其作用的染色质片断。洗脱后进行高通量测序,鉴定和定量分析结合区域。亲和探针的设计仅基于RNA序列,不需要事先了解RNA的结构或功能域,因此方便快捷。ChIRP-seq可为lncRNA-染色质互作的功能机制研究提供新的技术支撑。
CHART-seq是Simon MD等[82]开发的基于杂交的高通量测定内源性非编码RNA结合靶标的新技术。该方法使用互补寡核苷酸从可逆交联提取物中纯化RNA及其靶标,可用于绘制内源性RNA的全基因组结合位点。作者使用CHART-seq分析了Jpx结合的染色质位点,发现其倾向于结合启动子、转录起始临近区域或转录终止区等,尤其与转录活跃基因显示出高相关性。鉴于lncRNA在染色质结构和基因表达调控中的重要作用,CHART提供了一种有价值的工具来鉴定其在染色质上的结合位点。
4.2 数据库IncRNA与可变剪接相关数据库见表 2。
| 表 2 lncRNA与可变剪接相关数据库 Table 2 Long non coding RNA and alternative splicing related database |
SpliceAid-F是Giulietti M等[83]在2013年开发的剪接因子数据库,并于2014年进行了更新。研究人员通过人工筛选文献和数据库,收集整理了71种可与RNA结合的人类剪接调控蛋白相关实验数据。数据库还收录了实验验证的RNA-SF相互作用及其相关信息,包括结合位点序列、位点所在的基因、结合位点坐标、参考文献等。此外,数据库中还记录了剪接因子的功能结构域及其相互作用蛋白。该数据库中收集的RNA-剪接因子互作可为lncRNA调控可变剪接机制研究提供借鉴。
NPInter是Wu T等[84]创建的非编码RNA和生物大分子相互作用数据库。2022年Zheng Y等[85]将其更新至第五版,收录了200多万条非编码RNA功能相互作用信息。其中,新增添的RNA结合蛋白数据包含277个RNA结合蛋白,并提供细胞定位, RNA结合区域及生物学功能等信息。依据功能信息,用户可方便地检索到剪接调控相关RNA结合蛋白,并下载与其互作的lncRNA相关信息, 也可从数据库中直接下载lncRNA-蛋白质互作数据。
starBase v2.0是Li JH等[86]基于CLIP-seq等高通量数据构建的RNA互作数据库,包含410万个miRNA-ncRNA、290万个miRNA-mRNA,410万个RBP-RNA和150万个RNA-RNA相互作用,并且提供数据集可视化,分析和下载功能,极大地扩展了对非编码RNA功能及其调控网络的理解。其中,从lncRNA-RBP数据集中可以筛选得到lncRNA-剪接因子相互作用对。
4.2.2 lncRNA-TF-SF互作数据库ChIPBase是Yang JH等[87]开发的基于ChIP-Seq数据的转录因子数据库,用于全面注释和发现lncRNA和miRNA与转录因子的互作调控关系。目前,Huang J等[88]已将数据库更新到第三版,利用收集的来自CistromeDB, ReMap2022, ENCODE的ChIP-seq数据,来自ENCODE, TCGA, GTEx, CCLE, ICGC的RNA-seq数据以及其他来源的SNV,SNP和基因注释数据,绘制了mRNA和ncRNA的转录调控关系图谱。作者整合了在不同样本中基因-转录因子相互作用,构建了以基因为中心的调控网络。用户仅需输入官方基因名称,即可获得与查询基因结合的转录调节因子。
LncMAP数据库是Li Y等[89]构建的包含20种癌症中lncRNA介导的转录扰动资源数据库,主要包括lncRNA-TF-Gene、药物-lncRNA、生存分析等功能模块。用户可以根据癌症、lncRNA,转录因子或基因检索数据库,获取目标癌症的lncRNA-TF-Gene互作关系,也可下载相关数据自行筛选。该数据库提供了比ChIPBase增加了lncRNA-转录因子作用对的靶基因信息,可提供更详细的lncRNA-TF-gene互作信息。
Harmonizome是Rouillard AD等[90]于2016年创建的数据库,旨在整合基因、蛋白质及其关联信息,以实现更好的知识提取和数据重用。该数据库收集了来自72个公开资源的125个独特数据集,涵盖了关于哺乳动物基因和蛋白质的六大类信息:疾病和表型关联、基因组图谱、物理相互作用、蛋白质组图谱, 结构或功能注释和转录组图谱。在搜索界面输入“Transcription factor targets”即可获得转录因子靶标数据集,通过对靶基因的筛选可获得转录因子与剪接因子的互作关系。
Cistrome Cancer是Mei S等[91]为了深入了解调节癌症异常基因表达模式的转录和表观遗传因素开发的数据库。癌症相关数据资源来自TCGA数据库,包括转录组、DNA甲基化、拷贝数变异和临床生存信息。另外,作者也收集整理了GEO, ENCODE和Roadmap中表观基因组学ChIP-seq和染色质可及性数据。用户可在“Transcription Factor Targets in Cancer”功能页面左侧列表中,输入或选择目标转录因子获取转录因子靶标信息。
GTRD是Yevshin I等[92]于2017年构建的数据库,2021年Kolmykov S等[93]对数据库进行了更新。更新后的版本是目前真核生物转录调控数据的最大集成资源,包含与基因转录相关的测序数据:ChIP-seq,ChIP-exo,DNase-seq,MNase-seq,ATAC-seq和RNA-seq。用户通过高级浏览与搜索功能,可快速准确获取转录因子靶标信息,包括基因ID、基因名、转录本ID等。
4.2.3 miRNA-target相互作用miRTarBase数据库由Hsu SD等[94]于2011年创建,主要提供miRNA-靶标相互作用,历经五次修订,最新版本由Huang HY等[95]于2022年更新丰富,集成了数十种数据集,添加了miRNA表达谱及其生物学数据。目前,miRTarBase共收录了5 173 924个miRNA-靶标互作数据对,包含5 818个miRNA和33 071个靶标基因。用户可通过搜索功能,直接检索与指定基因(lncRNA,转录因子或剪接因子)结合的miRNA,且其互作信息提供证据来源,可筛选实验验证的互作关系对。
DIANA-LncBase是Paraskevopoulou MD等[96]于2013年构建的用于探索miRNA与lncRNA之间相互作用的在线数据库,在近期进行了更新[97]。该数据库通过整合多个公共数据源中的RNA-RNA相互作用信息,包含约24万个具有实验数据支撑的人类和小鼠组织,细胞类型特异性相互作用miRNA-lncRNA对。DIANA-LncBase允许用户采用不同的筛选标准组合进行搜索与浏览,并提供miRNA-lncRNA相互作用的详细信息,例如lncRNA表达谱、亚细胞定位、转录本基因组位置的图形绘制以及外部数据库信息链接等。
4.2.4 lncRNA-chromatin相互作用LnChrom是Yu F等[98]从文献中人工收集整理实验验证的lncRNA-染色质相互作用信息构建的数据库,总共包含了263种人类和小鼠细胞类型或组织中的382 743个lncRNA-染色质相互作用,涉及34 345个靶基因和2 390个lncRNA。该数据库除提供lncRNA-染色质相互作用图谱外,还提供“Detailed Annotation”, “Genome Brower”, “TF Co-occupancy”及“Cancer Exploration”等功能模块,用于提供详细数据和分析结果。其中,“Cancer Exploration”功能模块提供了来自TCGA的13种癌症的4 351个肿瘤样本和556个正常样本中lncRNA及其靶基因的表达,有助于阐述lncRNA-染色质相互作用的功能意义。
5 总结剪接失调被认为是驱动癌症发生发展的关键因素,而近来越来越多的证据表明lncRNA在可变剪接过程中发挥着重要的调控作用。研究发现,lncRNA可通过与剪接因子互作,与pre-mRNA形成双链结构以及染色体重塑等方式调节可变剪接,其表达失调与癌症中基因异常剪接事件的发生密切相关。目前,癌症相关研究大多聚焦于lncRNA与剪接因子间的相互作用,对于其他类型的剪接调控机制报道有限。此外,鉴于lncRNA调控机制的复杂性和多样性,其剪接调控模式也有待进一步的挖掘。尽管已有诸多生信工具和数据库可用于剪接调控相关lncRNA的识别及作用机制分析,但受限于当前高通量测序技术的固有缺陷及数据质量,预测结果的准确性及可靠性仍有待进一步提升。
| [1] |
LEWIS B P, SHIH I H, JONES-RHOADES M W, et al. Prediction of mammalian microRNA targets[J]. Cell, 2003, 115(7): 787-798. DOI:10.1016/s0092-8674(03)01018-3 (  0) 0) |
| [2] |
LIU Yunze, LIU Xin, LIN Changwei, et al. Noncoding RNAs regulate alternative splicing in Cancer[J]. Journal of Experimental & Clinical Cancer Research, 2021, 40(1): 11. DOI:10.1186/s13046-020-01798-2 (  0) 0) |
| [3] |
CAO Pengbo, JIN Qian, FENG Lan, et al. Emerging roles and potential clinical applications of noncoding RNAs in hepatocellular carcinoma[J]. Seminars in Cancer Biology, 2021, 75: 136-152. DOI:10.1016/j.semcancer.2020.09.003 (  0) 0) |
| [4] |
HERMAN A B, TSITSIPATIS D, GOROSPE M. Integrated lncRNA function upon genomic and epigenomic regulation[J]. Molecular Cell, 2022, 82(12): 2252-2266. DOI:10.1016/j.molcel.2022.05.027 (  0) 0) |
| [5] |
YUAN Kefei, LAN Jiang, XU Lin, et al. Long noncoding RNA TLNC1 promotes the growth and metastasis of liver cancer via inhibition of p53 signaling[J]. Molecular Cancer, 2022, 21(1): 105. DOI:10.1186/s12943-022-01578-w (  0) 0) |
| [6] |
ZHU Yiran, ZHOU Bingluo, HU Xinyang, et al. LncRNA LINC00942 promotes chemoresistance in gastric cancer by suppressing MSI2 degradation to enhance c-Myc mRNA stability[J]. Clinical and Translational Medicine, 2022, 12(1): e703. DOI:10.1002/ctm2.703 (  0) 0) |
| [7] |
GORDON M A, BABBS B, COCHRANE D R, et al. The long non-coding RNA MALAT1 promotes ovarian cancer progression by regulating RBFOX2-mediated alternative splicing[J]. Molecular Carcinogenesis, 2019, 58(2): 196-205. DOI:10.1002/mc.22919 (  0) 0) |
| [8] |
LI Jinglei, TONG Hanxing, LI Dongping, et al. The long non-coding RNA DKFZp434J0226 regulates the alternative splicing process through phosphorylation of SF3B6 in PDAC[J]. Molecular Medicine, 2021, 27(1): 95. DOI:10.1186/s10020-021-00347-7 (  0) 0) |
| [9] |
LI Bosheng, KANG Houyi, XIAO Yufeng, et al. LncRNA GAL promotes colorectal cancer liver metastasis through stabilizing GLUT1[J]. Oncogene, 2022, 41(13): 1882-1894. DOI:10.1038/s41388-022-02230-z (  0) 0) |
| [10] |
BARALLE F E, GIUDICE J. Alternative splicing as a regulator of development and tissue identity[J]. Nature Reviews Molecular Cell Biology, 2017, 18(7): 437-451. DOI:10.1038/nrm.2017.27 (  0) 0) |
| [11] |
WANG E T, SANDBERG R, LUO Shujun, et al. Alternative isoform regulation in human tissue transcriptomes[J]. Nature, 2008, 456(7221): 470-476. DOI:10.1038/nature07509 (  0) 0) |
| [12] |
DU Junxian, ZHU Guiqin, CAI Jialiang, et al. Splicing factors: Insights into their regulatory network in alternative splicing in cancer[J]. Cancer Letters, 2021, 501: 83-104. DOI:10.1016/j.canlet.2020.11.043 (  0) 0) |
| [13] |
TRIPATHI V, ELLIS J D, SHEN Zhen, et al. The nuclear-retained noncoding RNA MALAT1 regulates alternative splicing by modulating SR splicing factor phosphorylation[J]. Molecular Cell, 2010, 39(6): 925-938. DOI:10.1016/j.molcel.2010.08.011 (  0) 0) |
| [14] |
SI Zizhen, YU Lei, JING Haoyu, et al. Oncogenic lncRNA ZNF561-AS1 is essential for colorectal cancer proliferation and survival through regulation of miR-26a-3p/miR-128-5p-SRSF6 axis[J]. Journal of Experimental & Clinical Cancer Research, 2021, 40(1): 78. DOI:10.1186/s13046-021-01882-1 (  0) 0) |
| [15] |
DAVID C J, CHEN Mo, ASSANAH M, et al. HnRNP proteins controlled by c-Myc deregulate pyruvate kinase mRNA splicing in cancer[J]. Nature, 2010, 463(7279): 364-368. DOI:10.1038/nature08697 (  0) 0) |
| [16] |
BELTRAN M, PUIG I, PENA C, et al. A natural antisense transcript regulates Zeb2/Sip1 gene expression during Snail1-induced epithelial-mesenchymal transition[J]. Genes & Development, 2008, 22(6): 756-769. DOI:10.1101/gad.455708 (  0) 0) |
| [17] |
VILLAMIZAR O, CHAMBERS C B, RIBERDY J M, et al. Long noncoding RNA Saf and splicing factor 45 increase soluble Fas and resistance to apoptosis[J]. Oncotarget, 2016, 7(12): 13810-13826. DOI:10.18632/oncotarget.7329 (  0) 0) |
| [18] |
TENG Liu, FENG Yuchen, GUO Sutang, et al. The pan-cancer lncRNA PLANE regulates an alternative splicing program to promote cancer pathogenesis[J]. Nature Communications, 2021, 12(1): 3734. DOI:10.1038/s41467-021-24099-4 (  0) 0) |
| [19] |
ZHANG Guanxiong, LAN Yujia, XIE Aimi, et al. Comprehensive analysis of long noncoding RNA (lncRNA)-chromatin interactions reveals lncRNA functions dependent on binding diverse regulatory elements[J]. Journal of Biological Chemistry, 2019, 294(43): 15613-15622. DOI:10.1074/jbc.RA119.008732 (  0) 0) |
| [20] |
TANG Yanyan, WANG Jinpeng, LIAN Yu, et al. Linking long non-coding RNAs and SWI/SNF complexes to chromatin remodeling in cancer[J]. Molecular Cancer, 2017, 16(1): 42. DOI:10.1186/s12943-017-0612-0 (  0) 0) |
| [21] |
HUANG Mingyan, WANG Huamin, HU Xiang, et al. lncRNA MALAT1 binds chromatin remodeling subunit BRG1 to epigenetically promote inflammation-related hepatocellular carcinoma progression[J]. Oncoimmunology, 2019, 8(1): e1518628. DOI:10.1080/2162402X.2018.1518628 (  0) 0) |
| [22] |
PANDEY R R, MONDAL T, MOHAMMAD F, et al. Kcnq1ot1 antisense noncoding RNA mediates lineage-specific transcriptional silencing through chromatin-level regulation[J]. Molecular Cell, 2008, 32(2): 232-246. DOI:10.1016/j.molcel.2008.08.022 (  0) 0) |
| [23] |
LUCO R F, ALLO M, SCHOR I E, et al. Epigenetics in alternative pre-mRNA splicing[J]. Cell, 2011, 144(1): 16-26. DOI:10.1016/j.cell.2010.11.056 (  0) 0) |
| [24] |
SIMS R R, MILLHOUSE S, CHEN C F, et al. Recognition of trimethylated histone H3 lysine 4 facilitates the recruitment of transcription postinitiation factors and pre-mRNA splicing[J]. Molecular Cell, 2007, 28(4): 665-676. DOI:10.1016/j.molcel.2007.11.010 (  0) 0) |
| [25] |
CHO K R, SHIH I. Ovarian cancer[J]. Annual Review of Pathology-Mechanisms of Disease, 2009, 4: 287-313. DOI:10.1146/annurev.pathol.4.110807.092246 (  0) 0) |
| [26] |
SHOU Jian, ZHANG Chuanling, ZHENG Xiaoyu, et al. LncRNA HCP5 Facilitates the Progression of Ovarian Cancer by Interacting with the PTBP1 Protein[J]. Biochemical Genetics, 2024, 62(4): 3136-3154. DOI:10.1007/s10528-023-10558-8 (  0) 0) |
| [27] |
WU Jianfa, NI Xiaohong, YU Zhen, et al. CRNDE inducing cisplatin resistance through SRSF1/TIA1 signaling pathway in ovarian cancer[J]. Pathology Research and Practice, 2022, 235: 153957. DOI:10.1016/j.prp.2022.153957 (  0) 0) |
| [28] |
WEITZ J, KOCH M, DEBUS J, et al. Colorectal cancer[J]. Lancet, 2005, 365(9454): 153-165. DOI:10.1016/S0140-6736(05)17706-X (  0) 0) |
| [29] |
LAN Zhixian, YAO Xiang, SUN Kangyue, et al. The interaction between lncrna snhg6 and hnrnpa1 contributes to the growth of colorectal cancer by enhancing aerobic glycolysis through the regulation of alternative splicing of PKM[J]. Frontiers in Oncology, 2020, 10: 363. DOI:10.3389/fonc.2020.00363 (  0) 0) |
| [30] |
HU Zhiyan, WANG Xiaoyan, GUO Wenbin, et al. Long non-coding RNA MALAT1 increases AKAP-9 expression by promoting SRPK1-catalyzed SRSF1 phosphorylation in colorectal cancer cells[J]. Oncotarget, 2016, 7(10): 11733-11743. DOI:10.18632/oncotarget.7367 (  0) 0) |
| [31] |
KONG Jianlu, SUN Wenjie, LI Chen, et al. Long non-coding RNA LINC01133 inhibits epithelial-mesenchymal transition and metastasis in colorectal cancer by interacting with SRSF6[J]. Cancer Letters, 2016, 380(2): 476-484. DOI:10.1016/j.canlet.2016.07.015 (  0) 0) |
| [32] |
SMYTH E C, NILSSON M, GRABSCH H I, et al. Gastric cancer[J]. Lancet, 2020, 396(10251): 635-648. DOI:10.1016/S0140-6736(20)31288-5 (  0) 0) |
| [33] |
ZHANG Feifei, WANG Hui, YU Jiang, et al. LncRNA CRNDE attenuates chemoresistance in gastric cancer via SRSF6-regulated alternative splicing of PICALM[J]. Molecular Cancer, 2021, 20(1): 6. DOI:10.1186/s12943-020-01299-y (  0) 0) |
| [34] |
HE Qiuming, YANG Chaogang, XIANG Zhenxian, et al. LINC00924-induced fatty acid metabolic reprogramming facilitates gastric cancer peritoneal metastasis via hnRNPC-regulated alternative splicing of Mnk2[J]. Cell Death & Disease, 2022, 13(11): 987. DOI:10.1038/s41419-022-05436-x (  0) 0) |
| [35] |
ZHU Wenzhong, TAN Lulu, MA Tiantian, et al. Long noncoding RNA SNHG8 promotes chemoresistance in gastric cancer via binding with hnRNPA1 and stabilizing TROY expression[J]. Digestive and Liver Disease, 2022, 54(11): 1573-1582. DOI:10.1016/j.dld.2022.02.011 (  0) 0) |
| [36] |
FORNER A, REIG M, BRUIX J. Hepatocellular carcinoma[J]. Lancet, 2018, 391(10127): 1301-1314. DOI:10.1016/S0140-6736(18)30010-2 (  0) 0) |
| [37] |
GONZALEZ I, MUNITA R, AGIRRE E, et al. A lncRNA regulates alternative splicing via establishment of a splicing-specific chromatin signature[J]. Nature Structural & Molecular Biology, 2015, 22(5): 370-376. DOI:10.1038/nsmb.3005 (  0) 0) |
| [38] |
AMANN T, BATAILLE F, SPRUSS T, et al. Reduced expression of fibroblast growth factor receptor 2IIIb in hepatocellular carcinoma induces a more aggressive growth[J]. American Journal of Pathology, 2010, 176(3): 1433-1442. DOI:10.2353/ajpath.2010.090356 (  0) 0) |
| [39] |
LIN Y H, WU M H, LIU Y C, et al. LINC01348 suppresses hepatocellular carcinoma metastasis through inhibition of SF3B3-mediated EZH2 pre-mRNA splicing[J]. Oncogene, 2021, 40(28): 4675-4685. DOI:10.1038/s41388-021-01905-3 (  0) 0) |
| [40] |
MALAKAR P, SHILO A, MOGILEVSKY A, et al. Long Noncoding RNA MALAT1 Promotes Hepatocellular Carcinoma Development by SRSF1 Upregulation and mTOR Activation[J]. Cancer Research, 2017, 77(5): 1155-1167. DOI:10.1158/0008-5472.CAN-16-1508 (  0) 0) |
| [41] |
KLEIN A P. Pancreatic cancer epidemiology: Understanding the role of lifestyle and inherited risk factors[J]. Nature Reviews Gastroenterology & Hepatology, 2021, 18(7): 493-502. DOI:10.1038/s41575-021-00457-x (  0) 0) |
| [42] |
MENG L D, SHI G D, GE W L, et al. Linc01232 promotes the metastasis of pancreatic cancer by suppressing the ubiquitin-mediated degradation of HNRNPA2B1 and activating the A-Raf-induced MAPK/ERK signaling pathway[J]. Cancer Letters, 2020, 494: 107-120. DOI:10.1016/j.canlet.2020.08.001 (  0) 0) |
| [43] |
ZHANG Yong, FANG Yuan, MA Lijie, et al. LINC00857 regulated by ZNF460 enhances the expression of CLDN12 by sponging miR-150-5p and recruiting SRSF1 for alternative splicing to promote epithelial-mesenchymal transformation of pancreatic adenocarcinoma cells[J]. RNA Biology, 2022, 19(1): 548-559. DOI:10.1080/15476286.2021.1992995 (  0) 0) |
| [44] |
XU Shouping, WANG Peiyuan, ZHANG Jian, et al. Ai-lncRNA EGOT enhancing autophagy sensitizes paclitaxel cytotoxicity via upregulation of ITPR1 expression by RNA-RNA and RNA-protein interactions in human cancer[J]. Molecular Cancer, 2019, 18(1): 89. DOI:10.1186/s12943-019-1017-z (  0) 0) |
| [45] |
SINGH R, GUPTA S C, PENG W X, et al. Regulation of alternative splicing of Bcl-x by BC200 contributes to breast cancer pathogenesis[J]. Cell Death & Disease, 2016, 7(6): e2262. DOI:10.1038/cddis.2016.168 (  0) 0) |
| [46] |
DUAN Yuqing, JIA Yunlong, WANG Jiali, et al. Long noncoding RNA DGCR5 involves in tumorigenesis of esophageal squamous cell carcinoma via SRSF1-mediated alternative splicing of Mcl-1[J]. Cell Death & Disease, 2021, 12(6): 587. DOI:10.1038/s41419-021-03858-7 (  0) 0) |
| [47] |
ZHANG Ziqiang, ZHOU Nanjiang, HUANG Jianguo, et al. Regulation of androgen receptor splice variant AR3 by PCGEM1[J]. Oncotarget, 2016, 7(13): 15481-15491. DOI:10.18632/oncotarget.7139 (  0) 0) |
| [48] |
BERNARD A, BOIDOT R, VEGRAN F. Alternative splicing in cancer and immune cells[J]. Cancers, 2022, 14(7): 1726. DOI:10.3390/cancers14071726 (  0) 0) |
| [49] |
AUBREY B J, STRASSER A, KELLY G L. Tumor-suppressor functions of the TP53 pathway[J]. Cold Spring Harbor Perspectives in Medicine, 2016, 6: a026062. DOI:10.1101/cshperspect.a026062 (  0) 0) |
| [50] |
YAO Yiran, GU Xiang, XU Xiaofang, et al. Novel insights into RB1 mutation[J]. Cancer Letters, 2022, 547: 215870. DOI:10.1016/j.canlet.2022.215870 (  0) 0) |
| [51] |
CHEN C Y, CHEN Jingyu, HE Lina, et al. PTEN: Tumor suppressor and metabolic regulator[J]. Frontiers in Endocrinology, 2018, 9: 338. DOI:10.3389/fendo.2018.00338 (  0) 0) |
| [52] |
CALABRESE C, DAVIDSON N R, DEMIRCIOGLU D, et al. Genomic basis for RNA alterations in cancer[J]. Nature, 2020, 578(7793): 129-136. DOI:10.1038/s41586-020-1970-0 (  0) 0) |
| [53] |
BAO Guangyao, LI Tian, GUAN Xiaojiao, et al. Development of a prognostic alternative splicing signature associated with tumor microenvironment immune profiles in lung adenocarcinoma[J]. Frontiers in Oncology, 2022, 12: 880478. DOI:10.3389/fonc.2022.880478 (  0) 0) |
| [54] |
GUO Shuang, WANG Xinyue, ZHOU Hanxiao, et al. Identification and characterization of immunogene-related alternative splicing patterns and tumor microenvironment infiltration patterns in breast cancer[J]. Cancers, 2022, 14(3): 595. DOI:10.3390/cancers14030595 (  0) 0) |
| [55] |
LIU Weizhen, LI Dongfang, LU Ting, et al. Comprehensive analysis of RNA-binding protein SRSF2-dependent alternative splicing signature in malignant proliferation of colorectal carcinoma[J]. Journal of Biological Chemistry, 2023, 299(2): 102876. DOI:10.1016/j.jbc.2023.102876 (  0) 0) |
| [56] |
FUENTES-FAYOS A C, PEREZ-GOMEZ J M, G-GARCIA M E, et al. SF3B1 inhibition disrupts malignancy and prolongs survival in glioblastoma patients through BCL2L1 splicing and mTOR/ss-catenin pathways imbalances[J]. Journal of Experimental & Clinical Cancer Research, 2022, 41(1): 39. DOI:10.1186/s13046-022-02241-4 (  0) 0) |
| [57] |
GUO Jing, LI Chuzhong, FANG Qiuyue, et al. The SF3B1(R625H) mutation promotes prolactinoma tumor progression through aberrant splicing of DLG1[J]. Journal of Experimental & Clinical Cancer Research, 2022, 41(1): 26. DOI:10.1186/s13046-022-02245-0 (  0) 0) |
| [58] |
WANG Xianghu, SUN Mingjun, GAO Zhikui, et al. N-nitrosamines-mediated downregulation of LncRNA-UCA1 induces carcinogenesis of esophageal squamous by regulating the alternative splicing of FGFR2[J]. Science of the Total Environment, 2023, 855: 158918. DOI:10.1016/j.scitotenv.2022.158918 (  0) 0) |
| [59] |
HUAN Lin, GUO Tianan, WU Yangjun, et al. Hypoxia induced LUCAT1/PTBP1 axis modulates cancer cell viability and chemotherapy response[J]. Molecular Cancer, 2020, 19(1): 11. DOI:10.1186/s12943-019-1122-z (  0) 0) |
| [60] |
FU Rong, YANG Peng, AMIN S, et al. A novel miR-206/hnRNPA1/PKM2 axis reshapes the Warburg effect to suppress colon cancer growth[J]. Biochemical and Biophysical Research Communications, 2020, 531(4): 465-471. DOI:10.1016/j.bbrc.2020.08.019 (  0) 0) |
| [61] |
WU Haili, CUI Mimi, LI Chenglu, et al. Kaempferol reverses aerobic glycolysis via miR-339-5p-Mediated PKM alternative splicing in colon cancer cells[J]. Journal of Agricultural and Food Chemistry, 2021, 69(10): 3060-3068. DOI:10.1021/acs.jafc.0c07640 (  0) 0) |
| [62] |
LIU Zhiyong, LI Wei, PANG Yanan, et al. SF3B4 is regulated by microRNA-133b and promotes cell proliferation and metastasis in hepatocellular carcinoma[J]. Ebiomedicine, 2018, 38: 57-68. DOI:10.1016/j.ebiom.2018.10.067 (  0) 0) |
| [63] |
SHEN Shihao, PARK J W, LU Zhixiang, et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(51): E5593-E5601. DOI:10.1073/pnas.1419161111 (  0) 0) |
| [64] |
WU Jie, AKERMAN M, SUN S, et al. SpliceTrap: A method to quantify alternative splicing under single cellular conditions[J]. Bioinformatics, 2011, 27(21): 3010-3016. DOI:10.1093/bioinformatics/btr508 (  0) 0) |
| [65] |
SHI Yang, JIANG Hui. rSeqDiff: Detecting differential isoform expression from RNA-Seq data using hierarchical likelihood ratio test[J]. Plos One, 2013, 8(11): e79448. DOI:10.1371/journal.pone.0079448 (  0) 0) |
| [66] |
RYAN M C, CLELAND J, KIM R, et al. SpliceSeq: A resource for analysis and visualization of RNA-Seq data on alternative splicing and its functional impacts[J]. Bioinformatics, 2012, 28(18): 2385-2387. DOI:10.1093/bioinformatics/bts452 (  0) 0) |
| [67] |
RYAN M, WONG W C, BROWN R, et al. TCGASpliceSeq a compendium of alternative mRNA splicing in cancer[J]. Nucleic Acids Research, 2016, 44(D1): D1018-D1022. DOI:10.1093/nar/gkv1288 (  0) 0) |
| [68] |
LI Y I, KNOWLES D A, HUMPHREY J, et al. Annotation-free quantification of RNA splicing using LeafCutter[J]. Nature Genetics, 2018, 50(1): 151-158. DOI:10.1038/s41588-017-0004-9 (  0) 0) |
| [69] |
TRINCADO J L, ENTIZNE J C, HYSENAJ G, et al. SUPPA2: Fast, accurate, and uncertainty-aware differential splicing analysis across multiple conditions[J]. Genome Biology, 2018, 19(1): 40. DOI:10.1186/s13059-018-1417-1 (  0) 0) |
| [70] |
TANG A D, SOULETTE C M, VAN BAREN M J, et al. Full-length transcript characterization of SF3B1 mutation in chronic lymphocytic leukemia reveals downregulation of retained introns[J]. Nature Communications, 2020, 11(1): 1438. DOI:10.1038/s41467-020-15171-6 (  0) 0) |
| [71] |
LEWIS B P, SHIH I H, JONES-RHOADES M W, et al. Prediction of mammalian microRNA targets[J]. Cell, 2003, 115(7): 787-798. DOI:10.1016/s0092-8674(03)01018-3 (  0) 0) |
| [72] |
KRUGER J, REHMSMEIER M. RNAhybrid: microRNA target prediction easy, fast and flexible[J]. Nucleic Acids Research, 2006, 34(Web server issue): W451-W454. DOI:10.1093/nar/gkl243 (  0) 0) |
| [73] |
LI Jianwei, MA Wei, ZENG Pan, et al. LncTar: a tool for predicting the RNA targets of long noncoding RNAs[J]. Briefings in Bioinformatics, 2015, 16(5): 806-812. DOI:10.1093/bib/bbu048 (  0) 0) |
| [74] |
FUKUNAGA T, HAMADA M. RIblast: An ultrafast RNA-RNA interaction prediction system based on a seed-and-extension approach[J]. Bioinformatics, 2017, 33(17): 2666-2674. DOI:10.1093/bioinformatics/btx287 (  0) 0) |
| [75] |
ALKAN F, WENZEL A, PALASCA O, et al. RIsearch2: Suffix array-based large-scale prediction of RNA-RNA interactions and siRNA off-targets[J]. Nucleic Acids Research, 2017, 45(8): e60. DOI:10.1093/nar/gkw1325 (  0) 0) |
| [76] |
STICHT C, DE LA TORRE C, PARVEEN A, et al. miRWalk: An online resource for prediction of microRNA binding sites[J]. Plos One, 2018, 13(10): e206239. DOI:10.1371/journal.pone.0206239 (  0) 0) |
| [77] |
AGOSTINI F, ZANZONI A, KLUS P, et al. catRAPID omics: A web server for large-scale prediction of protein-RNA interactions[J]. Bioinformatics, 2013, 29(22): 2928-2930. DOI:10.1093/bioinformatics/btt495 (  0) 0) |
| [78] |
ARMAOS A, COLANTONI A, PROIETTI G, et al. catRAPID omics v2.0: Going deeper and wider in the prediction of protein-RNA interactions[J]. Nucleic Acids Research, 2021, 49(W1): W72-W79. DOI:10.1093/nar/gkab393 (  0) 0) |
| [79] |
YANG Cheng, YANG Longshu, ZHOU Man, et al. LncADeep: An ab initio lncRNA identification and functional annotation tool based on deep learning[J]. Bioinformatics, 2018, 34(22): 3825-3834. DOI:10.1093/bioinformatics/bty428 (  0) 0) |
| [80] |
LI Ying, SUN Hang, FENG Shiyao, et al. Capsule-LPI: A LncRNA-protein interaction predicting tool based on a capsule network[J]. Bmc Bioinformatics, 2021, 22(1): 246. DOI:10.1186/s12859-021-04171-y (  0) 0) |
| [81] |
CHU Ci, QU Kun, ZHONG F L, et al. Genomic maps of long noncoding RNA occupancy reveal principles of RNA-chromatin interactions[J]. Molecular Cell, 2011, 44(4): 667-678. DOI:10.1016/j.molcel.2011.08.027 (  0) 0) |
| [82] |
SIMON M D, WANG C I, KHARCHENKO P V, et al. The genomic binding sites of a noncoding RNA[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(51): 20497-20502. DOI:10.1073/pnas.1113536108 (  0) 0) |
| [83] |
GIULIETTI M, PIVA F, D'ANTONIO M, et al. SpliceAid-F: A database of human splicing factors and their RNA-binding sites[J]. Nucleic Acids Research, 2013, 41(Database issue): D125-D131. DOI:10.1093/nar/gks997 (  0) 0) |
| [84] |
WU Tao, WANG Jie, LIU Changning, et al. NPInter: The noncoding RNAs and protein related biomacromolecules interaction database[J]. Nucleic Acids Research, 2006, 34(Database issue): D150-D152. DOI:10.1093/nar/gkj025 (  0) 0) |
| [85] |
ZHENG Yu, LUO Huaxia, TENG Xueyi, et al. NPInter v5.0: ncRNA interaction database in a new era[J]. Nucleic Acids Research, 2023, 51(D1): D232-D239. DOI:10.1093/nar/gkac1002 (  0) 0) |
| [86] |
LI Junhao, LIU Shun, ZHOU Hui, et al. starBase v2.0: Decoding miRNA-ceRNA, miRNA-ncRNA and protein-RNA interaction networks from large-scale CLIP-Seq data[J]. Nucleic Acids Research, 2014, 42(Database issue): D92-D97. DOI:10.1093/nar/gkt1248 (  0) 0) |
| [87] |
YANG Jianhua, LI Junhao, JIANG Shan, et al. ChIPBase: A database for decoding the transcriptional regulation of long non-coding RNA and microRNA genes from ChIP-Seq data[J]. Nucleic Acids Research, 2013, 41(Database issue): D177-D187. DOI:10.1093/nar/gks1060 (  0) 0) |
| [88] |
HUANG Junhong, ZHENG Wujian, ZHANG Ping, et al. ChIPBase v3.0: The encyclopedia of transcriptional regulations of non-coding RNAs and protein-coding genes[J]. Nucleic Acids Research, 2023, 51(D1): D46-D56. DOI:10.1093/nar/gkac1067 (  0) 0) |
| [89] |
LI Yongsheng, LI Lili, WANG Zishan, et al. LncMAP: Pan-cancer atlas of long noncoding RNA-mediated transcriptional network perturbations[J]. Nucleic Acids Research, 2018, 46(3): 1113-1123. DOI:10.1093/nar/gkx1311 (  0) 0) |
| [90] |
ROUILLARD A D, GUNDERSEN G W, FERNANDEZ N F, et al. The harmonizome: A collection of processed datasets gathered to serve and mine knowledge about genes and proteins[J]. Database-the Journal of Biological Databases and Curation, 2016, 2016: baw100. DOI:10.1093/database/baw100 (  0) 0) |
| [91] |
MEI Shenglin, MEYER C A, ZHENG Rongbin, et al. Cistrome cancer: A web resource for integrative gene regulation modeling in cancer[J]. Cancer Research, 2017, 77(21): e19-e22. DOI:10.1158/0008-5472.CAN-17-0327 (  0) 0) |
| [92] |
YEVSHIN I, SHARIPOV R, VALEEV T, et al. GTRD: A database of transcription factor binding sites identified by ChIP-seq experiments[J]. Nucleic Acids Research, 2017, 45(D1): D61-D67. DOI:10.1093/nar/gkw951 (  0) 0) |
| [93] |
KOLMYKOV S, YEVSHIN I, KULYASHOV M, et al. GTRD: An integrated view of transcription regulation[J]. Nucleic Acids Research, 2021, 49(D1): D104-D111. DOI:10.1093/nar/gkaa1057 (  0) 0) |
| [94] |
HSU S D, LIN Fengmao, WU Weiyun, et al. miRTarBase: A database curates experimentally validated microRNA-target interactions[J]. Nucleic Acids Research, 2011, 39(Database issue): D163-D169. DOI:10.1093/nar/gkq1107 (  0) 0) |
| [95] |
HUANG H Y, LIN Y C, LI Jing, et al. miRTarBase 2020: Updates to the experimentally validated microRNA-target interaction database[J]. Nucleic Acids Research, 2020, 48(D1): D148-D154. DOI:10.1093/nar/gkz896 (  0) 0) |
| [96] |
PARASKEVOPOULOU M D, GEORGAKILAS G, KOSTOULAS N, et al. DIANA-LncBase: Experimentally verified and computationally predicted microRNA targets on long non-coding RNAs[J]. Nucleic Acids Research, 2013, 41(Database issue): D239-D245. DOI:10.1093/nar/gks1246 (  0) 0) |
| [97] |
KARAGKOUNI D, PARASKEVOPOULOU M D, TASTSOGLOU S, et al. DIANA-LncBase v3: Indexing experimentally supported miRNA targets on non-coding transcripts[J]. Nucleic Acids Research, 2020, 48(D1): D101-D110. DOI:10.1093/nar/gkz1036 (  0) 0) |
| [98] |
YU Fulong, ZHANG Guanxiong, SHI Aiai, et al. LnChrom: A resource of experimentally validated lncRNA-chromatin interactions in human and mouse[J]. Database-the Journal of Biological Databases and Curation, 2018, 2018: bay039. DOI:10.1093/database/bay039 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


