IRES是一种RNA内部顺式作用元件。在大多数非帽依赖翻译起始事件中,IRES直接招募40S核糖体亚基与起始密码子上游区域结合,或直接与起始密码子结合,启动蛋白质翻译。随着越来越多的IRES在真核细胞mRNA中被发现,IRES介导的非帽依赖翻译起始机制开始引起人们的注意。已有研究表明,细胞在处于内质网应激、缺氧、凋亡、有丝分裂或分化等压力环境时,帽依赖翻译起始机制会受到抑制,细胞开始通过IRES介导的翻译机制生成维持生存所必须的蛋白质。最近,大量环形RNA(circRNA)被发现可以作为信使RNA编码蛋白质[1-2],其中IRES元件在翻译起始的核糖体招募过程中扮演着极其重要角色[3-5]。目前研究揭示的IRES介导的翻译起始事件仅是冰山一角,且绝大多数已知IRES元件的翻译调控机制也仍然未知。因此本文对IRES起始翻译的相关研究进行了综述(图 1),并对目前现有的可用于识别或分析IRES的生物信息学工具进行了总结。
|
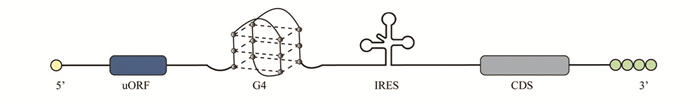
图 1 mRNA 5’UTR可能存在的结构 Figure 1 Possible structure of mRNA 5'UTR |
1991年,Macejak和Sarnow首次在细胞中发现IRES介导的非帽依赖翻译起始事件[6]。他们发现在脊髓灰质炎病毒感染细胞后,尽管宿主细胞mRNA的帽依赖翻译途径因真核起始因子eIF-4F的失活而被抑制,但仍检测到免疫球蛋白重链结合蛋白(Immunoglobulin heavy-chain binding protein, BIP)的生成。Northern印迹分析表明,未感染病毒和感染病毒细胞中BIP mRNA的稳态水平相同,因此BIP蛋白水平的增加仅可能是翻译水平上被诱导所致。双顺反子实验表明,BIP mRNA的5’UTR具有非帽依赖翻译活性,证实含有IRES元件。
之后,IRES元件被发现在疾病或应激反应引发的细胞凋亡中发挥着重要作用。例如,XIAP(X-linked inhibitor of apoptosis)是凋亡抑制剂家族的代表性成员,通过IRES介导的翻译机制维持蛋白质的稳定表达,增强细胞的抗凋亡能力[7]。在与细胞凋亡有关的其他mRNA(Apaf-1、c-myc等)中也发现IRES元件,表明IRES介导的翻译调控机制对维持细胞稳态的重要作用[8-9]。但是,Apaf-1可激活半胱氨酸蛋白酶(Caspase)进而诱导细胞凋亡,展现出与XIAP在细胞凋亡调节中截然不同的作用。因此,IRES的差异激活引起的关键调控因子翻译水平变化对于凋亡途径的进展以及细胞的生存死亡十分重要。
近来,Weingarten-Gabbay等[10]设计了一种高通量双顺反子实验分析方法来识别非帽依赖翻译起始调控元件,使迄今已知的具有帽独立翻译活性的序列数量增加了50倍,证实人类转录组中约10%的5’UTR含有IRES元件。根据IRES的序列结构特征,IRES元件被分成了两类,分别代表两种潜在的翻译调控机制。一类被称为“局部敏感型”IRES,其主要通过短序列基序(如ITAF结合位点)发挥作用,只有特定基序的突变会降低其活性;另一类则被称为“全局敏感型”IRES,其活性与其二级结构密切相关,不同位置的突变都有可能破坏其整体结构,导致活性降低。此外,研究还发现具有帽独立翻译活性的基因并没有在特定生物过程、细胞成分或分子功能当中富集,预示IRES介导的非帽依赖翻译是功能基因间共享的一种全局机制。
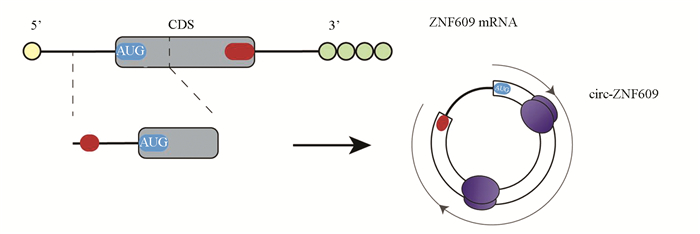
如今随着核糖体图谱分析及高通量测序技术的应用和发展,研究发现一些先前认为不具备编码能力的circRNA和lncRNA也可通过内部IRES元件翻译产生蛋白质或小肽。Legnini等[11]发现circ-ZNF609包含一个753-nt的开放阅读框,可通过UTR区中的IRES元件招募核糖体翻译产生蛋白(图 2)。此外,circ-FBXW7、circ-SHPRH、circPINTexon2、circβ-catenin、circAKT3等[4-5, 12-14]均已被实验证实可由其内部IRES元件介导翻译产生具有重要生物学功能的蛋白质。Carbonnele等[15]则在lncRNA MELOE上发现其序列中的两个小ORF通过IRES介导的翻译机制编码产生黑色素瘤抗原MELOE-1和MELOE-2,或可作为黑色素瘤免疫治疗的理想靶点。
|
图 2 circ-ZNF609翻译过程 Figure 2 Translation process of circ-ZNF609 |
LncRNA和circRNA作为进化保守且数量丰富的内源性RNA,已被证实在多种生物学过程中扮演着重要角色,但其分子作用机制在很大程度上仍有待探索。IRES介导的帽独立性翻译起始机制则为解释其细胞调控功能提供了一种新的可能,而对于已知可编码蛋白的circRNAs,IRES介导的翻译调控机制也有待进一步的阐明。
2 IRES翻译相关调控机制 2.1 IRES反式作用因子在非帽依赖翻译起始调控过程中,除一些标准的翻译起始因子(Eukaryotic initiation factors, eIFs)外,IRES还需要与相应的反式作用因子(IRES trans-acting factor,ITAF)结合,才能完成核糖体的招募工作。ITAF作为一种特殊的RNA结合蛋白,其在IRES介导的翻译调控中的确切作用机制尚不完全清楚。
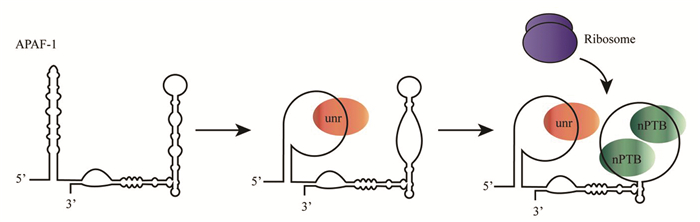
已有研究表明,ITAF大致有以下两种作用方式:1)重塑IRES周围的RNA结构,帮助其获得合适的构象状态,促进IRES与核糖体的结合;2)作为接头蛋白与核糖体或其他ITAFs相互作用,增加IRES和核糖体之间的结合亲和力[16]。Apaf-1(Apoptotic protease-activating factor 1)中的IRES元件具有高度的二级结构,其仅在unr(Upstream of N-ras)存在时,才可与PTB结合,发挥核糖体招募能力[17]。unr与Apaf-1中的IRES相结合,促使后者的茎环结构被打开,使得PTB得以与之结合,进而使IRES获得核糖体“着陆”的正确结构构象(图 3)。BAG-1(Bcl-2 associated athanogene 1)则需要PTB-1和PCBP1共同作用,才能起始IRES介导的翻译过程。其中,PCBP1的主要作用是改变IRES周围的RNA构象,让核糖体的招募不再受该区域二级结构的阻碍;而PTB-1则作为接头蛋白,是起始前复合物的重要组分[18-19]。
|
图 3 APAF-1 IRES通过与ITAF结合进行核糖体招募 Figure 3 APAF-1 IRES recruits ribosomes by binding to ITAF |
ITAF对IRES的影响可能是积极性的,也可能是抑制性的,目前已发现有11种ITAFs具有这种双重作用[20]。例如,PTB通过与IRES元件的相互作用,特异性地增强p27 mRNA的IRES活性[21];而在另一项研究中则发现PTB抑制Bip IRES介导的翻译,这种抑制也是特异性的,但对于其具体的工作机制目前仍不是很清楚[22]。大部分研究倾向于认为,作为负调节因子发挥作用的ITAF,可能保留或者进一步稳定了IRES的二级结构,从而抑制了后者的核糖体招募能力。此外,有些编码ITAF的mRNA本身也含有IRES。例如,unr蛋白在体内能够与自身mRNA相互作用,并且负调控unr的表达[23]。DAP5(Death-associated protein 5)也有类似报道,Henis-Korenblit等[24]则证其在细胞凋亡过程中可以通过一个正反馈回路,调节自身IRES的活性。
ITAFs间的相互作用除了可以是协同合作外,也可能是竞争性的。Sharathchandra等[25]证明Annexin A2和PSF均能与p53 IRES特异性结合,并且在IRES元件上有重叠的结合位点。通过紫外交联实验,他们发现随着PSF浓度的增加,Annexin A2与p53 IRES的相互作用逐渐减弱;增加Annexin A2,PSF与p53 IRES间的结合同样逐渐减少;以上结果表明这两种蛋白质相互竞争性结合p53中的IRES元件。此外,具有相反活动特性的ITAFs也可能存在竞争性作用关系。Takagi等[26]证明RPL26(Ribosomal protein L26)的过表达,会增强p53 mRNA与多聚体的关联,增加p53的翻译;相反,nucleolin的过表达则会抑制p53的翻译。并且,它们两者也在p53 IRES上竞争相同的结合位点,通过在MCF7细胞中强制表达nucleolin,p53的蛋白水平显著降低,而将RPL26转染这些细胞后,则逆转了nucleolin对p53的翻译抑制。
最近,Yang等[27]报道了一种lncRNA TRMP(TP53-regulated modulator of p27),可通过与PTBP1结合破坏其与p27 mRNA 5’UTR的相互作用,进而抑制p27的IRES依赖性翻译。虽然这种抑制作用是直接的,但具体机制还有待进一步研究,此外这可能不是唯一能够调节IRES翻译的lncRNA。
2.2 G-四链体G-四链体(G-quadruplex, G4)是富含鸟嘌呤的DNA或RNA形成的独特核酸结构。生物信息学分析显示,人类mRNA的5’UTR中有4 000多个G4基序[28-29],其通常会抑制帽依赖性翻译起始[30]。然而,当G4结构位于IRES元件附近或作为IRES元件的一部分时,则会促进非帽依赖性翻译起始。
FGF-2(Human fibroblast growth factor 2)mRNA包含四个CUG和一个AUG起始密码子,其中5’近端的CUG密码子依赖帽子结构招募核糖体,而其余四个起始密码子则由IRES介导核糖体招募。Bonnal等[31]通过化学探测和酶足迹实验确定FGF-2 IRES包含两个茎环结构和一个G4基序,每个结构域都有助于提高IRES活性,从而调控下游四个起始密码子的翻译起始。
hVEGF(Human vascular endothelial growth factor)mRNA包含两个独立的IRES元件,其中IRES-A可通过不同的鸟嘌呤G-延伸组合,切换不同的G4结构,引起其IRES活性变化,从而在不同程度上支持翻译起始。Morris等[32]通过双荧光素酶报告系统证实,IRES-A可以在缺氧条件下维持hVEGF的翻译,但当G4结构被破坏,则其活性丧失,蛋白翻译水平下降。
ARPC2(Actin-related protein 2/3 complex subunit 2)mRNA的一个亚型的IRES元件中富含鸟嘌呤的区域能形成G4结构,Al-Zeer等[33]通过引入突变破坏G4结构,发现其翻译效率显著降低约30%。虽然这种效应不如hVEGF报道的那么明显,但也显示出G4在IRES介导的翻译起始中有着促进作用。类似地,Koukouraki和Doxakis等[34]在SNCA(α-Synuclein)mRNA的5’UTR中同样发现了含有G4基序的IRES元件,且证实该G4结构对于IRES的功能不是绝对必需的,但能提高其活性。
此外,Jodoin等[35]在2019年首次报道了第一个可以同时影响帽依赖和非帽依赖翻译功能的BAG-1 RNA G4结构。该团队在之前的一项研究中发现BAG-1 5’UTR的第6-35核苷酸位置处存在一个G4结构,并证实此G4结构会抑制BAG-1所有亚型的帽依赖翻译功能[36]。而此次研究发现,该G4结构的突变还会导致BAG-1 IRES元件(5’UTR中第247-423核苷酸处)翻译起始调控活性的下降,即使它本身不属于IRES的结构部分。为了阐明这一现象,利用SHAPE技术分析发现G4结构的破坏显著改变了IRES区域的碱基配对规则,使得IRES所在区域的结构更加稳定和“封闭”,进而阻碍了其与招募40S核糖体至关重要的ITAF的结合,引起IRES元件功能失活。
2.3 uORF上游开放阅读框(Upstream open reading frame,uORF)位于mRNA主翻译区(Main open reading frame,mORF)起始密码子的上游,是控制蛋白质翻译的元件之一。在帽依赖翻译过程中,uORF因位置关系会先于mORF与核糖体结合,这通常导致后者翻译受阻。最近研究发现,uORF可与IRES一起协同调控细胞在不同状态下的蛋白质表达水平。
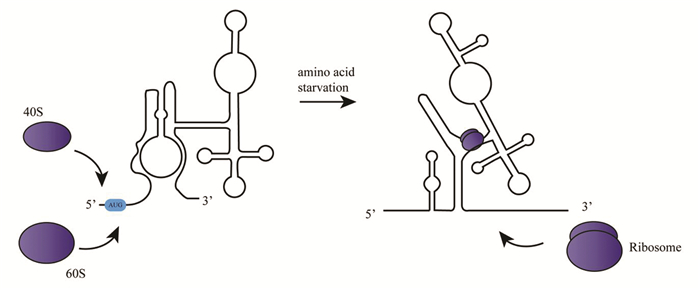
Yaman等[37]报道,在氨基酸饥饿诱发的细胞应激反应中,CAT-1(Arginine/Lysine transporter)mRNA的帽依赖翻译途径被抑制,转而通过其内部IRES元件招募核糖体,翻译产生蛋白质。研究发现,CAT-1 uORF是调节IRES元件活性的开关,uORF的翻译将激活后者的翻译起始调控活性。当uORF不翻译时,IRES元件受自身结构影响,被锁定在休眠状态,氨基酸饥饿亦无法诱发CAT-1的非帽依赖翻译;而当uORF翻译时,这种锁定结构被破坏,IRES元件转变为活性结构,CAT-1的非帽依赖翻译能力增加(图 4)。
|
图 4 CAT1 mRNA uORF的翻译调控IRES活性 Figure 4 Translation of CAT1 mRNA uORF regulates IRES activity |
Chen等[38]发现,在正常状态下,FGF9(Fibroblast growth factor 9)mRNA通过帽依赖翻译机制合成蛋白质,但受到uORF翻译的抑制,其蛋白表达水平较低。然而,当细胞处于缺氧环境时,FGF9由帽依赖转换为IRES依赖的翻译途径,mORF的翻译不再受到uORF的限制,蛋白质表达上调。核糖体分析实验发现,在低氧条件下,uORF起始密码子位置附近的核糖体分布显著减少,而CDS起始密码子位置附近的核糖体分布则显著增加。这一结果表明,在缺氧状态下,FGF9 mRNA由帽依赖翻译转换为IRES介导的非帽依赖翻译,进而摆脱uORF的翻译限制,促进蛋白质表达水平的上升。
与uORF调节IRES活性类似,IRES也可影响uORF的调控功能。Bastide等[39]研究证实,在VEGFA(Vascular endothelial growth factor A)mRNA中,病理条件下,缺氧反应启动子优先激活,从而允许VEGF 121的起始事件表达;相反,由于低氧反应启动子在常氧条件下活性较弱,此外当内部启动子激活时,位于IRES-A序列内的uORF以帽独立的方式翻译,从而抑制VEGF 121亚型的翻译表达。
随着越来越多的uORF和IRES的发现,它们在各种应激条件下的“开关”模式可能代表了在不断变化的环境中控制蛋白质合成的常见机制,但还需进一步研究确定它们的RNA结构以及相应的ITAF,从而解析两者的调控关系。
3 IRES元件相关生物信息学工具及数据库IRES实验验证方法需体外扩增待检序列,构建双顺反子载体,并设置阳性及阴性对照,最终根据报告基因的相对表达水平评估待检序列的IRES活性。此外,若想进一步确定IRES元件的精确位置,则需从两端依次截短待检序列,重复验证截短序列的IRES活性,直至IRES活性显著下降。IRES元件实验鉴定方法成本较高,费时费力,且常需设置多个对照实验以排除其他因素的干扰。开发IRES元件相关生物信息学识别方法,则有助于缩小实验验证的潜在序列范围,排除干扰因素,减少实验成本(表 1)。此外,构建数据库整合已知IRES元件相关数据资源,也有助于对其功能及作用机制的进一步研究(表 2)。
| 表 1 IRES识别工具 Table 1 IRES identification tools |
| 表 2 IRES数据库 Table 2 IRES database |
2009年,Wu等[40]为了获得更好的IRES预测结果,设计了一个IRES搜索系统,命名为IRSS。该系统主要包含两步:RNA二级结构预测、RNA二级结构比对,由两个主要的程序完成。其中,RNALfold程序用于通过最小自由能方法预测局部RNA二级结构,而RNA比对程序则将预测结构与已知IRES元件二级结构进行比较。在RNA比对程序中,根据距离分数(Distance score, DIST)和比对匹配长度(Alignment match length, ALEN)计算IRES预测得分(R=ALEN/DIST)。工具以Web服务的形式供用户使用,工具输入为FASTA或纯文本格式的序列,输出会列出所有R ≥ 1.5的IRES预测结果,包括RNA序列、二级结构及最小自由能。
2013年,Hong等[41]又在IRSS的基础上,增添了pknotsRG程序,用以提高病毒IRES元件的预测准确性,新方法被命名为VIPS。病毒IRES元件根据RNA二级结构可主要分为四种类型:第1种类型的代表为蟋蟀麻痹病毒(Cricket paralysis virus, CrPV)的IRES结构;第二种类型的代表为丙型肝炎病毒(Hepatitis C Virus, HCV)的IRES结构;第三种类型的代表为脑心肌炎病毒(Encephalomyocarditis virus, EMCV)的IRES结构;第四种类型的代表为脊髓灰质炎病毒(Poliovirus, PV)的IRES结构。VIPS对此四种类型IRES元件的预测准确率分别为98.53%、90.80%、82.36%和80.41%。与IRSS相比,使用RNA假结结构预测模块后,VIPS显著提高了第1及第4中类型IRES元件的预测敏感性和准确性。VIPS同样以Web服务的形式供用户使用,输入序列限制在5 000个核苷酸以下,输出则在IRSS结果的基础上增添了假结结构预测结果。不过,在IRSS和VIPS中,如果RNALfold程序预测的IRES元件不能匹配Rfam中的任何IRES模型或相关物种中至少两个同源IRES,那这个输入数据会被丢弃,所以该工具倾向于预测具有高保守性的候选IRES元件,发现新型IRES的能力较低;同时,它是基于最小自由能的方法进行预测,那对于较长的序列则会降低结果的准确性,所以对输入序列有长度限制;但近来VIPS及IRSS均已无法访问。
2016年,Kolekar等[42]基于支持向量机模型,开发了一个可用于病毒及细胞IRES元件的预测工具IRESPred。模型所用训练及测试数据集中的正样本来自IRESite数据库及其收集的经过实验验证的IRES序列(n=189),负样本则来自随机选择的看家基因5’UTR序列(n=97)、细胞CDS序列(n=46)和病毒CDS序列(n=46)。该预测模型共使用35个特征,其中8个为UTR的序列和结构特征,其余为UTR与27种小亚基核糖体蛋白(Small-subunit ribosomal proteins, SSRPs)的相互作用概率。116个实验验证的细胞IRES序列,IRESPred正确预测85个,准确率为73.2%;58个人类IRES序列,IRESPred正确预测44个,准确率为75.86%。IRESPred也以web服务形式供用户使用,每次最多接受10个长度在15到7 500个碱基之间的FASTA格式的核苷酸序列,输出包括IRES预测结果、二级结构及35个特征的值。众所周知,在预测模型的开发中起着决定性作用的是真实正负数据集的可用性,IRESPred缺乏实验验证的真实负样本数据,使用的是遵循帽依赖起始路径的看家基因序列,并且训练数据集的数量也比较少。此外,它的输入限制导致其也不适用于处理大量数据。
2018年,本课题组[44]提出了一种新的用于识别细胞IRES元件的工具IRESfinder。与以往工具不同,所用数据集均来自Weingarten-Gabbay等[10]的高通量IRES元件鉴定实验,包括实验验证具有IRES活性的583个人类序列(正样本)及大量不具有IRES活性的序列(负样本)。我们开发了一种新的framed k-mer方法来计算序列特征,从中筛选出由19个特征组成的最优集,构建逻辑回归分类模型,十折交叉验证的平均AUC值为0.825。在独立测试数据集上,IRESfinder展现出80%的预测精确率和73%的预测准确率。与IRESPred相比,IRESfinder除提供预测序列是否为IRES的判定结果外,还会给用户提供序列的概率评分值,据此用户可以对输入序列做进一步的筛选,并且具有更高的准确度和精确度。此外,特征分析发现IRES序列倾向于包含更多的T、TNNTT和TTNA,而非IRES序列倾向于包含更多CG和CGG,这一结果表明T(或U)核苷酸可能在IRES的功能中起着关键作用。
2019年,Wang等[45]结合序列与结构特征,开发了基于XGBoost的IRES分类器—IRESpy。其训练集同样来自Weingarten-Gabbay等[10]的高通量IRES元件鉴定实验,IRES活性分数高于600的序列被选作正样本,其余序列则被当作负样本。独立测试集则选用IRESite数据库中收录的IRES序列为正样本,随机挑选看家基因的5’UTR序列作为负样本。IRESpy主要使用了三种类型特征,序列特征、结构特征和序列-结构混合特征(即结合了一级序列和预测的碱基配对结构的三联体特征)。通过比较XGBoost模型预测的IRES与已定量的IRES实验活性,发现当两个数据集的预测阈值均为0.1时,13.47%的人类序列预计包含IRES,接近之前报告中报道的10%;同时对于高活性分数(>600)的IRES,预测的IRES正确概率显著高于活性分数接近基本水平(≤600)的IRES,表明XGBoost模型对高活性IRES的预测精度较高。
3.2 IRES元件数据库IRESdb是Bonnal等[47]于2003年开发的首个IRES元件数据库,包含30个病毒IRES和50个真核IRES。其中病毒IRES根据NCBI病毒分类法进行了归类,而细胞IRES则根据基因产物功能进行了归类。IRES的注释信息包括病毒或包含IRES的基因的缩写和全名、观察到IRES活性的细胞条件、与IRES发生物理相互作用的蛋白质(可能的反式作用因子)等。数据库地址为 http://ifr31w3.toulouse.inserm.fr/IRESdb/,目前已无法访问。
IRESite是Mokrejs等[48, 51]于2006年开发的IRES元件数据库,并于2010年进行了更新。该数据库不仅存储了IRES验证实验的主要结果(序列、结构、测量值),还存储了实验最重要的方法学特征。IRESite数据库主要包含两种类型的IRES元件,分别是存在于病毒或细胞mRNA中的天然IRES,和用于实验的人工构建的IRES。IRESite目前收录了68种病毒和115种真核细胞mRNA中的IRES,数据库网址为 http://iresite.org/。
IRESbase是本课题组[49]于2020年开发的一个IRES元件综合数据库,总共收录了1 328个IRES元件,其中554个病毒IRES,691个人类IRES以及83个来自其他真核生物的IRES,数据库网址为 http://reprod.njmu.edu.cn/iresbase/。该数据库提供了丰富的注释信息,特别是对于人类IRES元件,包括基因组位置信息、序列保守性、单核苷酸多态性(SNP)、RNA修饰、miRNA结合位点、宿主基因、宿主转录本(mRNA、lncRNA及circRNA三种类型)、GO注释、KEGG通路以及验证实验等相关信息。IRESbase对于同一区域内不同长度的IRES元件,仅最短的IRES元件作为核心序列被收录。IRESbase是首个收录circRNA和lncRNA中IRES元件的数据库,并且通过对mRNA来源的IRES元件进行重定位分析,发现2 191个circRNAs和168个lncRNAs包含部分或全长IRES序列。
Human IRES Atlas是Yang等[50]于2021年构建的人类IRES图谱数据库。其主要利用IRESfinder、PatSearch[52](可检测核苷酸序列中的特定模式和结构基序)和IRESpy三种工具对人类转录组范围的IRES元件进行预测,将预测结果中的重叠区域作为IRES序列,共识别出17 013个位于5’UTR中的IRES元件。并且,还单独利用IRESpy从CDS和3’UTR中识别出1 868个IRES。此外,还从IRESite、IRESbase及文献中收集了659个实验验证的IRES元件。该数据库提供八种类型的翻译起始特征数据,包括翻译起始位点、uORF、序列保守性、核糖体图谱数据、IRES元件结构信息、实验鉴定的ITAF靶点序列、双顺反子实验及其他IRES实验证据。该数据库旨在帮助推断IRES驱动的细胞翻译相关调节机制,网址为 http://cobishss0.im.nuk.edu.tw/Human_IRES_Atlas/。
4 总结蛋白质合成是维持细胞内稳态的关键调节过程,翻译机制的错误调控会导致多种疾病发生。翻译的调节发生在翻译过程的各个阶段,即起始、延伸和终止。但人们普遍认为,翻译起始是限速步骤,也是最严格的一步,这其中就包括IRES介导的调控起始。总的来说,虽然近年来对IRES的研究有了大幅深入,但细胞IRES相关的结构、调节机制及潜在的信号通路仍不清楚,以及现有工具的准确性也有待进一步提升。这其中一个限制性的因素就是实验鉴定的IRES元件的数量有限。一方面是因为相比于病毒IRES,细胞IRES含有更少的RNA结构,并且它们之间的序列保守性小,因此很难被分为不同的组,这也导致很难预测mRNA中新的内源性IRES。另一方面主要是因为用于测定IRES活性的翻译报告系统的局限性。在体外翻译系统中,如果没有补充额外的ITAF或者缺少细胞内必要的RNA修饰以及结合蛋白,一些IRES不具有活性或者活性很弱;如果转染到体内系统中进行检测,IRES的活性又会因为不同细胞类型中ITAF的差异而有不同的表现。不过随着高通量测序技术的发展,特别是核糖体测序技术的发展,相信会帮助我们发现翻译起始位点信息,并随着数据的积累使得预测的准确度也大幅提升。同时,IRES介导的翻译起始与癌症、心血管疾病、神经退行性疾病、肌肉萎缩和其他遗传病密切相关[20]。近年来,精准医学也得到了快速发展,基于RNA的疗法是其中潜力很大的实际应用区域,人们也对此进行研究并提出了新的治疗靶点和治疗方法,例如靶向IRES治疗法,使用RNA分子选择性的对靶点的翻译进行调控,从而更安全的让疾病得到改善;中断IRES-ITAF相互作用或改变ITAF表达水平[53]。因此,了解与IRES介导的翻译失调的相关机制,对于开发新药和促进精准医学的进展至关重要。
| [1] |
QU L, YI Z, SHEN Y, et al. Circular RNA vaccines against SARS-CoV-2 and emerging variants[J]. Cell, 2022, 185(10): 1728-1744. DOI:10.1016/j.cell.2022.03.044 (  0) 0) |
| [2] |
LIU X, ZHANG Y, ZHOU S, et al. Circular RNA: An emerging frontier in RNA therapeutic targets, RNA therapeutics, and mRNA vaccines[J]. Journal of Controlled Release: Official Journal of the Controlled Release Society, 2022, 348: 84-94. DOI:10.1016/j.jconrel.2022.05.043 (  0) 0) |
| [3] |
ZHANG J, CHEN J. Circular mRNAs: More stable RNAs lead to more persistent protein expression[J]. Molecular Therapy. Nucleic Acids, 2022, 30: 357-358. DOI:10.1016/j.omtn.2022.10.014 (  0) 0) |
| [4] |
XIA X, LI X, LI F, et al. A novel tumor suppressor protein encoded by circular AKT3 RNA inhibits glioblastoma tumorigenicity by competing with active phosphoinositide-dependent Kinase-1[J]. Molecular Cancer, 2019, 18(1): 131. DOI:10.1186/s12943-019-1056-5 (  0) 0) |
| [5] |
LIANG W C, WONG C W, LIANG P P, et al. Translation of the circular RNA circβ-catenin promotes liver cancer cell growth through activation of the Wnt pathway[J]. Genome Biology, 2019, 20(1): 84. DOI:10.1186/s13059-019-1685-4 (  0) 0) |
| [6] |
MACEJAK D G, SARNOW P. Internal initiation of translation mediated by the 5' leader of a cellular mRNA[J]. Nature, 1991, 353(6339): 90-94. DOI:10.1038/353090a0 (  0) 0) |
| [7] |
HOLCIK M, LEFEBVRE C, YEH C, et al. A new internal-ribosome-entry-site motif potentiates XIAP-mediated cytoprotection[J]. Nature Cell Biology, 1999, 1(3): 190-192. DOI:10.1038/11109 (  0) 0) |
| [8] |
COLDWELL M J, MITCHELL S A, STONELEY M, et al. Initiation of Apaf-1 translation by internal ribosome entry[J]. Oncogene, 2000, 19(7): 899-905. DOI:10.1038/sj.onc.1203407 (  0) 0) |
| [9] |
STONELEY M, CHAPPELL S A, JOPLING C L, et al. c-Myc protein synthesis is initiated from the internal ribosome entry segment during apoptosis[J]. Molecular and Cellular Biology, 2000, 20(4): 1162-1169. DOI:10.1128/MCB.20.4.1162-1169.2000 (  0) 0) |
| [10] |
WEINGARTEN-GABBAY S, ELIAS-KIRMA S, NIR R, et al. Systematic discovery of cap-independent translation sequences in human and viral genomes[J]. Science, 2016, 351(6270): 4939. DOI:10.1126/science.aad4939 (  0) 0) |
| [11] |
LEGNINI I, DI TIMOTEO G, ROSSI F, et al. Circ-ZNF609 is a circular RNA that can be translated and functions in myogenesis[J]. Molecular Cell, 2017, 66(1): 22-37. DOI:10.1016/j.molcel.2017.02.017 (  0) 0) |
| [12] |
YANG S, HUANG S, ZHANG M, et al. Novel role of FBXW7 circular RNA in repressing glioma tumorigenesis[J]. Journal of the National Cancer Institute, 2018, 110(3): 304-315. DOI:10.1093/jnci/djx166 (  0) 0) |
| [13] |
ZHANG M, HUANG N, YANG X, et al. A novel protein encoded by the circular form of the SHPRH gene suppresses glioma tumorigenesis[J]. Oncogene, 2018, 37(13): 1805-1814. DOI:10.1038/s41388-017-0019-9 (  0) 0) |
| [14] |
ZHANG M, ZHAO K, XU X, et al. A peptide encoded by circular form of LINC-PINT suppresses oncogenic transcriptional elongation in glioblastoma[J]. Nature Communications, 2018, 9(1): 4475. DOI:10.1038/s41467-018-06862-2 (  0) 0) |
| [15] |
CARBONNELLE D, VIGNARD V, SEHEDIC D, et al. The melanoma antigens MELOE-1 and MELOE-2 are translated from a bona fide polycistronic mRNA containing functional IRES sequences[J]. PloS One, 2013, 8(9): e75233. DOI:10.1371/journal.pone.0075233 (  0) 0) |
| [16] |
KING H A, COBBOLD L C, WILLIS A E. The role of IRES trans-acting factors in regulating translation initiation[J]. Biochemical Society Transactions, 2010, 38(6): 1581-1586. DOI:10.1042/BST0381581 (  0) 0) |
| [17] |
MITCHELL S A, SPRIGGS K A, COLDWELL M J, et al. The Apaf-1 internal ribosome entry segment attains the correct structural conformation for function via interactions with PTB and unr[J]. Molecular Cell, 2003, 11(3): 757-771. DOI:10.1016/s1097-2765(03)00093-5 (  0) 0) |
| [18] |
PICKERING B M, MITCHELL S A, EVANS J R, et al. Polypyrimidine tract binding protein and poly r(C) binding protein 1 interact with the BAG-1 IRES and stimulate its activity in vitro and in vivo[J]. Nucleic Acids Research, 2003, 31(2): 639-646. DOI:10.1093/nar/gkg146 (  0) 0) |
| [19] |
PICKERING B M, MITCHELL S A, SPRIGGS K A, et al. Bag-1 internal ribosome entry segment activity is promoted by structural changes mediated by poly(rc) binding protein 1 and recruitment of polypyrimidine tract binding protein 1[J]. Molecular and Cellular Biology, 2004, 24(12): 5595-5605. DOI:10.1128/MCB.24.12.5595-5605.2004 (  0) 0) |
| [20] |
GODET A C, DAVID F, HANTELYS F, et al. IRES trans-acting factors, key actors of the stress response[J]. International Journal of Molecular Sciences, 2019, 20(4): E924. DOI:10.3390/ijms20040924 (  0) 0) |
| [21] |
CHO S, KIM J H, BACK S H, et al. Polypyrimidine tract-binding protein enhances the internal ribosomal entry site-dependent translation of p27Kip1 mRNA and modulates transition from G1 to S phase[J]. Molecular and Cellular Biology, 2005, 25(4): 1283-1297. DOI:10.1128/MCB.25.4.1283-1297.2005 (  0) 0) |
| [22] |
KIM Y K, HAHM B, JANG S K. Polypyrimidine tract-binding protein inhibits translation of bip mRNA[J]. Journal of Molecular Biology, 2000, 304(2): 119-133. DOI:10.1006/jmbi.2000.4179 (  0) 0) |
| [23] |
DORMOY-RACLET V, MARKOVITS J, JACQUEMIN-SABLON A, et al. Regulation of Unr expression by 5'- and 3'-untranslated regions of its mRNA through modulation of stability and IRES mediated translation[J]. RNA Biology, 2005, 2(3): e27-e35. DOI:10.4161/rna.2.3.2203 (  0) 0) |
| [24] |
HENIS-KORENBLIT S, STRUMPF N L, GOLDSTAUB D, et al. A novel form of DAP5 protein accumulates in apoptotic cells as a result of caspase cleavage and internal ribosome entry site-mediated translation[J]. Molecular and Cellular Biology, 2000, 20(2): 496-506. DOI:10.1128/MCB.20.2.496-506.2000 (  0) 0) |
| [25] |
SHARATHCHANDRA A, LAL R, KHAN D, et al. Annexin A2 and PSF proteins interact with p53 IRES and regulate translation of p53 mRNA[J]. RNA Biology, 2012, 9(12): 1429-1439. DOI:10.4161/rna.22707 (  0) 0) |
| [26] |
TAKAGI M, ABSALON M J, MCLURE K G, et al. Regulation of p53 translation and induction after DNA damage by ribosomal protein L26 and nucleolin[J]. Cell, 2005, 123(1): 49-63. DOI:10.1016/j.cell.2005.07.034 (  0) 0) |
| [27] |
YANG Y, WANG C, ZHAO K, et al. TRMP, a p53-inducible long noncoding RNA, regulates G1/S cell cycle progression by modulating IRES-dependent p27 translation[J]. Cell Death & Disease, 2018, 9(9): 886. DOI:10.1038/s41419-018-0884-3 (  0) 0) |
| [28] |
GARANT J M, PERREAULT J P, SCOTT M S. G4RNA screener web server: User focused interface for RNA G-quadruplex prediction[J]. Biochimie, 2018, 151: 115-118. DOI:10.1016/j.biochi.2018.06.002 (  0) 0) |
| [29] |
KIKIN O, D'ANTONIO L, BAGGA P S. QGRS Mapper: A web-based server for predicting G-quadruplexes in nucleotide sequences[J]. Nucleic Acids Research, 2006, 34(Web server issue): W676-W682. DOI:10.1093/nar/gkl253 (  0) 0) |
| [30] |
LEPPEK K, DAS R, BARNA M. Functional 5' UTR mRNA structures in eukaryotic translation regulation and how to find them[J]. Nature Reviews. Molecular Cell Biology, 2018, 19(3): 158-174. DOI:10.1038/nrm.2017.103 (  0) 0) |
| [31] |
BONNAL S, SCHAEFFER C, CRÉANCIER L, et al. A single internal ribosome entry site containing a G quartet RNA structure drives fibroblast growth factor 2 gene expression at four alternative translation initiation codons[J]. The Journal of Biological Chemistry, 2003, 278(41): 39330-39336. DOI:10.1074/jbc.M305580200 (  0) 0) |
| [32] |
MORRIS M J, NEGISHI Y, PAZSINT C, et al. An RNA G-quadruplex is essential for cap-independent translation initiation in human VEGF IRES[J]. Journal of the American Chemical Society, 2010, 132(50): 17831-17839. DOI:10.1021/ja106287x (  0) 0) |
| [33] |
AL-ZEER M A, DUTKIEWICZ M, VON HACHT A, et al. Alternatively spliced variants of the 5'-UTR of the ARPC2 mRNA regulate translation by an internal ribosome entry site (IRES) harboring a guanine-quadruplex motif[J]. RNA Biology, 2019, 16(11): 1622-1632. DOI:10.1080/15476286.2019.1652524 (  0) 0) |
| [34] |
KOUKOURAKI P, DOXAKIS E. Constitutive translation of human α-synuclein is mediated by the 5'-untranslated region[J]. Open Biology, 2016, 6(4): 160022. DOI:10.1098/rsob.160022 (  0) 0) |
| [35] |
JODOIN R, CARRIER J C, RIVARD N, et al. G-quadruplex located in the 5'UTR of the BAG-1 mRNA affects both its cap-dependent and cap-independent translation through global secondary structure maintenance[J]. Nucleic Acids Research, 2019, 47(19): 10247-10266. DOI:10.1093/nar/gkz777 (  0) 0) |
| [36] |
JODOIN R, PERREAULT J P. G-quadruplexes formation in the 5'UTRs of mRNAs associated with colorectal cancer pathways[J]. PLoS One, 2018, 13(12): e0208363. DOI:10.1371/journal.pone.0208363 (  0) 0) |
| [37] |
YAMAN I, FERNANDEZ J, LIU H, et al. The zipper model of translational control: A small upstream ORF is the switch that controls structural remodeling of an mRNA leader[J]. Cell, 2003, 113(4): 519-531. DOI:10.1016/s0092-8674(03)00345-3 (  0) 0) |
| [38] |
CHEN T M, SHIH Y H, TSENG J T, et al. Overexpression of FGF9 in colon cancer cells is mediated by hypoxia-induced translational activation[J]. Nucleic Acids Research, 2014, 42(5): 2932-2944. DOI:10.1093/nar/gkt1286 (  0) 0) |
| [39] |
BASTIDE A, KARAA Z, BORNES S, et al. An upstream open reading frame within an IRES controls expression of a specific VEGF-A isoform[J]. Nucleic Acids Research, 2008, 36(7): 2434-2445. DOI:10.1093/nar/gkn093 (  0) 0) |
| [40] |
WU T Y, HSIEH C C, HONG J J, et al. IRSS: A web-based tool for automatic layout and analysis of IRES secondary structure prediction and searching system in silico[J]. BMC Bioinformatics, 2009, 10: 160. DOI:10.1186/1471-2105-10-160 (  0) 0) |
| [41] |
HONG J J, WU T Y, CHANG T Y, et al. Viral IRES prediction system - A web server for prediction of the IRES secondary structure in silico[J]. PLoS One, 2013, 8(11): e79288. DOI:10.1371/journal.pone.0079288 (  0) 0) |
| [42] |
KOLEKAR P, PATASKAR A, KULKARNI-KALE U, et al. IRESPred: Web server for prediction of cellular and viral internal ribosome entry site (IRES)[J]. Scientific Reports, 2016, 6: 27436. DOI:10.1038/srep27436 (  0) 0) |
| [43] |
EISENBERG E, LEVANON E Y. Human housekeeping genes, revisited[J]. Trends in Genetics: TIG, 2013, 29(10): 569-574. DOI:10.1016/j.tig.2013.05.010 (  0) 0) |
| [44] |
ZHAO J, WU J, XU T, et al. IRESfinder: Identifying RNA internal ribosome entry site in eukaryotic cell using framed k-mer features[J]. Journal of Genetics and Genomics, 2018, 45(7): 403-406. DOI:10.1016/j.jgg.2018.07.006 (  0) 0) |
| [45] |
WANG J, GRIBSKOV M. IRESpy: An XGBoost model for prediction of internal ribosome entry sites[J]. BMC Bioinformatics, 2019, 20(1): 409. DOI:10.1186/s12859-019-2999-7 (  0) 0) |
| [46] |
KOMAR A A, MAZUMDER B, MERRICK W C. A new framework for understanding IRES-mediated translation[J]. Gene, 2012, 502(2): 75-86. DOI:10.1016/j.gene.2012.04.039 (  0) 0) |
| [47] |
BONNAL S, BOUTONNET C, PRADO-LOURENÇO L, et al. IRESdb: The internal ribosome entry site database[J]. Nucleic Acids Research, 2003, 31(1): 427-428. DOI:10.1093/nar/gkg003 (  0) 0) |
| [48] |
MOKREJS M, MASEK T, VOPÁLENSKY V, et al. IRESite--A tool for the examination of viral and cellular internal ribosome entry sites[J]. Nucleic Acids Research, 2010, 38(Database issue): D131-D136. DOI:10.1093/nar/gkp981 (  0) 0) |
| [49] |
ZHAO J, LI Y, WANG C, et al. IRESbase: A comprehensive database of experimentally validated internal ribosome entry sites[J]. Genomics Proteomics Bioinformatics, 2020, 18(2): 129-139. DOI:10.1016/j.gpb.2020.03.001 (  0) 0) |
| [50] |
YANG T H, WANG C Y, TSAI H C, et al. Human IRES Atlas: An integrative platform for studying IRES-driven translational regulation in humans[J]. Database, 2021, 2021: baab025. DOI:10.1093/database/baab025 (  0) 0) |
| [51] |
MOKREJS M, VOPÁ LENSKỲV, KOLENATY O, et al. IRESite: The database of experimentally verified IRES structures (www.iresite.org)[J]. Nucleic Acids Research, 2006, 34(Database issue): D125-D130. DOI:10.1093/nar/gkj081 (  0) 0) |
| [52] |
GRILLO G, LICCIULLI F, LIUNI S, et al. PatSearch: A program for the detection of patterns and structural motifs in nucleotide sequences[J]. Nucleic Acids Research, 2003, 31(13): 3608-3612. DOI:10.1093/nar/gkg548 (  0) 0) |
| [53] |
MARQUES R, LACERDA R, ROMÃO L. Internal ribosome entry site (ires)-mediated translation and its potential for novel mrna-based therapy development[J]. Biomedicines, 2022, 10(8): 1865. DOI:10.3390/biomedicines10081865 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


