2. 中国农业大学 资源与环境学院,北京 100193
2. College of Resources and Environmental Sciences, China Agricultural University, Beijing 100193, China
微囊藻毒素(Microcystins, MCs)是蓝藻中出现频率最广和危害最大的一种单环七肽肝毒素[1],分子量在1 000左右,一般结构见图 1,其中A、B是可变氨基酸,Adda的分子式为3-氨基-9-甲氧基-2, 6, 8-三甲基-10-苯基-4, 6-二烯酸,是MCs潜在的毒性基团[2]。由于A、B的可变性以及其它氨基酸甲基化与去甲基化的差异,使得MCs具有多种亚型[3-4],到2017年已发现240余种[5-6],其中MC-LR、-RR、-YR(L,R,Y分别代表亮氨酸、精氨酸、酪氨酸)在天然水体中出现频率最高,MC-LR毒性最大,其次为-RR,-YR[7]。MCs主要作用器官是肝脏,对生态系统及人类健康存在极大威胁。生物降解去除MCs因其安全、高效、成本低、无污染等特点引起了国内外的广泛研究。
MCs的生物降解是在蛋白酶催化作用下完成的,目前的研究主要集中在基因功能方面。早期利用分子生物学手段研究发现,MCs降解菌Sphingomonas sp. ACM-3962 (MJ-PV) 在降解MC-LR的过程中,mlr基因簇发挥了重要作用,其中mlrA表达的水解酶(MlrA)通过断裂Adda-Arg键将环状MC-LR水解为线性,随后mlrB表达的酶(MlrB)将线性MC-LR的Ala-Leu键断裂,使其进一步裂解产生四肽,最终四肽在mlrC表达的酶(MlrC)的催化作用下裂解为更小片段[8-9]。后续通过异源表达的方式在其他降解菌中证实了mlrA、mlrB、mlrC的功能,同时表明MlrC能够直接降解线性MC-LR和-RR而无需MlrB的作用[10-12]。最初的研究表明,mlrD和mlrA组成一个操纵子,并推论mlrD基因能够编码一种转运蛋白[8],但到目前为止并没有进一步研究mlrD基因的功能。本课题组曾试图通过基因敲除的方式研究野生菌Novosphingobium sp. THN1中mlrD基因的功能,但因同源双交换难以发生以及敲除后的菌株很快死亡,导致后续功能验证无法继续进行。通过生物信息学的手段探究MlrD结构特征,预测其活性位点,可为进一步研究其功能奠定基础。
对蛋白质序列进行分析有助于了解其结构和功能,进而为研究生物学功能提供关键信息[13]。Bourne等在发现mlr基因簇的同时,对ACM-3962降解酶Mlr蛋白的性质进行了分析[8],表明MlrA包括336个氨基酸残基,分子量为36.6 kD,预测等电点为9.76,存在信号肽,裂解位置在第26和27个氨基酸之间,之后通过序列比对预测并确认了其活性位点H260AIHNE265[14]。Xu等构建了MlrA的三级结构,并通过分子对接和定点突变验证了其他活性位点(Glu172、Trp176、His205、Trp201)[15]。Bourne等[8, 16]对MlrC蛋白序列进行分析,发现并证实了活性位点Asp167、His169、His191。Wang等[17]通过定点突变确定了其他几个活性位点,同时结合MlrC的三级结构进一步分析了活性位点在空间上的分布,推测了其活性口袋。
早期研究表明MlrD包含422个氨基酸残基,分子量为45.4 kD,预测等电点为9.83,有12个跨膜结构域,序列相似性结果显示MlrD属于PTR2超家族[8]。MCs降解酶MlrA、MlrB和MlrC的功能已经被证实,但被认为是转运蛋白的MlrD的功能及结构依然是未知的。近年来,随着基因组学和蛋白质组学的发展,数以万计的蛋白质三维结构尚待分析,但蛋白质结构的确定非常困难[18]。随着计算机技术的发展和已知三维结构蛋白的增多,对未知结构蛋白的预测成为可能。平均50%~70%的典型基因组可以使用计算技术进行结构建模[19]。预测蛋白质结构将有助于探索其活性位点及作用方式,进一步多角度了解MCs的降解。本研究在前人研究的基础上,全面的分析mlrD的序列特征,构建其三级结构,预测活性位点,为进一步研究mlrD的功能及结构提供理论依据。
1 数据来源及研究方法 1.1 MlrD注释及其蛋白序列特征分析通过在NCBI数据库中检索并下载降解菌的ACM-3962的MlrD(MlrD-ACM-3962)蛋白序列,分别利用在线软件Cell-PLoc 2.0和Protscale分析该蛋白亚细胞定位和亲/疏水性。利用STRING数据库对蛋白相互作用关系进行分析。
在NCBI数据库搜索并下载MCs降解菌的MlrD及16S rDNA序列,同时下载外围基因的相应序列。用clusterW[20]进行多序列比对,MEGA7.0构建系统发育树,对mlrD亲缘关系和进化关系进行分析。
1.2 MlrD蛋白三级结构分析利用HMMTOP工具分析MlrD二级结构,在PDB数据库中检索与MlrD-ACM-3962高度相似的蛋白,筛选模板蛋白并下载其三级结构,选取easymodeller 4.0(Modeller 9.20)[21]软件建模。首先将模板蛋白导入软件,通过模板比对、模板与目的蛋白比对等过程,构建9个模型。构建好的模型用Ramachandran图和Verify-3D [22-23]程序进行评估。评分都通过的模型作为最终的蛋白结构模型,并在PyMOL2中分析展示。
1.3 MlrD蛋白活性位点分析在上述结构分析的基础上,进一步分析MlrD的保守结构,通过同一蛋白家族已有蛋白活性位点的分析,推测MlrD的活性位点。
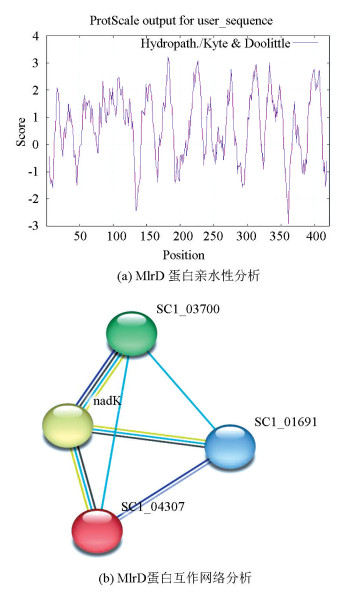
2 结果与分析 2.1 MlrD-ACM-3962蛋白基本性质分析对ACM-3962的MlrD蛋白的一级结构进行ProtScale分析,该多肽链的第182位异亮氨酸(Ile)和第361位的半胱氨酸(Cys)分别具有最高分值(1.500)和最低分(-2.867),即前者疏水性最强,后者亲水性最强(见图 2a)。利用ProtParam分析可知,氨基酸序列平均疏水指数为0.677,即该蛋白为疏水性蛋白。使用Cell-PLoc 2.0在线网站分析目标蛋白亚细胞定位,结果表明此蛋白质位于细胞内膜上。
|
图 2 MlrD蛋白亲水性及互作网络分析 Figure 2 Analysis of hydrophilicity and interaction network of MlrD protein |
蛋白互作网络中的SCI_04307为MlrD蛋白序列,SCI_03700、nadK和SCI_01691与MlrD相互作用(见图 2b),其中nadK得分最高说明其与MlrD蛋白相互作用更紧密。nadK参与调节NAD和NADP的胞内平衡,是NADP生物合成的关键酶,特异性催化NAD腺苷部分2'-羟基的磷酸化生成NADP;SCI_03700是NADH焦磷酸酶,属于Nudix水解酶家族;SCI_01691为二肽/三肽转运酶。根据互作网络推测,MlrD可能参与了NADH的合成,MCs的转运可能是在MlrD与SCI_01691的协同作用下完成的。
2.2 MlrD的同源性分析在ACM-3962菌株中首次鉴定出mlrD基因后,后续研究根据已经发现的序列,通过PCR或基因组测序,在其他降解菌中也发现了mlrD基因序列。本文通过在NCBI数据库中检索MCs降解菌的mlrD的基因序列,对降解菌及MlrD蛋白注释进行了总结(见表 1)。目前分离到的携带mlrD基因的降解菌共有17株,都属于α变形菌门,其中鞘氨醇盒菌属(Sphingopyxis sp.)有9株,占有最高的比例。由于有些菌株只测了mlrD基因的部分序列,所以在序列长度方面差异比较大。对mlrD的基因注释并不统一,可以分为三类,第一类(8株)注释为微囊藻毒素降解酶(包括THN1菌株中较为模糊地注释为参与了微囊藻毒素的降解);第二类注释为肽转运蛋白,其中6株为寡肽转运蛋白,1株(Novosphingobium sp. MD-1菌株)注释为二肽/三肽转运蛋白,1株(Sphingosinicella sp. strain JEZ-8L菌株)较为确定的注释为微囊藻毒素转运蛋白;第三类未进行注释(菌株Sphingopyxis sp. LH21和Sphingopyxis sp. C-1)。
| 表 1 MCs降解菌中MlrD的特征 Table 1 Characteristics of MlrD in diverse MCs-degradation bacteria |
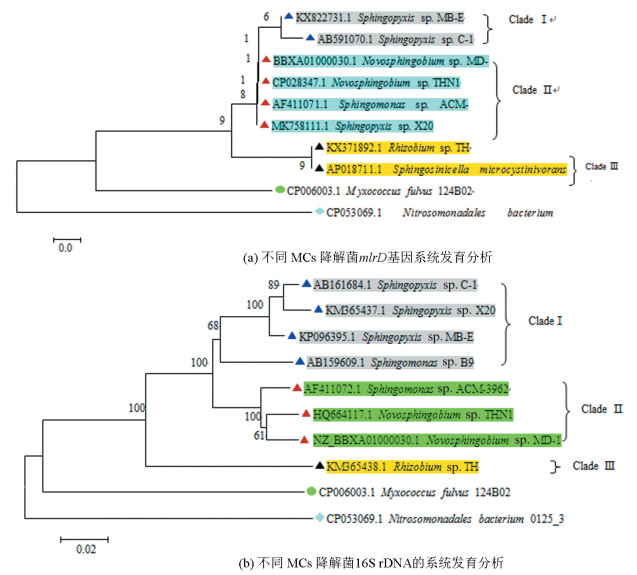
为进一步分析不同菌株之间MlrD的保守性,用clustal W进行了多序列比对,结果表明序列较短的MlrD是长序列中的一部分,不同菌株之间的MlrD是保守的。采用MEGA7.0 Neighbor-Joining法对mlrD基因及相应菌株的16S rDNA序列构建系统发育树,选取Myxococcus fulvus 124B02和Nitrosomonadales bacterium 0125_3作为外围基因,在mlr D基因发育树中,降解菌形成了三个分支。除Sphingopyxis sp.X20以外,Sphingopyxis属的降解菌形成了一个独立的分支(Clade Ⅰ),Sphingomonas属和Novosphingobium属的降解菌形成了一个分支(Clade Ⅱ)(见图 3a),这两个分支具有较近的亲缘关系,Sphingosinicella属和Rhizobium属形成了另一个分支(Clade Ⅲ)。16S rDNA基因发育树的结构(见图 3b)与mlrD基因相同,也形成了三个独立的分支,除Sphingopyxis sp.X20和Sphingosinicella microcystinivorans B9的分支位置不一致外,其余菌株分支与mlr D基因完全相同。
|
图 3 不同MCs降解菌系统发育分析 Figure 3 Phylogenetic analysis of various MCs-degrading bacteria |
在Bourne等[8]对MlrD-ACM-3962初步分析的基础上,构建了其三级结构模型。将MlrD-ACM-3962序列与PDB数据库中蛋白序列进行比较,根据比对的结果,发现与MlrD相似的蛋白较多,相似率在30%左右,大部分属于肽转运蛋白,充分利用较多的相似蛋白,采用Modeller多模板建模预测其三级结构。Modeller同源建模使用多个模板可以组合来自各个模板结构的信息,通常会提高模型的准确性[21]。
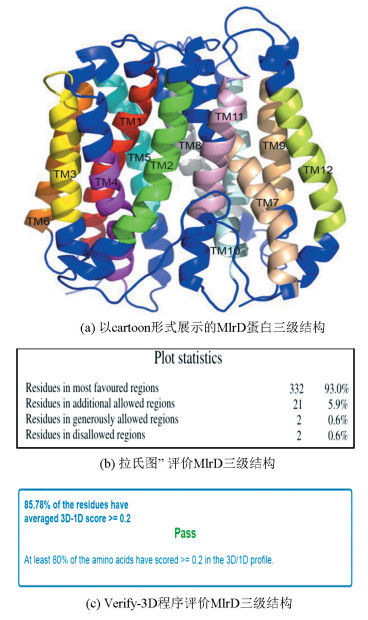
根据相似性,以4ikxa、4ikva、4q65a、4oh3a、4xnia、4apsa、5mmta、4lepa、4w6va、6exsa、2xuta、6ei3a、5a2na蛋白为模板,通过模板蛋白互相比对、与目的蛋白比对、主链生成、loop区建模、模型优化[32],用Modeller构建9个模型。9个模型分别用Ramachandran图和Verify-3D评分进行评价,Ranmachandran图在最允许范围内氨基酸所占比例达到90%以上,被认为是一个高质量的模型[33],Verify-3D分析原子模型与自身序列在位置和环境上的兼容性,如果80%的氨基酸残基得分大于等于0.2,可认为模型是合理的[34]。经过比较,选取评分较高的模型(见图 4a),从Ramachandran图统计结果(见图 4b)可以看出,模型中93.0%的氨基酸在最允许区域内,5.9%在额外允许区域。Verify-3D评估结果表明85.78%的残基3D-1D评分大于等于0.2(见图 4c),即相容性是通过的。综上MlrD的三维结构具有一定的理论意义和参考价值。
|
图 4 MlrD蛋白三级结构及其评价 Figure 4 The 3D structure of MlrD and its evaluation |
MlrD由422个氨基酸组成,HMMTOP二级结构预测表明由12个跨膜α-螺旋和无规则卷曲组成,没有β-折叠结构。在12个跨膜α-螺旋中,前6个与后6个跨膜α-螺旋结构域呈现出非常相似的拓扑结构,以一种假二次轴对称的方式存在,对称轴垂直于膜平面
2.4 MlrD蛋白功能及活性位点分析经过保守结构域的查找,发现MlrD蛋白属于质子依赖型寡肽转运蛋白PTR2(Peptide transport)超家族,这个家族的蛋白属于二肽/三肽渗透酶,参与氨基酸运输与代谢。
PTR2家族有两个保守基序(Motif)序列,分别是位于第2和3跨膜螺旋连接处的GXXXADXXXGKXXTI(X代表任意氨基酸)和位于第5个跨膜螺旋上的FYXXINXG(X代表任意氨基酸),第二个保守motif在肽转运家族的所有成员中都表现出绝对的保守性[35-36]。在MlrD序列中这两个motif与PTR2具有一些差异(见图 5),第1个保守序列位于第2和3跨膜螺旋连接处,位于MlrD-ACM-3962的69-83 bp,氨基酸序列为GGWIADRFIGRSAAI,即上述保守序列中的K和T分别变成了R和A;第2个保守序列在MlrD中不存在,比较相似的序列位于第5个跨膜螺旋上142 -150 bp,氨基酸序列为FYYLAVSAG。这两个序列中个别氨基酸的差异可能是由于底物的差异性造成的,这些序列在不同MCs降解菌之间是保守的(见图 5)。综上所述,位于第2和3跨膜螺旋连接处的Gly69、Ala73、Asp74、Gly78、Arg79、Ala82、Ile83和位于第五个跨膜螺旋上的Phe142、Tyr143、Ala146、Val147、Gly150可能为MlrD的活性位点。

|
图 5 微囊藻毒素降解菌MlrD氨基酸序列的比对 Figure 5 Alignment of MlrD sequence of MCs-degrading bacteria |
到目前为止,MlrD的功能并未通过实验证实,对其基因注释大多是根据序列保守性分析或依据前人的经验得到的,导致注释存在差异。
系统发育树分析表明mlrD和16S rDNA具有相同的拓扑结构。Qin等[25]的研究表明其他mlr基因构建的系统发育树都形成了三个主要分支,而且整体结构相同,表明mlrD基因与其他mlr基因具有相同的进化来源,整个mlr基因簇共同进行进化。经过多序列比对及系统发育树分析,表明MlrD在MCs降解菌之间是高度保守的,MlrD具有功能特异性。
Dziga等通过异源表达的方式研究MlrA、MlrB、MlrC的功能[10, 11, 14, 16-17, 37],表明在没有mlrD基因的情况下,菌株能够降解MCs及其产物,说明在异源表达菌株中mlrD基因对于MCs及其产物的转运和降解不是必需的,然而在异源表达菌株中的情况并不适应于野生菌。利用同源双交换敲除基因是研究野生菌基因功能的常用方法之一,但在对mlrD进行敲除时,因二次同源双交换难以发生,双交换菌株不稳定等原因,基因敲除没有成功,表明MlrD是菌株生长所必需的,大片段的缺失会导致菌株的死亡。通过生物信息学的方法预测MlrD活性位点,进一步采用活性位点碱基突变、敲除等方法,可在野生菌中进一步研究MlrD的功能。
保守结构域分析表明MlrD属于PTR2超家族,在膜转运蛋白分类数据库(TCDB)中,PTR是位于主要协助转运蛋白超家族(MFS)内的一个蛋白家族。MFS家族的蛋白大多数都含有400-600个氨基酸残基,大多数拥有12个跨膜α-螺旋[38],N端和C端分别由6个跨膜α螺旋组成,呈现出非常相似的拓扑结构,以假二次轴对称的方式存在[39-40]。MlrD的结构完全符合MFS家族的特性。MlrD的结构与上述特征相符合,从三级结构的角度说明MlrD属于MFS超家族。
MFS家族的蛋白都是次级转运蛋白的典型代表,利用膜内外底物浓度差产生的电化学势能转运底物[41-42],MCs及其产物可能通过浓度差在MlrD的作用下进出细胞。有研究表明,突变MFS上述两个保守序列会导致转运活性丧失[43-44],因此后续研究可以通过定点突变的或小片段基因敲除方法,突变或敲除GGWIADRFIGRSAAI和FYYLAVSAG这两个区域,深度了解MCs及其降解产物的转运。
4 结论MCs降解菌的mlrD和16S rDNA系统发育树有相同的拓扑结构,MlrD蛋白属于PTR2超家族,其二级结构由α-螺旋和无规则卷曲结构组成,且完全符合PTR2家族的特性,可能的活性位点为位于第2和3跨膜螺旋连接处的Gly69、Ala73、Asp74、Gly78、Arg79、Ala82、Ile83和位于第5个跨膜螺旋上的Phe142、Tyr143、Ala146、Val147、Gly150,可以通过突变或敲除这两个结构域来研究其功能。
| [1] |
徐慧敏, 闫海, 马松, 等. 鞘氨醇单胞菌USTB-05对微囊藻毒素的生物降解[J]. 中国环境科学, 2014, 34(5): 1316-1321. XU Huimin, YAN Hai, MA Song, et al. Biodegradation of microcystins by Sphingopyxis sp. USTB-05[J]. China Environmental Science, 2014, 34(5): 1316-1321. DOI:10.3969/j.issn.1000-6923.2014.05.044 (  0) 0) |
| [2] |
孔赟, 徐向阳, 朱亮, 等. 环境水体微囊藻毒素微生物降解技术研究进展[J]. 应用生态学报, 2011, 22(6): 1646-1652. DOI: CNKI:SUN:YYSB.0.2011-06-039. KONG Yun, XU Xiangyang, ZHU Liang, et al. Microbial degradation of microcystins in water environment: A review[J]. Chinese Journal of Applied Ecology, 2011, 22(6): 1646-1652. DOI: CNKI:SUN:YYSB.0.2011-06-039. (  0) 0) |
| [3] |
卫玮, 肖雯. 藻毒素的类型危害和防治[J]. 生物学通报, 2004, 39(8): 21-23. WEI Wei, XIAO Wen. Types, hazards and control of algal toxins[J]. Bulletin of Biology, 2004, 39(8): 21-23. DOI:10.3969/j.issn.0006-3193.2004.08.010 (  0) 0) |
| [4] |
HITZFELD B C, HOGER S J, DIETRICH D R. Cyanobacterial toxins: Removal during drinking water treatment, and human risk assessment[J]. Environmental Health Perspectives Supplements, 2000, 108(Suppl 1): 113-122. DOI:10.1289/ehp.00108s1113 (  0) 0) |
| [5] |
SVIRCEV Z, DROBAC D, TOKODI N, et al. Toxicology of microcystins with reference to cases of human intoxications and epidemiological investigations of exposures to cyanobacteria and cyanotoxins[J]. Archives of Toxicology, 2017, 91(2): 621-650. DOI:10.1007/s00204-016-1921-6 (  0) 0) |
| [6] |
MERILUOTO J, SPOOF L, CODD G A. A tables of microcystins and nodularins. In: Handbook of cyanobacterial monitoring and cyanotoxin analysis[M]. Hoboken, NJ, USA: Meriluoto, J., Spoof, L., Codd, G. A, 2017.
(  0) 0) |
| [7] |
张维昊, 徐小清, 丘昌强. 水环境中微囊藻毒素研究进展[J]. 环境科学研究, 2001, 14(2): 57-61. ZHANG WeiHao, XU Xiaoqing, QIU Changqiang. Advance in study on Microcystins in Aquatic Environment[J]. Research of Environmental Sciences, 2001, 14(2): 57-61. DOI:10.3321/j.issn:1001-6929.2001.02.017 (  0) 0) |
| [8] |
BOURNE D G, RIDDLES P, JONES G J, et al. Characterisation of a gene cluster involved in bacterial degradation of the cyanobacterial toxin microcystin LR[J]. Environmental Toxicology, 2001, 16(6): 523-534. DOI:10.1002/tox.10013 (  0) 0) |
| [9] |
BOURNE D G, JONES G J, BLAKELEY R L, et al. Enzymatic pathway for the bacterial degradation of the cyanobacterial cyclic peptide toxin microcystin LR[J]. Applied & Environmental Microbiology, 1996, 62(11): 4086-4094. DOI:10.1016/S0027-5107(96)00126-1 (  0) 0) |
| [10] |
DZIGA D, WASYLEWSKI M, SZETELA A, et al. Verification of the role of MlrC in microcystin biodegradation by studies using a heterologously expressed enzyme[J]. Chemical Research in Toxicology, 2012, 25(6): 1192-1194. DOI:10.1021/tx300174e (  0) 0) |
| [11] |
SHIMIZU K, MASEDA H, OKANO K, et al. Enzymatic pathway for biodegrade-ing microcystin LR in Sphingopyxis sp. C-1[J]. Journal of Bioscience and Bioengineering, 2012, 114(6): 630-634. DOI:10.1016/j.jbiosc.2012.07.004 (  0) 0) |
| [12] |
WANG Huasheng, YAN Hai, MA Song, et al. Characterization of the second and third steps in the enzymatic pathway for microcystin-RR biodegradation by Sphingopyxis sp. USTB-05[J]. Annals of Microbiology, 2014, 65(1): 495-502. DOI:10.1007/s13213-014-0885-0 (  0) 0) |
| [13] |
LEWIS T E, SILLITOE I, ANDREEVA A, et al. Genome3D: A UK collaborative project to annotate genomic sequences with predicted 3D structures based on SCOP and CATH domains[J]. Nucleic Acids Research, 2013, 41: D499-D507. DOI:10.1093/nar/gks1266 (  0) 0) |
| [14] |
DZIGA D, WLADYKA B, ZIELINSKA G, et al. Heterologous expression and characterisation of microcystinase[J]. Toxicon, 2012, 59(5): 578-586. DOI:10.1016/j.toxicon.2012.01.001 (  0) 0) |
| [15] |
XU Qianqian, FAN Jinhui, YAN Hai, et al. Structural basis of microcystinase activity for biodegrading microcystin-LR[J]. Chemosphere, 2019, 236: 124281. DOI:10.1016/j.chemosphere.2019.07.012 (  0) 0) |
| [16] |
DZIGA D, ZIELINSKA G, WLADYKA B, et al. Characterization of enzymatic activity of MlrB and MlrC proteins involved in bacterial degradation of cyanotoxins microcystins[J]. Toxins, 2016, 8(76): 1-13. DOI:10.3390/toxins8030076 (  0) 0) |
| [17] |
WANG Ruiping, LI Jieming, LI Ji, et al. Functional and structural analyses for MlrC enzyme of Novosphingobium sp. THN1 in microcystin-biodegradation: Involving optimized heterologous expression, bioinformatics and site-directed mutagenesis[J]. Chemosphere, 2020, 255: 126906. DOI:10.1016/j.chemosphere.2020.126906 (  0) 0) |
| [18] |
SCHWEDE T, KOPP J, GUEX N, et al. SWISS-MODEL: an automated protein homology-modeling server[J]. Nucleic Acids Research, 2003, 31(13): 3381-3385. DOI:10.1093/nar/gkg520 (  0) 0) |
| [19] |
MUKHERJEE S, SZILAGYI A, ROY A, et al. Genome-wide protein structure prediction, in multiscale approaches to protein modeling: Structure prediction, dynamics, thermodynamics and macromolecular assemblies[M]. Springer New York: Kolinski, Editor, 2011.
(  0) 0) |
| [20] |
LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948. DOI:10.1093/bioinformatics/btm404 (  0) 0) |
| [21] |
WEBB B, SALI A. Comparative protein structure modeling using modeller[J]. Current Protocols in Protein Science, 2016, 86(1): 2.9.1-2.9.37. DOI:10.1002/cpps.20 (  0) 0) |
| [22] |
BOWIE J U, LUTHY R F, EISENBERG D. A method to identify protein sequences that fold into a known three-dimensional structure[J]. Science, 1991, 253(5016): 164-170. DOI:10.1126/science.1853201 (  0) 0) |
| [23] |
LUTHY R, BOWIE J F, EISENBERG D. Assessment of protein models with three-dimensional profiles[J]. Nature, 1992, 356(6364): 83-85. DOI:10.1038/356083a0 (  0) 0) |
| [24] |
LEZCANO M A, MORON-LOPEZ J, AGHA R, et al. Presence or absence of mlr genes and nutrient concentrations co-determine the microcystin biodegradation efficiency of a natural bacterial Community[J]. Toxins (Basel), 2016, 8(318): 1-17. DOI:10.3390/toxins8110318 (  0) 0) |
| [25] |
QIN Lian, ZHANG Xiaoxing, CHEN Xiaoguo, et al. Isolation of a novel microcystin-degrading bacterium and the evolutionary origin of mlr gene cluster[J]. Toxins (Basel), 2019, 11(5): 269. DOI:10.3390/toxins11050269 (  0) 0) |
| [26] |
ZHU Xiaoyun, SHEN Yitian, CHEN Xiaoguo, et al. Biodegradation mechanism of microcystin-LR by a novel isolate of Rhizobium sp. TH and the evolutionary origin of the mlrA gene[J]. International Biodeterioration & Biodegradation, 2016, 115: 17-25. DOI:10.1016/j.ibiod.2016.07.011 (  0) 0) |
| [27] |
JIANG Yongguang, SHAO Jihai, WU Xingqiang, et al. Active and silent members in the mlr gene cluster of a microcystin-degrading bacterium isolated from Lake Taihu, China[J]. FEMS Microbiology Letters, 2011, 322(2): 108-114. DOI:10.1111/j.1574-6968.2011.02337.x (  0) 0) |
| [28] |
HO L, HOEFEL D, SAINT C P, et al. Isolation and identification of a novel microcystin-degrading bacterium from a biological sand filter[J]. Water Research, 2007, 41(20): 4685-4695. DOI:10.1016/j.watres.2007.06.057 (  0) 0) |
| [29] |
MAGHSOUDI E, FORTIN N, GREER C, et al. Cyanotoxin degradation activity and mlr gene expression profiles of a Sphingopyxis sp. isolated from Lake Champlain, Canada[J]. Environmental Science Processes & Impacts, 2016, 18(11): 1417-1426. DOI:10.1039/c6em00001k (  0) 0) |
| [30] |
谢维, 吴涓, 李玉成, 等. 一株微囊藻毒素降解菌的筛选及鉴定[J]. 生物学杂志, 2012, 29(6): 35-38. XEI Wei, WU Juan, LI Yucheng, et al. Screening and identification of microcystins degrading bacterial strain[J]. Journal of Biology, 2012, 29(6): 35-38. DOI:10.3969/j.issn.2095-1736.2012.06.035 (  0) 0) |
| [31] |
SOMDEE T, THUNDERS M, RUCK J, et al. Degradation of[Dha(7)]MC-LR by a microcystin degrading bacterium isolated from lake Rotoiti, New Zealand[J]. ISRN Microbiology, 2013, 2013: 596429. DOI:10.1155/2013/596429 (  0) 0) |
| [32] |
FISER A, SALI A. Modeller: Generation and refinement of homology-based protein structure models[J]. Methods in Enzymology, 2003, 374(374): 461. DOI:10.1016/s0076-6879(03)74020-8 (  0) 0) |
| [33] |
RAMACHANDRAN G N, RAMAKRISHNAN C, SASISEKHARAN V. Stereochemistry of polypeptide chain configurations[J]. Journal of Molecular Biology, 1963, 7(1): 95-99. DOI:10.1016/S0022-2836(63)80023-6 (  0) 0) |
| [34] |
LUTHY R, BOWIE J U, EISENBERG D. Assessment of protein models with three-dimensional profiles[J]. Methods in Enzymology, 1997, 277: 396-404. DOI:10.1016/S0076-6879(97)77022-8 (  0) 0) |
| [35] |
PAULSEN I T, SKURRAY R A. The POT family of transport proteins[J]. Trends in Biochemical Sciences, 1994, 19(10): 404. DOI:10.1016/0968-0004(94)90087-6 (  0) 0) |
| [36] |
DANIEL H, SPANIER B, KOTTRA G, et al. From bacteria to man: Archaic proton-dependent peptide transporters at work[J]. Physiology, 2006, 21: 93-102. DOI:10.1152/physiol.00054.2005 (  0) 0) |
| [37] |
WANG Ruiping, LI Jieming, JIANG Yongguang, et al. Heterologous expression of mlrA gene originated from Novosphingobium sp. THN1 to degrade microcystin-RR and identify the first step involved in degradation pathway[J]. Chemosphere, 2017, 184: 159-167. DOI:10.1016/j.chemosphere.2017.05.086 (  0) 0) |
| [38] |
SAIER M H. Genome archeology leading to the characterization and classification of transport proteins[J]. Current Opinion in Microbiology, 1999, 2(5): 555-561. DOI:10.1016/S1369-5274(99)00016-8 (  0) 0) |
| [39] |
孙林峰, 王佳伟, 颜宁. MFS超家族转运蛋白结构与分子机制的研究[J]. 生命科学, 2011, 23(11): 1052-1056. SUN Linfeng, WANG Jiawei, YAN Ning. Molecular mechanisms of the major facilitator superfamily transporters[J]. Chinese Bulletin of Life Sciences, 2011, 23(11): 1052-1056. (  0) 0) |
| [40] |
张伟欣, 李春阳, 陈秀兰. 细菌肽转运蛋白的研究进展[J]. 微生物学通报, 2014, 41(9): 1856-1863. ZHANG Weixin, LI Chunyang, CHEN Xiulan. Advances in bacterial peptide transporters[J]. Microbiology China, 2014, 41(9): 1856-1863. DOI:10.13344/j.microbiol.china.130787 (  0) 0) |
| [41] |
MALONEY P C. The molecular and cell biology of anion transport by bacteria[J]. Bioessays, 1992, 14(11): 757-762. DOI:10.1002/bies.950141106 (  0) 0) |
| [42] |
MALONEY P C. Microbes and membrane biology[J]. FEMS Microbiology Reviews, 1990, 87: 91-102. DOI:10.1016/0378-1097(90)90699-Q (  0) 0) |
| [43] |
HAUSER M, KAUFFMAN S, NAIDER F, et al. Substrate preference is altered by mutations in the fifth transmembrane domain of Ptr2p, the di/tri-peptide transporter of Saccharomyces cerevisiae[J]. Molecular Membrane Biology, 2005, 22(3): 215-227. DOI:10.1080/09687860500093248 (  0) 0) |
| [44] |
FEI Y J, LIU W, PRASAD P D, et al. Identification of the histidyl residue obligatory for the catalytic activity of the human H+/peptide cotransporters PEPT1 and PEPT2[J]. Biochemistry, 1997, 36(2): 452-460. DOI:10.1021/bi962058p (  0) 0) |
 2023, Vol. 21
2023, Vol. 21



