乳腺癌是当前全球范围内最常见的恶性肿瘤,发病率仍呈上升趋势。作为一种高度异质性的疾病,基于组织病理、基因表达或基因组-转录组数据有多种分类方法,临床工作中常根据雌激素受体(Estrogen receptor,ER)、孕激素受体(Progesterone receptor,PR)和人表皮生长因子受体2(Human epidermal growth factor receptor 2,HER2)的表达将其分为管腔型、HER2过表达型和三阴性乳腺癌[1]。不同分子亚型的乳腺癌具有独特的临床表现、组织病理学特征、治疗反应以及预后,深入了解其内在机制的差异对改进分类方法和发现潜在治疗靶点至关重要。微小RNA(microRNA,miRNA)是一类内源性非编码RNA,主要通过抑制翻译或降解靶mRNA调控基因表达,在癌症发病机制中发挥致癌或抑癌作用[2]。部分miRNA在乳腺癌中的表达存在异质性,与特定的分子亚型相关联,可用于肿瘤的风险判断和精准分类[3]。研究显示miR-449a在乳腺癌、宫颈癌、肺癌、鼻窦腺癌、结直肠癌等多种恶性肿瘤中异常表达,参与调控肿瘤增殖、迁移、侵袭等过程,但对其在不同亚型乳腺癌的作用了解有限[4-6]。本研究通过检测miR-449a在乳腺癌组织中的表达,分析其与临床病理特征的关系,并采用生物信息学方法探索其在肿瘤发生发展过程中可能的作用机制,以期为乳腺癌的研究提供一定的参考依据。
1 资料与方法 1.1 组织标本和临床资料选取本院2017年至2021年行手术切除的83例乳腺癌患者作为研究对象。纳入标准:①首诊患者,经病理组织学确诊;②临床病理资料完整,明确ER、PR和HER2蛋白的表达状态;③病灶无明显出血坏死区。排除标准:①术前接受过新辅助治疗;②合并其他器官恶性肿瘤。本研究经医院伦理委员会批准,患者或家属签署知情同意书。患者发病年龄27~81岁,平均年龄(50.48±12.11)岁,组织学类型包括浸润性导管癌70例、伴髓样特征的浸润性癌9例、化生性癌4例;分子亚型包括管腔型43例、HER2过表达型19例、三阴性乳腺癌21例。选择肿瘤石蜡包埋组织为观察组,距肿物边缘>2 cm的正常乳腺组织作为对照组。
1.2 方法 1.2.1 qRT-PCR检测miR-449a表达水平将石蜡标本以15 μm厚度切片,按石蜡组织切片miRNA提取试剂盒(北京天根生化科技公司)说明书提取RNA。按逆转录试剂盒(美国APExBIO公司)操作步骤将RNA逆转录为cDNA。miR-449a逆转录引物序列:GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACACCAGC。将合成的cDNA稀释为统一浓度后,按照荧光定量PCR试剂盒说明书(南京诺唯赞生物科技公司)进行操作,配制20 μL反应体系:cDNA模板2 μL、引物0.8 μL、SYBR Green PCR Master Mix 10 μL、ddH2O 7.2 μL。miR-449a上游引物:CGCGTGGCAGTGTATTGTTA,下游引物:AGTGCAGGGTCCGAGGTATT。反应条件:95 ℃预变性5 min;95 ℃变性15 s、60 ℃退火30 s、72 ℃延伸15 s,循环45次。记录Ct值,以U6为内参照,采用2-ΔΔCt法计算miR-449a的相对表达量。
1.2.2 生存数据分析使用Kaplan-Meier Plotter数据库(https://kmplot.com/analysis/)进行在线分析,选择METABRIC数据集和默认参数,比较miR-449a表达与不同亚型乳腺癌患者总生存率的关系。
1.2.3 靶基因预测和富集分析从ENCORI网站(https://starbase.sysu.edu.cn/index.php)下载miRNA-mRNA相互作用数据,选择TargetScan、miRanda和PicTar三个数据库共同的预测结果作为预测靶基因集合。应用metascape在线数据库(https://metascape.org/gp/index.html#/main/step1)对所预测的靶基因集合进行基因本体(Gene ontology,GO)分析、京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)富集分析。
1.2.4 编码蛋白互作网络分析使用string数据库(https://cn.string-db.org/)进行编码蛋白相互作用(Protein-protein interaction,PPI)网络分析。将数据结果导入Cytoscape 3.9.1软件,使用MCODE插件进行PPI子网络的聚类模块分析。
1.2.5 miR-449a与靶基因表达的相关性分析使用ENCORI数据库探索乳腺癌组织miR-449a与靶基因表达的相关性,mRNA和miRNA数据来源于癌症基因组图谱(The cancer genome atlas,TCGA),并经log2(FPKM/RPM + 0.01)转换。
1.3 统计学处理运用SPSS 26.0软件对数据进行统计处理,计量资料以均数±标准差表示,组间差异比较采用配对t检验,多组资料两两比较采用Games-Howell法,计数资料以例和百分率表示,行χ2检验。P<0.05表示差异具有统计学意义。
2 结果分析 2.1 乳腺癌和癌旁正常组织miR-449a表达水平比较乳腺癌组织中miR-449a表达水平(1.33±0.67)高于癌旁正常组织(1.02±0.41),差异具有统计学意义(t=3.801,P<0.001,见图 1a)。进一步比较每例样本癌旁组织和癌组织的表达,结果显示,与癌旁组织相比,miR-449a在38.6%(32/83)的乳腺癌组织中表达下调(见图 1b),其中包括13例管腔型乳腺癌、6例HER2过表达型乳腺癌和13例三阴性乳腺癌,说明miR-449a在乳腺癌组织中的表达存在明显的异质性。
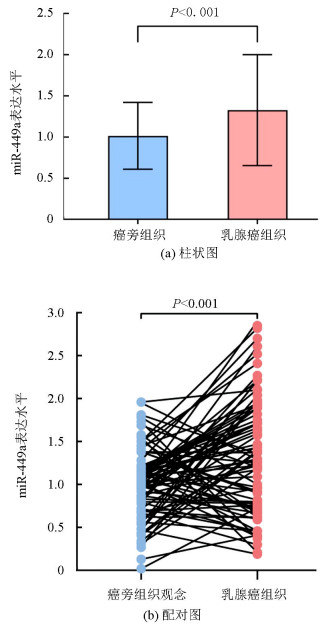
|
图 1 miR-449a在乳腺癌和癌旁组织中的表达水平 Figure 1 Expression level of miR-449a in breast cancer tissues and adjacent tissues |
进一步比较不同分子亚型乳腺癌miR-449a的表达,结果显示其在三阴性乳腺癌中表达水平最低(0.90±0.39),在管腔型乳腺癌中的表达水平最高(1.56±0.71),两组之间的差异具有统计学意义(P<0.001),而HER2过表达型乳腺癌(1.29±0.63)与其他亚型之间的差异不具有统计学意义(P>0.05,见图 2)。

|
图 2 miR-449a在不同分子亚型乳腺癌中的表达水平 Figure 2 Expression level of miR-449a in different molecular subtypes of breast cancer |
根据乳腺癌组织miR-449a表达水平的平均值,将患者分为低表达组43例,高表达组40例。分析miR-449a表达水平与乳腺癌患者临床病理特征的关系,结果显示其与肿瘤组织学级别、肿瘤大小以及ER、PR的表达状态有关(P<0.05),与患者年龄、肿瘤发生部位、淋巴结转移、TNM分期以及HER2表达状态无相关性(P>0.05,见表 1)。
| 表 1 miR-449a表达水平与乳腺癌患者临床病理特征的关系 n(%) Table 1 Relationship between the expression level of miR-449a and the clinicopathological features of patients with breast cancer n(%) |
使用Kaplan-Meier plotter数据库分析miR-449a表达水平与不同亚型乳腺癌患者总生存率的关系,结果显示,在管腔B型乳腺癌中miR-449a高表达组总生存率显著低于低表达组(P<0.01),在三阴性乳腺癌中miR-449a低表达组总生存率显著低于高表达组(P<0.05,见图 3),相反的趋势提示miR-449a在不同分子亚型乳腺癌的作用可能存在差别。此外,管腔A型、HER2过表达型乳腺癌患者的总生存率与miR-449a的表达水平无关(P>0.05)。
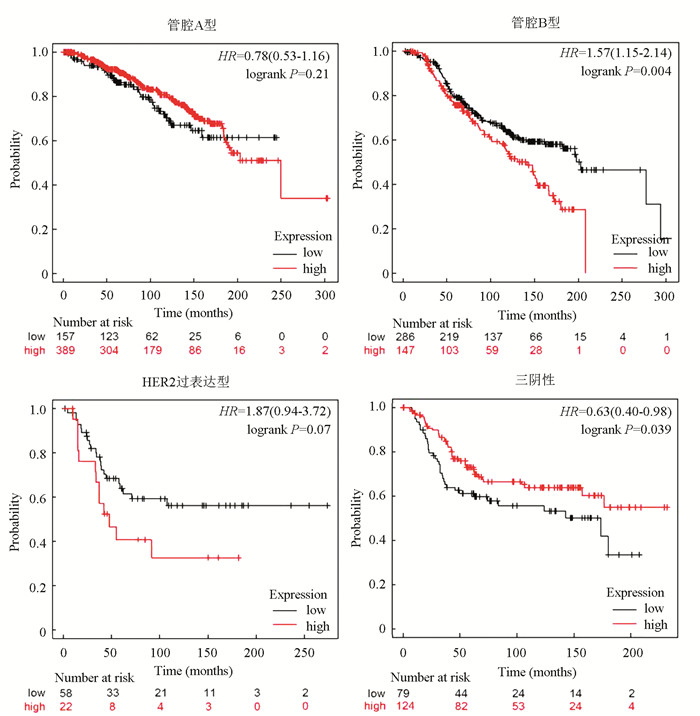
|
图 3 miR-449a表达水平与不同分子亚型乳腺癌患者预后的关系 Figure 3 Relationship between miR-449a expression level and prognosis of patients with different molecular subtypes of breast cancer |
预测结果筛选出186个miR-449a靶基因,GO分析提示蛋白功能主要涉及细胞成分形态发生、间充质细胞分化、器官发育、组蛋白修饰、细胞迁移的正向调控、囊泡介导的转运调控等生物学过程(见图 4)。KEGG分析结果显示miR-449a靶基因富集于内分泌抵抗、粘附连接、粘着斑、肌动蛋白细胞骨架调节以及NOTCH、TGF-β、Wnt、Ras、MAPK、PI3K-Akt等介导的信号通路,其中多个靶基因已知与乳腺癌关系密切(见表 2)。
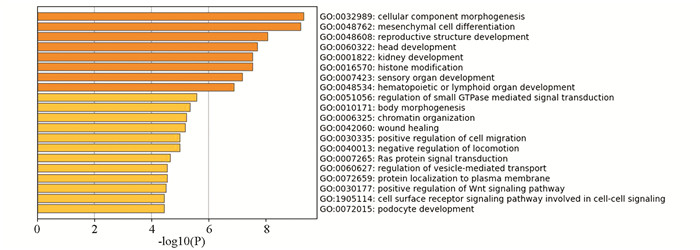
|
图 4 miR-449a预测靶基因GO分析结果 Figure 4 Result of GO analysis of predicted miR-449a target genes |
| 表 2 miR-449a预测靶基因KEGG通路分析结果 Table 2 Result of KEGG pathway anaysis of predicted miR-449a target genes |
将靶基因集合上传至string数据库构建PPI网络,该网络包含137个节点和276条边,平均局部聚类系数为0.357,数据导入Cytoscape进行可视化(见图 5a)。使用MCODE插件进行分析,筛选出以Notch受体1(Notch receptor 1,NOTCH1)、Notch配体1(Jagged canonical Notch ligand 1,JAG1)、细胞周期蛋白D1(cyclin D1,CCND1)等为核心的子网络(见图 5b)。
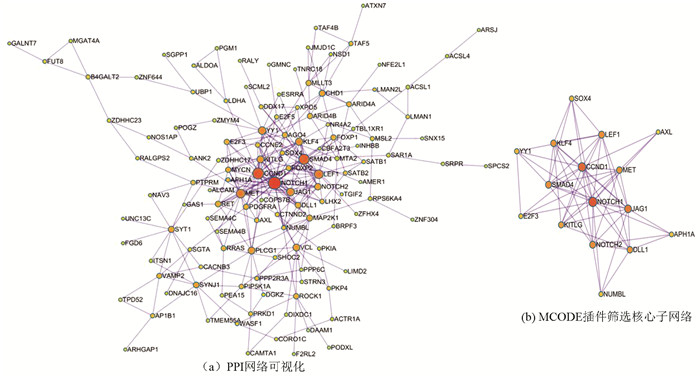
|
图 5 蛋白质互作网络分析结果 Figure 5 Result of analysis of protein-protein interaction network |
进一步分析miR-449a与NOTCH信号通路靶基因(见表 2)表达的相关性,结果显示miR-449a与NOTCH1在乳腺癌组织中的表达呈负相关(r=-0.186,P<0.001,见图 6)。
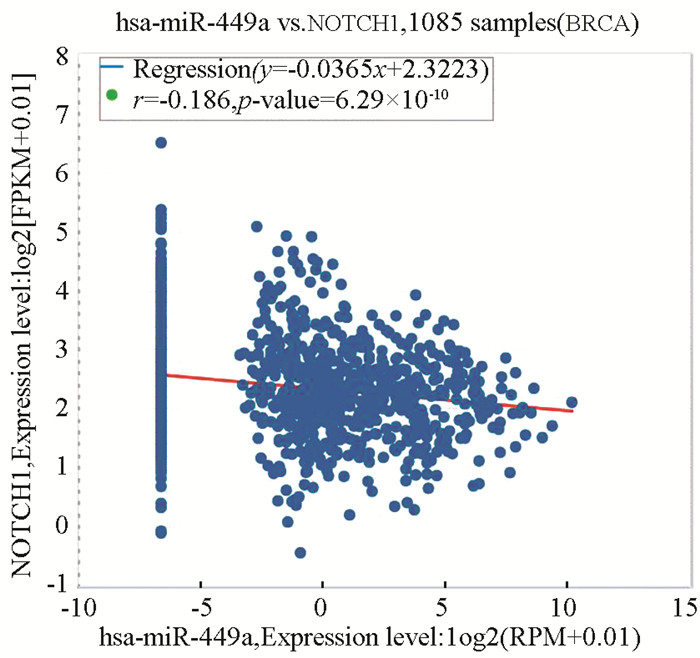
|
图 6 miR-449a与NOTCH1在乳腺癌组织中表达的相关性 Figure 6 Correlation of miR-449a expression with NOTCH1 in breast cancer tissues |
miR-449家族包括miR-449a、miR-449b、miR-449c,由位于染色体5q11.2的基因簇所编码,这一区域已被鉴定是癌症易感区域[7]。miR-449a在人类肺、气管和睾丸中高表达,在非小细胞肺癌、肝细胞癌、鼻窦腺癌等多种恶性肿瘤中常常下调表达,发挥抑癌作用[6-8]。目前miR-449a与乳腺癌相关的研究结论存在一些矛盾。有报道显示miR-449a在乳腺癌组织和细胞系表达下调,过表达miR-449a对乳腺癌细胞起抑制作用[9]。但也有研究发现miR-449a在淋巴结未转移的浸润性导管癌中高表达,其表达与患者复发率增加和总生存期、无病生存期降低显著相关,在体外可促进多种乳腺癌细胞增殖、迁移和侵袭[10]。本研究结果显示miR-449a在乳腺癌组织中的表达呈现出高度的异质性,虽然总体水平高于癌旁组织,但其中有38.6%的病例出现表达下调,因此不同研究结论的矛盾在一定程度上与纳入的样本有关。
本组资料显示miR-449a的表达与肿瘤组织学级别和大小有关,在高级别、体积大的乳腺癌中表达降低,提示其在乳腺癌进展中一定程度上发挥抑癌作用。乳腺癌是具有不同生物学和表型特征的异质性疾病,miR-449a的表达水平在不同分子亚型乳腺癌间存在差异,尤其是三阴性乳腺癌表达水平显著低于管腔型。三阴性乳腺癌是一种高侵袭性亚型,具有很强的增殖能力和高转移风险,上皮间充质转化(Epithelial-mesenchymal transition,EMT)相关的形态学和功能改变是其获得侵袭性的关键事件。靶基因及富集分析结果显示miR-449a的功能涉及间充质细胞分化、细胞迁移、粘附连接、粘着斑、肌动蛋白细胞骨架调节等生物学过程,提示miR-449a的功能与肿瘤获得侵袭性密切相关。体外实验表明在三阴性乳腺癌细胞系中上调miR-449a的表达后可抑制细胞迁移和EMT进程并促进细胞凋亡,从而发挥抗肿瘤作用[2]。生存分析结果显示miR-449a低表达的三阴性乳腺癌患者总生存率显著低于高表达者。
miR-449a在管腔型乳腺癌中高表达,表达水平与激素受体ER、PR阳性状态有关。先前有研究发现miR-449a在ER阳性的乳腺癌细胞株MCF-7中的表达显著高于正常乳腺上皮细胞,敲低后则会抑制癌细胞增殖和迁移[11]。管腔B型是ER阳性乳腺癌中侵袭性较强的类型,生存分析结果显示miR-449a在该亚型中表达较高的患者预后更差,提示miR-449a在该亚型肿瘤进展过程中可能发挥重要作用,但具体机制目前尚未阐明。有研究发现miR-449a表达增加与磷脂酰肌醇-3-激酶催化亚基α(Phosphatidylinositol 3-kinase catalytic subunit alpha,PIK3CA)突变状态有关,后者是ER阳性乳腺癌中最常见的体细胞突变癌基因[12]。PIK3CA突变导致PI3K-Akt信号通路的异常激活,可促进乳腺癌细胞的生长和侵袭[13]。靶基因预测结果也显示miR-449a调控PI3K-Akt信号通路的多个基因,因此miR-449a在乳腺癌中发挥抑癌或致癌作用,可能取决于不同亚型或发展阶段。
KEGG富集分析结果显示miR-449a参与NOTCH、TGF-β、Wnt、PI3K-Akt等多种信号通路,通过PPI网络分析筛选关键子网络,主要由NOTCH1、JAG1和cyclin D1等蛋白所构成。NOTCH信号通路是一种细胞间通信系统,可广泛调控细胞生物学特性,如干细胞维持、上皮细胞极性/粘附、细胞增殖和凋亡等,在器官发育和维持组织稳态中发挥重要作用[14]。异常激活的NOTCH信号与多种恶性肿瘤的迁移、侵袭和转移密切相关,NOTCH1作为NOTCH受体家族成员之一,在信号转导中起关键作用[15]。本研究基于TCGA数据发现miR-449a在乳腺癌组织中的表达与NOTCH1呈负相关。NOTCH1在三阴性乳腺癌中的表达最为显著,可能通过诱导多种EMT相关基因的表达,促进管腔细胞向基底型分化,从而参与三阴性乳腺癌的发生发展过程[16]。JAG1是NOTCH信号通路的重要配体,在乳腺癌中的表达水平与肿瘤侵袭进展、转移及不良预后有关[17]。JAG1表达阳性率在不同亚型的乳腺癌中同样也存在差异,在三阴性乳腺癌中表达率最高且与ER、PR的表达呈负相关[18]。Poddar等[19]已证实miR-449a可靶向作用于JAG1从而在骨骼肌细胞中调控NOTCH信号。cyclin D1在细胞周期控制中起核心作用,是NOTCH1的直接靶点,在三阴性乳腺癌中JAG1可介导NOTCH1对cyclin D1启动子的转录激活,从而促进肿瘤细胞增殖[20]。而在ER阳性的乳腺癌中,cyclin D1则是雌激素调控细胞周期进程的重要组成,并可直接结合ER的激素结合域,在雌激素缺乏的情况下也能介导基因转录,过表达与肿瘤的高增殖和不良预后相关[21-22]。
4 结论miR-449a的表达水平在三阴性和管腔型乳腺癌中存在显著差异,可通过影响多种信号通路参与肿瘤发展过程,调控NOTCH信号通路可能是其在乳腺癌中的重要机制。鉴于调控网络复杂,miR-449a在乳腺癌分型和靶向治疗方面的潜在价值仍有待深入研究。
| [1] |
MUELLER C, HAYMOND A, DAVIS J B, et al. Protein biomarkers for subtyping breast cancer and implications for future research[J]. Expert Review of Proteomics, 2018, 15(2): 131-152. DOI:10.1080/14789450.2018.1421071 (  0) 0) |
| [2] |
COPPOLA A, ILISSO C P, STELLAVATO A, et al. S-Adenosylmethionine inhibits cell growth and migration of triple negative breast cancer cells through upregulating mirna-34c and mirNA-449a[J]. International Journal of Molecular Sciences, 2020, 22(1): 286-304. DOI:10.3390/ijms22010286 (  0) 0) |
| [3] |
RICHARD V, DAVEY M G, ANNUK H, et al. micrornas in molecular classification and pathogenesis of breast tumors[J]. Cancers (Basel), 2021, 13(21): 5332-5351. DOI:10.3390/cancers13215332 (  0) 0) |
| [4] |
HUANG Guangcheng, ZHONG Xiaowu, YAO Lihua, et al. MicroRNA-449a inhibits cell proliferation and migration by regulating mutant p53 in MDA-MB-468 cells[J]. Experimental and Therapeutic Medicine, 2021, 22(3): 1020-1027. DOI:10.3892/etm.2021.10452 (  0) 0) |
| [5] |
HU Shan, CAO Peng, KONG Kangle, et al. MicroRNA-449a delays lung cancer development through inhibiting KDM3A/HIF-1alpha axis[J]. Journal of Translational Medicine, 2021, 19(1): 224-234. DOI:10.1186/s12967-021-02881-8 (  0) 0) |
| [6] |
RE M, TOMASETTI M, MONACO F, et al. miRNome analysis identifying miR-205 and miR-449a as biomarkers of disease progression in intestinal-type sinonasal adenocarcinoma[J]. Head & Neck, 2022, 44(1): 18-33. DOI:10.1002/hed.26894 (  0) 0) |
| [7] |
HE Yongming, JI Aijun, XU Xiaoyue, et al. miR-449a: A potential therapeutic agent for cancer[J]. Anticancer Drugs, 2017, 28(10): 1067-1078. DOI:10.1097/CAD.0000000000000555 (  0) 0) |
| [8] |
HAN Bing, HUANG Jiawei, YANG Zhenjie, et al. miR-449a Is related to short-term recurrence of hepatocellular carcinoma and inhibits migration and invasion by targeting notch1[J]. OncoTargets and Therapy, 2019, 2019(12): 10975-10987. DOI:10.2147/OTT.S216997 (  0) 0) |
| [9] |
XU Bin, ZHANG Xiping, WANG Shanshan, et al. miR-449a suppresses cell migration and invasion by targeting PLAGL2 in breast cancer[J]. Pathology, Research and Practice, 2018, 214(5): 790-795. DOI:10.1016/j.prp.2017.12.012 (  0) 0) |
| [10] |
SHI W, BRUCE J, LEE M, et al. miR-449a promotes breast cancer progression by targeting CRIP2[J]. Oncotarget, 2016, 7(14): 18906-18918. DOI:10.18632/oncotarget.7753 (  0) 0) |
| [11] |
王红磊, 肖奕, 武力, 等. miR-449a对人乳腺癌细胞MCF-7增殖及迁移能力的影响[J]. 中国应用生理学杂志, 2017, 33(6): 508-513. WANG Honglei, XIAO Yi, WU Li, et al. Effects of miR-449a on proliferation and migration of human breast cancer cell line MCF-7[J]. Chinese Journal of Applied Physiology, 2017, 33(6): 508-513. DOI:10.12047/j.cjap.5573.2017.121 (  0) 0) |
| [12] |
RAMIREZ-ARDILA D E, RUIGROK-RITSTIER K, HELMIJR J C, et al. LRG1 mRNA expression in breast cancer associates with PIK3CA genotype and with aromatase inhibitor therapy outcome[J]. Molecular Oncology, 2016, 10(8): 1363-1373. DOI:10.1016/j.molonc.2016.07.004 (  0) 0) |
| [13] |
NUNNERY S E, MAYER I A. Targeting the PI3K/AKT/mTOR pathway in hormone-positive breast cancer[J]. Drugs, 2020, 80(16): 1685-1697. DOI:10.1007/s40265-020-01394-w (  0) 0) |
| [14] |
JAISWAL A, MURAKAMI K, ELIA A, et al. Therapeutic inhibition of USP9x-mediated Notch signaling in triple-negative breast cancer[J]. Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(38): e2101592118. DOI:10.1073/pnas.2101592118 (  0) 0) |
| [15] |
GHARAIBEH L, ELMADANY N, ALWOSAIBAI K, et al. Notch1 in cancer therapy: possible clinical implications and challenges[J]. Molecular Pharmacology, 2020, 98(5): 559-576. DOI:10.1124/molpharm.120.000006 (  0) 0) |
| [16] |
MIAO K, LEI J H, VALECHA M V, et al. NOTCH1 activation compensates BRCA1 deficiency and promotes triple-negative breast cancer formation[J]. Nature Communications, 2020, 11(1): 3256-3270. DOI:10.1038/s41467-020-16936-9 (  0) 0) |
| [17] |
孙维廷, 刘彩霞. Notch配体JAG1在乳腺癌中的研究进展[J]. 肿瘤学杂志, 2021, 27(9): 766-771. SUN Weiting, LIU Caixia. Research progress on notch ligand jag1 in breast cancer[J]. Journal of Chinese Oncology, 2021, 27(9): 766-771. DOI:10.11735/j.issn.1671-170X.2021.09.B012 (  0) 0) |
| [18] |
李秀芹, 韩玉贞, 曾令瑞, 等. 乳腺癌组织Notch1和JAG1蛋白表达与分子分型相关性研究[J]. 中华肿瘤防治杂志, 2017, 24(5): 197-301. LI Xiuqin, HAN Yuzhen, ZENG Lingrui, et al. Expression and significance of Notch1 and JAG1 proteins in different subtypes of breast cancer[J]. Chinese Journal of Cancer Prevention and Treatment, 2017, 24(5): 197-301. DOI:10.16073/j.cnki.cjcpt.2017.05.003 (  0) 0) |
| [19] |
PODDAR S, KESHARWANI D, DATTA M. miR-449a regulates insulin signalling by targeting the Notch ligand, Jag1 in skeletal muscle cells[J]. Cell Communication and Signaling, 2019, 17(1): 84-93. DOI:10.1186/s12964-019-0394-7 (  0) 0) |
| [20] |
NASSER F, MOUSSA N, HELMY M W, et al. Dual targeting of Notch and Wnt/beta-catenin pathways: Potential approach in triple-negative breast cancer treatment[J]. Naunyn-Schmiedeberg's Archives of Pharmacology, 2021, 394(3): 481-490. DOI:10.1007/s00210-020-01988-x (  0) 0) |
| [21] |
AHLIN C, LUNDGREN C, EMBRETSÉN-VARRO E, et al. High expression of cyclin D1 is associated to high proliferation rate and increased risk of mortality in women with ER-positive but not in ER-negative breast cancers[J]. Breast Cancer Research and Treatment, 2017, 164(3): 667-678. DOI:10.1007/s10549-017-4294-5 (  0) 0) |
| [22] |
MONTALTO F I, DE AMICIS F. Cyclin D1 in cancer: a molecular connection for cell cycle control, adhesion and invasion in tumor and stroma[J]. Cells, 2020, 9(12): 2648-2662. DOI:10.3390/cells9122648 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


