2. 教育部癌变与侵袭原理重点实验室,上海 200032;
3. 上海市徐汇区中心医院,复旦大学附属中山徐汇医院, 上海 200032
2. Key Laboratory of Carcinogenesis and Cancer Invasion, Chinese Ministry of Education, Shanghai 200032, China;
3. Shanghai Xuhui Central Hospital, Zhongshan-Xuhui Hospital, Fudan University, Shanghai 200032, China
单细胞转录组测序(Single cell RNA sequencing,ScRNA seq)作为新兴生物技术,在解析组织复杂性、细胞异质性、基因表达调控等方面发挥重要作用,很大程度上弥补了普通转录组测序(Bulk RNA sequencing)的缺陷[1]。批次效应(Batch effects,BEs)是高通量生物学数据中源于技术因素的变异[2],单细胞数据通常需要多次实验来获得,不同批次实验的试剂、实验仪器、实验员、单细胞捕获时间,甚至是技术平台,均可能存在差异。因此,这些因素也就构成了单细胞数据的批次,数据整合过程中可能会干扰感兴趣生物学变异的发现和解释,如何有效去除批次效应在单细胞数据分析中有充分的必要性。
然而,单细胞数据批次效应较为复杂,且和具体的研究有关[3-4],有效处理能增加数据价值,反之则会可能会导致假阳性或假阴性分析结果[5]。批次效应可能是高度非线性的,常常和真正的生物学变异相互混杂,正确纠正不同批次单细胞数据的批次效应,同时正确保留关键生物学变异常常较为困难。为了解决上述问题,此前用于处理芯片数据批次效应的工具,包括Combat[6]和Limma等[7],也用于单细胞数据的批次效应纠正。由于单细胞数据的具有高缺失率(Dropout)以及基因捕获随机性的特点[8],和芯片数据存在显著不同,因此适用于单细胞数据的批次效应处理算法也在不断涌现。其中,代表性算法的包括通过识别相互最近邻来整合不同的数据集的MNNs(Mutual nearest neighbours)[9],建立在MNNs基础上的Scanorama[10]和BBKNN[11],整合于Seurat包中的MultiCCA算法[12],处理多种来源的批次效应具有显著优势的Harmony[13],将不同数据集间变异全部归于技术因素的LIGER[14],以及新型的深度学习算法[15],均在不同程度上去除了批次效应,促进了生物学规律的发现。
目前批次效应的处理已经是单细胞数据分析的常规流程(单细胞数据分析流程见图 1),但批次效应的评估是有效去除批次效应的基础,也常常是被忽视的一步[16]。实际上,选择合适的指标评测批次效应的来源、对数据变异的贡献度和对数据分析的影响有利于判断处理批次效应的必要性及选择合适的处理算法。如今跨平台、跨物种以及多模式单细胞数据的整合,包括单细胞表观组、基因组、转录组、蛋白组等[17],在研究生物学规律及疾病发生发展机制方面显示出巨大优势[18]。同时,数据量和数据来源的增加也使得批次效应更加复杂,准确地评估批次效应的来源、批次效应处理后的效果,对于数据整合尤为关键[19]。
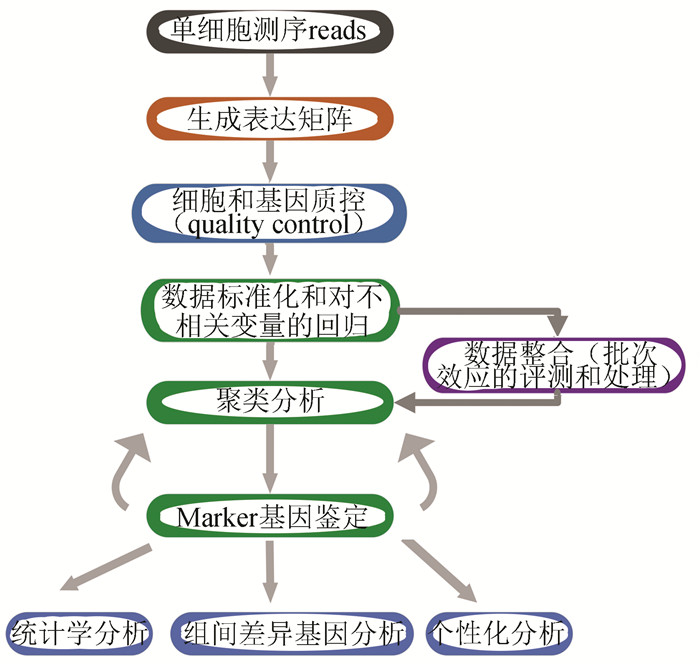
|
图 1 单细胞数据分析流程 Figure 1 Analysis pipeline of single cell data |
因此,本篇综述聚焦单细胞转录组数据的批次效应,依次论述了单细胞批次效应与普通转录组的区别,目前常用的评测算法的特点,最后总结了目前面临的挑战和未来发展方向。
1 单细胞转录组和普通转录组批次效应的区别分析目的及数据结构的不同是构成两者批次效应差异的基础。普通转录组主要目的是计算样本间的差异基因及分子分型,在宏观层面解析其基因表达改变[20]。单细胞转录组则是以细胞群为研究单位,通过降维、聚类和细胞类型注释来识别特定的、同质性的细胞群,并在细胞群及基因层面进行探索性分析[21-22]。数据结构方面,普通转录组数据是低维数据,数据结构较简单,无需做降维处理;而单细胞转录组数据为高维度数据,下游分析前需要多步骤降维处理。
基因缺失率(Dropout rates)是两者最大的不同。高质量普通转录组测序产生的缺失率低于20%,而基于微液滴或者微孔技术的单细胞转录组的缺失率可达80%。即使是测序深度高并且支持全长测序的Smart-seq2技术,缺失率也达到了50%。然而,基因缺失的产生并不完全随机,具有一定的基因偏倚性(Gene-based bias)、细胞偏倚性(Cell-based bias)和批次偏倚性(Batch-based bias)[2, 23]。
此外,普通转录组测序同质性较高,批次效应通常涉及不同研究来源及不同平台的数据,处理批次效应的流程较为固定。而单细胞转录组测序则涉及多个样本、多种细胞类型和成千上万的细胞,甚至百万级细胞量[24],批次效应的评测和处置都相对复杂且缺乏固定的流程。不同样本的细胞类型差异可能很大,甚至部分细胞类型是某些样本特有的,因此这种批次来源变异和生物来源变异的相互混杂是普通转录组未曾碰到过的难题。同时,为了消除多模式的单细胞数据的整合分析中批次效应的影响,则需要对来源于不同平台和不同样本的数据进行多步骤、多来源批次效应的评测和处置。
综上,由于两者测序技术、分析目的、数据结构以及数据量之间的差异,适用于普通转录组的工具并不能直接用于单细胞转录组数据。因此,在单细胞多组学时代,开发新型的批次效应评测及处置算法来整合海量单细胞数据,是正确揭示生物学规律和疾病发生发展机制的基础。
2 批次效应的定性评测:特征选择法特征选择可以评估批次效应对差异基因的影响。目前常用的差异基因工具包括t-test,limma[25],DESeq2[26]和edgeR[27],通过比较批次效应纠正前后的差异基因、差异基因的功能富集与某个已知表型的一致性来实现对批次效应的评测。特征选择并不限于基因或者蛋白质、代谢物等生物单位,也适用于主成分(Principle components,PCs),通过可视化解释度最大的几个主成分是否与细胞群或批次存在更强关联来评测批次效应,尤其是批次效应处理后。
主成分分析(Principle component analysis,PCA)联合散点图是常用的可视化方式[16]。但PCA对批次效应的评测的前提是前两个或者前三个主成分可以解释批次效应来源的变异。PCA是将数据的变异分解为正交因子,各个主成分之间相互独立,意味着用于可视化批次效应的PCA散点图取决于所使用的PC,并且不保留单细胞高维数据的局部结构。因此,通过PCA解析数据结构需要较多的数据分析技巧作为支撑。PCA是将高维数据的变异分解为正交因子而不是将高维数据嵌入到低维流形中,那么由K个因子构成的单细胞数据变异就需要K个主成分来解释,而K值实际上并不知晓。
t-SNE(t-distributed Stochastic neighbor embedding)[28]和UMAP(Uniform manifold approximation and projection)[29]是PCA的拓展和延伸,都是将单细胞高维数据嵌入低维度、非线性流形的算法[30]。与PCA不同,t-SNE和UAMP将高维数据嵌入低维空间并保留高维数据的局部结构,有利于直观展示样本分布和潜在的细胞群,在解析单细胞数据中的异质性和批次效应来源的变异方面具有优势。然而,t-SNE对单细胞高维数据的全局结构赋予的权重很小,使得降维后低维流形中细胞群间的相对空间距离和空间位置没有意义,因此利用t-SNE中的空间位置信息来推断细胞群之间的相似性或者其他任何关系均不可行。UMAP与t-SNE类似,但运行的速度更快,在保留单细胞高维数据的全局结构方面表现更好。
UMAP和t-SNE将单细胞高维数据嵌入到低维流形中时,依赖对细胞间距离的不同定义,而这些距离的定义具有一定的探索性,对于一些单细胞数据并不适用。因此应该慎重对待降维结果,Charia认为,即不加区别地应用这些算法可能会造成数据结构在多个维度上的改变,虽然保留了大部分的数据结构,但细胞、最近邻和细胞类型之间的量化关系被严重扭曲[31]。同时,得到的结果并不稳健,会随着参数的调整而发生显著的变化[32]。因此,对使用UMAP和t-SNE可视化的细胞群做生物学分析时,应结合其他局部指标,包括标记基因(Marker genes)的特异性、效应值大小或概率推断的结果[31]。
由于细胞被嵌入的空间缺乏生物学解释度,t-SNE和UMAP可能无法揭示关于批次效应的可分析信息[32]。t-SNE和UMAP都是建立在邻接图(Neighbor graph)基础上的算法,并不保证单细胞高维数据中细胞群间的距离关系被正确保留。同时,将单细胞的高维数据变异嵌入低维空间后,一些细微、不显著的批次效应干扰可能无法有效识别。
综上,上述方法均有一定局限性,无偏倚的实验设计对于减少批次效应的干扰仍是关键。若批次来源和细胞群来源的变异完全混杂,使用上述方法显然不合适,但当两者是正交关系时,PCA仍是一种系统评测批次效应的方法,通过对批次高度相关的主成分进行系统性检验来针对性地纠正或避免批次效应相关的数据特征,而UMAP和t-SNE无法实现这点[33]。
3 批次效应的定量评测:新型算法单细胞测序技术的发展催生出评测批次效应的新方法。尽管PCA散点图是一种评测批次效应的经典可视化算法,但新型算法工具已经展现出极大优势。
较为流行的kBET(k-nearest Neighbor batch effect test)由无监督机器学习算法(ML,Machine learning)改造而来[34],计算单个样本的每个细胞在不同批次间的最近邻,并评测每个批次间的混合方式及程度。该算法原理较为简单:若检测到每个细胞的最近邻在不同批次间是均匀分布的,那认为是不存在批次效应的。然而,如果批次间的细胞类型组成是不均匀的(比如某细胞类型的大多数细胞均来源于某一批次样本),kBET是不适用的。同样,数据存在明显离群值时(某一个细胞群内部,部分细胞所处的空间位置明显偏离该细胞群的主体空间位置)或数据有明显异质性时(多批次的样本内部有大量的细胞亚群,并且不同批次间的细胞亚群组成的异质性较大),kBET也无法有效地评测批次效应。LISI[13](Local inverse simpson's index)算法与kBET的原理类似,不仅评估批次间的细胞混合程度,还计算细胞谱系间的混杂程度。
另外一种方法是ASW(Average silhouette width),同样由无监督机器学习算法延伸而来,是用于评估高质量细胞群的聚类验证指数[35]。ASW通常与无监督聚类的算法联合使用(比如k-means)。在ASW中,由一群点聚集而成的轮廓是对高维数据的低维展示,代表一个细胞群。该算法通过细胞群内部及细胞群间的紧凑度进行量化,将量化值作为该单细胞数据的整体分群质量,然后判断整体分群情况与和批次之间是否存在关联。然而,ASW毕竟是对整体分群情况的一种量化,难以评估特定细胞群是否存在批次上的偏倚,并对离群值较敏感。除此之外,即使ASW提示细胞群的分群质量较高并与批次无相关性,也不意味着批次效应可以忽略。即使一些单细胞数据的细胞群之间区分度很高(分群时,群与群之间的细胞数目差别很大或者群与群之间的距离很近),也可能会产生低ASW值。这种较小的ASW值往往是错误地提示不同批次之间的细胞混合较好,导致细胞群与批次之间存在的关联无法被正确识别。
与上述三种算法的原理差别较大,兰德指数(RI,Rand index)评估对同一个数据应用不同聚类方法后所产生的聚类结果之间的关联性[36]。RI通过评估每个样本中的细胞如何聚到每个细胞群来判断批次效应纠正后对数据的影响,即如果人为产生了与批次效应相关联的细胞群,那就说明批次效应的纠正是对聚类的结果有影响的。而兰德矫正指数(ARI, Adjusted rand index)则是对兰德指数(RI)的一种随机性矫正,通过评估不同方法对之间的比较来获得他们之间的相似性,从而建立不同方法之间比较的基线标准。
然而,上述算法的前提条件是批次效应对聚类的影响要达到一定的程度才能在低维空间中识别出。如果批次间的样本混合较好,上述算法也会计算出一个较好的评测结果,但如果批次效应对聚类的结果影响很小,通过这些工具难以识别批次效应。相比之下,PCA是评测批次效应对数据影响程度的一种鲁棒性很高的方法[33],而在局部水平评估混合程度的算法(比如kBET)易受到纠正批次效应和保留有意义生物学信号之间的平衡的影响。
4 单细胞批次效应评测方法总结目前评测单细胞批次效应最常用的是PCA、tSNE、UMAP这些可视化的方法,尤其是数据量较小、细胞类型较少时,并且他们在直观展示分群结果以及发现新的细胞类型方面具有独特的优势。然而,当单细胞数据量增大、细胞类型更复杂时,这些可视化方法的主观性和局限性就较为突出了。差异基因分析(DEGs analysis)则更偏于对分析结果的直观评价和对生物学意义保留情况的判断。
不同的是,kBET、LISI、ARI和ASW都是相对客观的指标,但其计算结果可能与可视化的结果存在差异较大甚至相互矛盾的情况。实际应用中,kBET和LISI的结果较为一致,可能是因为两者都是对局部水平批次间混合程度的计算;反之,ARI和ASW都是对整体水平批次效应的评测,这可能是导致不同指标的结果不一致的原因。值得注意的是,这些评测指标对细胞类型纯度和各批次混合程度是分开计算的,实际应用中还需要采用合适的策略将细胞类型纯度和各批次混合程度进行整合。
评测单细胞批次效应的较好流程是,将可视化的方法和kBET等这些定量指标相互结合,综合判定批次效应的来源、对数据分析的影响程度以及批次效应的纠正效果(单细胞数据批次效应评测方法总结和对比见表 1)。
| 表 1 单细胞数据批次效应评测方法总结和对比 Table 1 Summary and comparison of batch effect evaluation methods for single cell data |
目前,单细胞中批次效应的评测还面临以下挑战,并且这些挑战很可能会延伸到新兴起的技术,比如说空间组学技术(Spatial omics technology)。
1) 纠正批次效应的算法通常是对批次间的细胞进行相似度计算,然后对批次间的细胞做混合聚类来纠正批次效应。过度纠正批次来源的变异会埋没部分生物学变异,导致某些样本特异的细胞类型或者罕见细胞类型不能通过聚类鉴定出来。然而,PCA及其他算法工具尚无法充分评估批次效应纠正和生物意义保留之间的平衡关系。同时,现有工具还无法精确评测不均衡实验设计(不同批次间样本的细胞类型差异较大)带来的批次效应,从而为不均衡实验批次效应的去除提供依据。
2) 纠正批次效应来源的变异与保留生物学变异之间的平衡。现有的评测批次效应的算法工具不能量化批次效应和生物学变异各自对数据总变异的解释度,以及计算出合适阈值在纠正批次效应的同时最大化保留生物学变异。PCA无法保留高维数据的局部结构以及判断生物学变异和批次的变异对数据的影响;t-SNE和UMAP虽然可以保留单细胞高维数据的局部结构,但是嵌入的低维空间并不能保证对生物学变异的解释度。
3) 对单细胞数据高缺失率造成的批次效应研究不足。由于测序深度、mRNA捕获效率等的限制,大量基因的count是零值,但基因缺失的产生并不完全随机,同样有一定的偏倚性。某些基因更易产生零值,导致某些高表达这些基因的细胞类型及富集这些细胞类型的某批次样本更易产生零值。基因零值是批次效应的重要来源,具体产生原因及数据分布特征尚不清楚,因此如何评测基因零值来源的批次效应并将其可视化,是评测批次效应的的重大挑战之一。
4) 不同单细胞批次效应评测指标可能出现结果不一致情况,并且与PCA、tSNE、UMAP这些可视化结果也可能差异较大。因此,如何将不同评测方法的评测方面进行统一化是面临的挑战之一。同时,如何很好地综合细胞类型纯度和各批次混合情况,也是评测工具未来需要解决的问题。
6 未来展望多模式的单细胞数据不断出现以及数据的开源性进一步增加,大数据整合分析已经是单细胞技术领域的重要环节,批次效应的评测仍然是生物信息学分析的热点领域,必将直接影响到单细胞数据分析质量和结果解读。目前常用的评测批次效应的定性或者定量算法工具,比如说PCA,t-SNE和UMAP,kBET,ASW,RI等,有效地评测了批次效应,但在应用方面均存在一定的局限性。因此,未来随着单细胞转录组测序技术的进步和分析流程的不断优化,基于机器学习或者深度学习的批次效应评测算法的开发和优化,将有希望开发出整合批次效应可视化和定量指标、区分不同来源批次效应的影响、综合细胞纯度和各批次混合程度的评测工具。
| [1] |
LI Yunjin, MA Lu, WU Duojiao, et al. Advances in bulk and single-cell multi-omics approaches for systems biology and precision medicine[J]. Briefings in Bioinformatics, 2021, 22(5): 1-8. DOI:10.1093/bib/bbab024 (  0) 0) |
| [2] |
HICKS S C, TOWNES F W, TENG M, et al. Missing data and technical variability in single-cell RNA-sequencing experiments[J]. Biostatistics, 2018, 19(4): 562-578. DOI:10.1093/biostatistics/kxx053 (  0) 0) |
| [3] |
TUNG P Y, BLISCHAK J D, HSIAO C J, et al. Batch effects and the effective design of single-cell gene expression studies[J]. Scientific Reports, 2017, 7: 39921. DOI:10.1038/srep39921 (  0) 0) |
| [4] |
LAFZI A, MOUTINHO C, PICELLI S, et al. Tutorial: guidelines for the experimental design of single-cell RNA sequencing studies[J]. Nature Protocols, 2018, 13(12): 2742-2757. DOI:10.1038/s41596-018-0073-y (  0) 0) |
| [5] |
GOH W W B, WANG W, WONG L. Why batch effects matter in omics data, and how to avoid them[J]. Trends in Biotechnology, 2017, 35(6): 498-507. DOI:10.1016/j.tibtech.2017.02.012 (  0) 0) |
| [6] |
JOHNSON W E, LI C, RABINOVIC A. Adjusting batch effects in microarray expression data using empirical Bayes methods[J]. Biostatistics, 2007, 8(1): 118-127. DOI:10.1093/biostatistics/kxj037 (  0) 0) |
| [7] |
SMYTH G K, SPEED T. Normalization of cDNA microarray data[J]. Methods, 2003, 31(4): 265-273. DOI:10.1016/s1046-2023(03)00155-5 (  0) 0) |
| [8] |
GAWAD C, KOH W, QUAKE S R. Single-cell genome sequencing: current state of the science[J]. Nature Reviews: Genetics, 2016, 17(3): 175-188. DOI:10.1038/nrg.2015.16 (  0) 0) |
| [9] |
HAGHVERDI L, LUN A T L, MORGAN M D, et al. Batch effects in single-cell RNA-sequencing data are corrected by matching mutual nearest neighbors[J]. Nature Biotechnology, 2018, 36(5): 421-427. DOI:10.1038/nbt.4091 (  0) 0) |
| [10] |
HIE B, BRYSON B, BERGER B. Efficient integration of heterogeneous single-cell transcriptomes using Scanorama[J]. Nature Biotechnology, 2019, 37(6): 685-691. DOI:10.1038/s41587-019-0113-3 (  0) 0) |
| [11] |
POLAŃSKI K, YOUNG M D, MIAO Z, et al. BBKNN: fast batch alignment of singlecelltranscriptomes[J]. Bioinformatics, 2020, 36(3): 964-965. DOI:10.1093/bioinformatics/btz625 (  0) 0) |
| [12] |
BUTLER A, HOFFMAN P, SMIBERT P, et al. Integrating single-cell transcriptomic data across different conditions, technologies, and species[J]. Nature Biotechnology, 2018, 36(5): 411-420. DOI:10.1038/nbt.4096 (  0) 0) |
| [13] |
KORSUNSKY I, MILLARD N, FAN J, et al. Fast, sensitive and accurate integration of single-cell data with Harmony[J]. Nature Methods, 2019, 16(12): 1289-1296. DOI:10.1038/s41592-019-0619-0 (  0) 0) |
| [14] |
WELCH J, KOZAREVA V, FERREIRA A, et al. Integrative inference of brain cell similarities and differences from single-cell genomics[J]. bioRxiv, 2018, 459891. DOI:10.1101/459891 (  0) 0) |
| [15] |
FLORES M, LIU Z, ZHANG T, et al. Deep learning tackles single-cell analysis-a survey of deep learning for scRNA-seq analysis[J]. Briefings in Bioinformatics, 2022, 23(1): 1-31. DOI:10.1093/bib/bbab531 (  0) 0) |
| [16] |
ANDREWS T S, KISELEV V Y, MCCARTHY D, et al. Tutorial: guidelines for the computational analysis of single-cell RNA sequencing data[J]. Nature Protocols, 2021, 16(1): 1-9. DOI:10.1038/s41596-020-00409-w (  0) 0) |
| [17] |
GOH W W B, WONG L. The birth of bio-data science: trends, expectations, and applications[J]. Genomics, Proteomics & Bioinformatics, 2020, 18(1): 5-15. DOI:10.1016/j.gpb.2020.01.002 (  0) 0) |
| [18] |
MA A, MCDERMAID A, XU J, et al. Integrative methods and practical challenges for Single-Cell Multi-omics[J]. Trends in Biotechnology, 2020, 38(9): 1007-1022. DOI:10.1016/j.tibtech.2020.02.013 (  0) 0) |
| [19] |
GOH W W B, WONG L. Dealing with confounders in omics analysis[J]. Trends inBiotechnology, 2018, 36(5): 488-498. DOI:10.1016/j.tibtech.2018.01.0133 (  0) 0) |
| [20] |
GIANCARLO R, ROMBO S E, UTRO F. Compressive biological sequence analysis and archival in the era of high-throughput sequencing technologies[J]. BriefingsinBioinformatics, 2014, 15(3): 390-406. DOI:10.1093/bib/bbt088 (  0) 0) |
| [21] |
CLARKE Z A, ANDREWS T S, ATIF J, et al. Tutorial: guidelines for annotating single-cell transcriptomic maps using automated and manual methods[J]. Nature Protocols, 2021, 16(6): 2749-2764. DOI:10.1038/s41596-021-00534-0 (  0) 0) |
| [22] |
LUECKEN M D, THEIS F J. Current best practices in single-cell RNA-seq analysis: atutorial[J]. MolecularSystems Biology, 2019, 15(6): e8746. DOI:10.15252/msb.20188746 (  0) 0) |
| [23] |
QIU Peng. Embracing the dropouts in single-cell RNA-seq analysis[J]. Nature Communications, 2020, 11: 1169. DOI:10.1038/s41467-020-14976-9 (  0) 0) |
| [24] |
JONES R C, KARKANIAS J, KRASNOW M A, et al. The Tabula Sapiens: A multiple-organ, single-cell transcriptomic atlas of humans[J]. Science, 2022, 376(6594): eabl4896. DOI:10.1126/science.abl4896 (  0) 0) |
| [25] |
RITCHIE M E, PHIPSON B, WU D, et al. limma powers differential expression analyses for RNA-sequencing and microarray studies[J]. Nucleic Acids Research, 2015, 43(7): e47. DOI:10.1093/nar/gkv007 (  0) 0) |
| [26] |
LOVE M I, HUBER W, ANDERS S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J]. Genome Biology, 2014, 15(12): 550. DOI:10.1186/s13059-014-0550-8 (  0) 0) |
| [27] |
ROBINSON M D, MCCARTHY D J, SMYTH G K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data[J]. Bioinformatics, 2010, 26(1): 139-140. DOI:10.1093/bioinformatics/btp616 (  0) 0) |
| [28] |
KOBAK D, BERENS P. The art of using t-SNE for single-cell transcriptomics[J]. Nature Communications, 2019, 10(1): 5416. DOI:10.1038/s41467-019-13056-x (  0) 0) |
| [29] |
BECHT E, MCINNES L, HEALY J, et al. Dimensionality reduction for visualizing single-cell data using UMAP[J]. Nature Biotechnology, 2018, 37(1): 38-44. DOI:10.1038/nbt.4314 (  0) 0) |
| [30] |
DO V H, CANZAR S. A generalization of t-SNE and UMAP to single-cell multi modal omics[J]. Genome Biology, 2021, 22(1): 130. DOI:10.1186/s13059-021-02356-5 (  0) 0) |
| [31] |
CHARI T, PACHTER L. The specious art of single-cell genomics[J]. bioRxiv, 2022, 457696. DOI:10.1101/2021.08.25.457696 (  0) 0) |
| [32] |
BREDA J, ZAVOLAN M, VAN NIMWEGEN E. Bayesian inference of gene expression states from single-cell RNA-seq data[J]. Nature Biotechnology, 2021, 39(8): 1008-1016. DOI:10.1038/s41587-021-00875-x (  0) 0) |
| [33] |
GOH W W B, SNG J C, YEE J Y, et al. Can Peripheral Blood-Derived Gene Expressions Characterize Individuals at Ultra-high Risk for Psychosis?[J]. Computational Psychiatry, 2017, 1: 168-183. DOI:10.1162/CPSY_a_00007 (  0) 0) |
| [34] |
BVTTNER M, MIAO Z, WOLF F A, et al. A test metric for assessing single-cell RNA-seq batch correction[J]. Nature Methods, 2019, 16(1): 43-49. DOI:10.1038/s41592-018-0254-1 (  0) 0) |
| [35] |
BATOOL F, HENNIG C. Clustering with the average silhouette width[J]. Computational Statistics & DataAnalysis, 2021, 158: 1-18. DOI:10.1016/j.csda.2021.107190 (  0) 0) |
| [36] |
WU Zhijin, WU Hao. Accounting for cell type hierarchy in evaluating single cell RNA-seq clustering[J]. Genome Biology, 2020, 21: 123. DOI:10.1186/s13059-020-02027-x (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


