2. 山西省农业科学院 谷子研究所, 山西 长治 046011;
3. 内蒙古农业大学 农学院, 呼和浩特 010019
2. Millet Research Institute, Shanxi Academy of Agricultural Sciences, Changzhi 046011, Shanxi, China;
3. College of Agronomy, Inner Mongolia Agricultural University, Hohhot 010019, China
MADS-box基因是真核生物中一类重要的转录因子,在动物、植物和真菌中都有报道[1]。MADS-box的名称来自于酵母Mini Chromosome Maintenance 1(MCM1)、拟南芥Agamous (AG)、金鱼草Deficiens (DEF)和人类的Serum Response Factor (SRF) 4种蛋白的首字母,这4种蛋白质结构里都有一个由大约60个氨基酸组成的高度保守的MADS盒,编码含有这种蛋白因子的基因称MADS-box转录因子[2]。MADS-box基因根据结构域的类型,可分为两个谱系,Ⅰ型和Ⅱ型。Ⅰ型MADS基因只含有保守的MADS结构域,并且在转录水平上丰度很低。Ⅱ型MADS基因又称为MIKC型基因,通常含有4个结构域,分别为:高度保守的MADS-box(M)、中度保守的Kertain-like domain (K)、较为保守的Intervening domain (I) 和多变的C-terminal domain (C),植物中大多数功能已知的MADS-box基因都属于typeⅡ型[3-4]。
MADS-box基因在植物生长发育过程中起重要的调控作用,已经在拟南芥、水稻、小麦、番茄、梨、菠萝等植物中进行了深入的研究 [5-13]。例如拟南芥的花形成的ABCDE模型中的MADS-box基因起着重要作用[14]。水稻的MADS-box基因SUPPRESSOR OF OVEREXPRESSION OF CONSTANS 1 (SOC1), SHORT VEGETATIVE PHASE (SVP), AGAMOUS-LIKE 24 (AGL24)和SEPALLATA 4 (SEP4) 基因共同调控水稻花序的枝梗分生组织的分化[15],而另一个水稻MADS-box基因Panicle phytomer 2(PAP2)基因正向调节小穗分生组织的形成[16]。另外拟南芥AGAMOUS-LIKE 67(AGL67)参与调控种子的萌发过程[17]。同时一些MADS转录因子参与植物逆境胁迫应答反应[18-19]。
谷子(Setaria italica (L.) Beauv.)是禾本科植物,一年生,具有适应性广、抗逆性强,产量稳定、粮草兼用等特点,且谷子全身是宝,各有用途,它的蛋白质和矿物质含量也非常高,不仅解决了人类营养饮食问题,还促进了饲牧业的发展,所以深受中国人民喜爱[20]。然而谷子平均产量比较低,一般谷子的亩产量在0.6~0.8 kg。当前如何提高谷子产量是育种工作者的重要任务。谷子的枝梗数和穗码数量与谷子的产量密切相关。因此研究调控与谷穗分生组织发育相关MADS-box基因的功能对于提高谷子产量有重要的意义。本研究利用生物信息学分析方法,对谷子MADS-box家族进行全面而系统分析,得到较为完整的谷子MADS基因家族信息。同时利用转录组测序技术对发育初期的谷穗和成熟期的谷穗进行了转录组测序分析。通过差异表达基因分析,筛选了数个与谷穗分生组织发育相关MADS-box基因。本研究为进一步揭示MADS-box基因在谷子穗发育过程中的作用奠定了重要的基础。
1 材料与方法 1.1 谷子SiMADS家族成员的鉴定首先在TAIR数据库(https://www.arabidopsis.org/index.jsp)下载107条拟南芥AtMADS转录因子蛋白序列[18],然后在Phytozome 13.0数据库(https://phytozome-next.jgi.doe.gov)的谷子全基因组数据库中进行序列检索,在E值< 1.0,得到全部的谷子SiMADS转录因子。为确保所获取的谷子SiMADS转录因子序列的可靠性和有效性,通过在线SMART数据库(http://smart.embl-heidelberg.de)对上述蛋白序列的结构域进行鉴定和冗余性分析,确定所得到的SiMADS转录因子蛋白序列都含有真实的MADS保守结构域,最终鉴定出68条谷子的SiMADS转录因子,根据它们在染色体上的位置顺序,分别将它们命名为SiMADS1~SiMADS68。
1.2 谷子SiMADS蛋白的理化性质分析利用在线软件ExPASy中的Protparam软件(https://web.expasy.org/protparam)分析谷子SiMADS蛋白质的相对分子量、理论等电点、稳定性等进行分析。
1.3 谷子SiMADS基因的染色体定位分析应用Mapinspect软件把获得的谷子SiMADS基因在染色体上的物理位置绘制成染色体的物理图谱。
1.4 谷子SiMADS蛋白系统进化树的构建利用MEGA7.0软件中的Clustal W对68条谷子SiMADS氨基酸序列和拟南芥的7条AtMADS氨基酸序列进行多序列比对,然后采用N-J邻接法构建系统发育进化树。
1.5 谷子SiMADS基因家族表达谱分析从NCBI的SRA下载谷子根、茎、叶、穗的转录组数据。利用Fastqc软件对数据进行筛选,选择Q20以上的reads,利用Galaxy数据分析平台与青狗尾草基因组序列进行比对,表达量的单位用RPKM值衡量,归一化处理后的数据用log2作为基因表达的数据,用HemI软件生成热图。
1.6 谷子穗发育转录组分析本试验的谷子品种“晋谷21”种植于山西农业大学谷子研究所试验田。采集谷子的幼嫩的谷穗(开花后1 d)和成熟的谷穗(开花后30 d),立即液氮处理,用干冰寄给北京诺禾致源科技股份有限公司,进行RNA的提取和转录组测序。样品RNA检测合格后,进行建库并运用illumina测序平台进行转录组测序。获得测序数据后,进行基因表达量分析。使用诺禾致源售后平台工具NovoMagic对差异基因的进行分析(https://magic.novogene.com/public/customer/main#/home)。
1.7 谷子SiMADS基因的荧光定量PCR表达分析采集晋谷21植株的叶片、根、幼嫩的谷穗和成熟期的谷穗,立即液氮处理,-80 ℃保存。采用TRIzol法标准流程提取样品总RNA。反转录合成cDNA,然后进行荧光定量PCR反应,具体步骤参照试剂盒说明(Takara,RR047A)。采用GenScript Real-time PCR引物设计工具(https://www.genscript.com/ssl-bin/app/primer)设计SiMADS的引物(见表 1)。采用ABI7500荧光定量PCR仪(Applied Biosystems,USA)进行PCR反应,热循环程序如下:1个循环(95 ℃,30 s); 40个循环(95 ℃, 15 s,57 ℃,30 s,72 ℃,30 s)。内参用谷子的18S RNA。实验进行了三次技术重复。实验结果用基因的相对表达量计算,计算公式:2-ΔΔct,ΔΔct=(Ct目的基因-Ct内参)-(Ct目的基因-Ct内参)。
| 表 1 SiMADS基因荧光定量PCR引物序列 Table 1 Primers sequences of qRT-PCR analysis of SiMADS genes |
本研究使用序列相似性检索,在Phytozome 13.0数据库中筛选并且鉴定出了68个谷子MADS家族成员,总共鉴定出68个SiMADS转录因子,SiMADS序列的平均长度为251.6 aa,其中氨基酸序列最长的是SiMADS17,为477 aa,而SiMADS68最短,只有61 aa。68个SiMADS经过SMART数据库检测,都含有真实的MADS保守结构域。此外利用在线软件Protparam对68条谷子SiMADS蛋白质的相对分子量、理论等电点(PI)、稳定性等进行分析,结果(见表 2)。
| 表 2 谷子SiMADS蛋白理化性质 Table 2 Physical and chemical properties of foxtail millet SiMADS proteins |
为了获得SiMADS家族所有基因在染色体上的物理位置,首先在Phytozome13.0数据库获得这些基因的物理位置,然后使用Mapinspect软件绘制了染色体的物理图谱(见图 1)。在染色体之间,SiMADS基因的分布不平衡。5号染色体上的基因数量最多,为13个,占总数的19%。8号染色体上的SiMADS基因数量为2,约占总数的3%,是最少的。
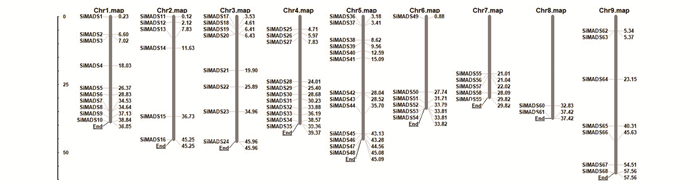
|
图 1 谷子SiMADS基因的染色体分布 Figure 1 Chromosome distribution of SiMADS genes in foxtail millet |
参考拟南芥的MADS家族基因的分类情况,对谷子SiMADS成员之间的进化关系进行研究。根据拟南芥的系统发育关系,Ⅰ型MADS蛋白质可进一步分为Mα、Mβ和Mγ三个亚组,Ⅱ型MADS蛋白质则含有Mδ和MIKC两个亚组。挑选拟南芥的MIKC(At2g03710、At2g45650、At3g54340),Mα(At5g27130),Mβ(At5g41200),Mγ(At1g65300)和Mδ(At1g18750)氨基酸序列作为参考序列,构建谷子SiMADS家族的系统发育树(见图 2)。17个SiMADS被归为Mα,3个被归为Mβ,5个被归为Mγ。13个Mδ和30个MIKC组成了SiMADSⅡ型蛋白。

|
图 2 谷子SiMADS的系统进化树及分类 Figure 2 Phylogenetic relationships of foxtail millet SiMADS proteins |
在SRA库中下载谷子的根、茎、叶和穗转录组数据集,用热图的形式来显示各个基因的表达情况,对谷子MADS基因家族所有成员的组织特异性表达情况进行分析(见图 3)。基因的表达量越大,其颜色就越深。其中13个SiMADS基因没有检测到基因表达。这13个基因中除了SiMADS15属于MIKC亚组以外,其他都属于Ⅰ型MADS。SiMADS家族基因表达的组织特异性较强。大部分基因在花器官中都有比较高的表达量。4个SiMADS基因(SiMADS29、41、49和SiMADS50)在根中的表达量高于其他器官。SiMADS53在叶中的表达量显著高于其他器官高,SiMADS54和SiMADS63在茎中的表达量比其他器官高。

|
图 3 谷子SiMADS基因的组织特异性表达 Figure 3 Tissue-specific expression pattern of foxtail millet SiMADS genes 注:橘红色代表表达量上调,蓝色代表表达量下调. |
为了研究调控谷子穗发育相关的基因,对幼嫩的谷穗(幼穗)和成熟期的谷穗(成熟穗)进行了转录组测序分析。根据基因在不同样品中的表达水平(FPKM值),识别差异表达基因,并使用诺禾致源售后平台工具NovoMagic对差异基因的进行分析,绘制热图(https://magic.novogene.com/public/customer/main#/home)。结果发现:有30个MADS基因在谷子幼穗和成熟穗中表达,其中4个基因在幼穗中有较高表达量,6个基因在成熟穗中有较高表达量,其余基因的表达量在幼穗和成熟穗中差异不大(见图 4)。

|
图 4 谷子SiMADS基因在谷穗发育过程中的表达情况的热图分析 Figure 4 Heatmap analysis of SiMADS genes expression during foxtail millet spike development 注:红色代表表达量上调,蓝色代表表达量下调. |
为了验证转录组数据的可靠性,随机选取5个基因,包括SiMADS27、SiMADS41、SiMADS44、SiMADS52和SiMADS62,利用荧光定量PCR,对这些在谷子根、叶、幼穗和成熟谷穗中表达情况进行分析。结果发现5个基因在幼穗和成熟穗中的表达量都高于在根和叶中的表达量,其中SiMADS44和SiMADS62在谷子幼穗的表达量高于成熟穗,而SiMADS41和SiMADS52在幼穗和成熟穗中的表达量没有明显差异(见图 5)。该结果与转录组测序结果一致,证明了转录组测序数据的可靠性。

|
图 5 5个谷子SiMADS基因在不同组织的表达分析 Figure 5 Expression analysis of five SiMADS genes in different tissues 注:实验结果用相对表达量表示。内参基因为谷子的18S RNA. |
MADS-box基因参与调控多项植物生长发育过程,到目前为止已经在多种植物进行了鉴定[5-13],其中在拟南芥、水稻、二穗短柄草中分别鉴定了107、75和57个基因[5-7]。本研究利用序列相似性检索(BLAST),对谷子基因组进行全面检索,鉴定出了68个SiMADS基因。尽管谷子的基因组(515 Mb)大于拟南芥的基因组(123 Mb),谷子MADS-box基因总数(68)小于拟南芥(107)。同样水稻和二穗短柄草的基因组(466 Mb和260 Mb)分别都大于拟南芥的基因组(123 Mb),而水稻和二穗短柄草的MADS-box基因数(75和57)都小于拟南芥(107)。研究发现拟南芥拟南芥Ⅰ型MADS-box基因存在大量基因复制[5],可能导致了拟南芥Ⅰ型MADS-box基因的总数(61)大于谷子和水稻Ⅰ型MADS-box基因数量(25和32)。
谷子全身是宝,各有用途,它的蛋白质和矿物质含量也非常高,不仅解决了人类营养饮食问题,还促进了饲牧业的发展,所以深受中国人民喜爱[21]。然而在优质小米受到市场追捧,价格一路攀升的市场背景下,谷子生产情况却不尽人意呢?其中一个主要原因就是谷子平均产量比较低,一般谷子的亩产量在0.6~0.8 kg。当前如何提高谷子产量是育种工作者的重要任务。谷子的花序总体上像是圆锥状,谷子穗子的性状从下往上分为主轴、分枝、小穗和花,并且谷子的穗轴上有枝梗,枝梗分为三个等级,小穗主要长在第三级枝梗上,俗称穗码[24]。谷子的枝梗数和穗码数量与谷子的产量密切相关。因此研究调控与谷子穗发育相关MADS-box基因的功能对于提高谷子产量有重要的意义。为了研究调控谷子穗发育相关的基因,对发育初期的谷穗(幼穗)和成熟期的谷穗(成熟穗)进行了转录组测序分析。结果发现有30个MADS基因在谷子幼穗和成熟穗中表达,其中4个基因在幼穗中有较高表达量,6个基因在成熟穗中有较高表达量。另外荧光定量PCR分析发现,SiMADS44和SiMADS62在谷子幼穗的表达量高于成熟穗(见图 4)。有趣的是最近的一项研究表明SiMADS34(本文中SiMADS62)基因调控谷穗的发育,突变体谷子产量降低[25]。推测SiMADS44可能为另一个参与调控谷穗发育的基因,需要进一步的功能验证。
| [1] |
FOLTER S D, ANGENENT G G. Trans meets cis in MADS science[J]. Trends in Plant Science, 2006, 11(5): 224-231. DOI:10.1016/j.tplants.2006.03.008 (  0) 0) |
| [2] |
SMACZNIAK C, IMMINK R G, ANGENENT G C, et al. Developmental and evolutionary diversity of plant MADS-domain factors: Insights from recent studies[J]. Development, 2012, 139: 3081-3098. DOI:10.1242/dev.074674 (  0) 0) |
| [3] |
BECKER A, THEIßEN G. The major clades of MADS-box genes and their role in the development and evolution of flowering plants[J]. Molecular Phylogenetics and Evolution, 2003, 29: 464-489. DOI:10.1016/s1055-7903(03)00207-0 (  0) 0) |
| [4] |
SAEDLER H, BECKER A, WINTER K U, et al. MADS-box genes are involved in floral development and evolution[J]. Acta Biochimica Polonica, 2001, 48: 351-358. (  0) 0) |
| [5] |
PARENICOVA L, DE FOLTER S, KIEFFER M, et al. Molecular and phylogenetic analyses of the complete MADS-box transcription factor family in Arabidopsis: new openings to the MADS world[J]. Plant Cell, 2003, 15: 1538-1551. DOI:10.1105/tpc.011544 (  0) 0) |
| [6] |
ARORA R, AGARWAL P, RAY S, et al. MADS-box gene family in rice: genome-wide identification, organization and expression profiling during reproductive development and stress[J]. BMC Genomics, 2007, 8(1): 242. DOI:10.1186/1471-2164-8-242 (  0) 0) |
| [7] |
WEI B, ZHANG R, GUO J, et al. Genome-wide analysis of the MADS-box gene family in Brachypodium distachyon[J]. PLoS One, 2014, 9(1): e84781. DOI:10.1371/journal.pone.0084781 (  0) 0) |
| [8] |
JIAN M, YANG Y, LUO W, et al. Genome-wide identification and analysis of the MADS-box gene family in bread wheat (Triticum aestivum L.)[J]. PLoS One, 2017, 12(7): e0181443. DOI:10.1371/journal.pone.0181443 (  0) 0) |
| [9] |
LESEBERG C H, LI A, KANG H, et al. Genome-wide analysis of the MADS-box gene family in Populus trichocarpa[J]. Gene, 2006, 378: 84-94. DOI:10.1016/j.gene.2006.05.022 (  0) 0) |
| [10] |
WANG Y, ZHANG J, HU Z, et al. Genome-wide analysis of the MADS-box transcription factor family in solanum lycopersicum[J]. The International Journal of Molecular Sciences, 2019, 20(12): 2961. DOI:10.3390/ijms20122961 (  0) 0) |
| [11] |
LIU J, ZHANG J, ZHANG J, et al. Genome-wide analysis of banana MADS-box family closely related to fruit development and ripening[J]. Scientific Reports, 2017, 7(1): 3467. DOI:10.1038/s41598-017-03897-1 (  0) 0) |
| [12] |
MENG D, CAO Y, CHEN T, et al. Evolution and functional divergence of MADS-box genes in Pyrus[J]. Scientific Reports, 2019, 9(1): 1266. DOI:10.1038/s41598-018-37897-6 (  0) 0) |
| [13] |
ZHANG X, FATIMA M, ZHOU P, et al. Analysis of MADS-box genes revealed modified flowering gene network and diurnal expression in pineapple[J]. BMC Genomics, 2020, 21(1): 8. DOI:10.1186/s12864-019-6421-7 (  0) 0) |
| [14] |
KRIZEK B A, FLETCHER J C. Molecular mechanisms of flower development: an armchair guide[J]. Nature Reviews Genetics, 2005, 6: 688-698. DOI:10.1038/nrg1675 (  0) 0) |
| [15] |
LIU C, TEO Z W, BI Y, et al. A conserved genetic pathway determines inforescence architecture in Arabidopsis and rice[J]. Developmental Cell, 2013, 24(6): 612-622. DOI:10.1016/j.devcel.2013.02.013 (  0) 0) |
| [16] |
YOSHIDA A, SASAO M, YASUNO N, et al. TAWAWA1, a regulator of rice inflorescence architecture, functions through the suppression of meristem phase transition[J]. Proceedings of the National Academy of Sciences, 2013, 110(2): 767-772. DOI:10.1073/pnas.1216151110 (  0) 0) |
| [17] |
LI P, ZHANG Q, HE D, et al. AGAMOUS-LIKE67 cooperates with the histone mark reader EBS to modulate seed germination under high temperature[J]. Plant Physiology, 2020, 184: 529-545. DOI:10.1104/pp.20.00056 (  0) 0) |
| [18] |
WU R, WANG T, WARREN B, et al. Kiwifruit SVP2 controls developmental and drought-stress pathways[J]. Plant Molecular Biology, 2017, 96: 233-244. DOI:10.1007/s11103-017-0688-3 (  0) 0) |
| [19] |
JIA J, ZHAO P, CHENG L, et al. MADS-box family genes in sheepgrass and their involvement in abiotic stress responses[J]. BMC Plant Biology, 2018, 18: 42. DOI:10.1186/s12870-018-1259-8 (  0) 0) |
| [20] |
DIAO X M. China's grain industry and industrial technology system[M]. Beijing: China's Agricultural Science and Technology Press, 2011.
(  0) 0) |
| [21] |
ZHANG G, LIU X, QUAN Z, et al. Genome sequence of foxtail millet (Setaria italica) provide sinsights into grasse volutionand biofuel potential[J]. Nature Biotechnology, 2012, 30(6): 549-554. DOI:10.1038/nbt.2195 (  0) 0) |
| [22] |
BENNETZEN J L, SCHMUTZ J, WANG H, et al. Reference genome sequence of the model plant Setaria[J]. Nature Biotechnology, 2012, 30(6): 555-561. DOI:10.1038/nbt.2196 (  0) 0) |
| [23] |
JIA G, HUANG X, ZHI H, et al. A haplotype map of genomic variations and genome-wide association studies of agronomic traits in foxtail millet (Setaria italica)[J]. Nature Genettic, 2013, 45: 957-961. DOI:10.1038/ng.2673 (  0) 0) |
| [24] |
PENSIERO J F, VEDETTI A C. Inflorescence typology in Setaria P. Beauv, (Poaceae, Paniceae)[J]. Feddes Repertorium, 2001, 112: 371-385. DOI:10.1002/fedr.4921120508 (  0) 0) |
| [25] |
HUSSIN S, WANG H, TANG S, et al. SiMADS34, an E-class MADS-box transcription factor, regulates inflorescence architecture and grain yield in Setaria italic[J]. Plant Molecular Biology, 2021, 105(4/5): 419-434. DOI:10.1007/s11103-020-01097-6 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


