真核生物RNA中存在多种甲基化修饰。在过去,大部分的修饰被发现存于高丰度的非编码RNA中。随着生物技术水平的提高,测序手段的改进,相对低丰度的信使RNA (Message RNA, mRNA)上的甲基化修饰也被陆续发现。m6A修饰是最为常见的一种动态甲基化修饰, 在真核细胞中,其含量占mRNA甲基化相关修饰含量的80%[1]。m6A修饰是指通过甲基化相关酶的作用,在RNA腺嘌呤的第6位氮原子上发生甲基化修饰,进而调控RNA代谢,包括mRNA前体的剪接、mRNA的核输出、3'非翻译区域序列(3'untranslated region, 3'UTR)加工、mRNA翻译、mRNA的稳定性等过程[2-4]。在模式植物拟南芥中,70%以上的mRNA存在m6A修饰,主要分布在mRNA终止密码子附近和3'UTR序列区域[5]。近年来,科学家们陆续对植物m6A修饰相关的酶进行了鉴定,并对m6A修饰在植物生长发育和植物抗逆境胁迫中的功能进行了研究[2, 4]。本文就m6A修饰相关酶的组成及其在植物生长发育和抗生物胁迫和非生物胁迫过程中的功能相关研究进展进行综述,同时对甘蓝型油菜m6A修饰相关的酶进行生物信息学分析。
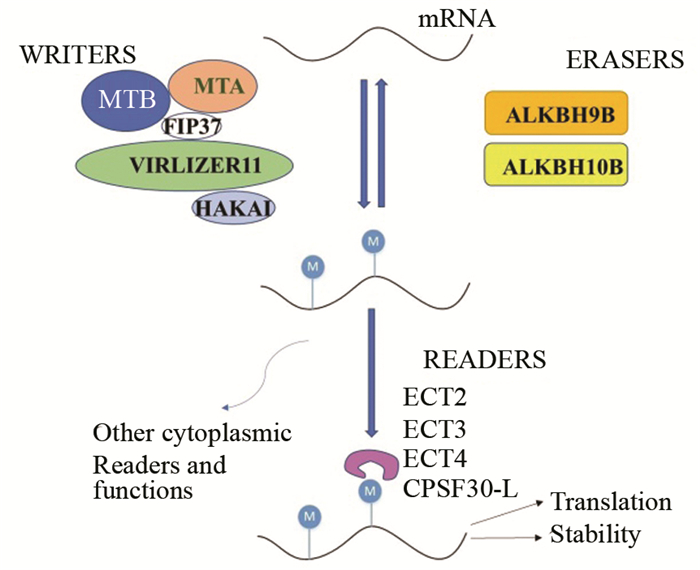
1 m6A修饰相关酶的组成m6A修饰相关的酶分为“writer”,“eraser”和“reader”三类。“writer”即m6A甲基转移酶,它以一个复合体的形式识别靶mRNA并进行甲基化。在动物体中该复合体由METTL3、METTL14、WTAP、KIAA1429和VIRILIZER等组分构成[6-11]。在模式植物拟南芥中m6A甲基转移酶包括MTA(METTL3人类同源蛋白)、MTB(METTL14人类同源蛋白)、FIP37(WTAP人类同源蛋白),VIRILIZER(KIAA1429人类同源蛋白)和E3泛素连接酶HAKAI等组分[12-14](见图 1)。“eraser”即m6A去甲基化酶。去甲基化酶FTO和ALKBH5都属于ALKBH家族,二者在动物体中先后被发现[15-17]。在模式植物拟南芥中与ALKBH5同源的去甲基化酶ALKBH9B和ALKBH10B已被证明能够去除m6A甲基化修饰[18-19](见图 1)。“reader”即m6A甲基化阅读蛋白,带有能够特异识别并绑定底物RNA的RNA绑定结构域。目前发现的阅读蛋白大部分都带有保守的YTH结构域,例如YTHDF1、YTHDF2和YTFDC1等[10, 20-21]。在拟南芥和水稻中分别鉴定到了13个和12个YTH结构域蛋白编码基因[22],其中拟南芥ETC2、ETC3、ETC4和CPSF30-L已被证明具有识别m6A甲基化修饰的功能[23-27](见图 1)。最近,科学家通过BLASTP和HMMER搜索,从22种代表性植物中鉴定到了159个m6A甲基转移酶组分,293个ALKB去甲基化酶同源蛋白家族成员,278个m6A阅读蛋白家族成员[2]。
|
图 1 拟南芥mRNA m6A甲基化修饰模式图 Figure 1 m6A methylation modification pattern of mRNA in Arabidopsis |
胚胎发生是高等植物发育过程中的一个重要环节。m6A修饰调控植物胚胎发育。拟南芥m6A甲基转移酶复合体组分FIP37敲除突变导致m6A修饰整体水平降低, 胚乳和胚胎发育延迟,fip37-4突变体表现出胚胎致死的表型[12, 28]。甲基转移酶复合体组分MTA突变也导致胚胎致死,使胚胎发育停滞在球形期[13, 29]。MTA在顶端分生组织、胚珠、花药和种子等组织器官中高效表达,mta突变体种子的mRNA m6A修饰整体水平降低[13]。MTA被证明在体外和体内都能够与FIP37互作[13],说明FIP37和MTA共同调控拟南芥的胚胎发育。拟南芥甲基转移酶复合体组分的另一成员VIR突变也导致胚胎表现出致死表型[14, 30]。最近的研究报道表明,水稻m6A甲基转移酶复合体的组分OsFIP和OsMTA2对孢子早期发生至关重要[31]。OsFIP能够介导一组苏氨酸蛋白酶和NTPase mRNA的m6A甲基化,并且在它们的表达和/或剪接过程中发挥重要作用。OsFIP敲除导致小孢子早期退化,前期I的减数分裂异常,进而影响育性[31]。以上这些研究表明,m6A修饰对胚胎和小孢子发育是必需的。
2.2 m6A修饰调控植物茎尖分生组织的分化及叶片的发生m6A修饰对顶端分生组织的分化具有重要调控作用。FIP37在茎尖、幼叶、花器官和种子等增殖活跃的组织器官以及茎尖分生组织中高效表达[12]。fip37-4突变体茎尖分生组织关键调控基因WUSCHEL (WUS) 和SHOOTMERISTEMLESS (STM) mRNA的m6A甲基化修饰水平降低,mRNA的丰度升高,因而幼苗茎尖分生组织过度增殖,叶片生成延迟。FIP37能够与STM和WUS的转录本直接结合,使它们的终止密码子和3'UTR区域序列发生m6A甲基化,从而加速转录本的降解; FIP37突变则导致STM和WUS mRNA的稳定性增强[12]。这说明FIP37可通过添加m6A修饰调控WUS和STM的mRNA稳定性,进而抑制顶端分生组织过度增殖。MTA也介导STM和WUS mRNA的m6A甲基化修饰,调控茎尖分生组织的增殖和发育[12]。这表明m6A甲基转移酶介导的m6A修饰对拟南芥茎尖分生组织的正常分化也是必需的。Li等分析水稻愈伤组织和叶片的RNA甲基化组,在愈伤组织和叶片中分别确定了1 792个和6 508个组织特异性的含m6A的基因[32]。
此外,m6A甲基化阅读蛋白ECT2、ECT3和ECT4被证明控制根、茎、叶及花组织的发生时间和叶片的形态[23, 33]。ECTs在茎尖、根尖以及叶片等组织器官原基中有特别高的表达,ect2-1 ect3-1 和ect2-3 ect3-2突变体产生第一片真叶的时间推迟[23],根茎的生长以及花器官的形成也延迟[33]。ect2/3/4突变体叶片和维管原基细胞分裂速率降低[33]。因此,m6A修饰对细胞分化、植物器官形成具有重要促进作用。
2.3 m6A修饰调控毛状体分支m6A修饰参与调控毛状体发育[23-24]。m6A甲基化阅读蛋白ECT2和ECT3在幼嫩的毛状体中高表达,ECT2或者ECT3突变导致毛状体分支数量显著增加,ect2 ect3双突变体毛状体的多分支表型更加明显,ECT4在此过程中则无明显调控作用[23]。拟南芥ECT2蛋白能够通过其YTH结构域与带有m6A修饰的mRNA结合[25]。ECT2结合靶mRNA的位点主要集中在3'UTR,调控mRNA 3'端加工和mRNA在细胞质中的稳定性[24]。ECT2缺失导致毛状体形态相关基因TTG1 (TRANSPARENTTESTA GLABRA1)、ITB1(IRREGULAR TRICHOMEBRANCH1) 和DIS2(DISTORTED TRICHOME 2)的转录本降解加速,丰度降低,从而使毛状体分支数量增加[24]。这表明ECT2通过识别m6A甲基化修饰,调节毛状体形态相关基因mRNA的稳定性,从而调控毛状体的形态结构。
MTA表达量下调或者FIP37过量表达也导致毛状体形态结构异常,致使毛状体分支数量增加到4-6个[28-29]。毛状体分支数与DNA倍性水平直接相关[28]。ect2突变体毛状体细胞中的DNA含量增加[25]。因此,m6A修饰还可通过控制毛状体细胞内DNA含量和倍性水平来影响毛状体分支。
2.4 m6A修饰调控植物开花m6A修饰参与调控植物开花。m6A去基甲化酶ALKBH10B突变导致拟南芥mRNA的m6A整体水平增加,开花推迟; 相反,ALKBH10B过量表达植物中mRNA的m6A整体水平则下降,植物开花提早[18]。alkbh10b-1突变体中开花关键基因FT (FLOWERING LOCUS T), SPL3 (SQUAMOSA PROMOTER BINDING PROTEIN-LIKE 3)和SPL9 mRNA的m6A修饰增加,稳定性下降,丰度降低,因而突变体开花时间推迟。m6A甲基化阅读蛋白ECT2, ECT3和ECT4也调控植物的开花时间[33]。ect2 ect3双突变体, ect2 ect3 ect4三突变体的开花时间推迟,但其调控机制尚不明确。
最近,CPSF30-L (Cleavage and Polyadenylation Specificity Factor 30)被证明属于拟南芥m6A甲基化阅读蛋白[26-27](见图 1)。CPSF30-L由CPSF30-S和m6A结合结构域YTH组成,主要通过YTH结构域识别mRNA上poly(A)位点信号序列FUE元件中的m6A修饰,招募聚腺苷酸化复合物,从而控制聚腺苷酸化位点的选择和3'端加工。CPSF30-L突变导致开花关键基因SOC1(SUPPRESSOR OF OVEREXPRESSION OF CONSTANS1),以及ABA信号相关基因RPN10(REGULATORY PARTICLE NON-ATPASE 10)和FYVE13 (FYVE-domain protein required for endosomal sorting 1)mRNA的聚腺苷酸化位点选择发生变化,致使3'UTR延长,转录本降解加速,因而cpsf30-l突变体的开花时间延迟,突变体的种子萌发和幼苗生长对ABA超敏性[27]。
此外,m6A修饰对维持植物昼夜节律具有重要作用[34]。m6A甲基转移酶vir-1突变体的昼夜节律周期延长,昼夜节律关键调节基因CCA1(CIRCADIAN CLOCK ASSOCIATED1)的表达量增加, 但具体调控机制并不清楚。
2.5 m6A修饰调控维管发育拟南芥m6A甲基转移酶复合体组分MTA、MTB、FIP37、VIRILIZER和E3泛素连接酶HAKAI都参与调控维管形成[14]。Vir-1、fip37-4、mtaABI3:MTA、MTB RNAi和hakai-1突变体都表现出维管发育缺陷,与野生型相比,突变体主根原生木质部的维管出现中断或者加倍的频率增加。因此,m6A整体水平降低会影响维管的正常发育,但具体调控机制尚不清楚。
2.6 m6A修饰调控果实成熟mRNA m6A甲基化状态调控典型呼吸跃变型番茄果实的成熟[35]。m6A是番茄果实mRNA的一个比较普遍的甲基化修饰,在终止密码子和3'UTR序列区域富集。番茄果实成熟缺陷突变体Colorless non-ripening (Cnr) 果实中,m6A去甲基化酶SlALKBH2的表达水平降低,果实mRNA的m6A整体水平升高。SlALKBH2能够与番茄成熟所必需的DNA去甲基化酶SlDML2的转录本结合。SlALKBH2突变导致SlDML2 mRNA的m6A水平增加,稳定性下降,丰度降低,因而果实成熟推迟。
典型的非呼吸跃变型草莓果实的成熟也受到m6A甲基化修饰的调节[36]。在草莓果实成熟初期,脱落酸(ABA)生物合成和信号通路相关的基因,包括NCED5(9-cis-epoxycarotenoid dioxygenase 5)、ABAR(putative ABA receptor)和AREB1(ABA-responsive element-binding protein 1)等的mRNA编码序列区域发生m6A超甲基化。m6A甲基转移酶MTA和MTB在果实成熟初期被诱导表达。MTA RNAi和MTB RNAi干扰突变体的果实成熟延迟,相反MTA和MTB过量表达果实成熟提早。MTA可直接与NCED5、ABAR和AREB1的转录本结合,MTA RNAi突变体中NCED5和AREB1转录本的降解加速,ABAR转录本的翻译效率降低。因此,MTA可通过对靶基因mRNA编码序列区域进行m6A修饰,增加mRNA的稳定性或者翻译效率,来促进果实成熟。
3 m6A修饰在植物抗逆境胁迫中的功能 3.1 m6A修饰调控生物胁迫响应m6A修饰参与调控病毒感染防御过程。研究报道,ALKBH9B与siRNA小体及P-小体(Cytoplasmic processing bodies, P-bodies)共定位,在胞质中聚集形成颗粒和细丝,表明ALKBH9B可能作为siRNA小体和P小体的新成员介导mRNA沉默或衰变过程[19]。ALKBH9B能够与苜蓿花叶病毒(AMV)外壳蛋白在细胞质中相互作用。ALKBH9B缺失可增加AMV RNA的m6A修饰丰度,降低病毒RNA水平,从而限制病毒RNA在植物各器官之间转移,进而减弱其对植物的系统侵染能力。这表明植物可通过去甲基化酶调控病毒m6A修饰水平,影响病毒的侵染能力。因此,m6A修饰可能是植物控制RNA病毒在胞质中繁殖的策略之一。
烟草花叶病毒(TMV)感染烟草叶片后,烟草叶片中m6A/G比值降低,m6A甲基转移酶组分如XM_009766347、XM_009766348和XM_009775897的表达量降低,与人ALKBH5同源的去甲基化酶XM_009801708的mRNA水平则升高[37]。这种m6A修饰的变化是由于病毒促进感染引起,还是由于宿主限制感染所致,或者是与宿主-病毒的相互作用有关?尚不明确。
3.2 m6A修饰调控非生物胁迫响应近来研究表明,大量盐、干旱或冷胁迫响应基因的mRNA被高度甲基化[12, 38-40]。Anderson等[38]研究表明,m6A修饰与盐胁迫响应紧密相关。与野生型相比,mta突变体中m6A修饰差异丰富的转录本主要编码盐胁迫响应相关蛋白。盐胁迫条件下,干旱、渗透和盐胁迫响应相关基因的转录本出现盐特异性的m6A修饰,mRNA丰度升高。研究人员还发现,没有被m6A修饰的靶位点上游4或5碱基处通常被剪切,从而导致转录本的稳定性降低。因此,研究人员提出MTA介导的盐胁迫特异性m6A修饰,可能抑制了修饰位点的剪切,使盐胁迫响应相关基因的转录本更加稳定,进而提高植物的抗盐胁迫能力。另有研究报道,m6A甲基转移酶MTA、MTB、VIR和FIP37的表达均受盐诱导,盐处理拟南芥幼苗mRNA的m6A整体水平升高[41]。vir-1、MTB RNAi和hakai突变体幼苗对盐胁迫敏感,幼苗和根的生长受抑制,幼苗的叶绿素含量、鲜重及存活率降低。VIR突变导致盐胁迫响应负调节子ATAF1、GI和GSTU17转录本的选择性多聚腺苷酸化发生变化,3’UTR变长,mRNA的稳定性增强,丰度升高。因此,m6A修饰可通过调节选择性多聚腺苷酸化和3’UTR的长度,从而调控盐胁迫负调节子转录本的降解和植物的抗盐胁迫能力。此外,m6A甲基化阅读蛋白ECT2在热应激下定位到细胞质应激颗粒(Stress Granules, SGs)中[25]。干旱和渗透胁迫应激下,ECT2和ECT4在细胞质中聚集, ECT3则较少发生聚集现象[23]。这种应激反应是否与植物的适应性相关,尚不清楚。
最近,科学家们对两种基因型甜高粱的耐盐性与m6A甲基化修饰之间的相关性进行了分析,发现m6A甲基化修饰在耐盐品种M-81E与盐敏感品种Roma之间存在显著差异,耐盐品种M-81E中5’UTR序列区域的m6A修饰高于Roma[39]。盐胁迫下,两种基因型甜高粱的m6A修饰都发生变化,尤其是盐敏感品种Roma耐盐基因转录本的m6A修饰增加,mRNA的稳定性增强,丰度增加。因此,m6A修饰的数量和程度可能是确定和评估农作物抗胁迫能力的重要因素。此外,干旱胁迫处理两个玉米近等基因系(耐旱Han21和干旱敏感B73)的转录组分析结果表明,干旱敏感近等基因系B73的m6A去甲基化酶ZmALKBH10a/ZmALKBH10b和m6A甲基化阅读蛋白ZmECTa/ZmeECTb编码基因的表达受干旱诱导,干旱胁迫下m6A修饰整体水平降低[40]。因此,m6A低度甲基化在干旱胁迫响应中可能起正调控作用。
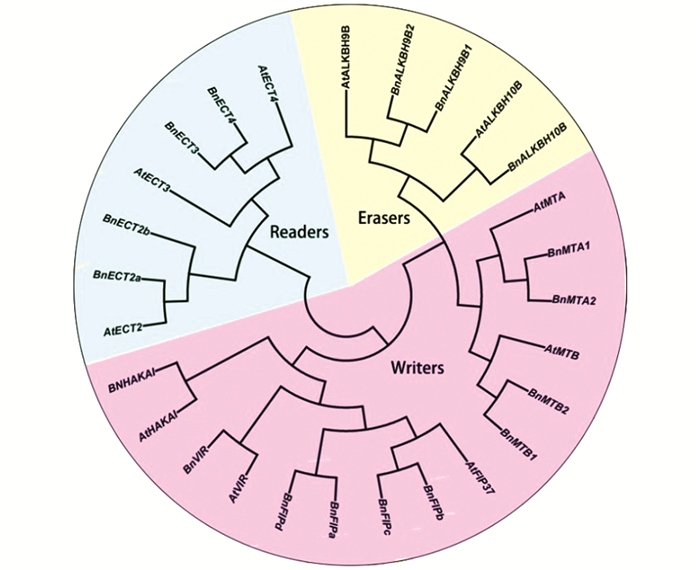
4 甘蓝型油菜m6A修饰相关酶的鉴定分析油菜是世界主要油料作物。油菜中m6A修饰相关酶及其功能尚未报道。采用BLAST方法(http://plants.ensembl.org/Multi/Tools/Blast),利用拟南芥m6A修饰相关酶的氨基酸序列在油菜数据库(The Brassicaceae Database (BRAD): http://brassicadb.cn)中进行比对,搜索甘蓝型油菜同源蛋白。通过序列比对,鉴定了甘蓝型油菜中FIP37、MTA、MTB、HAKAI、ALKBH9、ALKBH10和ECT蛋白家族(见表 1),并发现它们与拟南芥中的蛋白在进化上高度保守(见图 2)。
| 表 1 甘蓝型油菜和拟南芥中m6A甲基转移酶、去甲基化酶和甲基化阅读蛋白 Table 1 m6A writers, erasers, and readers in Brassica napus and Arabidopsis thaliana |
|
图 2 甘蓝型油菜和拟南芥m6A甲基转移酶、去甲基化酶及甲基化阅读蛋白进化分析 Figure 2 Phylogenetic relationship of m6A writers, erasers, and readers between Brassica napus and Arabidopsis thaliana |
在甘蓝型油菜中存在10个甲基转移酶组分,包括4个与AtFIP37同源的BnFIPa (BnaC08g25020D)、BnFIPb (BnaC06g15100D)、BnFIPc (BnaA09g34090D)和BnFIPd (BnaA07g16320D)(氨基酸序列一致性分别为90%, 89.6%, 88.8%和88.7%), 2个与AtMTA同源的BnMTA1 (BnaCnng25310D)和BnMTA2 (BnaA09g22350D)(氨基酸序列一致性分别为95.8%和95.5%), 2个与AtMTB同源的BnMTB1 (BnaA09g22720D)和BnMTB2 (BnaC09g24750D) (氨基酸序列一致性分别为97.1%和96.8%),1个与AtVIR同源的BnVIR (BnaA03g29130D)(氨基酸序列一致性为100%),1个与AtHAKAI同源的BnHAKAI(BnaCnng03840D)(氨基酸序列一致性为91.5%)(见图 2)。在甘蓝型油菜中含有3个m6A去甲基化酶,包括2个与AtALKBH9B同源的BnALKBH9B1 (BnaA07g02380D)和BnALKBH9B2 (BnaC07g06970D)(氨基酸序列一致性分别为88.1%和88.2%),1个与AtALKBH10B同源的BnALKBH10B(BnaA09g01150D)(氨基酸序列一致性为92.1%)(见图 2)。在甘蓝型油菜中含有4个和1个分别与AtECT2/3/4和AtCPSF30-L同源的m6A甲基化阅读蛋白(见图 2)。这为进一步研究m6A修饰在油菜生长发育中的功能奠定了基础。
5 m6A修饰相关的数据库及分析方法随着m6A测序技术的发展,越来越多的包含m6A修饰信息转录组数据在各项研究中被公开发表。而对于这些数据的整合与分析的需求促使各种类型的m6A数据库随之出现,为科研人员提供了便利的工具。m6A数据库有许多种类,其中一部分是针对转录组m6A修饰位点的数据库,还有一部分数据库包含m6A修饰相关的其他信息。m6A-Altas是一个用于解开m6A表观转录组的综合数据库。与现有其他数据库相比,m6A-Atlas具有高可信度的m6A位点集合,支持m6A的查询、可视化、共享以及下游过程(TF结合、RBP结合、微小RNA相互作用和剪接位点)和其他已知RNA修饰的注释信息[42]。m6A WER靶基因数据库(m6A2Target)是一个获取有关WER及其靶标的相关信息的全面、便捷的平台。它集成了WER及其目标的基本信息还有来自权威站点的基因注释信息。此外,m6A2Target还提供了有关WER与其靶标之间关联的信息,例如蛋白质-DNA、蛋白质-RNA、蛋白质-蛋白质相互作用、基因表达水平的变化、m6A修饰水平或翻译效率和可变剪接事件以及WER的扰动[43]。该数据库对m6A研究领域的功能和机制研究起着积极的促进作用。m6AVar (http://m6avar.renlab.org)是一个综合数据库,其中包含可能影响m6A修饰的m6A相关的变异。该数据库搜集了多种来源的人类和小鼠的m6A相关变异,通过检查它们是否位于具有RBP结合位点以及miRNA靶点和剪接位点来进一步确认其m6A修饰位点并进行注释、可视化和探索[44]。此外,来自GWAS和ClinVar数据库的疾病相关数据也被集成到m6AVar中,这使得用户可以利用此数据库探索m6A机制与疾病之间的潜在关系。m6Avar现在已经更名为RMVar (http://rmvar.renlab.org),相比原来增加了更多种类的RNA修饰变异的信息[45],更方便研究者使用其探究表观遗传学与疾病的关系。与此类似的还有RMBase v2.0 (http://rna.sysu.edu.cn/rmbase)。这是一个整合了表观转录组测序数据的综合数据库,用于探索RNA的转录后修饰及其与miRNA结合、疾病相关的单核苷酸多态性(SNP) 以及RNA结合蛋白(RBP) 的关系[46]。
6 展望迄今为止,科学家们陆续发现m6A修饰参与调控植物胚胎发生、茎尖分生组织分化、毛状体以及维管的正常发育。同时,在调控植物开花时间,应对病毒感染以及抗非生物胁迫方面也发挥重要作用。这些研究大多数都是表明m6A修饰影响植物自身的生长发育,但是关于m6A修饰如何对外界环境的变化做出响应,从而去调控植物的生长发育过程,目前的研究还较少,因此,研究m6A修饰水平与植物响应外界环境信号如光、温度等之间的联系是未来的一个研究方向。此外,m6A修饰编辑作为一种改变植物表观遗传的主要方式在作物育种方面的潜力也逐渐受到人们的重视[3]。因此,进一步鉴定重要农作物的m6A修饰相关酶,可为m6A修饰编辑提供新的特异靶点。
| [1] |
KIERZEK E, KIERZEK R. The thermodynamic stability of RNA duplexes and hairpins containing N6-alkyladenosines and 2-methylthio-N6-alkyladenosines[J]. Nucleic Acids Research, 2003, 31(15): 4472-4480. DOI:10.1093/nar/gkg633 (  0) 0) |
| [2] |
YUE H, NIE X, YAN Z, et al. N6-methyladenosine regulatory machinery in plants: composition, function and evolution[J]. Plant Biotechnology Journal, 2019, 17(7): 1194-1208. DOI:10.1111/pbi.13149 (  0) 0) |
| [3] |
ZHENG H, SUN X, ZHANG X, et al. m6A editing: new tool to improve crop quality?[J]. Trends in Plant Science, 2020, 25(9): 859-867. DOI:10.1016/j.tplants.2020.04.005 (  0) 0) |
| [4] |
ARRIBAS-HERNÁNDEZ L, BRODERSEN P. Occurrence and functions of m6A and other covalent modifications in plant mRNA[J]. Plant Physiology, 2020, 182(1): 79-96. DOI:10.1104/pp.19.01156 (  0) 0) |
| [5] |
WAN Y, TANG K, ZHANG D, et al. Transcriptome-wide high-throughput deep m6A-seq reveals unique differential m6A methylation patterns between three organs in Arabidopsis thaliana[J]. Genome Biology, 2015, 16(1): 272. DOI:10.1186/s13059-015-0839-2 (  0) 0) |
| [6] |
BOKAR J A, SHAMBAUGH M E, POLAYES D, et al. Purification and cDNA cloning of the AdoMet-binding subunit of the human mRNA (N6-adenosine)-methyltransferase[J]. RNA, 1997, 3(11): 1233-1247. DOI:10.1002/(SICI)1097-0134(199711)29:3<391::AID-PROT12>3.0.CO;2-I (  0) 0) |
| [7] |
LIU J, YUE Y, HAN D, et al. A METTL3-METTL14 complex mediates mammalian nuclear RNA N6-adenosine methylation[J]. Nature Chemical Biology, 2014, 10(2): 93-95. DOI:10.1038/nchembio (  0) 0) |
| [8] |
PING X, SUN B, WANG L, et al. Mammalian WTAP is a regulatory subunit of the RNA N6-methyladenosine methyltransferase[J]. Cell Research, 2014, 24(2): 177-189. DOI:10.1038/cr.2014.3 (  0) 0) |
| [9] |
SCHWARTZ S, MUMBACH M R, JOVANOVIC M, et al. Perturbation of m6A writers reveals two distinct classes of mRNA methylation at internal and 5' sites[J]. Cell Reports, 2014, 8(1): 284-296. DOI:10.1016/j.celrep.2014.05.048 (  0) 0) |
| [10] |
KAN L, GROZHIK A V, VEDANAYAGAM J, et al. The m6A pathway facilitates sex determination in Drosophila[J]. Nature Communications, 2017, 8: 15737. DOI:10.1038/ncomms15737 (  0) 0) |
| [11] |
HILFIKER A, AMREIN H, DUBENDORFER A, et al. The gene virilizer is required for female-specific splicing controlled by Sxl, the master gene for sexual development in Drosophila[J]. Development (Cambridge, England), 1995, 121(2): 4017-4026. DOI:10.1111/j.1365-2303.1995.tb00491.x (  0) 0) |
| [12] |
SHEN L, LIANG Z, GU X, et al. N6-Methyladenosine RNA modification regulates shoot stem cell fate in Arabidopsis[J]. Developmental Cell, 2016, 38(2): 186-200. DOI:10.1016/j.devcel.2016.06.008 (  0) 0) |
| [13] |
ZHONG S, LI H, BODI Z, et al. MTA is an Arabidopsis messenger RNA adenosine methylase and interacts with a homolog of a sex-specific splicing factor[J]. The Plant Cell, 2008, 20(5): 1278-1288. DOI:10.1105/tpc.108.058883 (  0) 0) |
| [14] |
RUZICKA K, ZHANG M, CAMPILHO A, et al. Identification of factors required for m6A mRNA methylation in Arabidopsis reveals a role for the conserved E3 ubiquitin ligase HAKAI[J]. New Phytologist, 2017, 215(1): 157-172. DOI:10.1111/nph.14586 (  0) 0) |
| [15] |
JIA G, FU Y, ZHAO X, et al. N6-Methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO[J]. Nature Chemical Biology, 2011, 7(12): 885-887. DOI:10.1038/nchembio.687 (  0) 0) |
| [16] |
ZHENG G, DAHL J A, NIU Y, et al. ALKBH5 is a mammalian RNA demethylase that impacts RNA metabolism and mouse fertility[J]. Molecular Cell, 2013, 49(1): 18-29. DOI:10.1016/j.molcel.2012.10.015 (  0) 0) |
| [17] |
JIA G, FU Y, HE C. Reversible RNA adenosine methylation in biological regulation[J]. Trends in Genetics, 2013, 29(2): 108-115. DOI:10.1016/j.tig.2012.11.003 (  0) 0) |
| [18] |
DUAN H, WEI L, ZHANG C, et al. ALKBH10B is an RNA N6-Methyladenosine demethylase affecting Arabidopsis floral transition[J]. The Plant Cell, 2018, 29(2): 2995-3011. DOI:10.1016/j.tig.2012.11.003 (  0) 0) |
| [19] |
MARTÍNEZ-PÉREZ M, APARICIO F, LÓPEZ-GRESA M P, et al. Arabidopsis m6A demethylase activity modulates viral infection of a plant virus and the m6A abundance in its genomic RNAs[J]. Proceedings of the National Academy of Sciences, 2017, 114(40): 10755-10760. DOI:10.1073/pnas.1703139114 (  0) 0) |
| [20] |
WANG X, ZHAO B S, ROUNDTREE I A, et al. N6-methyladenosine modulates messenger RNA translation efficiency[J]. Cell, 2015, 161(6): 1388-1399. DOI:10.1016/j.cell.2015.05.014 (  0) 0) |
| [21] |
XIAO W, ADHIKARI S, DAHAL U, et al. Nuclear m6A Reader YTHDC1 regulates mRNA splicing[J]. Molecular Cell, 2016, 61(4): 507-519. DOI:10.1016/j.tig.2016.03.006 (  0) 0) |
| [22] |
LI D, ZHANG H, HONG Y, et al. Genome-wide identification, biochemical characterization, and expression analyses of the YTH domain-containing RNA-binding protein family in Arabidopsis and Rice[J]. Plant Molecular Biology Reporter, 2014, 32(6): 1169-1186. DOI:10.1007/s11105-014-0724-2 (  0) 0) |
| [23] |
ARRIBAS-HERNÁNDEZ L, BRESSENDORFF S, HANSEN M H, et al. An m6A-YTH module controls developmental timing and morphogenesis in Arabidopsis[J]. The Plant Cell, 2018, 30(5): 952-967. DOI:10.1105/tpc.17.00833 (  0) 0) |
| [24] |
WEI L, SONG P, WANG Y, et al. The m6A reader ECT2 controls trichome morphology by affecting mRNA stability in Arabidopsis[J]. The Plant Cell, 2018, 30(5): 968-985. DOI:10.1105/tpc.17.00934 (  0) 0) |
| [25] |
SCUTENAIRE J, DERAGON J, JEAN V, et al. The YTH domain protein ECT2 is an m6A reader required for normal trichome branching in Arabidopsis[J]. The Plant Cell, 2018, 30(5): 986-1005. DOI:10.1105/tpc.17.00854 (  0) 0) |
| [26] |
HOU Y, SUN J, WU B, et al. CPSF30-L-mediated recognition of mRNA m6A modification controls alternative polyadenylation of nitrate signaling-related gene transcripts in Arabidopsis[J]. Molecular Plant, 2021, 14(4): 688-699. DOI:10.1016/j.molp.2021.01.013 (  0) 0) |
| [27] |
SONG P, YANG J, WANG C, et al. Arabidopsis N6-methyladenosine reader CPSF30-L recognizes FUE signals to control polyadenylation site choice in liquid-like nuclear bodies[J]. Molecular Plant, 2021, 14(4): 571-587. DOI:10.1016/j.molp.2021.01.014 (  0) 0) |
| [28] |
VESPA L, VACHON G, BERGER F, et al. The Immunophilin-interacting protein AtFIP37 from Arabidopsis is essential for plant development and is involved in trichome endoreduplication[J]. Plant Physiology, 2004, 134(4): 1283-1292. DOI:10.1104/pp.103.028050 (  0) 0) |
| [29] |
BODI Z, ZHONG S, MEHRA S, et al. Adenosine methylation in Arabidopsis mRNA is associated with the 3'end and reduced levels cause developmental defects[J]. Frontiers in Plant Science, 2012, 3(48): 1-10. DOI:10.3389/fpls.2012.00048 (  0) 0) |
| [30] |
TZAFRIR I, PENA-MURALLA R, DICKERMAN A, et al. Identification of genes required for embryo development in Arabidopsis[J]. Plant Physiology, 2004, 135(3): 206-1220. DOI:10.1104/pp.104.045179 (  0) 0) |
| [31] |
ZHANG F, ZHANG Y, LIAO J, et al. The subunit of RNA N6-methyladenosine methyltransferase OsFIP regulates early degeneration of microspores in rice[J]. PLOS Genetics, 2019, 15(5): e1008120. DOI:10.1371/journal.pgen.1008120 (  0) 0) |
| [32] |
LI Y, WANG X, LI C, et al. Transcriptome-wide N6-methyladenosine profiling of rice callus and leaf reveals the presence of tissue-specific competitors involved in selective mRNA modification[J]. RNA Biology, 2014, 11(9): 1180-1188. DOI:10.4161/rna.36281 (  0) 0) |
| [33] |
ARRIBAS-HERNÁNDEZ L, SIMONINI S, HANSEN M H, et al. Recurrent requirement for the m6A-ECT2/ECT3/ECT4 axis in the control of cell proliferation during plant organogenesis[J]. Development, 2020, 147(14). DOI:10.1242/dev.189134 (  0) 0) |
| [34] |
SIMPSON G G, BARTON G J, PARKER M T, et al. Nanopore direct RNA sequencing maps an Arabidopsis N6-methyladenosine epitranscriptome[J]. bioRxiv, 2019, DOI 10.1101/706002.
(  0) 0) |
| [35] |
ZHOU L, TIAN S, QIN G. RNA methylomes reveal the m6A-mediated regulation of DNA demethylase gene SlDML2 in tomato fruit ripening[J]. Genome Biology, 2019, 20(156): 1-23. DOI:10.1186/s13059-019-1771-7 (  0) 0) |
| [36] |
ZHOU L, TANG R, LI X, et al. N6-methyladenosine RNA modification regulates strawberry fruit ripening in an ABA-dependent manner[J]. Genome Biology, 2021, 22(186): 1-32. DOI:10.1101/2021.03.04.433875 (  0) 0) |
| [37] |
LI Z, SHI J, YU L, et al. N6-methyl-adenosine level in Nicotiana tabacum is associated with tobacco mosaic virus[J]. Virology Journal, 2018, 15(87): 1-10. DOI:10.1186/s12985-018-0997-4 (  0) 0) |
| [38] |
ANDERSON S J, KRAMER M C, GOSAI S J, et al. N6-Methyladenosine inhibits local ribonucleolytic cleavage to stabilize mRNAs in Arabidopsis[J]. Cell Reports, 2018, 25(1): 1146-1157. DOI:10.1186/s12985-018-0997-4 (  0) 0) |
| [39] |
ZHENG H, SUN X, LI J, et al. Analysis of N6-methyladenosine reveals a new important mechanism regulating the salt tolerance of sweet sorghum[J]. Plant Science, 2021, 304(110801): 1-14. DOI:10.1016/j.plantsci.2020.110801 (  0) 0) |
| [40] |
MIAO Z, ZHANG T, QI Y, et al. Evolution of the RNA N6-methyladenosine methylome mediated by genomic duplication[J]. Plant Physiology, 2020, 182(1): 345-360. DOI:10.1104/pp.19.00323 (  0) 0) |
| [41] |
HU J, CAI J, PARK S J, et al. N6-Methyladenosine mRNA methylation is important for salt stress tolerance in Arabidopsis[J]. The Plant Journal, 2021, 106(6): 1759-1775. DOI:10.1111/tpj.15270 (  0) 0) |
| [42] |
TANG Y, CHEN K, SONG B, et al. m6A-Atlas: a comprehensive knowledgebase for unraveling the N6-methyladenosine (m6A) epitranscriptome[J]. Nucleic Acids Research, 2021, 49: D134-D143. DOI:10.1093/nar/gkaa692 (  0) 0) |
| [43] |
DENG S, ZHNAG H, ZHU K, et al. M6A2Target: a comprehensive database for targets of m6A writers, erasers and readers[J]. Briefings in Bioinformatics, 2020, 22(3): 1-11. DOI:10.1093/bib/bbaa055 (  0) 0) |
| [44] |
ZHENG Y, NIE P, PENG D, et al. m6AVar: a database of functional variants involved in m6A modification[J]. Nucleic Acids Research, 2017, 46: D139-D145. DOI:10.1093/nar/gkx895 (  0) 0) |
| [45] |
LUO X, LI H, LIANG J, et al. RMVar: an updated database of functional variants involved in RNA modifications[J]. Nucleic Acids Research, 2020, 49: D1405-D1412. DOI:10.1093/nar/gkaa811 (  0) 0) |
| [46] |
XUAN J, SUN W, LIN P, et al. RMBase v2.0: deciphering the map of RNA modifications from epitranscriptome sequencing data[J]. Nucleic Acids Research, 2017, 46: D327-D334. DOI:10.1093/nar/gkx934 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


