2. 福建省海洋酶工程重点实验室,福州 350116;
3. 福州大学 生物科学与工程学院,福州 350100
2. Fujian Key Laboratory of Marine Enzyme Engineering, Fuzhou 350116, China;
3. College of Biological Science and Engineering, Fuzhou University, Fuzhou 350100, China
蛋白质是细胞生命活动的主要承担者之一,是生物生长发育、遗传繁殖以及行使生物学功能的主要物质基础[1]。蛋白质酶是一种重要的生物分子,在科学研究以及工业应用中都扮演着重要的角色[2]。近十几年来,随着蛋白质和酶序列数据的大规模快速增长,其功能分析以也从传统分子生物学实验转向实验结合计算机技术协同进行的路径,以此来结合实验和计算模拟的双重优势,以期更好地探索酶以及其他生物分子相应的生物分子机制[3]。随着蛋白质和酶功能预测方法的研究得到广泛关注,相应的计算和分析方法在生物信息学领域持续不断发展和应用。例如,基于三维空间结构和功能的基础知识对酶分子进行设计和改造也是近几年的研究热点之一[4]。酶分子的设计有两个重要的研究方向:提高酶的亲和力以及稳定性。酶的亲和力一般是指酶和底物的结合能力,也就是酶的催化活力。酶的稳定性包括热稳定性、以及耐酸耐碱性等。在一些经典的酶分子设计案例中,新突变体可以同时优化酶的活力和稳定性。在我们课题组的前期工作中,分别在两个实例中对特定的酶进行改造,提高了酶的催化活力[5],以及提高了酶的热稳定性[6]。
酶与蛋白质的三维空间结构是分析其功能的重要信息之一。通常来说,酶和蛋白质的三维结构在很大程度上决定了其生物学功能。在PDB结构数据库[7]中,不少酶分子含有两条或多条多肽链,每一条多肽链通常都有完整的三级结构,称为酶结构的亚基(Subunit)。亚基之间的结合力主要是疏水作用,其次是氢键和离子键等作用力。酶分子中各亚基的空间排布及亚基接触部位的布局和相互作用称为酶的四级结构。酶功能的分析和其四级结构有着紧密联系。在结构生物学界,通常使用X射线衍射法、核磁共振以及电镜等实验手段来解析其结构以及测定相应功能,但结构生物学的实验成本太高以及耗时太长,进而难以开发出高通量的结构与功能的实验测定方法。从总体上看,酶功能分析从上个世纪以来一直是生物信息学家们面临的巨大科学挑战之一,科学家们付出了巨大努力进行探索,进展十分迅速但是仍然存在不少科学问题亟待解决。
简而言之,数以万种的物种已实现顺利测序,而相应的蛋白质和酶序列却没有得到高质量的功能注释,若直接投入湿实验分析需要投入大量的人力、经费及时间等资源[8]。因此,正逢高性能计算技术的飞速发展,学术界与工业界皆希望借助计算机与模拟技术来实现蛋白质以及酶的结构与功能自动分析与预测,以取代繁琐的手工预测或者成为现有人工注释的互补性方法。这已成为新兴的生物信息学科的重要研究方向之一。科学家们正在使用序列搜索、结构匹配和机器学习等尖端技术,通过结构已知的蛋白质和未知蛋白质的对比分析等方法来预测和分析未知酶和蛋白质的结构以及功能特点,从而一定程度上解决酶功能分析的难题。基于此,本文对酶功能分析与预测方法进行系统性总结。
1 蛋白质功能预测的主要方法随着国际上对蛋白质和酶功能的持续关注,国际蛋白质功能预测竞赛(Critical Assessment of Function Annotation,CAFA)应运而生。CAFA的竞赛网站为https://www.biofunctionprediction.org/cafa/。CAFA竞赛的目标是大批量地测试现有计算方法预测功能的性能,主要是系统性地测试不同算法预测GO(Gene Ontology)词条的能力。GO词条预测一般包括分子功能、细胞过程以及细胞组分等预测和评价。另外,CAFA竞赛也系统性测试不同方法对结合位点(Binding site)的预测性能。CAFA竞赛设计原则为对不同方法非偏向性(Unbiased)的评价。这个竞赛内容包括两个类型:1)真核生物类型的预测;2)原核生物类型的预测。不同的类型都提供一定的测试样本,参与者在规定时间内对样本进行预测并得到结果反馈给组织者,组织者将根据一定的指标对结果进行评价。CAFA竞赛中对蛋白质功能分析的方法可以归为以下几类[9]:
1.1 基于同源性的功能预测方法这种方法的理论基础是同源蛋白质以及酶通常具有相似的功能。由大量分子生物学研究可知,序列相似的蛋白以及酶具有较高的同源性具有类似的生物学功能。
一般而言,可以使用BLAST[10]和CLUSTAL[11]等比对工具找寻潜在的同源序列,从而进行蛋白质或者酶同源性的比对及识别。其主要方法是通过上述序列对比工具寻找与未知功能的蛋白质或酶相近的序列,从而把已知酶(或者蛋白质)序列的功能映射到未知功能的酶序列上, 这样完成基于序列比对的功能信息注释。
但需要注意的是,紧密相关的酶并非总是具有相同的功能,例如旁系同源酶可以进化出差距较大的功能,故而同源性方法虽然简单但需要有较多的专家或者背景知识介入。一般而言,这种方法比较适用于与功能已知的酶有较高同源性的新酶的功能预测,在序列保守区稳定的酶功能分析中也可以使用这个方法。并随着序列之间相似性的降低,依靠序列相似性建立起来模型的误差可能将增加。现如今,还没有明确的序列相似性阈值可用于界定酶功能的相似性。
在图 1中,列出了对三条漆酶(Laccase)包括1KYA、3PXL以及3X1B序列的多序列比对结果。这里的多序列比对由CLUSTAL软件产生,并通过Bioedit[12]软件编辑。文献中报道漆酶中有10个组氨酸(His)会与4个铜原子进行相互作用[13],因此漆酶中一般有10个组氨酸为保守氨基酸。在这个例子中,我们通过多序列比对结果分析,找出了与铜离子相互作用的10个组氨酸(图 1中红色三角形标注位置)。从图中可以看出,三条漆酶的序列是不太相似的,但是10个保守的组氨酸功能位点恰好比对上。通过本例进一步论证了可以通过序列比对寻找酶中的保守和关键区域。在这个多序列比对中,虽然其他位置也有共同的组氨酸比对上,但是其他位置的组氨酸并非是铜离子相互作用的组氨酸。通过序列寻找同源序列以及分析功能目前已经是学术界通用的一种方法。
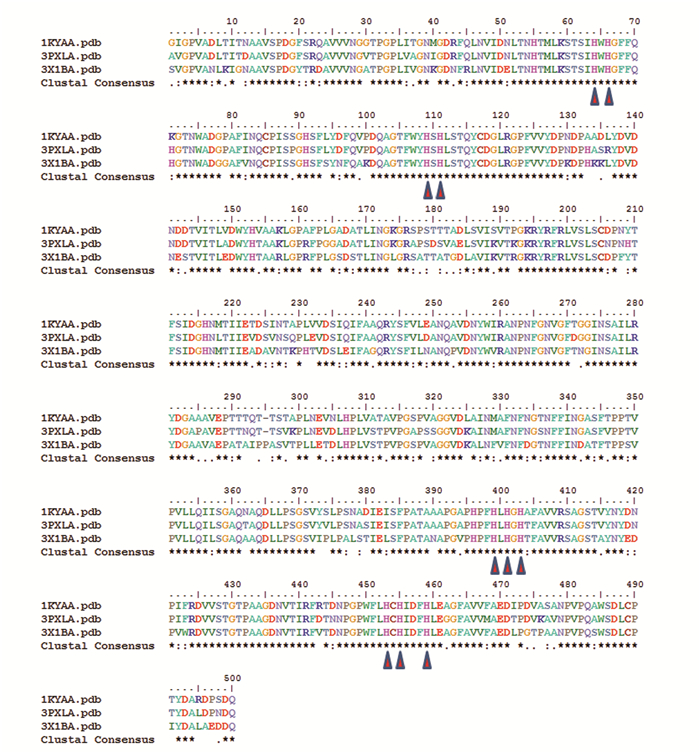
|
图 1 3个漆酶序列的多序列比对 Figure 1 Multiple sequence alignment of sequences of three laccases |
基序有时也称为模体,一般是指蛋白质结构域(Domain)中表示特定氨基酸序列的亚单元,是与特定的功能相关的多肽短片段,并体现结构域的多种生物学作用。序列基序是生物序列中具有一定生物学功能的短片段,具有生物学意义的多重序列中出现的子序列。序列基序可以是完全不变的,也可以是部分非保守区域可变的。编程中的正则表达式与生物序列中的基序有类似的概念,不少数据库(例如Prosite[14])采用正则表达式进行基序表示。
基序可以用来预测蛋白质在细胞中合成后被送到哪里,即蛋白质的亚细胞定位。总的来说,基于基序的酶功能预测分析方法可以归为一种序列局部特征分析的方法。序列局部特征分析方法又称为从头预测方法,该方法不依赖于蛋白质序列的全局比对,而是通过统计一组已知的具有相同功能的蛋白质序列的组成及生化特性等局部特征并建立分类模型,将具有相近或相似特征的序列看作同源序列再划为同一类,从而进行蛋白质和酶功能预测。
一些数据库,例如Pfam[15](Protein Families Database) 以及Prosite,可以用于分析蛋白质以及酶的结构域和基序, 这为深入分析酶功能分析提供一定的依据。考虑到序列突变以及随机性的情况,通过多序列比对识别基序可能比直接通过正则表达式匹配的方法更可靠,可以获得更少的假阳性。因为,多序列比对可以分析多条酶序列之间共同的保守区域和模体,这样得到的结果通常更加可靠。若一个模体在同一个多序列比对的多个酶中同时出现,则其为真阳性的概率显然更高。
在图 2中,把蛋白质1N55(PDB号)的序列输入网站https://myhits.sib.swiss/cgi-bin/PFSCAN进行模体搜索,搜索到1N55序列中的AYEPLWSIGTG(序列位置为166-176)匹配上Prosite数据库中的模体PS00171,这与已有的文献报道基本一致[16]。图 2中这个模体对应的区域标注为红色。
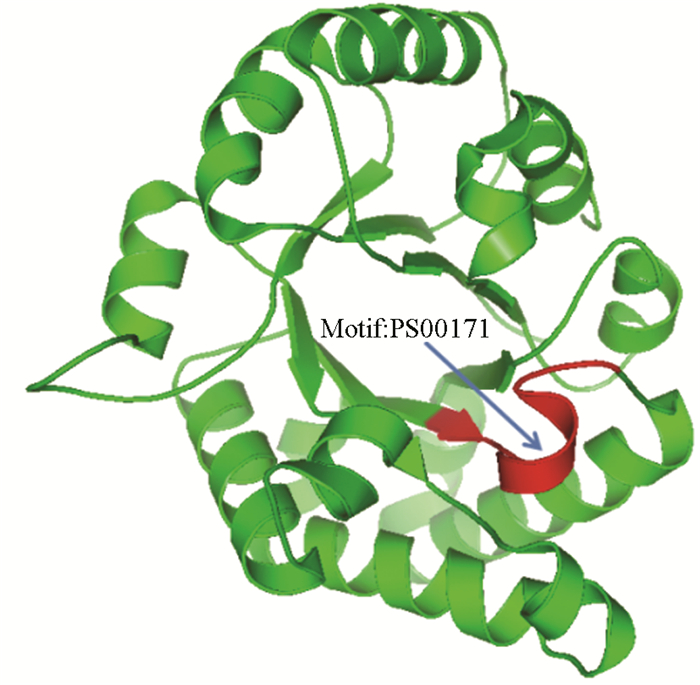
|
图 2 蛋白1N55中包含的一个功能模体PS00171 Figure 2 A functional motif PS00171 in 1N55 protein |
由于蛋白质三维结构(也包括酶的结构)通常比其序列更为保守,因此结构相似性是两种或两种以上酶功能相似的良好指标。针对许多酶没有实验结构的情况,可以采用预测的三维模型。在许多情况下,代表活性位点或结合位点的特定基序的三维结构可以被靶向结构比对,这种情况下可以不用整个蛋白质结构的信息。通过已知靶点的模板数据库的结构比对,可以使用新的蛋白质和酶结构来预测特定的功能位点。
结构比对可以用于判断两个蛋白质或者酶之间是否有相似的功能。在图 3中,列出了SCOPe[17]数据库中两个蛋白质d1w0ma_和d2vxna_之间结构比对的结果, 在图中d1w0ma_与d2vxna_分别以红色和绿色渲染。这两个蛋白(d1w0ma_和d2vxna_为SCOPe数据库中蛋白质的名称)同属于SCOPe数据库中的同一个家族(家族编号为c.1.1.1), 同属于磷酸丙糖异构酶。一般而言,在SCOPe数据库中属于同一个家族的蛋白之间,是具有结构以及功能上的相似性的。从图 3中,可以清晰地看出,这两个蛋白质之间结构是非常相似的。
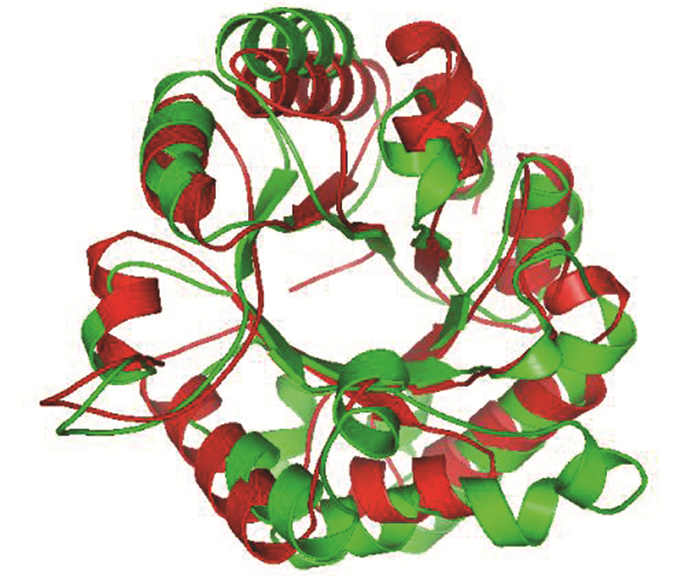
|
图 3 2个磷酸丙糖异构酶之间的结构比对 Figure 3 Structural alignment of two triose phosphate isomerases |
蛋白质结构分类(SCOPe)数据库主要是根据蛋白质结构域的结构和氨基酸序列的相似性对蛋白质结构域进行人工分类。这种分类的动机是确定蛋白质和酶的进化关系。
1.4 基于基因组背景的功能预测方法基于基因组背景的方法一般广泛应用于预测蛋白质以及酶的进化关系,也称为系统发育树的方法。两个或多个酶在多个物种的蛋白质组中频繁共同出现。这可能表示这些酶之间具有生物学功能上的关联性或者一致性,这与机器学习算法中的关联分析类似。同源性的方法可以用于识别蛋白的分子功能,而基因组背景的方法可以用于预测蛋白参与的细胞或者生物学过程。例如,参与物种中参与信号转导的蛋白可能具有相似的基因组背景。
1.5 基于机器学习的功能分析算法除了以上提到的4种方法之外,学术界还常常通过从序列层次上直接预测蛋白质或者酶的功能。这类方法一般直接通过机器学习[18]算法进行建模。这类方法一般可以分为以下几个主要步骤: 类标签定义、特征提取和筛选、训练模型和实际分类预测等。其中,类标签定义就是把功能预测问题转化为一个分类问题。特征提取,就是构造和酶功能分类相关的一些序列和结构参数,这个过程主要涉及序列特征的定义和提取,常用的特征包括氨基酸组成、二级结构、溶剂表面可及性、结构域、序列模体等;特征选择主要是对特征提取阶段提取的特征集进行数值化,同时去除噪声和去冗余等操作。这部分的内容在数据建模过程中称为特征工程;之后,利用机器学习方法建立一个分类模型,并对模型的准确率进行评估,最终使用该模型对未知功能的酶序列进行功能预测。
这类基于机器学习的算法与前面四种方法有一定的互补性。然而这类方法也有一定的局限性,特定分类问题的特征选取策略、以及正负样本比例选取策略对分类模型的性能都有一定的影响。在实际建立分类模型时常常发现,已知的具有某一功能的酶序列较少(正样本)、已知的不具有该功能的蛋白质序列不确定,训练模型时正样本和负样本集比例不平衡对模型的实际预测性能的影响也比较大。另外,不少酶可能同时具有多个功能,简单将功能预测问题看成二分类或者简单多类分类问题,会忽略个体酶功能多样性的特点。
2 通过分子对接分析酶与底物相互作用一般可以通过结构分析、模板信息以及文献调研等方式分析出酶的结合位点(Binding sites), 之后结合活性口袋信息进行进一步的分子对接(Molecular docking)。
分子对接是在找到酶结合位点后进一步分析其与底物相互作用的一种方法,也就是通过计算模拟的方法推测出酶与底物之间的构象关系。分子对接是分子模拟的重要方法之一,是两个或多个分子之间通过空间匹配和能量匹配的相互识别,形成分子复合物,并对复合物结构进行预测的过程。其核心概念是对受体和配体分子形成复合结构进行预测。分子对接方法在药物设计、材料设计等领域均具有广泛的应用。且由于在整体上考虑配体与受体的结合效果,可以较好地避免了局部作用优、整体欠佳的情况。
配体与受体相互作用是分子识别的过程,主要包括静电作用、氢键作用、疏水作用、范德华作用等。通过计算,可以预测两者间的结合模式和亲和力,从而进行药物的虚拟筛选。从对接方法的角度来,分子对接可以分为以下三类:1)刚性对接;2)半柔性对接;3)全柔性对接。刚性对接是指在对接的过程中,参与对接的受体与配体分子构象不发生变化,仅改变分子的空间位置与姿态。如锁匙模型,受体和配体均为刚性,进行刚性对接。刚性对接通常用于考察大分子之间的对接,如蛋白质-蛋白质(例如,酶与抑制剂之间的对接)之间的作用和蛋白质-核酸之间的作用。半柔性对接是指允许对接过程中配体(例如,小分子)构象发生一定程度的变化,但通常会固定大分子的构象,也即受体刚性,配体柔性。全柔性对接是指允许研究体系的构像发生自由变化,由于变量随着体系的原子数呈几何级数增长,因此柔性对接方法的计算量非常大,消耗计算机时很多,适合精确考察分子间识别情况。如诱导契合模型,其受体和配体均为柔性。不少软件(例如Autodock vina[19])通过设置,可以进行半柔性或者全柔性对接。不过,由于全柔性的时间耗时长,所以在实际的全柔性对接中也仅仅选择受体上的几个关键氨基酸进行可以自由旋转的全柔性对接。表 1中列出了几个经典以及具有代表性的分子对接程序。
| 表 1 几种代表性的分子对接程序 Table 1 Several representative molecular docking programs |
分子对接在生物信息学中有诸多的应用。计算机辅助药物设计的方法学可以分为基于配体的药物设计和基于受体的药物设计。基于受体的药物设计方法是通过受体的特征及受体和药物之间的相互作用方式来进行药物设计的方法。主要方法为分子对接和从头设计。分子对接的应用范围包括:1)虚拟筛选;2)机理解释;3)先导优化。一般可以通过分子对接进一步深入地分析蛋白质酶的生物学功能。分子对接模型最早可以追溯到1894年Fischer提出的锁匙模型。锁和钥匙相互识别的首要条件是它们在空间形状上要完美匹配,但药物分子和靶蛋白之间的识别比锁匙模型更为复杂,再结合过程中,受体和配体需要适应对方,已达到诱导契合。实验和计算得到的模型与生物体中真实的蛋白结构都存在一定误差,因此在对接过程中的一些区域采取柔性对接,这样处理有可能修正相应的结构误差。
“酶-蛋白质”与“酶-核酸”大分子间的对接最常用的为Zdock[20]和Rosetta[21]等程序。这些程序可以从单个酶结构出发,用计算的手段确定酶与底物的复合物的空间结构。Zdock[20]是一种基于快速傅里叶变换的刚性蛋白对接程序,由波士顿大学Weng研究组创建,Weng研究组相继开发出优化程序RDOCK和单独的打分程序ZRANK。Zdock采用快速傅里叶转化,根据蛋白质间形状匹配(去溶剂作用、静电作用)搜索分子间结合模式的刚性对接。主要预测领域为:蛋白-蛋白相互作用;抗原抗体相互作用;酶-DNA相互作用的结合预测等。
Zdock软件的使用流程如下:产生对接构象,使用Zdock软件进行打分,形状互补性越好,打分越高;过滤聚类对接构象,通过计算静电、范德华力、去溶剂化能等,使用ZRANK对对接构象进行冲打分,能量值越低,结构越稳定,得分越低。
FlexPepDock [22]是基于Rosetta软件包中开发出的一种蛋白质与多肽对接方法,这种方法可以提供一种高分辨率的多肽分子对接。FlexPepDock方法的网站为http://flexpepdock.furmanlab.cs.huji.ac.il/index.php。Autodock vina算法主要用于大分子和小分子对接(例如,酶和底物之间的对接)。分子对接完成之后,可以分析酶和底物之间的相互作用关系。例如可以通过Ligplus[23]等软件进行相互作用的可视化以及分析等。
对于分子对接而言,可以分为酶-小分子、酶与其他大分子、以及酶与多肽之间的对接过程。由于对接体系的不同,由于使用到的能量函数不同,所以研究人员需要选择特定的程序进行对接。目前还未形成一个统一的能量体系或者计算公式,以及未存在可以智能地选择特定对接能量体系的程序。另外,对接过程中,界面氨基酸(Interface residues)以及结合口袋的选择往往是决定对接结果与实验结果是否接近的一个关键因素,目前这个部分主要依靠科研人员根据实验背景以及经验选择,所以具有一定的不确定性以及可以改进的空间。
3 通过分子动力学模拟进一步深入解释酶作用机理分子对接得到的复合物(例如酶和底物的复合物)是一种静态的结构。另外,结构生物学目前通过实验手段大多数情况下也只能获得静止的蛋白质和酶的三维结构构象。为了深入揭示相应生物分子机制,就要不断获得中间态的三维结构构象信息,获得的这种情况的三维动态构象越多,相应分子机制则解释得越精准及深入。分子动力学就是通过计算机模拟得到这种中间态构象的一种计算模拟方法。
分子动力学模拟一般主要分为体系准备、能量最小化、加热、平衡、采样以及后续动态结构分析等步骤。动力学模拟是研究酶的作用机制的重要方法之一。常用的分子动力学方法有Amber[24]、Gromacs[25]以及Namd[26]等。为了更深入地表示蛋白和氨基酸残基的相互作用,一般文献中会用相互作用能量和氨基酸残基数的一个关系图表示,可以把每个残基对结合能的贡献算出来。每个分子结构不是静止的,基本都像个小机器,有一定的变化,通过动力学模拟的方法可以一定程度上分析这种动态状态。虽然经过几十年的发展,分子动力学模拟还是存在一些待改进的方面和可持续发展的方向。例如,大多数的分子动力学模拟软件包的命令行数量较多以及使用也相对复杂,另外模拟时间一般较长,对计算机的软件和硬件要求都相对较高。未来若能开发出使用更加简单易用的分子动力学模拟软件,也许是可以让更多实验科学使用分子动力学方法的可行途径之一。
在酶机理的解释中,分子动力学模拟往往是关键的一个重要步骤。但是动力学模拟由于计算量大以及后续的分析需要较为专业的计算模拟基础知识,对于实验科学家而言,应用起来有一定的门槛。更重要的是,由于计算量大,目前学术界较为缺少可以直接提交数据就可以进行动力学模拟的网站和服务器。生物信息学界开发并提供更多的可以直接提交数据就可以全自动进行动力学模拟的程序和网站也许是具有发展前景的一个重要方面。
4 酶分子设计是功能分析的实际应用学术界分析以及预测酶的功能是为了进一步改进其功能。酶分子设计是酶工程和科学研究的主要内容之一。酶工程就是科学家们在了解酶空间结构及其功能关系,并且在熟练掌握基因操作技术的基础上,设计和改造酶,借以改善酶的物理和化学性质以满足特定的科学研究以及工业应用的需求。例如,提高酶的热稳定性、酶的专一性等,使之更好地为人类所用。酶分子设计就是为有目的的酶质改造提供设计方案。按照改造部位多与少可分为三类:小改、中改以及大改。小改一般是指可通过几个位点的定位突变或化学修饰来实现;中改一般是对来源于不同蛋白的结构域进行拼接组装以达到酶性质的优化组合;大改则是完全从头设计新的酶。提高酶的亲和力是酶分子设计的一个重要方面。一般可以把一些非关键氨基酸突变为丙氨酸或者甘氨酸,以减少小分子进入相应位置的空间阻力。另外,考虑到酶中与底物距离较近的氨基酸可能为关键氨基酸。所以学术界常常以分子对接结果为输入;在对接结果的复合物中,选择底物(通常为小分子)3-5Angstrom范围的氨基酸;把选择的氨基酸突变为另外19种氨基酸重新计算结合能量,若所计算得到的结合能变小,那么这个突变则可以通过实验的方法进一步验证。提高酶的热稳定性是酶分子设计的另外一个重要方向。对提高酶热稳定性有一个经验性策略: 把远离酶活性位点的无规则卷曲(LOOP)区域的甘氨酸突变为谷氨酸。这个策略的主要原理为谷氨酸相比甘氨酸刚性更强,以及可以形成更多的分子键。另外,可以考虑把一些空间距离比较近的氨基酸同时突变为CYS让酶形成更多二硫键。在没有合适的方案情况下,也可以采用随机突变的方法寻找合适的突变位点,一般可以通过易错PCR的实验方式进行随机突变。有时为了同时提高亲和力和热稳定性,会采用双靶点或者多靶点共同改进以提高酶的性能。
5 总结酶功能分析中一般综合利用基于同源性方法、模体、三维空间结构、基因组以及蛋白质组上下文、文献调研以及实验等进行酶功能的基础分析。之后分析复合物结构,根据相应体系选择合适的对接程序进行复合物结构的构象预测。分子对接完成之后,利用分子动力学模拟算法进行深入分析和机理解释。酶是一个复杂的体系,不少酶具有多靶点、整体性以及动态性等特点。酶行使功能过程中一般由多个部分协同发挥作用,重要和不重要的区域都只是相对而言的。
分子对接可以分析酶与底物的相互作用关系。动力学模拟可以更深入地解释相应的作用机理。简而言之,分子对接和动力学模拟在酶功能分析方面都有重要应用,但也都存在一定的可改进空间。功能分析是酶分子设计的基础。酶分子设计通常采用点突变或者改变相应氨基酸片段提高酶亲和力与热稳定性,这需要对酶结构的每个基团以及功能结构域系统分析和准确定位。酶分子设计时一般需要准确分析出活性位点,之后常常采用在重要区域(例如,Loop区)再设计提高其催化效率和亲和力。酶分子设计的策略还包括通过对底物、产物的分子通道和路径再设计来减少产物抑制,同时减少空间位阻来提高催化效率和亲和力;通过对蛋白质内部氢键、盐桥、二硫键再设计来改善蛋白质的热稳定性;采用多种方法拓宽蛋白质分子的最适温度和pH范围;若可以同时提高酶的热稳定性和亲和力则为优良的分子设计策略。
本文系统性地总结了酶功能分析和预测的主要方法,包括酶结合位点、分子对接、分子动力学模拟以及分子设计等。由于近些年来学术界在酶结构预测方面的持续进展,特别是AlphaFold等高精度结构预测方法的出现,可以让更多的实验人员依靠计算方法获得相对可靠的蛋白质结构。这让基于结构的酶功能分析成为未来发展的一种方法。综上所述,功能分析也许会是未来学术界对酶研究的一个重要方面。
| [1] |
GLASGOW A A, HUANG Y M, MANDELL D J, et al. Computational design of a modular protein sense-response system[J]. Science, 2019, 366(6468): 1024-1028. DOI:10.1126/Science.Aax8780 (  0) 0) |
| [2] |
ROTHLISBERGER D, KHERSONSKY O, WOLLACOTT A M, et al. Kemp elimination catalysts by computational enzyme design[J]. Nature, 2008, 453(7192): 190-195. DOI:10.1038/Nature06879 (  0) 0) |
| [3] |
GHIRLANDA G. Computational biology: A recipe for ligand-binding proteins[J]. Nature, 2013, 501(7466): 177-178. DOI:10.1038/Nature12463 (  0) 0) |
| [4] |
BURKE A J, LOVELOCK S L, FRESE A, et al. Design and evolution of an enzyme with a non-canonical organocatalytic mechanism[J]. Nature, 2019, 570(7760): 219-223. DOI:10.1038/S41586-019-1262-8 (  0) 0) |
| [5] |
ZHANG Y, LANG B, ZENG D, et al. Truncation of kappacarrageenase for higher kappacarrageenan oligosaccharides yield with improved enzymatic characteristics[J]. International Journal of Biological Macromolecules, 2019, 130: 958-968. DOI:10.1016/J.Ijbiomac.2019.02.109 (  0) 0) |
| [6] |
SU B M, XU X Q, YAN R X, et al. Mutagenesis on the surface of a beta-agarase from vibrio Sp. ZC-1 increased its thermo-stability[J]. Enzyme and Microbial Technology, 2019, 127: 22-31. DOI:10.1016/J.Enzmictec.2019.04.006 (  0) 0) |
| [7] |
BERMAN H M, GIERASCH L M. How the protein data bank changed biology: An introduction to the JBC Reviews Thematic Series, Part 1[J]. The Journal of Biological Chemistry, 2021, 100608. DOI:10.1016/J.Jbc.2021.100608 (  0) 0) |
| [8] |
LANDER E S, LINTON L M, BIRREN B, et al. Initial sequencing and analysis of the human genome[J]. Nature, 2001, 409(6822): 860-921. DOI:10.1038/35057062 (  0) 0) |
| [9] |
RADIVOJAC P, CLARK W T, ORON T R, et al. A large-scale evaluation of computational protein function prediction[J]. Nature Methods, 2013, 10(3): 221-227. DOI:10.1038/Nmeth.2340 (  0) 0) |
| [10] |
ALTSCHUL S F, GISH W, MILLER W, et al. Basic local alignment search tool[J]. Journal of Molecular Biology, 1990, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2 (  0) 0) |
| [11] |
SIEVERS F, HIGGINS D G. The clustal omega multiple alignment package[J]. Methods in Molecular Biology (Clifton, NJ), 2021, 2231: 3-16. DOI:10.1007/978-1-0716-1036-7_1 (  0) 0) |
| [12] |
TORKTAZ I, ETEMADIFAR Z, DERIKVAND P. Comparative modeling of dszc, an enzyme in biodesulfurization, and performing in silico point mutation for increasing tendency to oil[J]. Bioinformation, 2012, 8(5): 246-250. DOI:10.6026/97320630008246 (  0) 0) |
| [13] |
REN D, WANG Z, JIANG S, et al. Recent environmental applications of and development prospects for immobilized laccase: A review[J]. Biotechnology & Genetic Engineering Reviews, 2020, 36(2): 81-131. DOI:10.1080/02648725.2020.1864187 (  0) 0) |
| [14] |
HULO N, BAIROCH A, BULLIARD V, et al. The 20 years of PROSITE[J]. Nucleic Acids Research, 2008, 36(Database Issue): D245-249. (  0) 0) |
| [15] |
MISTRY J, CHUGURANSKY S, WILLIAMS L, et al. Pfam: The protein families database in 2021[J]. Nucleic Acids Research, 2021, 49(D1): D412-D419. DOI:10.1093/Nar/Gkaa913 (  0) 0) |
| [16] |
SI J N, YAN R X, WANG C, et al. TIM-Finder: A new method for identifying tim-barrel proteins[J]. BMC Structural Biology, 2009, 9(1): 73. (  0) 0) |
| [17] |
FOX N K, BRENNER S E, CHANDONIA J M. Scope: Structural classification of proteins--extended, integrating scop and astral data and classification of new structures[J]. Nucleic Acids Research, 2014, 42(Database Issue): D304-309. DOI:10.1093/Nar/Gkt1240 (  0) 0) |
| [18] |
LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521(7553): 436-444. DOI:10.1038/Nature14539 (  0) 0) |
| [19] |
GAILLARD T. Evaluation of autodock and autodock vina on the CASF-2013 benchmark[J]. Journal of Chemical Information and Modeling, 2018, 58(8): 1697-1706. DOI:10.1021/Acs.Jcim.8b00312 (  0) 0) |
| [20] |
VREVEN T, VANGAVETI S, BORRMAN T M, et al. Performance of ZDOCK and IRAD in CAPRI rounds 39-45[J]. Proteins, 2020, 88(8): 1050-1054. DOI:10.1002/Prot.25873 (  0) 0) |
| [21] |
RAMAN S, VERNON R, THOMPSON J, et al. Structure prediction for CASP8 with all-atom refinement using rosetta[J]. Proteins, 2009, 77(Suppl 9): 89-99. DOI:10.1002/Prot.22540 (  0) 0) |
| [22] |
LONDON N, RAVEH B, COHEN E, et al. Rosetta flexpepdock web server--high resolution modeling of peptide-protein interactions[J]. Nucleic Acids Research, 2011, 39(Web Server Issue): W249-W253. DOI:10.1093/Nar/Gkr431 (  0) 0) |
| [23] |
LASKOWSKI R A, SWINDELLS M B. Ligplot+: Multiple ligand-protein interaction diagrams for drug discovery[J]. Journal of Chemical Information and Modeling, 2011, 51(10): 2778-2786. DOI:10.1021/Ci200227u (  0) 0) |
| [24] |
CASE D A, CHEATHAM T E, DARDEN T, et al. The amber biomolecular simulation programs[J]. Journal of Computational Chemistry, 2005, 26(16): 1668-1688. DOI:10.1002/Jcc.20290 (  0) 0) |
| [25] |
PRONK S, PALL S, SCHULZ R, et al. GROMACS 4.5: A high-throughput and highly parallel open source molecular simulation toolkit[J]. Bioinformatics (Oxford, England), 2013, 29(7): 845-854. DOI:10.1093/Bioinformatics/Btt055 (  0) 0) |
| [26] |
PANG Y T, MIAO Y, WANG Y, et al. Gaussian accelerated molecular dynamics in NAMD[J]. Journal of Chemical Theory and Computation, 2017, 13(1): 9-19. DOI:10.1021/acs.jctc.6b00931 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


