阿尔兹海默症(Alzheimer's disease,AD)是一种慢性神经退行性疾病,在临床上主要表现为记忆丧失与日常行动功能障碍。据阿尔兹海默协会(Alzheimer's Association)报道[1],截至2018年,阿尔兹海默症是造成美国65岁以上人群死亡的第五大原因。根据中国疾病预防控制中心发布的数据显示[2],作为一种高致残率、影响独立生活能力的疾病,AD所导致的死亡人数在中国所有疾病中排名第五。AD的病因众说纷纭,但确切的发病机制尚未阐明,迄今也无特效治疗或逆转疾病进展的药物。
随着高通量测序技术和全基因组关联分析研究的发展,多个与AD患病相关的基因被发现,其中已证实有三个基因——淀粉样前体蛋白(APP)、早老素1(PSEN1)和早老素2(PSEN2)是家族性阿尔兹海默症(Familial Alzheimer disease,FAD)的致病基因。目前针对AD的研究主要集中在高加索人群,而对亚洲人群以及中国人群的研究相对较少。Jia等[3]招募了404个家系的1 330例AD或轻度认知障碍(Mild cognitive impairment,MCI)患者并检测PSENs / APP突变,鉴定了11个新的突变位点,新的PSENs / APP突变表明中国人与其他种族之间AD发病机制可能存在异质性。Zheng等[4]对汉族FAD患者进行了全基因组测序,鉴定出了汉族人特有的基因外显子错义突变rs3792646,表明Complement C7是汉族人患AD的新风险基因。Han等人指出[5],CLU rs11136000多态性与白种人AD发病显著相关,而它与亚洲人群AD发病并没有显著性关联。由此我们猜测,不同人种之间的AD遗传/发病机制存在差异,因此有必要对研究相对匮乏的亚洲人群进行研究,以挖掘亚洲人群特有的AD致病基因、生物标志物以及风险因素。
microRNA(miRNA)是一类保守的非编码RNA小分子,它们主要通过抑制靶mRNA翻译或促使其降解实现对靶基因的转录后调控[6]。miRNA在神经发育、组织分化和突触形成过程中起着重要作用[7]。此外,越来越多的证据表明血浆miRNA含量可能作为潜在的疾病标志物,比如血浆miRNA-206水平升高可预测认知能力下降和痴呆程度[8]。目前,有关miRNA在亚洲人群AD发生过程中的作用研究甚少,而且关于亚洲人AD的表达谱研究往往只针对mRNA或者miRNA表达谱,并没有将二者进行关联分析,结合miRNA及mRNA表达谱的亚洲人AD系统分析尚未见报道。
因此,收集现有公开的亚洲人AD与正常人mRNA/miRNA表达谱数据,通过生物信息学手段,筛选核心差异基因与差异miRNAs,构建miRNA-mRNA的差异表达网络。从转录组水平系统的对亚洲人群可能的AD发生机制进行分析,为亚洲人AD的诊疗提供新的潜在靶标。
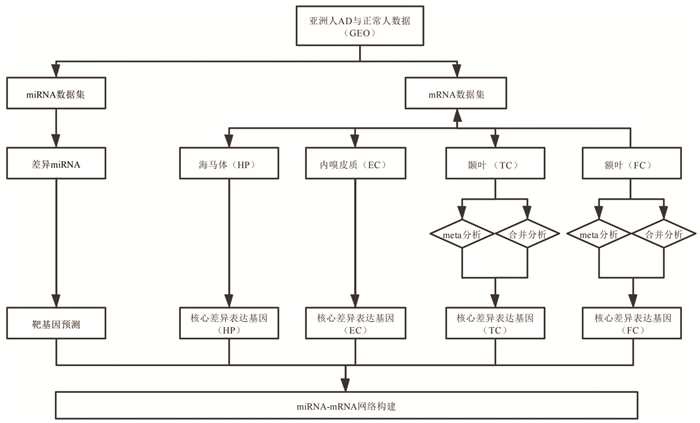
1 材料与方法分析流程主要包含以下环节:数据收集与下载,数据集预处理与单个数据集差异分析,可合并脑区的荟萃分析(Meta分析)和合并分析,核心差异基因选取,潜在靶基因预测,核心miRNA-mRNA
网络构建,差异基因/靶基因富集分析,靶基因相关蛋白的互作分析(见图 1)。
|
图 1 分析流程图 Figure 1 Analysis flow chart |
亚洲人AD患者与正常人的表达谱数据来自基因表达综合数据库(Gene Expression Omnibus, GEO)[9] (https://www.ncbi.nlm.nih.gov/geo/)。通过限定关键词“Alzheimer AND HAN”或者“Alzheimer AND ASIAN”,选取了包含额叶(Frontal Cortex, FC)、颞叶(Temporal Cortex,TC)、海马体(Hippocampus,HP) 与内嗅皮质(Entorhinal Cortex,EC) 等AD发病相关脑区的数据。截止2020年6月,一共收集4个表达谱数据集,分别是GSE131617[10]、GSE36980[11]、GSE139384[12]和GSE120584[13]。为确保数据质量,剔除了小于50岁的样本以及在主成分分析中明显离群的样本。最终一共获得了212个AD患者脑组织mRNA数据(76个FC,71个TC,58个EC以及7个HP)与92个正常人(Control,CT)脑组织mRNA数据(34个FC,35个TC,13个EC以及10个HP)。此外,miRNA血清数据集中包含1 021个AD以及288个CT。4个数据集的具体信息(见表 1)。
| 表 1 从GEO收集的亚洲人AD患者与正常对照的表达谱数据集 Table 1 Expression data sets for AD patients and normal controls in Asian populations collected from GEO |
首先用R包GEOquery[14]下载数据集,利用探针注释的文件对表达矩阵进行探针ID转换。接着利用R包limma[15]的线性模型,对单个表达谱数据进行归一化处理、性别校正与差异分析:其中miRNA数据集进行Benjamini-Hochberg(BH)法[16]校正,P校正值<0.05为显著差异;mRNA数据集取P值<0.05为显著差异。由于GSE131617数据集根据布拉克分期(Braak Staging)[17]将AD样本分为多组,将该数据集中布拉克分期为0的样本划分为正常人,将其余布拉克分期样本统一合并为AD患者。
1.3 FC/TC脑区多个mRNA数据集整合分析由于多个数据集中均包含FC和TC两个脑区的数据,同时采用以下两种算法进行整合分析:1)合并同脑区多个数据集,去除批次效应后进行差异分析;2)meta分析中的鲁棒排名聚集算法(Robust Rank Aggregation,RRA)[18]。
1.3.1 FC/TC脑区mRNA数据集的合并分析将不同研究的芯片数据合并进行归一化,可以移除不同研究的批次效应,同时保留真实的生物学差异[19]。通过R包sva中的ComBat函数[20],将三个mRNA数据集的表达矩阵进行合并,并且去除批次效应,然后用limma的线性模型对合并之后的表达矩阵进行性别校正,最后对合并之后的表达矩阵进行差异分析。
1.3.2 FC/TC脑区mRNA数据集的RRA分析对单个数据集中FC或TC脑区得到的差异基因(P<0.05),按照绝对值表达差异倍数(|logFC|)从大到小进行排列,并进行RRA运算,以RRA score<0.05作为筛选阈值,获得三个mRNA数据集数据中排名相对靠前的差异基因。
1.4 核心差异基因选取对于存在于多个数据集中的脑区(FC/TC),通过1.3中两种方法的交集来获得核心差异基因。对于仅存在于单个数据集的脑区(EC/HP),通过进一步降低P值与增加绝对值表达差异倍数(|logFC|)的筛选阈值来获得核心差异基因。
1.5 差异miRNA潜在靶基因预测利用miRWalk在线工具[21](http://zmf.umm.uniheidelberg.de/apps/zmf/mirwalk/)进行差异miRNA的靶基因预测。选择靶基因3端UTR作为miRNA的作用区域,并选择PicTar5[22]、miRWalk2.0、TargetScan[22]、DIANA[23]、miRDB[24]以及miRanda[25]等6个靶基因数据库,选取至少被其中2个靶基因数据库收录的靶基因作为潜在靶基因。
1.6 靶基因选取、miRNA-mRNA网络构建、靶基因蛋白互作分析将1.5得到的潜在靶基因与1.4得到的各脑区核心差异基因进行交集,得到四个脑区的靶基因。将靶基因与对应的差异miRNA以及差异表达倍数等导入Cytoscape软件[26],构建四个脑区的miRNA-mRNA差异表达网络。用geneMANIA[27](https://genemania.org/)数据库,对靶基因进行蛋白互作分析。
1.7 基因本体论(GO)以及京都基因组百科全书数据库(KEGG)通路富集分析通过R包clusterprofiler[28]对四个脑区的差异基因与靶基因进行GO以及KEGG通路富集分析。
2 结果 2.1 AD患者与正常对照组miRNA/mRNA差异表达结果 2.1.1 miRNA差异表达结果以P校正值<0.05和|logFC|>0.5作为差异miRNA的选择标准,共筛选出5个差异miRNAs,其中4个(hsa-mi-22、hsa-miR-24、hsa-miR-125b-1-3p、hsa-mi-125-3p)在AD组下调,1个(hsa-miR-208a-5p)在AD组上调。
2.1.2 mRNA差异表达结果(差异基因与核心差异基因)差异基因与核心差异基因的具体筛选要求及差异个数统计数据(见表 2)。对于存在于多个数据集中的脑区(FC/TC),采用方法1.31的合并分析进行差异基因筛选,以上差异基因再与1.32中RRA分析结果取得交集获得核心差异基因;对于仅存在于单个数据集的脑区(EC/HP),通过常规筛选条件获得差异基因,并通过提高筛选条件来获得核心差异基因。
| 表 2 差异基因与核心差异基因的筛选条件与个数统计 Table 2 Thresholds for DEGs and KDEGs selections and summary statistics |
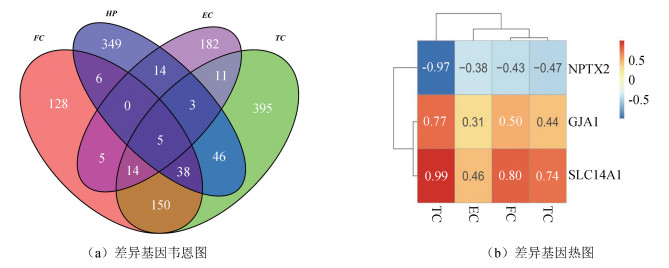
如表 2所示,额叶、颞叶、海马体与内嗅皮质中分别存在346、662、461与234个差异基因(见图 2),研究发现NPTX2、SLC14A1与GJA1在四个脑区中均为差异基因,且上述基因在AD患者(与正常对照相比)的四个脑区中呈现出相同的上调或下调表达趋势。额叶、颞叶、海马体与内嗅皮质中分别存在15、29、63与20个核心差异基因(见表 2)。SLC14A1是唯一一个在四个脑区中都被选入核心差异基因的基因。此外,NPAS4同时出现在FC、TC与EC脑区;GMPR、ITGB4、APLNR与NPTX2同时出现在FC、TC与HP脑区。
|
图 2 差异基因韦恩图和热图 Figure 2 Venn plot and heatmap for DEGs |
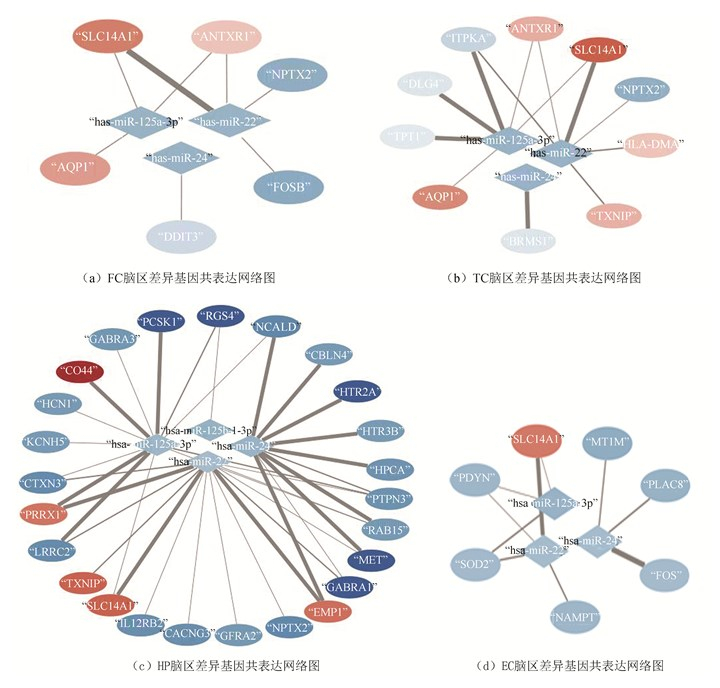
利用miRWalk等6个数据库,对5个差异miRNAs进行潜在靶基因预测,并与前文所述四个脑区的核心差异基因进行交集,获得靶基因。共有4个差异miRNAs的潜在靶基因与核心差异基因存在交集,分别是hsa-mi-22、hsa-miR-24、hsa-miR-125b-1-3p、hsa-mi-125-3p。4个差异miRNAs与四个脑区靶基因的调控关系见miRNA-mRNA网络(见图 3)。
|
图 3 四个脑区(FC/TC/HP/EC)筛选出的差异miRNAs与对应靶基因(mRNAs)的基因共表达网络图 Figure 3 Gene co-expression network diagram of selected differential miRNAs and corresponding target genes (mRNAs) in four brain regions (FC/TC/HP/EC) |
额叶、颞叶、海马体与内嗅皮质中分别存在6、10、25与7个靶基因(见表 3)。其中,SLC14A1同时出现在四个脑区的miRNA-mRNA网络中,NPTX2出现在FC、TC与HP脑区的网络中,AQP1与ANTXR1出现在FC与TC脑区的网络中,TXNIP出现在TC与HP脑区的网络中。此外,DDIT3、FOSB为FC脑区特有靶基因;HLA-DMA、TPT1、BRMS1、DLG4、ITPKA为TC脑区特有靶基因;CD44、EMP1、PRRX1、HCN1、KCNH5、HTR3B、CBLN4、RAB15、GFRA2、GABRA3、HPCA、PTPN3、IL12RB2、CACNG3、CTXN3、LRRC2、NCALD、HTR2A、GABRA1、RGS4、PCSK1、MET为HP脑区特有靶基因;FOS、SOD2、PDYN、NAMPT、PLAC8、MT1M为EC脑区特有靶基因。靶基因的GeneMANIA结果表明,四个脑区中的靶基因间均存在复杂的相互作用,包括共表达相关性、共享蛋白结构域、基因相互作用、物理相互作用与共同通路等等。值得注意的是,HP靶基因数目最多、差异倍数更大、具有更复杂的蛋白相互作用关系、且多在HP中特异表达;EC靶基因与其他脑区靶基因交集最少,SLC14A1为唯一交集。
| 表 3 靶基因统计以及geneMANIA蛋白互作分析结果 Table 3 Target genes and geneMANIA results of the PPI network analyses |
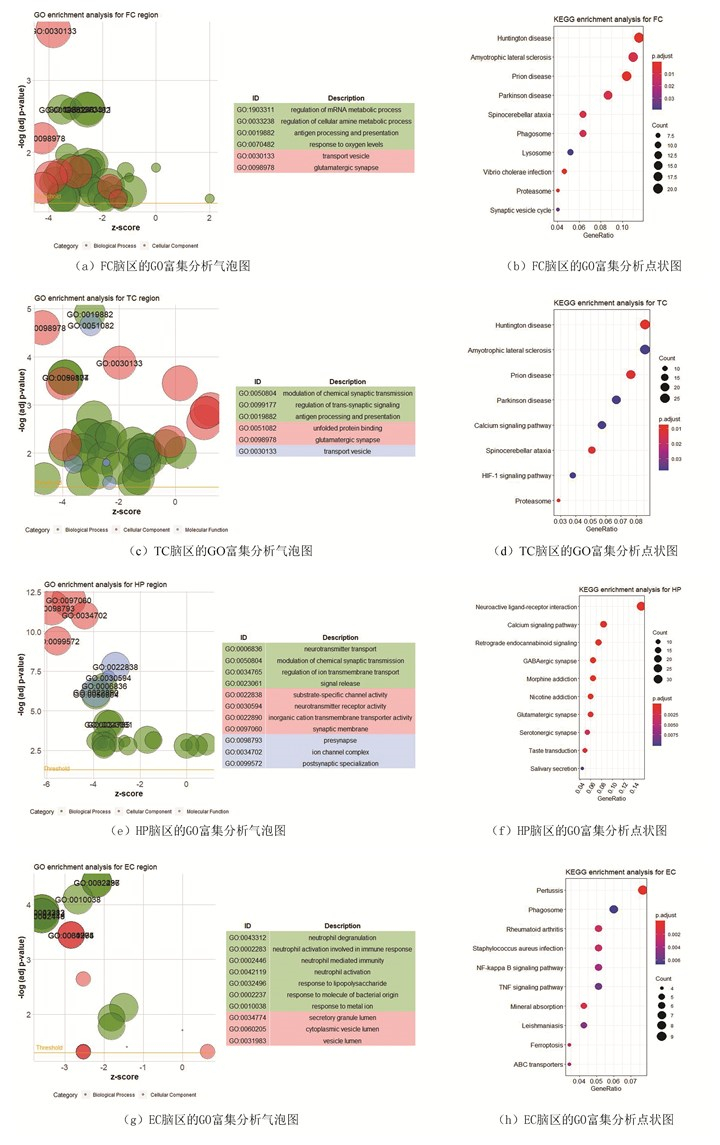
四个脑区差异基因的GO与KEGG富集结果(见图 4)。结果表明FC、TC与HP脑区的GO富集结果比较接近,主要集中在神经递质传导、化学突触传递等神经信号传递相关通路,而EC脑区的GO富集结果更偏向于免疫相关,如中性粒细胞脱颗粒等。KEGG分析中,FC与TC脑区都富集到了神经性退行性疾病通路,如亨廷顿氏病、渐冻症、帕金森症等,HP富集到了神经递质传导相关通路,如神经活性配体受体相互作用信号通路、钙离子信号通路、GABA能突触信号、谷氨酸能突触信号,而EC主要富集在炎症、免疫相关通路。
|
图 4 四个脑区差异基因的GO/KEGG富集分析图 Figure 4 GO/KEGG plots for DEGs in four brain regions |
四个脑区靶基因的GO与KEGG富集结果(见图 5a, 5b)。从GO分析结果来看,四个脑区各自的靶基因可被同时富集到转运相关的生物学过程中;FC、TC与HP的靶基因被同时富集到神经递质受体活性调控的生物学过程;HP的靶基因可被特异性地富集到离子跨膜运输调控过程中;EC的靶基因可被特异性地富集到低温应激、衰老、神经元死亡、活性氧应激等4个生物学过程中。从KEGG分析结果来看,四个脑区的靶基因未能富集到同一通路上,但FC、TC与EC脑区的靶基因能被同时富集到可卡因成瘾通路。
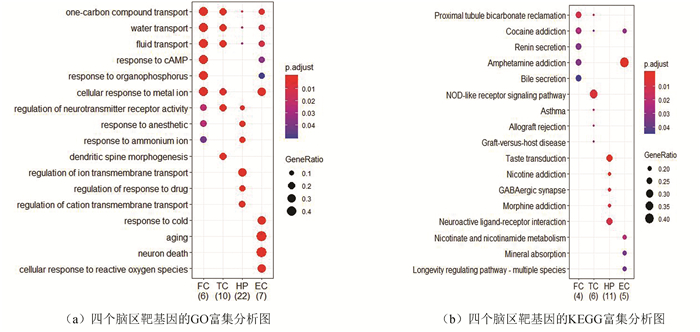
|
图 5 四个脑区靶基因GO/KEGG富集分析 Figure 5 GO/KEGG enrichment results for target genes in four brain regions |
本研究收集了GEO数据库中亚洲人AD患者与正常人mRNA/miRNA数据,通过荟萃分析与合并分析,从转录组水平系统地对亚洲人群可能的AD发病机制进行了分析,首次构建了亚洲人四个脑区AD的miRNA-mRNA差异表达网络。
目前有多项生信研究利用已有AD表达谱数据对AD机制进行了系统性研究。Xu等[29]运用合并分析方法,整合了20个表达谱数据,构建了AD患者四个脑区的mRNA转录谱,发现了YAP1等AD上游调控因子。Shi等[30]通过分析1个RNA-seq数据集,鉴定了AD患者上游调控的lncRNA,构建了lncRNA-mRNA共表达网络。Moradifard等[31]分析了1个miRNA以及6个mRNA芯片数据,通过荟萃分析发现了AD中重要的mRNA/miRNA相互作用关系。以上工作往往只采用一种整合分析方法,如荟萃分析或合并分析,或者只进行了单组学分析。本研究对多数据集脑区的数据同时应用了两种整合分析方法,可以更加准确地得到多数据集脑区中的差异基因。此外,上述生信研究的表达谱数据均为高加索人群,而不同人种之间的AD遗传/发病机制可能存在差异,因此本研究旨在收集亚洲人AD表达谱数据,构建亚洲人AD的miRNA-mRNA差异表达网络。
发现5个差异miRNAs,已有文献报道miR-22与miR-24在AD病人中低表达,与本研究中的发现一致:Jovicic等[32]指出miR-22在阿尔兹海默症患者中低表达;Hu等[33]通过荟萃分析发现miR-24在AD患者的脑脊液中低表达。尽管Yılmaz等[34]指出hsa-mi-125-3p和hsa-miR-125b-5p在AD患者中(相比于正常人)并没有显著差异,但是其研究样本较少(AD 172个,CT 109个),且亚洲人群占比较少。因此,这两个差异miRNAs及其靶基因在亚洲人群AD发生发展中的作用值得进一步的研究。此外,本研究发现NTPX2、GJA1与SLC14A1在四个脑区中均为差异基因。Xiao等[35]表明NPTX2在AD患者皮质中大量减少,且与患者认知能力和海马体积密切相关。Kajiwara等[36]指出星形胶质细胞特异性表达的GJA1在AD病人大脑上调,并发现GJA1与AD淀粉样蛋白、tau病理以及认知功能密切相关。Kerstin等[37]指出,SLC14A1的表达在AD病人和小鼠模型中均显著上调。上述基因在本研究的表达趋势与文献报道一致,进一步证明了本研究分析策略和结果的可靠性。
SLC14A1(尿素转运蛋白1)是唯一在四个脑区中共有的靶基因。通过蛋白互作分析,发现SLC14A1和AQP1(水通道蛋白1)存在蛋白相互作用,同时AQP1在AD患者中表达上调,且四个脑区靶基因的GO都富集到液体转运等生物学过程。有研究表明SLC14A1在亨廷顿舞蹈病中显著增高,从而破坏大脑中的尿素平衡,导致纹状体神经元大量死亡[38]。此外,Hansmannel等[39]通过AD患者额叶皮层的转录组测序,指出脑中尿素平衡与AD发病相关。因此猜测SLC14A1与AQP1在AD患者中的表达上调与相互作用,可能会引起脑中尿素循环与水循环紊乱,从而造成神经损伤并诱导AD发病。此外,通过艾伦大脑图谱(Allen Brain Map)[40]中的单细胞转录组数据,发现AQP1、SLC14A1都在星形胶质细胞(Astro L1-6 FGFR3)中特异表达。Misawa等[41]也指出,星形胶质细胞中AQP1上调可能与AD患者脑中Aβ沉积相关。因此本研究进一步推测,AD患者星形胶质细胞SLC14A1、AQP1的差异表达,将破坏星形胶质细胞的水平衡与尿素平衡,从而引起细胞肿胀破裂,最终影响神经元功能。
通过整合四个脑区靶基因和miRNA-mRNA的差异网络构建,对单脑区的特异性靶基因进行了分析,特别是HP脑区,其靶基因数目最多、差异倍数更大、多数在海马中特异表达、并且主要是配体门控性离子通道相关基因,包括GABRA1(A型γ-氨基丁酸受体亚基Alpha1),GABRA3(A型γ-氨基丁酸受体亚基Beta3),HTR3B(5-羟色胺受体3B)与HTR2A (5-羟色胺受体2A)等。Agenor等[42]表明GABA受体α1、β3亚型在AD患者的海马中表达下调。Garcia-Alloza等[43]发现胆碱能与5-羟色胺能系统之间的不平衡,可能与AD的认知障碍相关。此外,Pedro等[44]证明Cbln4参与形成并维持GABA能连接,敲除Cbln4后神经元GABA能连接大大减少,且Cbln4在AD小鼠的海马区中表达显著下调。上述基因在本研究中的HP脑区中均表达下调,因此,本研究推断亚洲人群AD患者的海马体GABA受体亚型与5-羟色胺能受体亚型表达下调,可能破坏兴奋性能与抑制性能神经元之间信号平衡,最终造成海马体中神经元的功能障碍。
此外,EC脑区靶基因与其他脑区靶基因交集只有SLC14A1,且其GO/KEGG富集通路与其他三个脑区有较大区别。Flynn等[45]表明SOD2(线粒体超氧化物歧化酶2)可以将超氧化物还原为过氧化氢,保护神经元细胞免受谷氨酸诱导的氧化应激和细胞毒性。Xing等[46]指出,NAMPT(烟酰胺磷酸核糖基转移酶)减少导致NAD +减少,从而导致NAD+ / NADH的比率降低与线粒体功能障碍。在本研究中,SOD2、NAMPT等基因在EC中下调,因此本研究猜测亚洲人群AD患者EC中SOD2与NAMPT的下调可能引起线粒体功能障碍,继而造成突触受损、神经细胞凋亡,最终促使AD的病情发展。
本研究主要基于GEO数据库中的微阵列芯片数据,因此具有一些局限性。首先,微阵列芯片数据受制于其技术的局限性,例如技术动态范围较窄、灵敏度较低等。其次,本研究使用的芯片数据来自不同条件下的多个研究,样本量不均并且许多患者临床细节未知(如用药情况等),增加了本研究不确定性。尽管本研究采用两种整合分析方法,也无法完全消除各项研究的异质性。此外,本研究受限于亚洲人数据集的样本量,某些脑区(如海马体)样本量较少,其结果可能不具备普适性。值得注意的是,上述芯片数据的基因表达量为单脑区所有细胞的平均值,并没有达到单细胞精确度,因此我们无法直接证实SLC14A1是否在AD患者的星形胶质细胞特异差异表达,并影响AD患者星形胶质细胞的功能。最后,本研究没有详细探究FC与TC脑区特异性变化的靶基因,可能忽略了这些靶基因在上述脑区的特异性作用。本研究虽然对SLC14A1在亚洲人AD中的潜在作用进行了推测,但仍然需要构建相应的细胞和动物模型进一步研究其确切的作用机制。此外,本研究希望通过单细胞转录组技术,核实本研究的推测,并进一步研究星形胶质细胞在亚洲人AD中的作用机制。
4 结论收集现有公开的亚洲人AD与正常人mRNA/miRNA表达谱数据,通过生物信息学手段,从转录组水平系统地对亚洲人群可能的AD发生机制进行了分析,最终筛选出差异miRNAs与核心差异基因,首次构建了亚洲人四个脑区的关键miRNA-mRNA差异表达网络,发现了SLC14A1基因在四个脑区均差异表达。本研究推测SLC14A1和AQP1蛋白相互作用,影响了AD患者星形胶质细胞的体液转运功能,最终影响神经元细胞,从而诱导AD的发病。此外,研究发现亚洲人AD海马体与内嗅皮质中的特异表达靶基因,分别与神经元间信号传递与线粒体功能障碍相关。以上研究为亚洲人AD的诊疗提供新的潜在靶标,也为组学分析提供了新的思路。
| [1] |
ZHAO L. 2020 Alzheimer's Disease facts and figures[J]. Alzheimer's & Dementia: the Journal of the Alzheimer's Association, 2020, 16(3): 391-460. DOI:10.1002/alz.12068 (  0) 0) |
| [2] |
JIA Li, QUAN Meina, FU Yue, et al. Dementia in China: epidemiology, clinical management, and research advances[J]. The Lancet Neurology, 2020, 19(1): 81-92. DOI:10.1016/s1474-4422(19)30290-x (  0) 0) |
| [3] |
JIA Longfei, FU Yue, SHEN Luxi, et al. PSEN1, PSEN2, and APP mutations in 404 Chinese pedigrees with familial Alzheimer's disease[J]. Alzheimer's & Dementia: the Journal of the Alzheimer's Association, 2020, 16(1): 178-191. DOI:10.1002/alz.12005 (  0) 0) |
| [4] |
ZHANG Dengfei, FAN Yu, XU Min, et al. Complement C7 is a novel risk gene for Alzheimer's disease in Han Chinese[J]. National science review, 2019, 6(2): 257-274. DOI:10.1093/nsr/nwy127 (  0) 0) |
| [5] |
HAN Zhijie, QU Jiaojiao, ZHAO Jielong, et al. Analyzing 74, 248 samples confirms the association between CLU rs11136000 polymorphism and Alzheimer's disease in Caucasian but not Chinese population[J]. Scientific Reports, 2018, 8(1): 11062. DOI:10.1038/s41598-018-29450-2 (  0) 0) |
| [6] |
GRAVES P, ZENG Yan. Biogenesis of mammalian microRNAs: a global view[J]. Genomics, Proteomics & Bioinformatics, 2012, 10(5): 239-245. DOI:10.1016/j.gpb.2012.06.004 (  0) 0) |
| [7] |
HU Zhonghua, LI Zheng. miRNAs in synapse development and synaptic plasticity[J]. Current Opinion in Neurobiology, 2017, 45: 24-31. DOI:10.1016/j.conb.2017.02.014 (  0) 0) |
| [8] |
KENNY A, MCARDLE H, CALERO M, et al. Elevated plasma microRNA-206 levels predict cognitive decline and progression to dementia from mild cognitive impairment[J]. Biomolecules, 2019, 9(11): 734. DOI:10.3390/biom9110734 (  0) 0) |
| [9] |
BRAZMA A, PARKINSON H, SARKANS U, et al. ArrayExpress-a public repository for microarray gene expression data at the EBI[J]. Nucleic Acids Research, 2003, 31(1): 68-71. DOI:10.1093/nar/gkg091 (  0) 0) |
| [10] |
MIYASHITA A, HATSUTA H, KIKUCHI M, et al. Genes associated with the progression of neurofibrillary tangles in Alzheimer's disease[J]. Translational Psychiatry, 2014, 4(6): e396. DOI:10.1038/tp.2014.35 (  0) 0) |
| [11] |
HOKAMA M, OKA S, LEON J, et al. Altered expression of diabetes-related genes in Alzheimer's disease brains: the Hisayama study[J]. Cerebral Cortex (New York, NY: 1991), 2014, 24(9): 2476-2488. DOI:10.1093/cercor/bht101 (  0) 0) |
| [12] |
MORIMOTO S, ISHIKAWA M, WATANABE H, et al. Brain transcriptome analysis links deficiencies of stress-responsive proteins to the pathomechanism of Kii ALS/PDC[J]. Antioxidants (Basel, Switzerland), 2020, 9(5): 423. DOI:10.3390/antiox9050423 (  0) 0) |
| [13] |
SHIGEMIZU D, AKIYAMA S, ASANOMI Y, et al. Risk prediction models for dementia constructed by supervised principal component analysis using miRNA expression data[J]. Communications Biology, 2019, 2: 77. DOI:10.1038/s42003-019-0324-7 (  0) 0) |
| [14] |
DAVIS S, MELTZER P. GEOquery: a bridge between the Gene Expression Omnibus (GEO) and BioConductor[J]. Bioinformatics (Oxford, England), 2007, 23(14): 1846-1847. DOI:10.1093/bioinformatics/btm254 (  0) 0) |
| [15] |
RITCHIE M, PHIPSON B, WU Di, et al. limma powers differential expression analyses for RNA-sequencing and microarray studies[J]. Nucleic Acids Research, 2015, 43(7): e47. DOI:10.1093/nar/gkv007 (  0) 0) |
| [16] |
FERREIRA J, ZWINDERMAN A. Approximate sample size calculations with microarray data: an illustration[J]. Statistical Applications in Genetics and Molecular Biology, 2006, 5: 25. DOI:10.2202/1544-6115.1227 (  0) 0) |
| [17] |
BRAAK H, BRAAK E. Staging of Alzheimer's disease-related neurofibrillary changes[J]. Neurobiology of Aging, 1995, 16(3): 271-284. DOI:10.1016/0197-4580(95)00021-6 (  0) 0) |
| [18] |
KOLDE R, LAUR S, ADLER P, et al. Robust rank aggregation for gene list integration and meta-analysis[J]. Bioinformatics (Oxford, England), 2012, 28(4): 573-580. DOI:10.1093/bioinformatics/btr709 (  0) 0) |
| [19] |
TAMINAU J, LAZAR C, MEGANCK S, et al. Comparison of merging and meta-analysis as alternative approaches for integrative gene expression analysis[J]. ISRN Bioinformatics, 2014, 2014: 345106. DOI:10.1155/2014/345106 (  0) 0) |
| [20] |
LEEK J, JOHNSON W, PARKER H, et al. The SVA package for removing batch effects and other unwanted variation in high-throughput experiments[J]. Bioinformatics (Oxford, England), 2012, 28(6): 882-883. DOI:10.1093/bioinformatics/bts034 (  0) 0) |
| [21] |
DWEEP H, STICHT C, PANDEY P, et al. miRWalk--database: prediction of possible miRNA binding sites by "walking" the genes of three genomes[J]. Journal of Biomedical Informatics, 2011, 44(5): 839-847. DOI:10.1016/j.jbi.2011.05.002 (  0) 0) |
| [22] |
KREK A, GRVN D, POY M, et al. Combinatorial microRNA target predictions[J]. Nature Genetics, 2005, 37(5): 495-500. DOI:10.1038/ng1536 (  0) 0) |
| [23] |
KARAGKOUNI D, PARASKEVOPOULOU M, CHATZOPOULOS S, et al. DIANA-TarBase v8: a decade-long collection of experimentally supported miRNA-gene interactions[J]. Nucleic Acids Research, 2018, 46(D1): D239-45. DOI:10.1093/nar/gkx1141 (  0) 0) |
| [24] |
WANG Xiaowei. miRDB: a microRNA target prediction and functional annotation database with a wiki interface[J]. RNA (New York, NY), 2008, 14(6): 1012-1017. DOI:10.1261/rna.965408 (  0) 0) |
| [25] |
JOHN B, ENRIGHT A, ARAVIN A, et al. Human MicroRNA targets[J]. PLoS Biology, 2004, 2(11): e363. DOI:10.1371/journal.pbio.0020363 (  0) 0) |
| [26] |
SU Gang, MORRIS J, DEMCHAK B, et al. Biological network exploration with Cytoscape 3[J]. Current Protocols in Bioinformatics, 2014, 47: 8.13.1-24. DOI:10.1002/0471250953.bi0813s47 (  0) 0) |
| [27] |
WARDE-FARLEY D, DONALDSON S, COMES O, et al. The GeneMANIA prediction server: biological network integration for gene prioritization and predicting gene function[J]. Nucleic Acids Research, 2010, 38: W214-W220. DOI:10.1093/nar/gkq537 (  0) 0) |
| [28] |
YU Guangchuang, WANG Ligen, HAN Yanyan, et al. clusterProfiler: an R package for comparing biological themes among gene clusters[J]. Omics: a Journal of Integrative Biology, 2012, 16(5): 284-287. DOI:10.1089/omi.2011.0118 (  0) 0) |
| [29] |
XU Min, ZHANG Dengfeng, LUO Rongcan, et al. A systematic integrated analysis of brain expression profiles reveals YAP1 and other prioritized hub genes as important upstream regulators in Alzheimer's disease[J]. Alzheimer's & Dementia: the Journal of the Alzheimer's Association, 2018, 14(2): 215-229. DOI:10.1016/j.jalz.2017.08.012 (  0) 0) |
| [30] |
SHI Yuchen, LIU Hui, YANG Changbo, et al. Transcriptomic analyses for identification and prioritization of genes associated with Alzheimer's disease in humans[J]. Frontiers in Bioengineering and Biotechnology, 2020, 8: 31. DOI:10.3389/fbioe.2020.00031 (  0) 0) |
| [31] |
MORADIFARD S, HOSEINBEYKI M, GANJI S, et al. Analysis of microRNA and gene expression profiles in Alzheimer's disease: a meta-analysis approach[J]. Scientific Reports, 2018, 8(1): 4767. DOI:10.1038/s41598-018-20959-0 (  0) 0) |
| [32] |
JOVICIC A, ZALDIVAR JOLISSAINT J, MOSER R, et al. MicroRNA-22 (miR-22) overexpression is neuroprotective via general anti-apoptotic effects and may also target specific Huntington's disease-related mechanisms[J]. PloS One, 2013, 8(1): e54222. DOI:10.1371/journal.pone.0054222 (  0) 0) |
| [33] |
HU Yongbo, LI Chunbo, SONG Ning, et al. Diagnostic value of microRNA for Alzheimer's disease: a systematic review and meta-analysis[J]. Frontiers in Aging Neuroscience, 2016, 8: 13. DOI:10.3389/fnagi.2016.00013 (  0) 0) |
| [34] |
YıLMAZ Ş, ERDAL M, ÖZGE A, et al. Can peripheral MicroRNA expression data serve as epigenomic (upstream) biomarkers of Alzheimer's disease?[J]. Omics: a Journal of Integrative Biology, 2016, 20(8): 456-461. DOI:10.1089/omi.2016.0099 (  0) 0) |
| [35] |
XIAO Meifang, XU Desheng, CRAIG M, et al. NPTX2 and cognitive dysfunction in Alzheimer's Disease[J]. eLife, 2017, 6: e23798. DOI:10.7554/eLife.23798 (  0) 0) |
| [36] |
KAJIWARA Y, WANG Erming, WANG Minghui, et al. GJA1 (connexin43) is a key regulator of Alzheimer's disease pathogenesis[J]. Acta Neuropathologica Communications, 2018, 6(1): 144. DOI:10.1186/s40478-018-0642-x (  0) 0) |
| [37] |
WIRZ K, BOSSERS K, STARGARDT A, et al. Cortical beta amyloid protein triggers an immune response, but no synaptic changes in the APPswe/PS1dE9 Alzheimer's disease mouse model[J]. Neurobiology of Aging, 2013, 34(5): 1328-1342. DOI:10.1016/j.neurobiolaging.2012.11.008 (  0) 0) |
| [38] |
HANDLEY R, REID S, BRAUNING R, et al. Brain urea increase is an early Huntington's disease pathogenic event observed in a prodromal transgenic sheep model and HD cases[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(52): E11293-E302. DOI:10.1073/pnas.1711243115 (  0) 0) |
| [39] |
HANSMANNEL F, SILLAIRE A, KAMBOH M, et al. Is the urea cycle involved in Alzheimer's disease?[J]. Journal of Alzheimer's Disease: JAD, 2010, 21(3): 1013-1021. DOI:10.3233/jad-2010-100630 (  0) 0) |
| [40] |
SUNKIN S, NG L, LAU C, et al. Allen Brain Atlas: an integrated spatio-temporal portal for exploring the central nervous system[J]. Nucleic Acids Research, 2013, 41: D996-1008. DOI:10.1093/nar/gks1042 (  0) 0) |
| [41] |
MISAWA T, ARIMA K, MIZUSAWA H, et al. Close association of water channel AQP1 with amyloid-beta deposition in Alzheimer disease brains[J]. Acta neuropathologica, 2008, 116(3): 247-260. DOI:10.1007/s00401-008-0387-x (  0) 0) |
| [42] |
LIMON A, REYES-RUIZ J, MILEDI R. Loss of functional GABA(A) receptors in the Alzheimer diseased brain[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(25): 10071-10076. DOI:10.1073/pnas.1204606109 (  0) 0) |
| [43] |
GARCIA-ALLOZA M, GIL-BEA F, DIEZ-ARIZA M, et al. Cholinergic-serotonergic imbalance contributes to cognitive and behavioral symptoms in Alzheimer's disease[J]. Neuropsychologia, 2005, 43(3): 442-449. DOI:10.1016/j.neuropsychologia.2004.06.007 (  0) 0) |
| [44] |
CHACÓN P, DEL MARCO Á, ARÉVALO Á, et al. Cerebellin 4, a synaptic protein, enhances inhibitory activity and resistance of neurons to amyloid-β toxicity[J]. Neurobiology of Aging, 2015, 36(2): 1057-1071. DOI:10.1016/j.neurobiolaging.2014.11.006 (  0) 0) |
| [45] |
FLYNN J, MELOV S. SOD2 in mitochondrial dysfunction and neurodegeneration[J]. Free Radical Biology & Medicine, 2013, 62: 4-12. DOI:10.1016/j.freeradbiomed.2013.05.027 (  0) 0) |
| [46] |
XING Sanli, HU Yiran, HUANG Xujiao, et al. Nicotinamide phosphoribosyltransferase-related signaling pathway in early Alzheimer's disease mouse models[J]. Molecular Medicine Reports, 2019, 20(6): 5163-5171. DOI:10.3892/mmr.2019.10782 (  0) 0) |
 2022, Vol. 20
2022, Vol. 20

