肺癌是一种常见的癌症,其早期表现是肺部CT图像中出现的结节。如果能尽早的发现结节并进行及时治疗则可以有效降低发展为肺癌的几率。目前主要是通过临床医生观察CT图像来检测肺结节,随着肺癌筛查普及,产生了大量的CT扫描,该方法不仅给医生带来了巨大的工作量而且其主要依赖于医生的临床经验,容易导致医生疲劳,从而导致误诊和漏诊的发生。因此,有研究者提出将计算机辅助诊断算法应用于肺结节的检测中。
传统肺部计算机辅助诊断检测结节主要是通过图像增强技术将结节分割后再进行识别,此类增强方法有可能改变原有的CT数据,进而干扰后续特征提取及检测环节[1]。如Teramoto [2]、Aresta [3]等人均关注结节的轮廓,并对其进行增强来进行肺结节的分割;John [4]、Leemput [5]等人则关注于找到合适的阈值来提取结节;Ogul [6]等人则使用了多尺度的方法来检测结节;齐守良[7]等人利用人工确定种子点,采用多阈值结合距离变换的方法提取结节。虽然传统的方法可以实现肺结节的检测,但是需要对图像进行复杂的预处理来人工提取特征,然后再对其进行识别和分类。该方法条件苛刻,若检测数量增加,则需要投入庞大的计算资源。
自2006年深度学习的概念被正式提出[8],研究者不断地探索和实验验证,证实了其在图像识别和分类方面应用的优势[9-10]。其中,卷积神经网络技术可以直接从输入的图像数据中提取特征并进行后续分类,相比于传统方法避免了复杂的预处理过程,在图像方面的分类和识别研究方面被广泛应用[11-14]。Setio等人[15]将二维卷积神经网络应用于三维立体图像的研究,针对不用形态的结节,借鉴已有的技术,设计了三种探测器,并针对每个候选结节对象,提取了多个二维视图,通过融合三个探测系统的结果,达到了较好的效果。其虽然考虑了结节的空间信息,结合了多个角度进行决策,但其模型仍是基于二维卷积神经网络实现的,(二维的缺陷)。Kim等人[16]将以结节为中心的多尺寸区域放缩到相同的尺寸,并以不同的顺序进行组合,其将各个分支的输出结合,网络结构十分复杂。Ding等人[17]将肺部CT切片轴向上连续的三层作为输入,训练了三维卷积神经网络模型用于降低假阳性,考虑了空间信息但仅使用了三层切片。以上研究均为对肺结节初筛后再降低结果中假阳性,耗时较长并且准确度不高。
综上,本文旨在提出一种基于三维卷积神经网络的肺结节探测方法,能够直接在CT数据中探测结节,并将结节进行标记。此外,设计一套完整的测试流程,为在CT图像中对肺结节进行识别和定位提供参考。
1 数据本研究选用美国2016年肺结节分析(Lung Nodule Analysis 2016, LUNA16)挑战赛开源数据集(https://luna16.grand-challenge.org)作为源数据。LUNA16挑战是一个完全开放的挑战,数据来自现有的最大的公共肺结节参考数据库LIDC/IDRI数据集。LIDC/IDRI数据集包括1 018份CT图像和附带相关的XML文件,并且由4名经验丰富的放射科医生对结节进行注释。LUNA16丢弃了切片厚度大于3 mm的扫描,还排除了有缺失或不一致切片的CT,最终得到888个CT扫描。其参考标准为由4名放射科医生中至少3名认为是大于等于3 mm的结节,这样的结节共有1 186个。未包含在参考标准中的注释(非结节、小于3 mm的结节和仅由1或2名放射科医生注释的结节)被称为不相关的发现,在官方附件中提供了不相关的注释文件。此外本研究还使用了官方给出的用于假阳性减少比赛的包含约55万个初筛的注释。
2 方法 2.1 卷积神经网络卷积神经网络是一种包含特征提取器的多层神经网络,由LeCun等人[18]在1998年首次正式提出,主要由卷积层、池化层,全连接层组成,网络多个层之间的组合能够对图像进行不同层次的抽象处理和特征提取[19],随着卷积神经网络被广泛地应用,后来陆续有新的网络层被开发出来对其不断地进行补充和优化,如为防止梯度消失的批归一化层,防止过拟合的随机丢弃层等等。
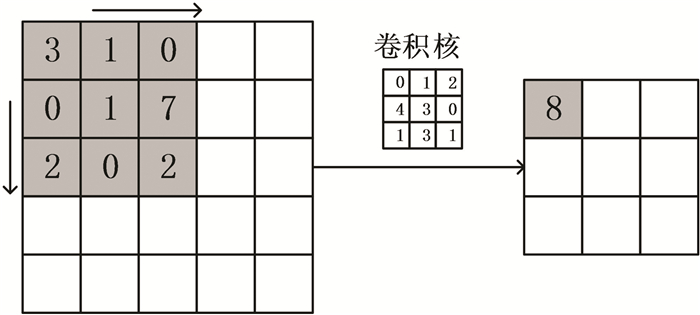
2.1.1 卷积层卷积层的功能主要是提取特征,层输出由卷积核实现,卷积核的数量与输出特征图的数量相同,卷积核的大小和移动步长决定了特征图的特征分布和精度,卷积过程中卷积核共享参数,但不同卷积核的参数不一定相同。深度学习中卷积操作指卷积核的每个值与卷积区域的对应位置的值相乘再求和,以一定的移动幅度遍历整个图像的过程,二维卷积过程(见图 1)。
|
图 1 二维卷积过程示意图 Figure 1 Schematic diagram of a 2D convolution process |
图中卷积前图像大小为5×5,经3×3的卷积核进行步长为1的卷积处理后图像大小变为3×3。卷积前在图像周围补“0”,可防止丢失图像边缘像素信息,还可使卷积前后图像大小保持一致。三维卷积原理与二维卷积类似,不同的是其卷积核是三维卷积核,卷积运算在三个维度中进行,因此输出的特征图也为三维形式。
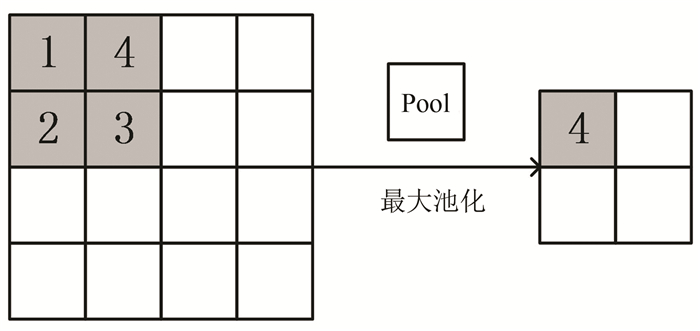
2.1.2 池化层池化层在于对特征图进行降维压缩,以减少参数数量,防止过拟合,并保持图像的平移不变性。目前常用的方法为最大池化和平均池化,根据图像特点和需求,本研究选用最大池化方法,即每次选取池化核邻域内最大的像素,如图 2中阴影部分所示。此外,池化运算的步长一般与池化窗口宽度相同,即移动时不与上一步重叠,以达到压缩特征图的目的。二维池化过程(见图 2)。
|
图 2 二维池化过程示意图 Figure 2 Schematic diagram of a 2D max-pooling process |
图中池化窗口尺寸为2×2,移动步长为2,因此输出特征图尺寸由输入时的4×4减小为2×2。三维池化原理与二维相同,不同的是池化核在三维邻域进行取值操作。该方法可以有效降低特征图的大小,降低计算压力,一般与卷积层成对设置。
2.1.3 全连接层全连接层将经过多个卷积层和池化层提取的特征进行整合后经Softmax分类函数进行分类,得到基于输入的概率分布[20]。前层输出的特征图在经过一维化后输入全连接层,经过加权后将分布式的特征映射到样本标记空间。
2.2 数据预处理 2.2.1 像素归一化与坐标转换LUNA16数据集中的CT图像由不同仪器扫描产生,成像因素造成灰度的差异。为了防止灰度值分布增加特征量而对后续研究产生影响[21],需要对图像进行像素归一化处理。根据人体肺部附近组织及空气CT值的分布范围,本研究使用线性变换将CT切片(-1 000, 400)的亨氏值归一化到(0, 1)。LUNA16数据集给出的坐标值是世界坐标,单位长度为mm,其数据存储均规范到512×512的大小,每个扫描在其头文件中给出了空间分辨率,因此需要将其转换为体素坐标。转换如公式(1) 所示。
| $ \begin{array}{l} {\rm{(coordX, coordY, coordZ) = (}}\frac{{{\rm{|coordx - origin\_x|}}}}{{{\rm{spacing\_x}}}}{\rm{, }}\\ \;\;\frac{{{\rm{|coordy - origin\_y|}}}}{{{\rm{spacing\_y}}}}{\rm{, }}\frac{{{\rm{|coordZ - origin\_z|}}}}{{{\rm{spacing\_z}}}}{\rm{)}} \end{array} $ | (1) |
公式中(coordx, coordy, coordz)为待检测目标的世界坐标,(origin_x, origin_y, origin_z)为扫描机器对应的世界坐标原点,(spacing_x, spacing_y, spacing_z)为存储时的空间分辨率,(coordX, coordY, coordZ)为转换后的像素坐标。
2.2.2 数据划分在深度学习中需要将数据按一定比例随机划分为训练集、验证集和测试集,训练集用于训练模型,验证集用于在训练过程中对模型进行调优,测试集不参与网络的训练,用于评估模型。本研究在包含888套扫描的十个无序文件夹中,取前九个作为训练源数据,共包含1 081个结节,最后一个文件夹中的图像作为独立的测试数据,包含105个结节。LUNA16比赛数据给出的1 186个肺结节直径均小于40 mm且其中90%小于15 mm,将所有图像进行像素归一化和坐标转换处理后,将结节位置和轴向同位置的邻近切片以五张40×40的连续切片存放到h5py格式的文件中,生成用于训练网络的3D数据集。
2.2.3 数据扩充与采样对于单次扫描而言,结节数量是远远小于正常样本数量的。模型具有高准确率的前提是对正负样本都能准确识别,这取决于模型是否全面地学习到了正负样本的特征信息。在LUNA16数据集中,正样本的注释相对于负样本是非常少的,因此需要扩充正样本,使训练数据集中有足够数量的正样本;而负样本数目庞大,因此需要进行下采样来减少数量,尽可能使有限数量的集合全面包含负样本特征信息。
本研究分别使用三种不同的正负样本选择与扩充方式得到了三个训练集。其中,训练集1和训练集2使用相同的方法扩充正样本,规则如下:正样本的样本中心在40×40的范围内随机平移,平移范围由结节直径大小约束,再经过90°、180°旋转以及水平翻转扩充数据。在训练集1中,假设被标记为正样本之外的区域都为负样本,对正样本关于水平中线、竖直中线和反斜对角线对称的位置进行采样,然后利用旋转、翻转扩增负样本。训练集2的负样本是我们排除官方提供的不相关注释列表中的点位,从剩余非结节点位中随机选取一部分获得的,数量与正样本数量相当。
为降低随机移动的偶然性,训练集3将随机平移的范围约束在40×40大小的四个10×10的矩形内,进行多次扩增,这些正样本相对均匀地在各方向上均得到了扩充。训练集3结合训练集1和训练集2的负样本采样方式,将采样到的负样本随机混合,并控制其数量与正样本数量相当。得到的三个训练集的样本数量分布(见表 1)。
| 表 1 训练集样本数量表 Table 1 The number of samples |
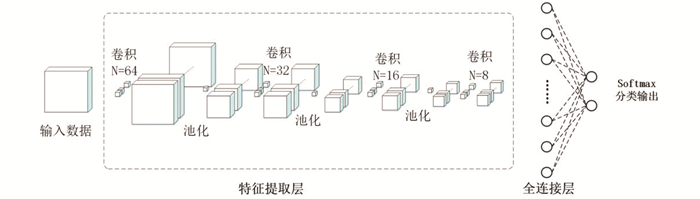
本研究基于Windows10操作系统,利用python编程语言,软件版本使用Tensorflow-gpu V1.7.0和KerasV2.1.6,搭配CUDAV9.0.176和cudnn V7.4并使用NVIDIAQuadro K2200 GPU搭建了基于3D-CNN的肺结节检测网络并进行训练。训练过程中根据验证集准确率和损失值的变化调整网络的结构和参数。经过大量实验得到3D-CNN结构框图(见图 3)。平均模型训练时间约0.5 h。
|
图 3 3D-CNN结构示意图 Figure 3 Structure diagram of 3D-CNN |
如图 3所示,本文搭建的3D-CNN包含4个卷积层、3个池化层、1个全连接层、1个输入层和1个输出层。其中,卷积层中卷积核的尺寸均为3×3×3,步长为1,填充方式为“same”,即卷积前在图像之外填充0以保证卷积前后图像大小不变,激活函数为ReLU激活函数。前3个卷积层后分别设置了步长为2的池化层以减少参数个数,降低网络计算压力。全连接层设置16个神经元,用于整合前层的特征,激活函数为ReLU激活函数,最后由softmax分类输出层输出结果。
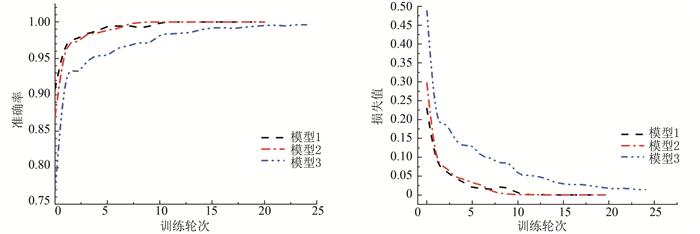
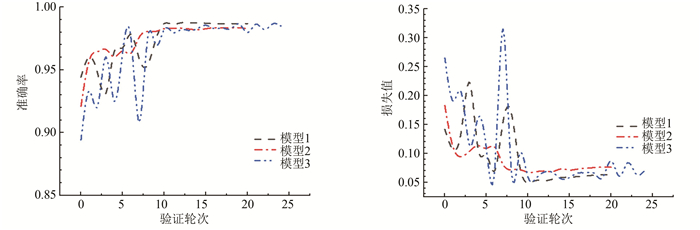
训练过程中通过可视化工具Tensorboard观察模型在训练集和验证集上的准确率变化情况,据此来调整模型的结构和参数。在此展示模型1-3在训练集和验证集上的准确率和损失值的变化情况(见图 4、图 5)。其中,图 4为模型1-3在训练集上准确率和损失值的变化,图 5为模型1-3在验证集上准确率和损失值的变化。
|
图 4 训练集上准确率和损失值曲线图 Figure 4 Gurves of accuracy and loss on training set |
|
图 5 验证集上准确率和损失值曲线图 Figure 5 Gurves of accuracy and loss on validation |
由图 4和图 5可以看出在网络进行训练20轮次左右时模型1和模型2收敛,准确率和损失值基本保持稳定。模型3的则需要更多的训练轮次才达到稳定,这是因为在数据扩充与采样时,训练集3相较于训练集1和训练集2数据更为多样化,网络学习特征的难度更大,从而训练模型到收敛的过程更长。为使结果更加可靠,我们对每个训练集分别进行了多次训练。
2.4 模型评价指标本研究中模型的性能主要通过敏感性、特异性来衡量。敏感性与特异性的计算方式如公式(2)、(3)所示。
| $ TPR = \frac{{TP}}{{TP + FN}} $ | (2) |
| $ TNR = \frac{{TN}}{{FP + TN}} $ | (3) |
式中真阳性比例(true positive rate, TPR)表示敏感性,真阴性比例(true negative rate, TNR)表示特异性。
测试集中包含88套CT扫描,在官方初筛注释中筛除掉不相关点位后对应32 620个点位,其中由于105个医生标注的结节中有些被多次检出,因此32 620个点位包括125个候选结节点位和32 495个非结节点位,我们以此为评估标准对模型进行测试。
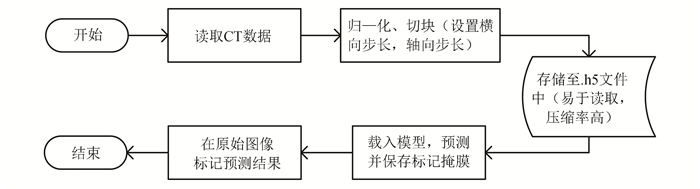
2.5 自动检测流程设计了系统检测流程(见图 6),为增加位置精确度,要以较小的步长遍历整套CT,在每一层扫描所有的位置,因此时间的花费主要集中在数据处理和存储,以及结果预测。
|
图 6 自动检测流程图 Figure 6 Flow chart of automatic detection |
为降低CNN训练过程中的随机因素对结果的影响,对每个训练集分别进行了三次训练,将对应的模型在测试集上的评价指标取均值作为最终的评估结果。评估结果(见表 2)。其中,第1、2和3组实验分别计算了由训练集1、训练集2和训练集3训练得到表 2的模型性能。
| 表 2 评估结果 Table 2 Results of evaluation |
由表 2可以看出,第1组具有较高的敏感性,但特异度非常低,这是因为训练集1的负样本选取方式不确定性较强,网络不能较为全面地学习到负样本的特征,对于肺结节检测,负样本显然数量远远大于正样本,假阳性率高使得结果易混淆,较低的特异性使结果参考意义大打折扣;第2组明显提高了特异度,但敏感性下降较多,说明在训练集2上网络对负样本特征过度拟合,对肺结节检测而言敏感性低意味着漏检率高,在肺癌筛查中是应当尽力避免的;与前两组相比第3组的结果同时具有较高的敏感性和特异性,说明后期对负样本的扩增方法是非常有效的,训练集3对样本进行了混合与均衡,网络得以充分学习到多样化的样本空间信息,因此可以在略微牺牲敏感性的基础上保证较高的特异度。并以多视图的方式展示了本文模型对部分样本预测的结果(见图 7),图中每一行图下方的数值表示模型预测该样本为结节的概率值。

|
图 7 部分样本预测结果 Figure 7 Part of prediction results of samples |
由图 7可以看出,本文模型对正负样本有较强的区分能力,在LUNA16提供的包含1 186个结节的候选点位上测试第3组实验模型,同样采用三个模型分别进行预测然后取平均值作为最终的预测结果(见表 3),与汪洋等人[22]将残差网络与传统SSD结合的检测算法相比,使用同样的测试数据,本文的结果明显更具优势。
| 表 3 与其他检测算法的对比 Table 3 Comparison with other detection algorithm |
本研究在模拟实际筛查任务中进行了测试,检测过程中每个样本选取包含待检测目标的40~50张CT切片,按照2.5的检测流程进行全局扫描检测,对检测为结节的区域中心标记一个点。因此针对单层切片的单个结节周围会出现多个标记,以及此结节涉及到的所有切片的区域会有多层标记。以像素为单位对带有标记点的掩膜进行叠加融合,连续双层的标记点颜色加深,连续三层的标记点置为红色。使用该方法在测试集中随机选取的CT样本中进行了检测实验,并以多视图的方式展示三维空间的可视化结果(见图 8),标注了检测目标的原始图像和本研究的检测方法检测出的标记示意。
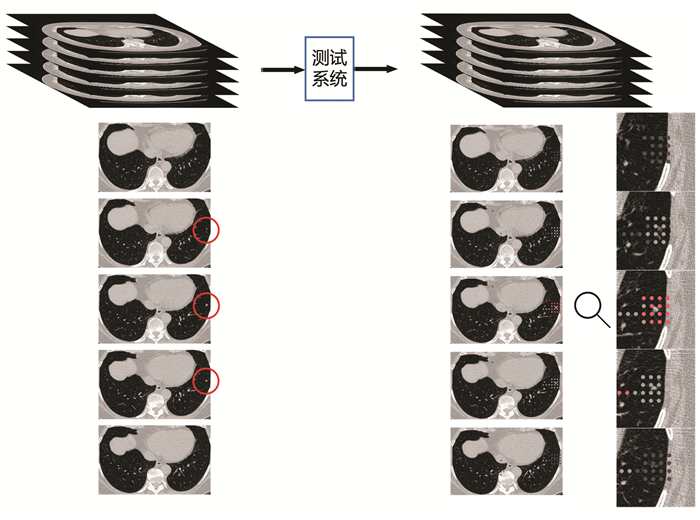
|
图 8 标记结果示意图 Figure 8 Schematic diagram of marking results |
对目标区域进行了放大显示,如图 8所示,红点密集的地方为检出区域。我们的模型在结节所在切片及邻近的切片均检测出了异常,在遍历整个CT数据块时能较为准确地识别结节所在区域,结果表明本研究的方法是切实可行的。在本文的硬件环境下,我们的模型对每个样本点位的预测时间约为1.5×10-3 s,则检测一套300张512×512的CT所需时间约为20~30 min。
4 结论本研究提出了基于三维卷积神经网络的肺结节探测方法,并通过实验取得了较好的检测模型和结果。为了提高肺结节初步筛查的准确率,采用不同的方法对正样本进行扩充、对负样本进行降维采样,构建了三个样本数据集。搭建了3D-CNN并分别使用三个数据集训练网络得到对应的分类模型,对比了三组数据训练的模型的效果,发现负样本的多样性在很大程度决定着模型的性能,多样化程度越高,模型的泛化能力越强,鲁棒性越高。最后从得到的肺结节检测模型中进行多次测试并将结果进行平均以降低偶然性误差对模型性能的影响,模型的敏感性为93.03%,特异性为97.39%。结果表明,本研究的肺结节检测方法可以有效的检测肺结节。此外,还设计了完整的结节自动化检测流程,充分利用了数据的空间特征,有效地提高了检测的精准度,大大降低了肺结节初筛的假阳性率。后续研究或将增大负样本采集数量和正样本扩充数量,进一步提升模型对肺结节的检出率。
| [1] |
赵愉, 鲁雯, 王远军, 等. 肺结节计算机辅助检测技术研究概述[J]. 生物医学工程学杂志, 2014, 5: 1172-1177. ZHAO Yu, LU Wen, WANG Yuanjun, et al. A review on the research progress of the computer-aided detection of pulmonary nodule[J]. Journal of Biomedical Engineering, 2014, 5: 1172-1177. DOI:10.7507/1001-5515.20140222 (  0) 0) |
| [2] |
TERAMOTO A, FUJITA H, YAMAMURO O, et al. Automated detection of pulmonary nodules in PET/CT images: Ensemble false-positive reduction using a convolutional neural network technique[J]. Medical Physics, 2016, 43(6): 2821-2827. DOI:10.1118/1.4948498 (  0) 0) |
| [3] |
ARESTA G, ANTÓNIO C, AURÉLIO C H. Detection of juxta-pleural lung nodules in computed tomography images[C]// SPIE Medical Imaging: Computer-Aided Diagnosis. Orlando: SPIE, 2016. DOI: 10.1117/12.2252022.
(  0) 0) |
| [4] |
JOHN J, MINI M G. Multilevel thresholding based segmentation and feature extraction for pulmonary nodule detection[J]. Procedia Technology, 2016, 24: 957-963. DOI:10.1016/j.protcy.2016.05.209 (  0) 0) |
| [5] |
LEEMPUT S V D, DORSSERS F, BEJNORDI B E. A novel spherical shell filter for reducing false positives in automatic detection of pulmonary nodules in thoracic CT scans[C]// Medical Imaging: Computer-Aided Diagnosis. Medical Imaging 2015: Computer-Aided Diagnosis. Orlando: SPIE, 2015. DOI: 10.1117/12.2082298.
(  0) 0) |
| [6] |
BURÇIN B O, POLAT K U, AHMET Ö, et al. Lung nodule detection in x-ray images: a new feature set[C]//6th European Conference of The International Federation for Medical and Biological Engineering. [S.L. ]: Springer, 2015, 45: 150-155. DOI: 10.1007/978-3-319-11128-5_38.
(  0) 0) |
| [7] |
齐守良, 司广磊, 岳勇, 等. 基于胸部CT图像的肺结节分割[J]. 北京生物医学工程, 2014(1): 33-38+44. QI Shouliang, SI Guanglei, YUE Yong, et al. Lung nodule segmentation based on thoracic CT images[J]. Beijing Biomedical Engineering, 2014(1): 33-38+44. DOI:10.3969/j.issn.1002-3208.2014.05 (  0) 0) |
| [8] |
HINTON G E, SALAKHUTDINOV R R. Reducing the dimensionality of data with neural networks[J]. Science, 2006, 313(5786): 504-507. DOI:10.1126/science.1127647 (  0) 0) |
| [9] |
王成弟, 郭际香, 杨阳, 等. 利用深度学习技术辅助肺结节的人工智能检测[J]. 中国呼吸与危重监护杂志, 2019, 18(3): 288-294. WANG Chengdi, GUO Jixiang, YANG Yang, et al. Artificial intelligence detection of pulmonary nodules using deep learning technology[J]. Chinese Journal of Respiratory and Critical Care Medicine, 2019, 18(3): 288-294. DOI:10.7507/1671-6205.201802042 (  0) 0) |
| [10] |
HE K, ZHANG X, REN S, et al. Deep residual learning for image recognition[C]//2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016, 1: 770-778. DOI: 10.1109/CVPR.2016.90.
(  0) 0) |
| [11] |
KRIZHEVSKY A, SUTSKEVER I, HINTON G E. ImageNet classification with deep convolutional neural networks[J]. Communications of the ACM, 2017, 60(6): 84-90. DOI:10.1145/3065386 (  0) 0) |
| [12] |
SUZUKI K. Overview of deep learning in medical imaging[J]. Radiological Physics and Technology, 2017, 10(3): 257-273. DOI:10.1007/s12194-017-0406-5 (  0) 0) |
| [13] |
SHAHRIMAN W N S, PHANG C R, NUMAN F, et al. Classification of brain functional connectivity using convolutional neural networks[J]. IOP Conference Series: Materials Science and Engineering, 2020, 884: 12003. DOI:10.1088/1757-899X/884/1/012003 (  0) 0) |
| [14] |
ISLAM R, IMRAN S, ASHIKUZZAMAN M, et al. Detection and classification of brain tumor based on multilevel segmentation with convolutional neural network[J]. Journal of Biomedical Science and Engineering, 2020, 13(4): 45-53. DOI:10.4236/jbise.2020.134004 (  0) 0) |
| [15] |
SETIO A A A, CIOMPI F, LITJENS G, et al. Pulmonary nodule detection in CT images: False positive reduction using multi-view convolutional networks[J]. IEEE Transactions on Medical Imaging, 2016, 35(5): 1160-1169. DOI:10.1109/TMI.2016.2536809 (  0) 0) |
| [16] |
KIM B C, YOON J S, CHOI J S, et al. Multi-scale gradual integration CNN for false positive reduction in pulmonary nodule detection[J]. Neural Networks, 2019, 115: 1-10. DOI:10.1016/j.neunet.2019.03.003 (  0) 0) |
| [17] |
DING Jia, LI Aoxue, HU Zhiqiang, et al. Accurate pulmonary nodule detection in computed tomography images using deep convolutional neural networks[J]. Lecture Notes in Computer Science, 2017, 10435: 559-567. DOI:10.1007/978-3-319-66179-7_64 (  0) 0) |
| [18] |
LECUN Y, BOTTOU L, BENGIO Y, et al. Gradient-based learning applied to document recognition[J]. Proceedings of the IEEE, 1998, 86(11): 2278-2324. DOI:10.1109/5.726791 (  0) 0) |
| [19] |
ZEILER M D, FERGUS R. Visualizing and understanding convolutional networks[C]// European Conference on Computer Vision. Cham: Springer, 2014. DOI: 10.1007/978-3-319-10590-1_53.
(  0) 0) |
| [20] |
李彦冬, 郝宗波, 雷航. 卷积神经网络研究综述[J]. 计算机应用, 2016, 36(9): 2508-2515, 2565. LI Yandong, HAO Zongbo, LEI Hang. Survey of convolutional neural network[J]. Journal of Computer Applications, 2016, 36(9): 2508-2515, 2565. DOI:10.11772/j.issn.1001-9081.2016.09.2508 (  0) 0) |
| [21] |
彭文献, 彭天舟, 叶小琴, 等. CT扫描参数对人体组织CT值影响的研究[J]. 中华放射医学与防护杂志, 2010, 30(1): 79-81. PENG Wenxian, PENG Tianzhou, YE Xiaoqin, et al. Effect of CT scanning parameters on CT number[J]. Chinese Journal of Radiological Medicine and Protection, 2010, 30(1): 79-81. DOI:10.3760/cma.j.issn.0254-5098.2010.01.026 (  0) 0) |
| [22] |
汪洋, 李建锋. 结合深度残差网络的SSD肺部结节检测方法[J]. 佳木斯大学学报(自然科学版), 2020, 38(6): 96-100. WANG Yang, LI Jianfeng. SSD lung nodule detection method combined with deep residual network[J]. Journal of Jiamusi University(Natural Science Edition), 2020, 38(6): 96-100. (  0) 0) |
 2022, Vol. 20
2022, Vol. 20


