肾细胞癌(简称肾癌)是一类常见的恶性肿瘤,起源于肾小管上皮,约占成人肿瘤发病率的2%~3%[1],主要病理类型有肾透明细胞癌、肾乳头状癌、肾嫌色细胞癌以及集合管癌等[2]。其中,肾透明细胞癌(Kidney renal clear cell carcinoma, KIRC)占肾癌的80%~90%,是最主要的病理类型[3]。随着肿瘤检测手段与治疗方法的进步,肾透明细胞癌患者的存活率得到显著提高,但总体生存期以及无进展生存期仍然较低,超过1/3的病人在诊断时已经发生转移,因此迫切需要新的早期诊断标志物以及治疗靶点来改善患者的生存预后情况[4]。微小RNA(microRNA,miRNA)是一类广泛存在于动植物体内的短链非编码RNA,仅有21~23个核苷酸序列,通过抑制翻译或促进细胞质中mRNA降解来调控转录后的基因表达[5]。大量研究证实miRNA在癌症的发生、发展、转移和患者生存中发挥重要作用,作为一种可能的非侵入性生物标志物,其预后价值在多种肿瘤中被广泛研究[6-8]。但相关研究报道在肾透明细胞癌中较少,仅有的几个研究也只是从miRNA的角度进行预后评估[9-10],未能联系下游mRNA,进而绘制miRNA-mRNA调控网络,以miRNA-mRNA关系对的形式深入挖掘其在肾透明细胞癌中的作用,为相关诊断与治疗提供参考,而这恰恰成为本研究的主要目的。
1 材料与方法 1.1 数据获取与整理于2020年7月12日检索癌症基因组图谱数据库(The Cancer Genome Atlas, TCGA, https://portal.gdc.cancer.gov/)并下载KIRC的mRNA与miRNA测序信息和相应临床数据,其中包括539例KIRC患者的肿瘤组织测序信息和72例正常组织测序信息。利用perl语言(perl 5.30.2, http://www.perl.org/)分别将mRNA与miRNA测序数据合并为矩阵文件;根据Ensembl数据库(http://www.ensembl.org/)将mRNA文件中“Ensembl_Stable_ID”转换为“Gene Symbol”,后整理为独立文件;根据miRbase数据库(http://www.mirbase.org/)将miRNA文件中的“MIMAT_ID”转换为“hsa-miR_ID”,后整理为独立文件。由于TCGA数据库属于公共的开放获取数据,故本研究不需要相关伦理学审核与批准。
1.2 差异分析利用R语言(R 3.6.3, https://www.r-project.org/)中的edgeR包对mRNA与miRNA分别进行差异分析,设置FDR(BH)矫正后的阈值P.adj<0.05,对数差异表达倍数变化绝对值|log2FC|>2;利用pheatmap包分别绘制表达上调或下调的前20个基因的热图。
1.3 预后模型构建利用perl语言将差异表达的miRNA同生存时间与生存状态进行合并,删除临床数据不完整以及生存时间小于30天的样本;首先设置生存分析过滤条件:P.adj<0.05,后利用R语言中的caret包将样本随机分为实验组(Train组)和验证组(Test组);利用survival包对Train组进行单因素与多因素Cox回归分析,筛选与患者预后密切相关的miRNA并构建预后风险评分模型,模型计算公式为:风险值(risk score)=风险基因表达量1×coef1+风险基因表达量2×coef2+...+风险基因表达量n×coefn(coef为风险系数);利用预后模型分别计算Train组与Test组各样本的风险值。将各组样本的风险值从低到高排序,依据中位数将患者分为低风险组和高风险组,风险值越大,生存率越低。利用survminer与survival ROC包对Train组与Test组进行Kaplan-Meier (K-M) 生存分析并计算ROC曲线的AUC值(设置Train组:P.adj<0.01,AUC>0.70;Test组:P.adj<0.01,AUC>0.69),如分组不满足设置条件,则重新利用caret包进行分组并循环。
1.4 预后模型评价利用survival包输出纳入模型的miRNA的生存曲线,验证其生存情况与coef的关系;同时利用OncoLnc网页工具(http://www.oncolnc.org/)输出模型中miRNA的生存曲线,验证数据准确性(截断值:50%);根据预后模型计算所有样本、Train组以及Test组的风险值,由低到高排序后根据中位数将患者分为高低风险两组,输出各组中模型的生存曲线,验证模型的准确性;利用survival ROC包输出所有样本、Train组以及Test组的ROC曲线,评价模型的预测能力。将所有样本的生存时间、生存状态、年龄、性别、WHO分级、分期、TNM分期及风险评分整理为数值数据,其中:生存状态(0,存活;1,死亡)、性别(0,女性;1,男性),利用survival包进行单因素与多因素的独立预后分析,从而评估风险值是否能作为患者的独立预后因子(评价标准:P.adj<0.01)。
1.5 miRNA-mRNA调控网络构建利用miRDB(http://mirdb.org/)、miRTarBase(http://mirtarbase.cuhk.edu.cn/)与TargetScan(http://www.targetscan.org/)数据库对纳入模型的miRNA进行靶基因预测,筛选同时被两个及以上数据库收录的靶基因与1.2中差异表达的mRNA取交集,明确其靶关系以及上下调表达关系,利用Cytoscape 3.7.2软件(https://cytoscape.org/)绘制miRNA-mRNA调控网络。利用perl语言将纳入网络的mRNA同临床数据进行合并,利用R语言中的survival包进行生存分析(设置P.adj<0.05),筛选与患者生存预后密切相关的mRNA。为了进一步筛选与患者生存预后相关的miRNA-mRNA调控关系对,根据miRNA对mRNA的功能发挥抑制作用这一生物学基础,制定关系对筛选标准:“高表达,生存率低”的miRNA对应的mRNA应符合“低表达,生存率低”;“低表达,生存率低”的miRNA对应的mRNA应符合“高表达,生存率低”。这样的miRNA-mRNA调控关系对具有重要生物学意义,通过选择性调控上游miRNA的表达,进而影响mRNA的表达,可改善肾透明细胞癌患者的生存情况。
2 结果 2.1 差异分析相比于正常组织,在KIRC肿瘤组织中共有3 613个差异表达的mRNA(上调2 603个,下调1 010个),上调与下调最显著的前20个mRNA(见图 1a)(C代表正常对照组,T代表肿瘤组);共有49个差异表达的miRNA(上调28个,下调21个),上调与下调最显著的前20个miRNA(见图 1b)。
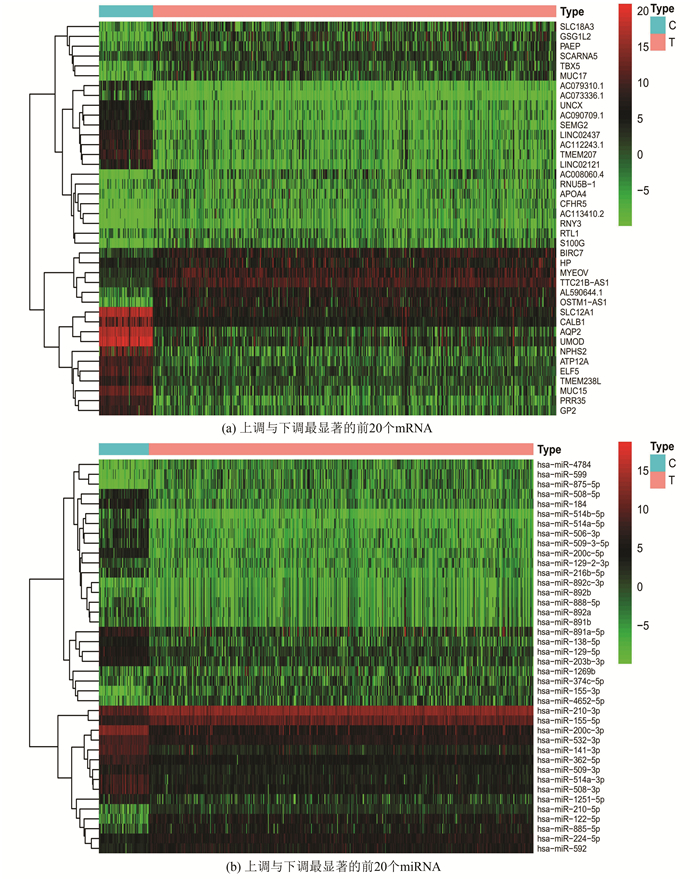
|
图 1 差异表达的mRNA与miRNA(C代表正常对照组,T代表肿瘤组) Figure 1 Differentially expressed mRNAs and miRNAs(C represents control group, T represents tumor group) |
删除临床数据不完整以及生存时间小于30天的样本,共有499个样本的miRNA表达数据被纳入分析。单因素Cox回归分析显示,共有3个miRNA的表达情况与患者的生存预后明显相关(见表 1);多因素Cox回归分析显示,共有2个miRNA被纳入预后风险评分模型(见表 2),分别是hsa-miR-21-5p和hsa-miR-1251-5p;预后风险评分模型计算公式为:风险值(risk score)=hsa-miR-21-5p表达量×0.603+hsa-miR-1251-5p表达量×-0.093。模型构建所基于的Train组共有251个样本,Test组共有248个样本。
| 表 1 单因素Cox回归分析 Table 1 Univariate Cox regression analysis |
| 表 2 多因素Cox回归分析 Table 2 Multivariate Cox regression analysis |
hsa-miR-21-5p生存曲线(见图 2a)显示,高表达组,生存率低;hsa-miR-1251-5p生存曲线(见图 2b)显示,低表达组,生存率低。根据预后模型计算所有样本、Train组以及Test组的风险值,所有样本(见图 2c)、Train组(见图 2d)以及Test组(见图 2e)的生存曲线均显示,高风险组生存率明显低于低风险组,模型准确。所有样本(见图 2f)、Train组(见图 2g)以及Test组(见图 2h)的ROC曲线显示,模型具备一定预测能力。利用OncoLnc网页工具输出hsa-miR-21-5p(见图 3a)与hsa-miR-1251-5p(见图 3b)在KIRC中的生存曲线,结果与本研究一致。单因素(见图 4a)与多因素(见图 4b)独立预后分析显示,年龄与风险值均可作为患者生存预后的独立预测因子,且随着二者的增大,患者生存率逐渐降低。

|
图 2 生存曲线与ROC曲线 Figure 2 Survival curves and ROC curves |
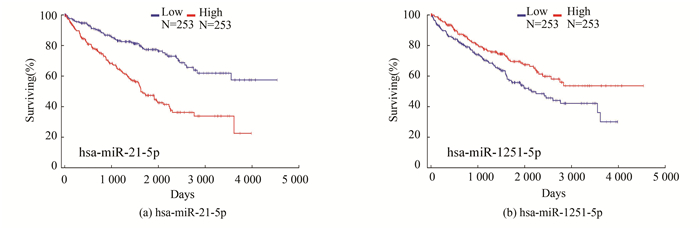
|
图 3 OncoLnc网页工具 Figure 3 OncoLnc web tool |

|
图 4 单因素与多因素独立预后分析 Figure 4 Univariate and multivariate independent prognostic analyses |
利用miRDB、miRTarBase与TargetScan三个数据库对纳入模型的2个miRNA,hsa-miR-21-5p与hsa-miR-1251-5p进行靶基因预测,共预测到12 354个靶基因,其中同时被两个及以上数据库收录的靶基因有991个,与1.2中差异表达的mRNA取交集,共得到31个靶基因,所构建miRNA-mRNA调控网络(见图 5)(图中红色代表表达上调,绿色代表表达下调)。对网络中的mRNA进行生存分析,共有8个mRNA的表达与患者生存预后相关,分别是:AIF1L、ALX1、CLIC5、COBLL1、FCGR1A、FCGR1B、FGF1、NIPAL1,生存曲线(见图 6)。根据制定的miRNA-mRNA关系对筛选标准,共有7个关系对具有重要生物学意义,通过调控miRNA的表达,可影响下游mRNA水平的变化,进而改善患者的生存预后情况(见表 3)。

|
图 5 miRNA-mRNA调控网络 Figure 5 miRNA-mRNA regulatory network |
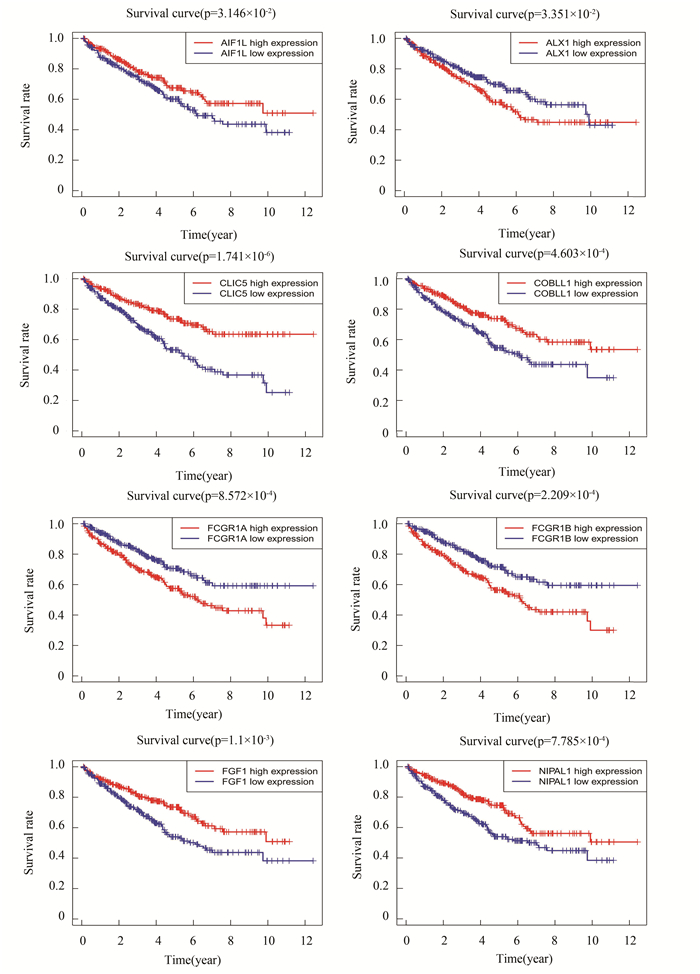
|
图 6 mRNA生存曲线 Figure 6 Survival curves of mRNAs |
| 表 3 miRNA-mRNA调控关系对 Table 3 miRNA-mRNA regulatory pairs |
miRNA作为一类具有调控功能的非编码RNA,被证明几乎参与肿瘤发生进展的全过程,包括持续增殖、无限复制、回避生长抑制、抵抗凋亡、血管生成以及肿瘤微环境构建等,在不同类型肿瘤中表达异常的miRNA通过作用于特定的靶基因mRNA,发挥抑癌或促癌作用[11]。由于其分子量较小,结构稳定不易降解,包裹在外泌体中的miRNA极易进入循环系统,可稳定存在于血清、血浆、脑脊液、唾液以及尿液等,这些特点使其逐步成为一种可靠的生物标志物,被广泛应用在各类肿瘤的诊断与预后评估中[12]。而最新研究表明,一种新型的细胞内活检技术,仅需约10分钟,即可从活细胞中分离出目标miRNA,大大缩短检测所需时间,同时操作简单,灵敏度和准确性都得以提升,这项技术为miRNA在体内的检测提供便利,同样有助于癌症的早期诊断与评估[13]。
本研究所构建的预后模型共纳入2个miRNA,分别是hsa-miR-21-5p与hsa-miR-1251-5p。miR-21是一个广泛表达的miRNA,在多种肿瘤细胞中增高,可通过抑制PTEN或其他抑癌基因来发挥其致癌作用。研究发现,在肾透明细胞癌组织中,miR-21-5p的过表达可抑制SATB1的表达,进而导致患者的不良预后[14];另一项研究则对石蜡包埋的肾透明细胞癌组织中miRNA的表达与患者生存情况之间的关系进行回顾性队列研究,结果发现miR-21-5p与miR-210-3p表达上调最显著,且与患者的不良预后密切相关[15],这些研究均提示miR-21-5p可成为该肿瘤的有效预后评价指标,构建预后模型具有一定临床意义。一项有关卵巢癌的研究发现,miR-1251-5p在癌组织中高表达,可通过靶向抑癌基因TBCC,进而促进癌细胞的增殖和自噬的发生[16]。而在本研究发现,miR-1251-5p在KIRC组织中低表达,但其高表达组生存率更高,且模型中coef值为负,即表达越多,患者的风险值越低,生存预后情况越好,提示过表达miR-1251-5p在KIRC中可能发挥抑制癌组织的作用,这与卵巢癌中的研究不一致。由于同一miRNA在不同组织中往往结合不同靶基因发挥不同功能,因此miR-1251-5p与KIRC生存预后的关系仍有待考证,具有一定研究价值。
由于一个miRNA的靶基因往往有多个,一个靶基因又可能和多个miRNA相互作用,组成复杂的调控网络,而单纯纠正某个miRNA异常可能会引起许多副作用,只有从miRNA-mRNA调控关系对的角度出发,精准调控mRNA的表达,才能实现对肿瘤的靶向治疗。因此本研究通过对KIRC肿瘤组织与正常组织mRNA的表达情况进行差异分析,筛选到3 613个差异表达的mRNA,后与miRNA的靶基因进行取交集,得到31个mRNA,对这些mRNA进行生存分析并根据关系对筛选标准,筛选到7个miRNA-mRNA进行后续研究。
其中,与miR-21-5p结合的mRNA共有5个,分别是AIF1L、CLIC5、COBLL1、FGF1、NIPAL1,且在KIRC组织中均低表达,生存分析均显示低表达组的生存率较低,提示其均可能发挥抑癌作用。(1)AIF1L(allograft inflammatory factor 1 like,同种异体移植炎症因子1样)是一类具有EF手型模序结构的钙结合蛋白,其在肾脏组织中高表达。一项有关乳腺癌的研究表明,肿瘤组织中AIF1L低表达且高度甲基化,其可导致患者的不良预后,过表达AIF1L则可抑制细胞的扩散,改变细胞形态,减少突起的形成[17]。(2)CLIC5(chloride intracellular channel 5,细胞内氯离子通道5)所编码的蛋白质与肌动蛋白为基础的细胞骨架结构相关,同时参与肾小球足细胞和内皮细胞的维持。研究表明,在肝癌细胞Huh7中抑制CLIC5表达,可导致肿瘤细胞迁移和侵袭能力下降[18];而在小儿急性淋巴细胞白血病中发现,过表达CLIC5可促进氧化应激诱导的DNA损伤积累,从而促进白血病的发生[19]。(3)COBLL1(cordon-bleu WH2 repeat protein like 1,蓝带WH2重复蛋白1)在肾脏组织中高表达,其具有肌动蛋白结合结构域,在临床前列腺癌组织中表达上调,并与前列腺癌患者不良预后有关[20];在慢性髓系白血病的急性转化期,COBLL1高表达,且与生存率降低显著相关[21]。(4)FGF1(fibroblast growth factor 1,成纤维细胞生长因子1)是成纤维细胞生长因子家族的一员,具有广泛的有丝分裂和细胞存活活性,参与多种生物过程,包括胚胎发育、细胞生长、形态形成、组织修复、肿瘤生长和侵袭等。研究证实,FGF家族成员在功能上参与了肾透明细胞癌的进展,通过自分泌和(或)旁分泌的作用直接刺激细胞增殖[22]。(5)NIPAL1(NIPA like domain containing 1,NIPA类域包含子1)在食管癌组织中表达降低,过表达该基因通过阻滞细胞G2/M期,抑制细胞有丝分裂及减少细胞骨架中丝状伪足和板状伪足的形成,抑制癌细胞的增殖和迁移[23];而在口腔鳞状细胞癌中的研究则发现,NIPAL1可调节肿瘤细胞和内皮细胞的生长和粘附,发挥促癌作用[24]。因此,AIF1L极有可能是KIRC的一个抑癌基因,成为有效治疗靶点;NIPAL1在不同肿瘤类型中发挥促癌或抑癌作用,其与KIRC的关系有待进一步研究与考证;而CLIC5、COBLL1、FGF1三个mRNA在几类癌症中均发挥促癌作用,与我们的假设相异。
与miR-1251-5p结合的mRNA有2个,分别是FCGR1A(Fc fragment of IgG receptor Ia,免疫球蛋白G Fc段受体1α)与FCGR1B(Fc fragment of IgG receptor Iβ,免疫球蛋白G Fc段受体1β),它们是位于1号染色体上相互关联的两个基因,该家族共有三个亚型,共同编码高亲和力的Fc -γ受体。二者在KIRC组织中均高表达,且生存分析显示高表达组生存率较低,提示其可能发挥促癌作用。一项前瞻性研究发现,FCGR1A的高表达与三阴性乳腺癌患者的不良预后有关[25];另一项研究则发现,抗癌基因miR-29可通过靶向FCGR1B,降低KIRC肿瘤细胞的迁移和侵袭能力,改善患者的生存预后情况[26]。
本研究发现一个新的miRNA与KIRC生存预后相关,即hsa-miR-1251-5p;发现7个miRNA-mRNA调控关系对可为肾透明细胞癌的研究与治疗提供靶点,其中miR-21-5p-AIF1L、miR-21-5p-NIPAL1、miR-1251-5p-FCGR1A与miR-1251-5p-FCGR1B,此4个关系对均未在KIRC中研究报道,具有较大研究价值与可信度。本研究的特色之处在于,利用miRNA进行预后模型构建,作为一种无创性检查手段具有一定应用价值;所分析的miRNA与mRNA数据均来源于同一批患者,具有一定说服力;在miRNA-mRNA调控网络的基础上,进一步进行mRNA的生存分析,筛选到更具研究价值的调控关系对。本研究的不足之处在于,未在其他数据集中对预后模型进行验证与评估;所筛选到的mRNA表达与生存情况未在蛋白质层面进行分析;未对有研究价值的调控关系对进行功能实验验证;这些将在后续研究中进一步完善。
4 结论1) 通过对TCGA数据库中KIRC的mRNA与miRNA数据进行差异分析,结合临床数据通过miRNA表达量有关的单因素与多因素Cox回归分析,成功构建可预测患者生存预后情况的风险评分模型,计算公式为:风险值(Risk score)=hsa-miR-21-5p表达量×0.603+hsa-miR-1251-5p表达量×-0.093。
2) 通过对纳入模型的miRNA进行靶基因预测,结果与差异表达的mRNA取交集,成功构建miRNA-mRNA调控网络,对网络中的mRNA进行生存分析,筛选到7个重要的调控关系对,可为相关研究与治疗提供理论支持。
| [1] |
SIEGEL R L, MILLER K D, JEMAL A. Cancer statistics, 2018[J]. CA: A Cancer Journal for Clinicians, 2018, 68(1): 7-30. DOI:10.3322/caac.21442 (  0) 0) |
| [2] |
RUPP N J, MOCH H. Characterization of different renal cell carcinoma entities[J]. Urologe A, 2020, 59(2): 135-141. DOI:10.1007/s00120-019-01108-5 (  0) 0) |
| [3] |
LJUNGBERG B, BENSALAH K, CANFIELD S, et al. EAU guidelines on renal cell carcinoma: 2014 update[J]. European Urology, 2015, 67(5): 913-924. DOI:10.1016/j.eururo.2015.01.005 (  0) 0) |
| [4] |
LINEHAN W M, RICKETTS C J. The cancer genome atlas of renal cell carcinoma: Findings and clinical implications[J]. Nature Reviews Urology, 2019, 16(9): 539-552. DOI:10.1038/s41585-019-0211-5 (  0) 0) |
| [5] |
LU T X, ROTHENBERG M E. MicroRNA[J]. Journal of Allergy and Clinical Immunology, 2018, 141(4): 1202-1207. DOI:10.1016/j.jaci.2017.08.034 (  0) 0) |
| [6] |
张皓旻, 杨波, 陈红飞, 等. 肺腺癌预后相关miRNA生物信息学筛选及其临床意义[J]. 中国应用生理学杂志, 2018, 34(6): 530-535. ZHANG Haomin, YANG Bo, CHEN Hongfei, et al. Prognosis-related miRNA bioinformatics screening of lung adenocarcinoma and its clinical significance[J]. Chinese Journal of Applied Physiology, 2018, 34(6): 530-535. DOI:10.12047/j.cjap.5755.2018.118 (  0) 0) |
| [7] |
TANG J, MA W, LUO L. Establishment of the prognosis predicting signature for endometrial cancer patient[J]. Medical Science Monitor, 2019, 25: 8248-8259. DOI:10.12659/MSM.917813 (  0) 0) |
| [8] |
TAKASHIMA Y, KAWAGUCHI A, IWADATE Y. miR-101, miR-548b, miR-554, and miR-1202 are reliable prognosis predictors of the miRNAs associated with cancer immunity in primary central nervous system lymphoma[J]. PLoS One, 2020, 15(2): e0229577. DOI:10.1371/journal.pone.0229577 (  0) 0) |
| [9] |
LUO Y, CHEN L, WANG G, et al. Identification of a three-miRNA signature as a novel potential prognostic biomarker in patients with clear cell renal cell carcinoma[J]. Journal of Cell Biochemistry, 2019, 120(8): 13751-13764. DOI:10.1002/jcb.28648 (  0) 0) |
| [10] |
XIE M, LV Y, LIU Z. Identification and validation of a four-miRNA (miRNA-21-5p, miRNA-9-5p, miR-149-5p, and miRNA-30b-5p) prognosis signature in clear cell renal cell carcinoma[J]. Cancer Management & Research, 2018, 10: 5759-5766. DOI:10.2147/CMAR.S187109 (  0) 0) |
| [11] |
IORIO M V, CROCE C M. MicroRNA dysregulation in cancer: diagnostics, monitoring and therapeutics. A comprehensive review[J]. EMBO Molecular Medicine, 2012, 4(3): 143-159. DOI:10.1002/emmm.201100209 (  0) 0) |
| [12] |
DUFRESNE S, REBILLARD A, MUTI P, et al. A review of physical activity and circulating mirna expression: implications in cancer risk and progression[J]. Cancer Epidemiol Biomarkers Preview, 2018, 27(1): 11-24. DOI:10.1158/1055-9965.EPI-16-0969 (  0) 0) |
| [13] |
WANG Z, QI L, YANG Y, et al. High-throughput intracellular biopsy of microRNAs for dissecting the temporal dynamics of cellular heterogeneity[J]. Science Advances, 2020, 6(24): eaba4971. DOI:10.1126/sciadv.aba4971 (  0) 0) |
| [14] |
KOWALCZYK A E, KRAZINSKI B E, GODLEWSKI J, et al. SATB1 is down-regulated in clear cell renal cell carcinoma and correlates with mir-21-5p overexpression and poor prognosis[J]. Cancer Genomics Proteomics, 2016, 13(3): 209-17. (  0) 0) |
| [15] |
PETROZZA V, CARBONE A, BELLISSIMO T, et al. Oncogenic micrornas characterization in clear cell renal cell carcinoma[J]. International Journal of Molecular Sciences, 2015, 16(12): 29219-29225. DOI:10.3390/ijms161226160 (  0) 0) |
| [16] |
SHAO Y, LIU X, MENG J, et al. MicroRNA-1251-5p Promotes carcinogenesis and autophagy via targeting the tumor suppressor TBCC in ovarian cancer cells[J]. Molecular Therapy: The Journal of the American Society of Gene Therapy, 2019, 27(9): 1653-1664. DOI:10.1016/j.ymthe.2019.06.005 (  0) 0) |
| [17] |
LIU P, LI W, HU Y, et al. Absence of AIF1L contributes to cell migration and a poor prognosis of breast cancer[J]. Onco Targets Therapy, 2018, 11: 5485-5498. DOI:10.2147/OTT.S165874 (  0) 0) |
| [18] |
FLORES-TELLEZ T N, LOPEZ T V, VASQUEZ GARZON V R, et al. Co-expression of ezrin-clic5-podocalyxin is associated with migration and invasiveness in hepatocellular carcinoma[J]. PLoS One, 2015, 10(7): e0131605. DOI:10.1371/journal.pone.0131605 (  0) 0) |
| [19] |
NEVEU B, SPINELLA J F, RICHER C, et al. CLIC5: A novel ETV6 target gene in childhood acute lymphoblastic leukemia[J]. Haematologica, 2016, 101(12): 1534-1543. DOI:10.3324/haematol.2016.149740 (  0) 0) |
| [20] |
TAKAYAMA K I, SUZUKI T, FUJIMURA T, et al. COBLL1 modulates cell morphology and facilitates androgen receptor genomic binding in advanced prostate cancer[J]. Proceedings National Academy of Sciences, 2018, 115(19): 4975-4980. DOI:10.1073/pnas.1721957115 (  0) 0) |
| [21] |
HAN S H, KIM S H, KIM H J, et al. Cobll1 is linked to drug resistance and blastic transformation in chronic myeloid leukemia[J]. Leukemia, 2017, 31(7): 1532-1539. DOI:10.1038/leu.2017.72 (  0) 0) |
| [22] |
RAMP U, REINECKE P, GABBERT H E, et al. Differential response to transforming growth factor (TGF)-alpha and fibroblast growth factor (FGF) in human renal cell carcinomas of the clear cell and papillary types[J]. European Journal of Cancer, 2000, 36(7): 932-941. DOI:10.1016/s0959-8049(00)00030-7 (  0) 0) |
| [23] |
李蒙妍. NIPAL1和GRHL1在食管鳞状细胞癌的临床意义及其作用机制初步研究[D]. 郑州: 郑州大学, 2018. LI Mengyan. Clinical significance and mechanism of NIPAL1 and GRHL1 in esophageal squamous cell carcinoma[D]. Zhengzhou: Zhengzhou University, 2018. (  0) 0) |
| [24] |
SASAHIRA T, NISHIGUCHI Y, KURIHARA-SHIMOMURA M, et al. NIPA-like domain containing 1 is a novel tumor-promoting factor in oral squamous cell carcinoma[J]. Journal of Cancer Research & Clinical Oncology, 2018, 144(5): 875-882. DOI:10.1007/s00432-018-2612-x (  0) 0) |
| [25] |
JIANG Y Z, LIU Y R, XU X E, et al. Transcriptome analysis of triple-negative breast cancer reveals an integrated mRNA-lncRNA signature with predictive and prognostic value[J]. Cancer Research, 2016, 76(8): 2105-2114. DOI:10.1158/0008-5472.CAN-15-3284 (  0) 0) |
| [26] |
YAMADA Y, SUGAWARA S, ARAI T, et al. Molecular pathogenesis of renal cell carcinoma: Impact of the anti-tumor miR-29 family on gene regulation[J]. International Journal of Urology: Official Journal of the Japanese Urological Association, 2018, 25(11): 953-965. DOI:10.1111/iju.13783 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


