2. 西北工业大学 医学研究院,西安 710072
2. Institute of Medical Research, Northwestern Polytechnical University, Xi'an 710072, China
甲状腺癌(Thyroid cancer, THCA)是内分泌系中最常见的恶性肿瘤,易受饮食、遗传、环境等多种因素的影响[1]。近年来,中国的甲状腺癌的发病率呈上升趋势且女性高于男性[2]。基于甲状腺癌术前诊断率低且晚期患者预后差的特点,探索其发病机制并寻找新型分子标志物,对于早发现、早诊断、早治疗具有重要意义[3]。近年来,随着高通量测序技术及基因芯片技术的进步,其在生命科学领域的应用愈加广泛。利用生物信息学方法在庞大的基因数据库中筛选癌症诊断的生物标志物方法的有效性已经被大量的临床数据证实[4]。
目前已有学者[3]应用基因表达综合数据库(The gene expression omnibus, GEO)对甲状腺癌潜在的miRNAs生物学标志物及靶基因功能和信号通路进行分析。Choi等通过肿瘤基因组图谱(The Cancer Genome Atlas, TCGA)建立了一个12个基因预测模型(包括BCC8,CHI3L1,CLCNKA,FAM155B,GABRG1,LUM,MRO,MT1G,MT1H,SELV,SLC4A4和TMEM92),用于预测甲状腺乳头状瘤(Papillary thyroid carcinoma, PTC)中的淋巴结转移[5]。此外,Lin等人使用肿瘤基因组图谱(The Cancer Genome Atlas, TCGA)中与免疫相关的7个基因建立预后预测模型(包括AGTR1,CTGF,FAM3B,IL11,IL17C,PTH2R和SPAG11A)用于预测PTC预后情况[6]。因此,进一步探索公共数据库,将为寻找THCA发生发展的分子机制及挖掘疾病新型生物标志物提供依据。本研究整合了TCGA中的THCA基因表达数据,应用edgeR和limma两种算法对诊断甲状腺癌具有潜在应用价值的基因标志物做出预测,后续通过双聚类分析及ROC分析进一步验证预测基因的可靠性。通过生物信息学分析鉴定出了11个THCA的差异表达基因(Differentially expressed genes, DEGs)及与疾病诊断相关的基因,以期为探索THCA发生发展的分子机制及挖掘疾病新型生物标志物提供依据[2, 7]。
1 资料与方法 1.1 数据来源及数据处理通过UCSC xean网站下载TCGA数据库中的甲状腺癌基因表达数据(https://gdc.xenahubs.net/download/TCGA-THCA.htseq_counts.tsv.gz),该数据为Log2标准化后的数据。该数据集包含了510例肿瘤样本和58例正常对照样本。
在UCSC xean网站下载THCA对应的ID/Gene Mapping (https://gdc.xenahubs.net/download/gencode.v22.annotation.gene.probeMap),将基因ID与基因名称进行匹配,当有多个ID对应同一个基因名称时,求多个ID的平均表达值。
1.2 统计学分析分别运用R/Bioconductor中的edgeR包[8]和limma包[9]对预处理过后的THCA数据提取差异表达基因。选取肿瘤与正常对照组间表达差异倍数(Fold change,FC)大于2,P < 0.05的基因作为差异表达基因(Differentially expressed genes, DEGs),将两种算下的DEGs取交集。运用R中的pheatmap包对DEGs进行双聚类。运用Medcalc19.0.4统计软件分析,检验所筛选的DEGs在鉴别肿瘤样本和正常对照样本的应用效果,获取敏感性、特异性、曲线下面积等指标。
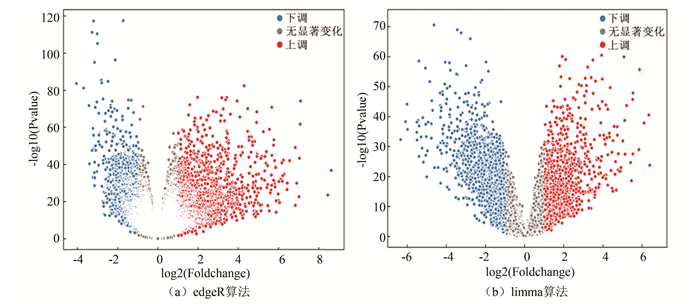
2 结果分析 2.1 THCA中差异表达基因的筛选首先选取肿瘤与正常对照组间倍数改变大于2,P < 0.05的基因。其中,利用edgeR包得到差异基因共2 768个(上调1 765个,下调1 003个);利用limma包得到差异基因共2 699个(上调1 080个,下调1 619个)(见图 1)。将上述两种算法的结果求交集并去除表达趋势不一致的基因,最终得到差异基因共1 945个(上调1 033个,下调912个)。进一步分析显示,随着组间差异倍数增大,差异基因主要表现为在肿瘤组织中上调(见图 2)。
|
图 1 肿瘤组与正常对照组间DEGs火山图 Figure 1 Volcanic diagram of DEGs between tumor group and normal control group |

|
图 2 不同倍数改变的DEGs统计 Figure 2 DEGs statistics with different multiples |
分析显示,随着组间差异倍数的增大,肿瘤组织中DEGs绝大部分表现为上调的模式,我们进一步筛选出组间差异倍数在32倍(log2(FC)=5)以上的DEGs进行后续分析。该11个差异基因在两种算法中的计算结果(见表 1)。对11个DEGs和样本进行双聚类分析,可以看出,基于组间的DEGs表达能够较好的将肿瘤样本和正常对照样本进行区分(见图 3)。
| 表 1 筛选出的DEGs汇总 Table 1 Summary of screened DEGs |

|
图 3 DEGs和样本的双聚类分析 Figure 3 Biclustering analysis of DEGs and samples 注: 横坐标为样本(红色代表癌症组, 蓝色代表正常组), 纵坐标为差异表达基因. |
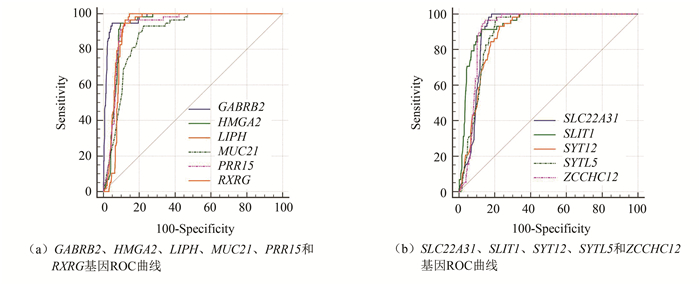
进一步对筛选出的11个候选差异基因进行显示,基于基因表达值鉴别肿瘤组与对照组的敏感性和特异性均在70%以上,曲线下面积均大于0.8(见图 4及表 2)。提示上述基因可以较好地鉴别THCA肿瘤组和正常组。
|
图 4 基于候选基因鉴别肿瘤样本与正常对照组的ROC曲线 Figure 4 ROC curves of tumor samples and normal control group based on candidate genes |
| 表 2 基于候选基因鉴别肿瘤样本与正常对照组的应用效果 Table 2 Application effects of differentiating tumor samples from normal control group based on candidate genes |
THCA是内分泌系统常见的恶性肿瘤之一,寻找潜在的分子标志物对于临床与科研工作至关重要。TCGA作为全球最大的癌症基因数据库,其大量且规范的样本及基因表达数据为研究探索THCA的发病机制及基因标志物提供了平台[10]。本文基于TCGA数据库中的THCA基因表达数据,对edgeR算法和limma算法的处理结果取交集并选择fold change>2、P < 0.05且差异表达变化趋势一致的基因为DEGs,最终得到了1 945个DEGs。且随着差异倍数的不断增大,肿瘤组织中DEGs主要表现为表达上调的改变模式。ROC结果显示,11个差异显著的DEGs在鉴别肿瘤与正常组具有较好的结果。预期由这11个表达差异的DEGs组合将为TCGA的诊断、预后及复发风险评估有一定的应用价值。
Jin Y等人发现GABRB2基因在甲状腺肿瘤组织中过度表达,通过与正常组织为对照组的队列研究中显示GABRB2在PCT中过表达与淋巴结转移相关,体外实验表明GABRB2下调会显著抑制三种PCT细胞系的集落形成,迁徙和侵袭[11]。说明其有作为分子诊断标志物的潜力。HMGA2是一种非组蛋白的转录因子,可影响包括细胞周期过程、DNA损伤修复、细胞凋亡、衰老等生物学过程。Chiappetta G等人通过免疫组织化学和定量RT-PCR分析,认为HMGA2表达与人类甲状腺肿瘤中的恶性表型相关[12]。Ivan Šamija通过对细针穿刺甲状腺结节中HMGA2分析认为其可以作为区分恶性和良性甲状腺结节的辅助生物标志物[13]。MUC21是一种从TA3-Ha细胞中鉴定出一种新型粘蛋白。它在甲状腺癌中通过mRNA水平和抗体结合被发现,但在相邻的正常上皮中却没有,这就进一步说明这种粘蛋白有用作甲状腺癌的组织或血清标志物[14]。SYT12有相关研究证明,SYT12在甲状腺癌中具有一定的预后意义,SYT12可用于PCT患者的病情进展预测的过表达与癌症的转移有关。但SYT12子癌症中的分子生物学作用仍不清楚[15]。一些研究表明ZCCHC12基因与某些疾病有关,但ZCCHC12在甲状腺癌中的功能尚未确定。Wang O的结论证明:ZCCHC12的表达在甲状腺癌中显著上调,该基因过表达与淋巴结转移相关,说明该基因具有重要的生物学功能,并有作为甲状腺癌症中与转移相关的癌基因的潜在价值[16]。
Li YDENG等研究发现,LIPH在甲状腺癌组织中的高表达与淋巴结转移密切相关,其细胞功能实验表明,LIPH与甲状腺癌细胞系的恶性行为呈正相关,这可以作为甲状腺癌诊断标志物的有力证据[17]。Jarzab B在应用基因芯片方法对23例甲状腺癌患者基因表达谱分析中也明确RXRG的表达有显著升高,但是该基因在甲状腺癌发生发展中发挥具体作用的机制还未明确[18]。
除了以上7种预测基因在甲状腺癌中的相关报道,目前尚未有对于PRR15、SLC22A31、SLIT1和SYTL5 4种基因在甲状腺癌作用机制的报道,但是SYTL5和PRR15基因表达上调在其他癌症中的有多次报道。Wright PK等人通过免疫组化显示SYTL5在正常乳腺导管上皮细胞、原位导管癌和浸润性乳腺癌细胞中表达[19]。Meunier D等人研究表明PRR15在小鼠和人类胃肠道肿瘤中高表达,可能APC蛋白的缺失有关[20]。预测的11个基因中发现了4个以往没有报道与甲状腺癌相关的基因值得进一步研究,但是这些基因用于甲状腺癌诊断的可靠性还有待更加深入的机制研究。
综上,本研究通过分析TCGA甲状腺癌表达数据,鉴定出了与THCA发生发展相关的11种生物标志物,鉴于此,在今后的临床研究中可以以这些显著表达差异的基因作为药物治疗的靶向治疗点。本研究不足在于缺乏更深入的机制研究,首先转录组学的分析并不能完全代表机体总体变化,其次,由于缺乏体内或体外试验,该分子预测结果还需要进一步的临床样本验证。
4 结论分析了TCGA中的甲状腺癌表达谱数据,鉴定出了与疾病诊断显著相关的11个差异表达基因,并通过双聚类分析及ROC分析进一步验证显示预测基因的可靠性,这将为探索甲状腺肿瘤发生发展机制及寻找新型分子标志物提供依据。
| [1] |
董芬, 张彪, 单广良. 中国甲状腺癌的流行现状和影响因素[J]. 中国癌症杂志, 2016, 26(1): 47-52. DONG Fen, ZHANG Biao, SHAN Guangliang. Distribution and risk factors of thyroid cancer in China[J]. China Oncology, 2016, 26(1): 47-52. DOI:10.3969/j.issn.1007-3969.2016.01.008 (  0) 0) |
| [2] |
赵建英, 王玄, 李云慧, 等. 生物信息学方法筛选甲状腺癌潜在的miRNAs生物学标志物及靶基因功能和信号通路分析[J]. 解放军医药杂志, 2017, 29(12): 5-9. ZHAO Jianying, WANG Xuan, LI Yunhui, et al. Bioinformatics method in screening of potential miRNAs biological marker, target gene function and signal pathways in thyroid cancer[J]. Medical & Pharmaceutical Journal of Chinese People's Liberation Army, 2017, 29(12): 5-9. DOI:10.3969/j.issn.2095-140X.2017.12.002 (  0) 0) |
| [3] |
袁小艳, 梁韡, 张舒, 等. 基于GEO甲状腺癌芯片数据的生物信息学分析[J]. 重庆医学, 2018, 47(36): 4619-4622+4627. YUAN Xiaoyan, LIANG Wei, ZHANG Shu, et al. Bioinformatics analysis of thyroid cancer genome microarray based on GEO database[J]. Chongqing Medicine, 2018, 47(36): 4619-4622+4627. DOI:10.3969/j.issn.1671-8348.2018.36.014 (  0) 0) |
| [4] |
邹小龙, 董雪松, 孙学溥. 结肠癌中核内miRNA的激活调控作用研究[J]. 生物信息学, 2019, 17(02): 111-115. ZOU Xiaolong, DONG Xuesong, SUN Xuepu. Activation regulation of nuclear miRNA regulation in colon cancer[J]. Chinese Journal of Bioinformatics, 2019, 17(02): 111-115. DOI:10.12113/j.issn.1672-5565.201903009 (  0) 0) |
| [5] |
CHOI K Y, KIM J H, PARK I S, et al. Predictive gene signatures of nodal metastasis in papillary thyroid carcinoma[J]. Cancer Biomark, 2018, 22(1): 35-42. DOI:10.3233/CBM-170784 (  0) 0) |
| [6] |
LIN P, GUO Y N, SHI L, et al. Development of a prognostic index based on an immunogenomic landscape analysis of papillary thyroid cancer[J]. Aging, 2019, 11(2): 480-500. DOI:10.18632/aging.101754 (  0) 0) |
| [7] |
汤喜, 张成瑶, 周晓红, 等. 甲状腺癌非编码RNA与mRNA差异表达谱及竞争性内源RNA调控网络分析[J]. 中国普通外科杂志, 2018, 27(11): 1409-1416. TANG Xi, ZHANG Chengyao, ZHOU Xiaohong, et al. Analysis of differential expression profiles of non-coding RNAs and mRNAs and competing endogenous RNA regulatory network in thyroid cancer[J]. Chinese Journal of General Surgery, 2018, 27(11): 1409-1416. DOI:10.7659/j.issn.1005-6947.2018.11.007 (  0) 0) |
| [8] |
ROBINSON M D, MCCARTHY D J, SMYTH G K. edgeR: a bioconductor package for differential expression analysis of digital gene expression data[J]. Bioinformatics, 2010, 26(1): 139-140. DOI:10.1093/bioinformatics/btp616 (  0) 0) |
| [9] |
RITCHIE M E, PHIPSON B, WU D, et al. limma powers differential expression analyses for RNA-sequencing and microarray studies[J]. Nucleic Acids Research, 2015, 43(7): e47. DOI:10.1093/nar/gkv007 (  0) 0) |
| [10] |
CHATTERJEE A, STOCKWELL P A, RODGER E J, et al. scan_tcga tools for integrated epigenomic and transcriptomic analysis of tumor subgroups[J]. Epigenomics, 2016, 8(10): 1315-1330. DOI:10.2217/epi-2016-0063 (  0) 0) |
| [11] |
JIN Y, JIN W, ZHENG Z, et al. GABRB2 plays an important role in the lymph node metastasis of papillary thyroid cancer[J]. Biochemical and Biophysical Research Communications, 2017, 492(3): 323-330. DOI:10.1016/j.bbrc.2017.08.114 (  0) 0) |
| [12] |
CHIAPPETTA G, FERRARO A, VUTTARIELLO E, et al. HMGA2 mRNA expression correlates with the malignant phenotype in human thyroid neoplasias[J]. European Journal of Cancer, 2008, 44(7): 1015-1021. DOI:10.1016/j.ejca.2008.02.039 (  0) 0) |
| [13] |
ŠAMIJAI, MATEŠAN, KOŽAJS, 等. HMGA2 gene expression in fine-needle aspiration samples of thyroid nodules as a marker for preoperative diagnosis of thyroid cancer[J]. Applied Immunohistochemistry & Molecular Morphology, 2019, 27(6): 471-476. DOI:10.1097/PAI.0000000000000637 (  0) 0) |
| [14] |
KYOKO O, MIKA K S, YUICHI I, et al. Abstract #4188: Epiglycanin/MUC21 as a potential marker for thyroid carcinomas[J]. Cancer Research, 2009, 69(9): 18-22. (  0) 0) |
| [15] |
JONKLAAS J, MURTHY S, LIU D, et al. Novel biomarker SYT12 may contribute to predicting papillary thyroid cancer outcomes[J]. Future Science OA, 2017, 4(1): FSO249. DOI:10.4155/fsoa-2017-0087 (  0) 0) |
| [16] |
WANG O, ZHENG Z, WANG Q, et al. ZCCHC12, a novel oncogene in papillary thyroid cancer[J]. Journal of Cancer Research and Clinical Oncology, 2017, 143(9): 1679-1686. DOI:10.1007/s00432-017-2414-6 (  0) 0) |
| [17] |
LI Y, ZHOU X, ZHANG Q, et al. Lipase member H is a downstream molecular target of hypoxia inducible factor-1α and promotes papillary thyroid carcinoma cell migration in BCPAP and KTC-1 cell lines[J]. Cancer Management and Research, 2019, 11: 931-941. DOI:10.2147/CMAR.S183355 (  0) 0) |
| [18] |
JARZAB B, WIENCH M, FUJAREWICZ K, et al. Gene expression profile of papillary thyroid cancer: sources of variability and diagnostic implications[J]. Cancer Research, 2005, 65(4): 1587-1597. DOI:10.1158/0008-5472.CAN-04-3078 (  0) 0) |
| [19] |
WRIGHT P K, MAY F E, DARBY S, et al. Estrogen regulates vesicle trafficking gene expression in EFF-3, EFM-19 and MCF-7 breast cancer cells[J]. International Journal of Clinical and Experimental Pathology, 2009, 2(5): 463-475. (  0) 0) |
| [20] |
MEUNIER D, PATRA K, SMITS R, et al. Expression analysis of proline rich 15(Prr15) in mouse and human gastrointestinal tumors[J]. Molecular Carcinogenesis, 2011, 50(1): 8-15. DOI:10.1002/mc.20692 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


