2. 桂林医学院 基础医学院, 桂林541100;
3. 桂林医学院 附属口腔医院, 桂林 541004
2. College of Basic Medicine, Guilin Medical University, Guilin 541100, China;
3. Affiliated Stomatology Hospital of Guilin Medical University, Guilin 541004, China
人类糖脂转运蛋白(Glycolipid transfer protein, GLTP)是一种能调节细胞内膜系统间鞘糖脂膜间运输的可溶性蛋白[1-2]。与其它脂类转运蛋白不同,人类GLTP蛋白的空间结构是由α-螺旋构成的独特的两层“三明治”状,这种“三明治”状结构被认为是真核生物GLTP蛋白家族的结构基础及原型蛋白[3]。GLTP蛋白家族的重要成员包括人类四磷酸适配器蛋白2 (FAPP2)、人类1-磷酸神经酰胺转运蛋白(Ceramide-1-phosphate transfer protein, CPTP, 即GLTPD1)、拟南芥促细胞死亡蛋白11(ACD11)等[1, 3]。
GLTP蛋白过表达能上调细胞周期蛋白依赖性激酶抑制因子1A(p21)及1B(p27),下调细胞周期蛋白依赖性激酶2(CDK2)及4(CDK4)、细胞周期蛋白E及D1,从而阻滞细胞周期于G1/S检查点,诱导细胞程序性坏死从而抑制人类结直肠癌HT-29细胞的生长[2]。尽管GLTP蛋白家族其它成员的结构、功能的研究已有很多报道[1-2,4-7],但是,近年内发现的GLTP蛋白家族的一个新成员人类GLTPD2,其功能尚不清楚。
本研究的目的在于通过在线生物信息学分析工具,对人类GLTPD2蛋白进行鉴定,并对其理化性质、结构域、表达特征、结构比较及潜在功能进行分析,为进一步深入研究人类GLTPD2蛋白的生物学功能及其在疾病发生发展中的作用奠定理论基础和实验依据。
1 材料与方法 1.1 序列的获得利用美国国家生物技术信息中心NCBI (https://www.ncbi.nlm.nih.gov/)检索人类GLTPD2,获得其核酸序列号为NM_001014985.3,蛋白序列号为NP_001014985.3,并作进一步的分析。
1.2 人类GLTPD2的结构及其蛋白的理化性质通过UCSC Genome Browser (http://www.genome.ucsc.edu/)分析人类GLTPD2的结构及其组蛋白修饰水平在不同细胞中的差异,并获取其基因组序列[8],再将人类GLTPD2的基因组序列输入在线分析网站EMBOSS(https://www.ebi.ac.uk/Tools/emboss/)的Cpgplot工具中[9],预测人类GLTPD2的CpG岛所分布的位置,通过UniProt在线服务器(https://www.uniprot.org/)预测人类GLTPD2蛋白的结构域[10]以及利用ExPASy在线服务器(https://web.expasy.org)中的在线工具ProtParam (https://web.expasy.org/protparam/)分析人类GLTPD2蛋白的理化性质[11],通过SignalP-5.0 (http://www.cbs.dtu.dk/services/SignalP/)预测人类GLTPD2蛋白的信号肽[12]以及利用TMHMM Server v. 2.0在线分析工具(http://www.cbs.dtu.dk/services/TMHMM-2.0/)预测人类GLTPD2蛋白的跨膜区[13]。
1.3 蛋白质的翻译后修饰预测通过NetPhos 3.1 Server (http://www.cbs.dtu.dk/services/NetPhos/)分析人类GLTPD2蛋白氨基酸序列上的磷酸化修饰位点[14],并通过NetOGlyc 4.0 Server (http://www.cbs.dtu.dk/services/NetOGlyc/)及NetNGlyc 1.0 Server在线工具(http://www.cbs.dtu.dk/services/NetNGlyc/)分别分析人类GLTPD2蛋白的氨基酸序列上O-连接与N-连接糖基化修饰位点[15-16]。此外,通过JASSA v4 (http://www.jassa.fr/)与GPS-SUMO-2.0 Online Service(http://sumosp.biocuckoo.org/)在线服务器分别对人类GLTPD2蛋白进行苏木化修饰位点的预测[17-18]。
1.4 序列比对及同源建模利用Vector NTI Suite 7.1应用软件包中子程序Align X将人类GLTPD2蛋白(序列号为NP_001014985.3)与人类CPTP蛋白(序列号NP_001025056.1)进行序列比对分析[19]。并将人类GLTPD2蛋白的氨基酸序列输入在线分析工具SWISS-MODEL (https://swissmodel.expasy.org/)中检索得到与人类GLTPD2蛋白同源性最高的三维结构文件(人类CPTP蛋白, 检索号: 4k85.1.A),以此为模板建立人类GLTPD2蛋白GLTP结构域的三维结构[20],并利用PyMol 0.97对模板及人类GLTPD2蛋白的GLTP结构域的空间结构进行比较分析,进一步通过Ras top 2.0软件对人类CPTP及GLTPD2蛋白中的脂转运相关的关键残基进行比较分析。
1.5 基因的表达分析通过在Human Protein Atlas (http://www.proteinatlas.org/)分析人类GLTPD2蛋白在不同细胞、组织中的表达情况以及亚细胞定位[21]。
1.6 蛋白质的相互作用分析及共表达分析利用STRING (https://string-db.org/)在线分析工具分析人类GLTPD2蛋白的潜在相互作用蛋白质[22]。并通过结合STRING及GeneCards (https://www.genecards.org/)数据库对获得的潜在相互作用蛋白的主要功能进行分析[23]。此外,通过Coexpedia(http://www.coexpedia.org/)查询与人类GLTPD2蛋白存在共表达关系的蛋白[24],并利用GeneCards对其功能进行分析。
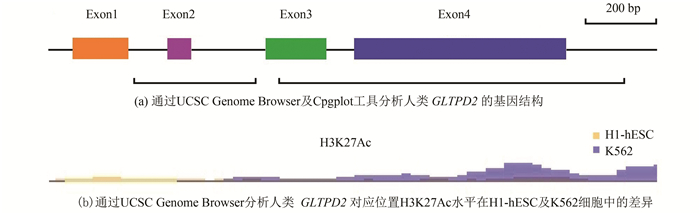
2 结果分析 2.1 人类GLTPD2的基因结构及理化性质通过UCSC Genome Browser及EMBOSS的子程序Cpgplot对人类GLTPD2进行分析,结果表明人类GLTPD2包括四个外显子,编码291个氨基酸。CpG岛分析结果表明人类GLTPD2(含上下游500 bp)含有2个CpG岛,值得注意的是该基因5’端上游无明显CpG岛,但在中下游区间出现了两个明显的CpG岛(见图 1a)。组蛋白修饰分析结果表明人类GLTPD2在K562细胞中H3K27Ac的水平显著高于H1-hESC细胞(见图 1b)。以上结果提示,人类GLTPD2的中下游区间也可能参与了基因的转录调控。此外,利用UniProt在线服务器和ExPASy中的分析工具SignalP-5.0、TMHMM Server v. 2.0及ProtParam对人类GLTPD2蛋白的理化性质及结构特征进行分析,结果表明人类GLTPD2蛋白的GLTP结构域位于102-252氨基酸残基,1-36氨基酸残基为N端信号肽,13-32氨基酸残基为跨膜区(见图 2),分子量大小为31.6 kDa,理论等电点为10.19。
|
图 1 人类GLTPD2结构的特殊性 Figure 1 Particularity of human GLTPD2 structure 注: (a) 黑色框线表示2个CpG岛位置; (b)H3K27Ac为组蛋白3第27位赖氨酸乙酰化, H1-hESC为人类胚胎干细胞H1细胞系, K562为人类红白血病细胞系. |
通过在线服务器NetPhos 3.1 Server、NetOGlyc 4.0 Server及NetNGlyc 1.0 Server分别对人类GLTPD2蛋白的磷酸化修饰位点、O-连接和N-连接糖基化修饰位点进行预测,结果表明在人类GLTPD2蛋白的氨基酸序列中有多个丝氨酸、苏氨酸、酪氨酸残基磷酸化修饰位点(见图 2),O-连接糖基化修饰位点分别为T53、S62、T64、S92、T126、T134残基,第276位氨基酸残基为N-连接糖基化修饰位点(见图 2)。此外,利用JASSA v4及GPS-SUMO-2.0 Online Service在线服务器对人类GLTPD2蛋白进行苏木化修饰位点的预测,结果表明人类GLTPD2蛋白发生苏木化修饰的潜在位点为K94残基(见图 2)。

|
图 2 人类GLTPD2蛋白氨基酸序列及翻译后修饰位点 Figure 2 Amino acid sequence and post-translational sites of human GLTPD2 protein 注: 粗线方框表示预测的人类GLTPD2蛋白的信号肽, 粗横线表示预测的跨膜区, 细线表示预测的结构域, *表示预测的磷酸化修饰位点, #表示预测的O-连接糖基化位点, 加粗加大的字体N为预测的N-连接糖基化修饰位点, 细线方框表示预测的苏木化修饰位点. |
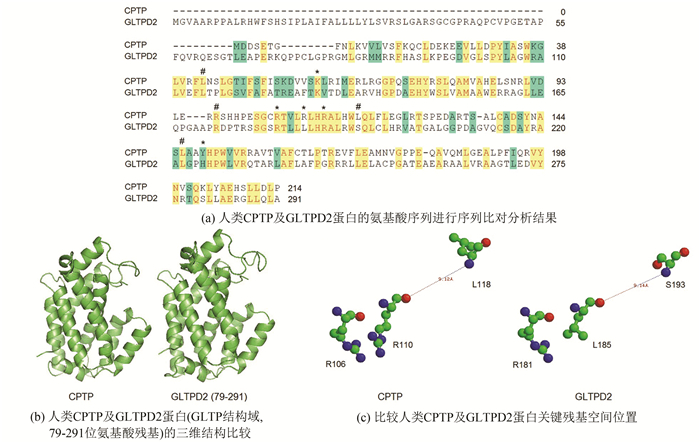
通过Vector NTI Suite 7.1软件中的Align X子程序比对人类CPTP和GLTPD2蛋白的氨基酸序列(见图 3a),结果表明人类CPTP蛋白和GLTPD2蛋白序列间相似性较高,其中人类CPTP蛋白的关键残基包括L43、K60、R97、R106、R110、R113、L118、L146、Y149,在人类GLTPD2蛋白相对应的残基分别为L115、K132、R172、R181、L185、R188、S193、L222、H225。
|
图 3 人类CPTP及GLTPD2蛋白的序列及结构比较 Figure 3 Comparison of sequence and structure of human CPTP and GLTPD2 protein 注: (a)黄色框表示相同的残基, 绿色框表示弱相似的残基, 红字母表示非相似/非保守残基, #表示突变后活性下降的残基, *表示突变后失去活性的关键残基; 蓝色球表示氮原子, (c)绿色球表示氢原子, 红色球表示氧原子. |
通过SWISS-MODEL服务器构建了人类GLTPD2蛋白的GLTP结构域的空间结构(见图 3b),并与人类CPTP蛋白的结构进行比较,分析结果表明人类CPTP蛋白的结构与GLTPD2蛋白的GLTP结构域具有相似的空间结构,均具有脂类结合的疏水口袋。
通过Ras top 2.0软件分析了人类CPTP及GLTPD2蛋白关键残基的空间结构(见图 3c),以人类CPTP蛋白的R106残基与GLTPD2蛋白的R181残基空间位置作为对照,人类CPTP蛋白的R110残基对应于GLTPD2蛋白的L185残基,人类CPTP蛋白的L118残基对应于GLTPD2蛋白的S193残基。这些关键残基尽管不同,但表现出相似的结构。同时,人类CPTP蛋白的R110与L118残基之间的距离和GLTPD2蛋白的L185与S193残基间的距离也非常接近(见图 3c)。
2.4 基因的表达分析通过Human Protein Atlas(https://www.proteinatlas.org)检索人类GLTPD2,综合HPA (Human Protein Atlas)、GTEx(Genotype-Tissue Expression)以及FANTOM 5(Functional Annotation Of the Mammalian Genome 5)三个数据库分析所获得的结果表明人类GLTPD2在组织中广谱表达,其中在肝、肠、肾等组织中的表达相对较高(见图 4a~c)。此外,人类GLTPD2在不同的肿瘤细胞中均有表达,其中在Caco-2 (肠癌细胞)、HepG2 (肝癌细胞)等细胞系中的表达较高(见图 4d)。这些结果提示人类GLTPD2与肝、肠、肾等组织的功能及疾病的发生密切相关。此外,亚细胞定位分析结果表明,人类GLTPD2蛋白主要分布于细胞内的囊泡中,这提示人类GLTPD2蛋白可能是一个分泌蛋白。

|
图 4 人类GLTPD2的表达分析 Figure 4 Expression analysis of human GLTPD2 注: (a~c)将HPA数据库、GETx数据库及FANTOM 5数据库中分析得到的数据分别输入GraphPad Prism 5中绘制人类GLTPD2组织表达图谱(a~c) 将HPA数据库中的数据输入GraphPad Prism 5中绘制人类GLTPD2细胞表达图谱(d) 误差线为标准差. |
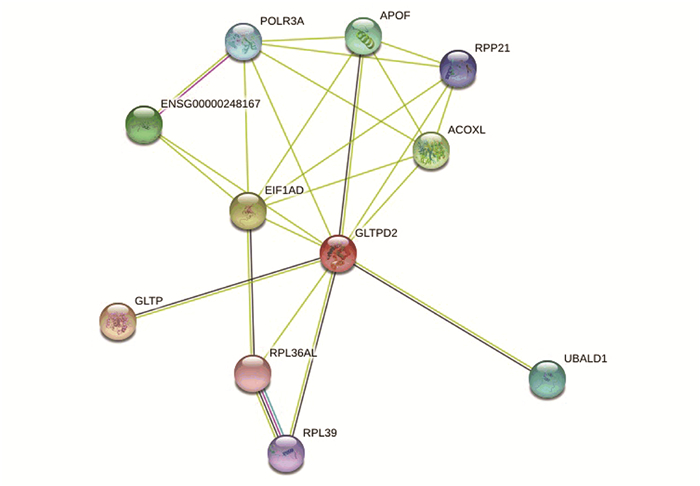
通过STRING数据库对人类GLTPD2蛋白进行蛋白质相互作用网络分析,获得与其有潜在相互作用的蛋白质(见图 5),并通过STRING及GeneCards数据库联合分析这些潜在相互作用蛋白的主要功能。结果表明人类GLTPD2蛋白与APOF、ACOXL、RPP21、EIF1AD等蛋白之间有潜在相互作用。其中载脂蛋白F (Apolipoprotein F, APOF)为胆固醇转运过程中的重要调控因子,能抑制胆固醇酯转运蛋白活性。糖脂转移蛋白(Glycolipid transfer protein, GLTP)可加速各种糖脂和糖鞘脂在膜间转移,但不加速磷脂的转移。酰基辅酶A氧化酶类(Acyl-CoA oxidase like, ACOXL)为脂肪酸结合蛋白,具有酰基辅酶A氧化酶和氧化还原酶的活性。核糖核酸酶P蛋白亚基p21 (Ribonuclease P protein subunit p21, RPP21)为核糖核酸酶P的组成部分,是一个通过切割其5’末端产生成熟tRNA分子并与锌等金属离子结合的蛋白复合物。真核生物翻译结构域1A起始因子(Eukaryotic translation initiation factor 1A domain containing, EIF1AD)可抑制细胞增殖,并参与细胞对氧化应激的反应。RNA聚合酶III亚基A (RNA polymerase III subunit A, POLR3A)是RNA聚合酶III最大的催化核心成分,可合成小RNA,如5S RNA和tRNA,与第二大亚基一起构成聚合酶活性中心。核糖体蛋白L39 (Ribosomal rotein L39, RPL39)为RNA结合蛋白,是核糖体结构组成部分。泛素结构域1结合蛋白(Ubiquitin-associated like domain containing 1, UBALD1)有增加痘苗病毒(Vaccinia virus, VACV)感染几率,减少IL-8分泌等功能。
|
图 5 人类GLTPD2蛋白的潜在相互作用蛋白网络 Figure 5 Potential protein interaction network of human GLTPD2 protein 注: 该图通过STRING在线分析工具获得, 图中不同颜色的圆圈代表不同蛋白质, 直线代表蛋白质之间相互作用. |
通过Coexpedia数据库检索人类GLTPD2蛋白及其共表达蛋白,获得共表达蛋白1个,即酪氨酸激酶-4 (SH3 and PX domain-containing protein 2B,SH3PXD2B)。利用在线工具GeneCards进一步分析其功能,结果表明该基因编码的适配蛋白酪氨酸激酶-4具有一个PX结构域(Phox homology domain)和4个SH3结构域(Src homology 3 domain),参与过氧化物歧化酶的形成和细胞外基质的降解,在脂肪细胞分化的早期阶段中促进有丝分裂的克隆扩增。
蛋白质相互作用及共表达分析的结果表明人类GLTPD2蛋白可能参与了转运脂质、基因的转录、细胞增殖与分化等过程。
3 讨论近年来发现了两个GLTP蛋白家族的新成员,包括人类CPTP (GLTPD1)及GLTPD2。GLTP蛋白家族的重要成员人类CPTP蛋白的功能已有许多研究[1-3,7]。研究表明人类CPTP蛋白的K60、R106、R110残基对于蛋白识别和结合1-磷酸神经酰胺(Ceramide-1-phosphate, C1P)是必须的,R113、Y149残基对于维持CPTP蛋白的空间结构是必要的,当这些关键残基发生突变时,蛋白原有活性丧失[7]。人类CPTP蛋白的L43、L118、L146残基的突变会显著降低C1P的转运活性,而R97残基的主要作用在于吸附脂类分子的头部基团[7]。本研究将人类CPTP蛋白的空间结构与人类GLTPD2蛋白的GLTP结构域空间结构及脂转运相关的关键残基进行了比较,结果表明无论是空间结构上还是在关键残基上,二者均具有较大的相似性(见图 3a)。此外,人类CPTP蛋白的R110残基对应于人类GLTPD2蛋白的L185残基,人类CPTP蛋白的L118残基对应于人类GLTPD2蛋白的S193残基(见图 3a)。这些关键残基尽管不同,但表现出相似的结构(见图 3c)。这些结果表明人类GLTPD2蛋白可能也具有转运脂类(尽管目前尚不能确定其转运脂类种类)的功能。
通过基因的细胞及组织表达谱分析,结果表明人类GLTPD2在肝、肠、肾等组织表达较高,在Caco-2 (肠癌细胞)、HepG2 (肝癌细胞)等细胞系中的表达也较高。这提示人类GLTPD2很可能与肝、肠、肾等组织的功能及疾病的发生密切相关。通过蛋白相互作用分析及共表达分析也表明人类GLTPD2蛋白可能参与转运脂质、基因的转录、细胞增殖与分化等生物学过程。
经研究初步鉴定了人类GLTP蛋白家族的一个新成员,GLTPD2,通过生物信息学分析工具分析了该蛋白的理化性质、结构域、结构比较、表达特征及潜在功能,迄今国内外尚无相关研究的文献报道。本研究为进一步深入研究人类GLTPD2蛋白的生物学功能及其在疾病发生发展中的作用提供了重要的理论基础和实验依据。
| [1] |
MALININA L, SIMANSHU D K, ZHAI X, et al. Sphingolipid transfer proteins defined by the GLTP-fold[J]. Quarterly Reviews of Biophysics, 2015, 48(3): 281-322. DOI:10.1017/S003358351400016X (  0) 0) |
| [2] |
MISHRA S K, STEPHENSON D J, CHALFANT C E, et al. Upregulation of human glycolipid transfer protein (GLTP) induces necroptosis in colon carcinoma cells[J]. Biochimica et Biophysica Acta-Molecular and Cell Biology of Lipids, 2019, 1864(2): 158-167. DOI:10.1016/j.bbalip.2018.11.002 (  0) 0) |
| [3] |
MALININA L, PATEL D J, BROWN R E. How α-helical motifs form functionally diverse lipid-binding compartments[J]. Annual Review of Biochemistry, 2017, 86: 609-636. DOI:10.1146/annurev-biochem-061516-044445 (  0) 0) |
| [4] |
KENOTH R, ZOU X, SIMANSHU D K, et al. Functional evaluation of tryptophans in glycolipid binding and membrane interaction by HET-C2, a fungal glycolipid transfer protein[J]. Biochimca et Biophysica Acta Biomembranes, 2018, 1860(5): 1069-1076. DOI:10.1016/j.bbamem.2018.01001 (  0) 0) |
| [5] |
MISHRA S K, GAO Y G, DENG Y, et al. CPTP: A sphingolipid transfer protein that regulates autophagy and inflammasome activation[J]. Autophagy, 2018, 14(5): 862-879. DOI:10.1080/15548627.2017.1393129 (  0) 0) |
| [6] |
OCHOA-LIZARRALDE B, GAO Y G, POPOV A N, et al. Structural analyses of 4-phosphate adaptor protein 2 yield mechanistic insights into sphingolipid recognition by the glycolipid transfer protein family[J]. Journal of Biological Chemistry, 2018, 293(43): 16709-16723. DOI:10.1074/jbc.RA117.000733 (  0) 0) |
| [7] |
SIMANSHU D K, KAMLEKAR R K, WIJESINGHE D S, et al. Non-vesicular trafficking by a ceramide-1-phosphate transfer protein regulates eicosanoids[J]. Nature, 2013, 500(7463): 463-467. DOI:10.1038/nature12332 (  0) 0) |
| [8] |
KAROLCHIK D, BAERTSCH R, DIEKHANS M, et al. The UCSC Genome Browser Database[J]. Nucleic Acids Research, 2003, 31(1): 51-54. DOI:10.1093/nar/gkg129 (  0) 0) |
| [9] |
COOK C E, BERGMAN M T, COCHRANE G, et al. The European Bioinformatics Institute in 2017: Data coordination and integration[J]. Nucleic Acids Research, 2018, 46(D1): D21-D29. DOI:10.1093/nar/gkx1154 (  0) 0) |
| [10] |
UniProt Consortium. UniProt: A worldwide hub of protein knowledge[J]. Nucleic Acids Research, 2019, 47(D1): D506-D515. DOI:10.1093/nar/gky1049 (  0) 0) |
| [11] |
ARTIMO P, JONNALAGEDDA M, ARNOLD K, et al. ExPASy: SIB bioinformatics resource portal[J]. Nucleic Acids Research, 2012, 40: W597-W603. DOI:10.1093/nar/gks400 (  0) 0) |
| [12] |
ALMAGRO ARMENTEROS J J, TSIRIGOS K D, SØNDERBY C K, et al. SignalP-5.0 improves signal peptide predictions using deep neural networks[J]. Nature Biotechnology, 2019, 37: 420-423. DOI:10.1038/s41587-019-0036-z (  0) 0) |
| [13] |
KROGH A, LARSSON B, VON HEIJNE G, et al. Predicting transmembrane protein topology with a hidden markov model: application to complete genomes[J]. Journal of Molecular Biology, 2001, 305(3): 567-580. DOI:10.1006/jmbi.2000.4315 (  0) 0) |
| [14] |
BLOM N, GAMMELTOFT S, BRUNAK S. Sequence and structure-based prediction of eukaryotic protein phosphorylation sites[J]. Journal of Molecular Biology, 1999, 294(5): 1351-1362. DOI:10.1006/jmbi.1999.3310 (  0) 0) |
| [15] |
DAI M M, YU C M, FANG T, et al. Identification and functional characterization of glycosylation of recombinant human platelet-derived growth factor-BB in pichia pastoris[J]. PloS One, 2015, 10(12): e0145419. DOI:10.1371/journal.pone.0145419 (  0) 0) |
| [16] |
STEENTOFT C, VAKHRUSHEV S Y, JOSHI H J, et al. Precision mapping of the human O-GalNAc glycoproteome through SimpleCell technology[J]. EMBO Journal, 2013, 32(10): 1478-1488. DOI:10.1038/emboj.2013.79 (  0) 0) |
| [17] |
ZHAO Q, XIE Y, ZHENG Y Y, et al. GPS-SUMO: A tool for the prediction of sumoylation sites and SUMO-interaction motifs[J]. Nucleic Acids Research, 2014, 42(W1): W325-W330. DOI:10.1093/nar/gku383 (  0) 0) |
| [18] |
BEAUCLAIR G, BRIDIER-NAHMIAS A, ZAGURY J F, et al. JASSA: A comprehensive tool for prediction of SUMOylation sites and SIMs[J]. Bioinformatics, 2015, 31(21): 3483-3491. DOI:10.1093/bioinformatics/btv403 (  0) 0) |
| [19] |
LU Guoqing, MORIYAMA E N. Vector NTI, a balanced all-in-one sequence analysis suite[J]. Briefings in Bioinformatics, 2004, 5(4): 378-388. DOI:10.1093/bib/5.4.378 (  0) 0) |
| [20] |
WATERHOUSE A, BERTONI M, BIENERT S, et al. SWISS-MODEL: Homology modelling of protein structures and complexes[J]. Nucleic Acids Research, 2018, 46(W1): W296-W303. DOI:10.1093/nar/gky427 (  0) 0) |
| [21] |
UHLÉN M, FAGERBERG L, HALLSTRÖM B M, et al. Tissue-based map of the human proteome[J]. Science, 2015, 347(6220): 1260419. DOI:10.1126/science.1260419 (  0) 0) |
| [22] |
SZKLARCZYK D, GABLE A L, LYON D, et al. STRING v11: Protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets[J]. Nucleic Acids Research, 2019, 47(D1): D607-D613. DOI:10.1093/nar/gky1131 (  0) 0) |
| [23] |
STELZER G, ROSEN N, PLASCHKES I, et al. The genecards suite: from gene data mining to disease genome sequence analysis[J]. Current Bioinformatics, 2016, 54(1): 1.30.1-1.30.33. DOI:10.1002/cpbi.5 (  0) 0) |
| [24] |
YANG S, KIM C Y, HWANG S, et al. COEXPEDIA: Exploring biomedical hypotheses via co-expressions associated with medical subject headings (MeSH)[J]. Nucleic Acids Research, 2017, 45(D1): D389-D396. DOI:10.1093/nar/gkw868 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


