2. 山西医科大学 药学院,太原 030000;
3. 山西医科大学 第一临床医学院,太原 030000
2. College of Pharmacy, Shanxi Medical University, Taiyuan 030000, China;
3. The First Clinical College, Shanxi Medical University, Taiyuan 030000, China
肝细胞癌(Hepatocellular carcinoma, HCC)是原发于肝脏的上皮性恶性肿瘤,约占原发性肝癌的90%以上[1]。2018年我国HCC患者新增39.3万例,死亡36.9万例,是中国癌症死亡的第二大原因[2-3]。由于HCC发病较为隐匿,大多数患者就诊时已是晚期,对其生存预后的评估主要依靠临床有创性检查,即TNM分期系统。其主要缺点为T期根据原发肿瘤的大小来划分,未能反映原发灶的厚度对淋巴结转移的影响,并且由于HCC高度的异质性,分期系统的预测准确性降低,不足以精确评估HCC患者的预后情况,急需新的预后标志物指导HCC患者的个体化治疗。自噬是依赖溶酶体系统对损伤或无用的细胞器与蛋白质等进行降解的一种途径,产生的能量可供应细胞存活和细胞器的更新[4]。近年来自噬与肿瘤之间的关系受到广泛关注,一方面自噬在血管生成较少的肿瘤中心部位发挥促进肿瘤形成的作用[5];另一方面自噬通过减少受损或无用的细胞器与蛋白质的积累,限制氧化应激等过程,从而抑制癌症的发生[6]。自噬与HCC的研究表明,自噬广泛参与该肿瘤的发生与侵袭性生长[7-9],其可通过激活Wnt /β-catenin信号传导来诱导MCT1表达,从而促进HCC细胞的转移和糖酵解;不同的肿瘤微环境中,通过肿瘤和免疫成分的协同作用可调节癌细胞自噬,从而诱导炎性单核细胞促进HCC的发生。
由于自噬与肿瘤的相互作用受到许多自噬相关基因(ATGs)的调控,瘤组织中ATGs的表达情况可以用来评估患者的生存预后,相关研究已在非小细胞肺癌、多形性胶质母细胞瘤以及肾透明细胞癌等报道[10-12]。因此,构建包含有多个ATGs的风险评分模型来预测HCC患者的生存预后,既可填补该方法在HCC中的研究空白,又能实现对HCC患者更精准的预后评估,为其个性化治疗提供重要参考。本研究联合TCGA数据库与人类自噬基因数据库,通过构建自噬相关基因的预后模型预测HCC患者的生存预后情况,为相关临床治疗提供诊疗依据与治疗靶点。
1 材料与方法 1.1 数据资源首先绘制本研究设计流程图,之后利用癌症基因组图谱数据库(The Cancer Genome Atlas, TCGA, https://portal.gdc.cancer.gov/)下载肝细胞癌(Hepatocellular carcinoma, HCC)的3级RNA测序信息和相应的临床数据,其中包括374例HCC患者的肿瘤组织测序信息和50例正常肝组织测序信息。利用perl语言(perl 5.30.2)进行数据提取,之后将表达矩阵文件中的“Ensembl_Stable_ID”转换为“Gene Symbol”,从人类自噬基因数据库(Human Autophagy Database, HADb, http://www.autophagy.lu/)下载232个与自噬有关的基因信息。将表达矩阵中的基因与232个自噬相关基因取交集,重新整理为自噬相关基因的表达矩阵,并绘制Venn图。由于本研究所用数据来源于TCGA数据库,故不需要相关伦理学审核与批准。
1.2 自噬相关基因差异分析使用R语言(R3.6.3)中的limma包,筛选差异表达的基因,筛选标准为:错误发现率(False discovery rate, FDR)BH法矫正后的阈值P.adj<0.01,对数差异表达倍数变化绝对值|log2FC|>1。使用pheatmap包绘制差异基因的火山图,使用ggpubr包绘制差异基因箱线图。
1.3 GO富集分析和KEGG通路分析利用R语言对差异表达的自噬相关基因进行基因本体功能(Gene Ontology, GO)富集分析和京都基因与基因组百科全书通路(Kyoto Encyclopedia of Genes and Genomes pathway,KEGG pathway)分析。首先下载clusterProfiler、enrichplot和ggpggplot2三个数据包,阈值设置为P<0.01、Q<0.01,之后与基因组背景对比。通过GO富集分析,得到显著富集的GO功能词条(Term),其中生物过程(Biological process,BP)、细胞成分(Cellular component,CC)和分子功能(Molecular function,MF)为主要的分析对象。通过KEGG通路分析,得到显著性富集的pathway。
1.4 预后风险评分模型构建利用perl语言将差异表达的自噬相关基因表达量与生存时间进行合并,剔除生存信息不完整的样本。利用R语言中的survival包对差异基因进行单变量Cox回归分析,设置阈值为P<0.001,明确基因表达水平与患者生存时间的关系,并绘制森林图。之后挑选P<0.001的基因,利用survival包进行多因素Cox回归分析,得到风险基因,构建可以预测生存时间的风险评分模型,并输出纳入模型基因的风险评分系数(Coef)。所构建的预后风险评分模型计算公式为:风险值(risk score)=风险基因表达量1×coef1+风险基因表达量2×coef2+...+风险基因表达量n×coefn。
1.5 预后风险评分模型评价根据风险评分模型计算患者的风险值,从低到高排序后依据中位数将患者分为低风险组和高风险组。利用survival包和survminer包进行Kaplan-Meier (K-M) 生存分析,明确风险值和生存时间之间的关系,并绘制生存曲线。下载pheatmap包绘制高低风险组的风险曲线、生存状态图和风险基因热图。将各组患者的生存时间、生存状态、年龄、性别、WHO分级、分期、TNM分期及风险评分整理为数值数据,其中:生存状态(0,存活;1,死亡)、性别(0,女性;1,男性),剔除临床数据不完整的样本,利用survival包进行单因素与多因素的独立预后分析,并绘制森林图。利用survival ROC包绘制各项独立预后指标的ROC曲线,从而评估其预测的准确性。将各项临床数据整理为二分类变量,整理标准为:生存时间(<中位生存期;>中位生存期;单位:天)、生存状态(0,存活;1,死亡)、年龄(<=65岁;>65岁)、性别(男性;女性)、WHO分级(G1-2;G3-4)、分期(StageⅠ-Ⅱ;StageⅢ-Ⅳ)、T(T1-2;T3-4)、M(M0;M1)、N(N0、N1-3)。利用beeswarm包进行风险基因表达量与风险值同临床数据之间的相关性分析,以评估所筛选基因及构建模型是否同临床数据具有良好的相关关系。
1.6 风险基因外部数据验证利用GEPIA2.0网页工具中有关HCC的数据对模型中的风险基因进行验证,输出表达量统计图和生存曲线图,利用人类蛋白图谱(The Human Protein Atlas, HPA, https://www.proteinatlas.org/)中的临床样本来验证风险基因在正常与肿瘤组织中的的表达情况。
2 结果分析 2.1 自噬相关基因差异分析利用TCGA数据库下载到374例患者和50例正常肝组织样本共计55 268个基因的表达数据,与自噬基因取交集绘制Venn图(见图 1a),差异分析后得到61个基因(表达上调57个,下调4个),差异基因火山图(见图 1b),肿瘤和非肿瘤组织中差异基因的相对表达量整理为箱线图(见图 1c)。
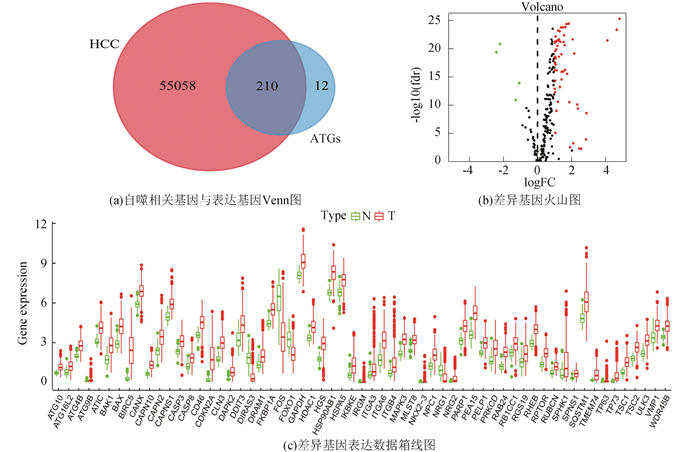
|
图 1 自噬相关基因差异分析 Figure 1 Differential analysis of autophagy-associated genes |
将61个自噬相关差异基因进行GO富集分析,共富集到225个GO terms,包括192个生物过程(BP),15个细胞成分(CC)和18个分子功能(MF)。按照p值由小到大分别筛选各组前10个GO terms。在生物过程方面,差异基因主要富集在自噬、自噬机制过程和巨噬细胞吞噬的调控;在细胞成分方面,主要富集在泡膜、自噬体和溶酶体膜;在分子功能方面,富集在蛋白激酶调节剂活性、激酶调节剂活性和泛素样蛋白连接酶结合等。差异基因的KEGG通路富集分析共得到46条信号通路,按照p值由小到大排序筛选前10个通路,结果主要富集在自噬和细胞凋亡通路上。具体富集分析结果(见表 1)。
| 表 1 GO富集与KEGG通路分析 Table 1 GO enrichment and KEGG pathway analyses |
剔除临床信息不完整的样本,共有370例HCC患者的表达数据被纳入。61个自噬相关差异基因的单因素Cox回归分析提示,共有12个基因和患者的生存预后明显相关,整理为森林图(见图 2)。多因素Cox回归分析提示,有4个基因和患者的生存预后明显相关,可作为风险基因用来构建预测模型(见表 2),分别是SQSTM1(HR 1.203,P=0.030)、HDAC1(HR 1.465,P=0.019)、RHEB(HR 1.526,P=0.026)和ATIC(HR 1.550,P=0.006)。所建立的预后风险评分模型公式为:风险值(risk score)=SQSTM1表达量×0.185+HDAC1表达量×0.382+RHEB表达量×0.423+ATIC表达量×0.438。
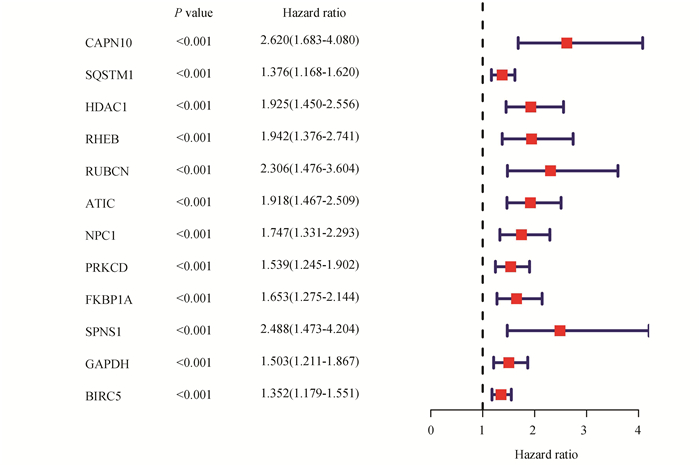
|
图 2 单因素Cox分析森林图 Figure 2 Forest map for univariate Cox analysis |
| 表 2 多因素Cox分析 Table 2 Multivariate Cox analysis |
利用风险评分模型计算公式计算370例患者的风险值,共有185例患者属于低风险,185例患者为高风险。Kaplan-Meier (K-M) 生存分析(见图 3a)显示,高风险患者的生存率明显低于低风险患者(P=2.967×10-4)。高风险患者1年生存率为72.0%,低风险为92.0%;3年生存率前者为50.0%,后者为71.0%。风险曲线(见图 3b)表明从低到高排序后,患者的风险值逐渐增大;生存状态图(见图 3c)表明高风险组死亡个体数多于低风险组;风险基因热图显示(见图 3d)高风险组中SQSTM1、HDAC1、RHEB和ATIC的表达量均高于低风险组,证明4个基因均为高风险基因。单因素独立预后分析(见图 3e)与多因素独立预后分析(见图 3f)联合说明,仅有风险值可作为患者生存预后的危险因子,随着风险值增高,患者的生存率逐渐降低。独立预后分析相关的ROC曲线(见图 3g)显示,风险值的AUC值为0.751,具有一定预测能力。风险基因表达量与风险值同临床数据之间的相关性分析表明,HDAC1(见图 4a)、ATIC(见图 4b)与风险值(见图 4c)同生存时间呈负相关关系;SQSTM1(见图 4d)、HDAC1(见图 4e)、RHEB(见图 4f)与ATIC(见图 4g)的表达及风险值(见图 4h)在死亡个体中均高于存活个体。因此,4个风险基因及风险值同患者的生存数据具有良好的相关关系,所构建模型可用来评估患者的生存预后情况。
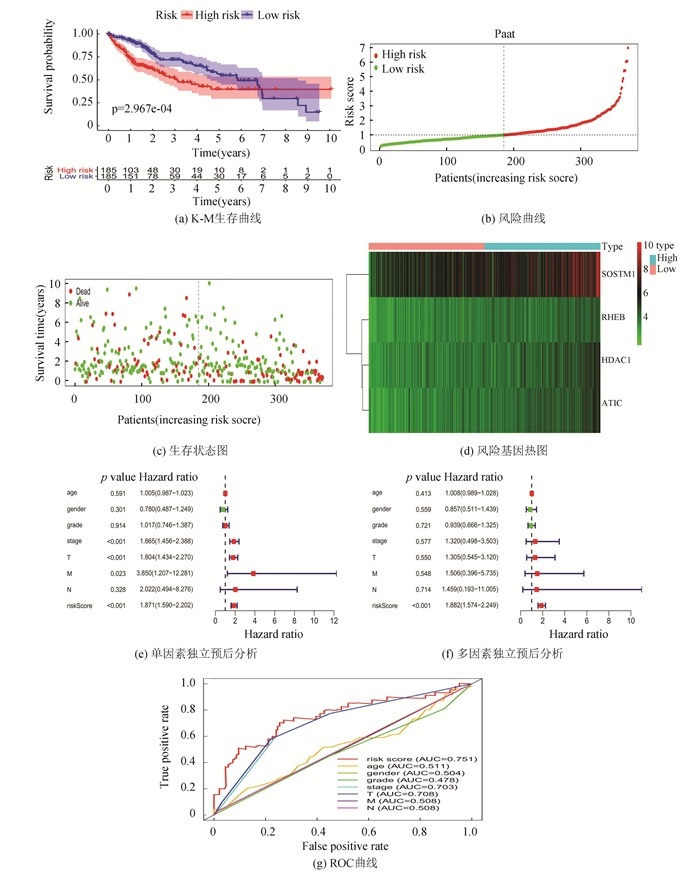
|
图 3 预后风险评分模型评价 Figure 3 Evaluation of prognostic risk score model |
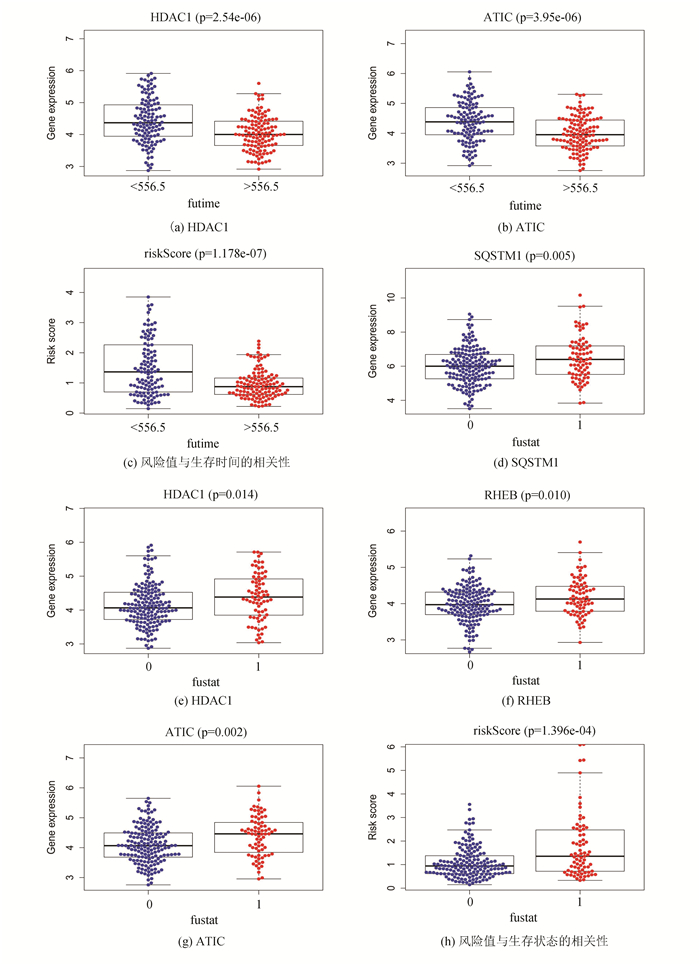
|
图 4 临床相关性分析 Figure 4 Clinical correlation analysis |
GEPIA2.0网页工具中共有369例HCC患者数据和160例正常组织样本,分别输出SQSTM1(见图 5a)、HDAC1(见图 5b)、RHEB(见图 5c)和ATIC(图见5d)的表达量统计图(红色代表肿瘤组织,黑色代表正常组织),可见4个基因在肿瘤中表达量均高于正常组织样本。输出SQSTM1(见图 5e)、HDAC1(见图 5f)、RHEB(见图 5g)和ATIC(见图 5h)的生存曲线图,发现高表达4个风险基因组的生存率均低于低表达组。利用HPA数据库中的临床样本病理学与组织学免疫组化染色图发现,SQSTM1(见图 6a)、HDAC1(见图 6b)、RHEB(见图 6c)和ATIC(见图 6d)在肿瘤中的表达量均高于正常组织,这与GEPIA2.0中得到的统计图结果一致。
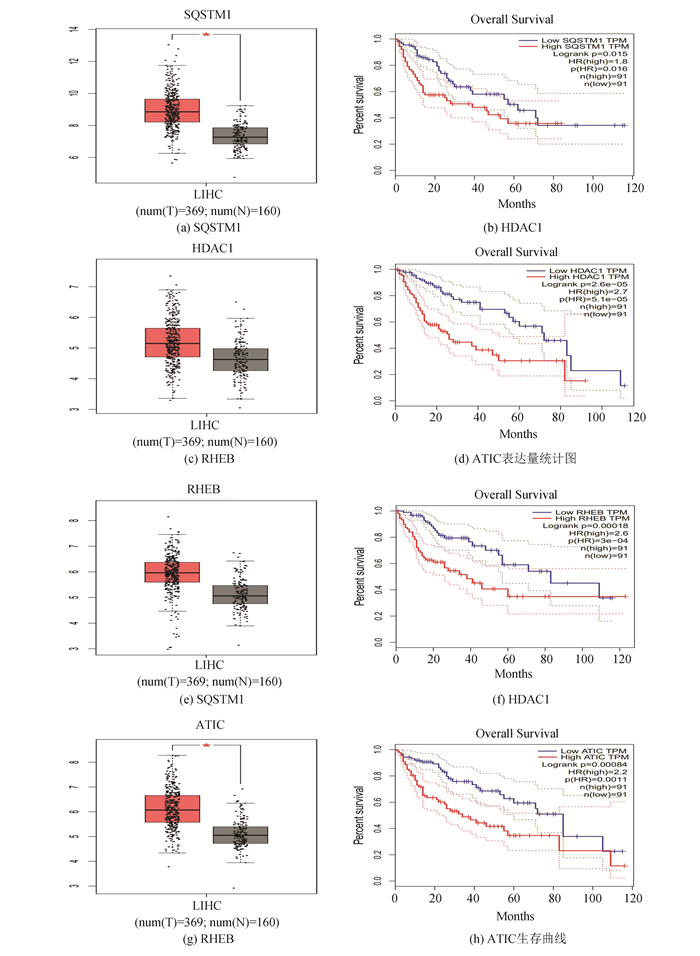
|
图 5 GEPIA网页工具 Figure 5 GEPIA web tool |
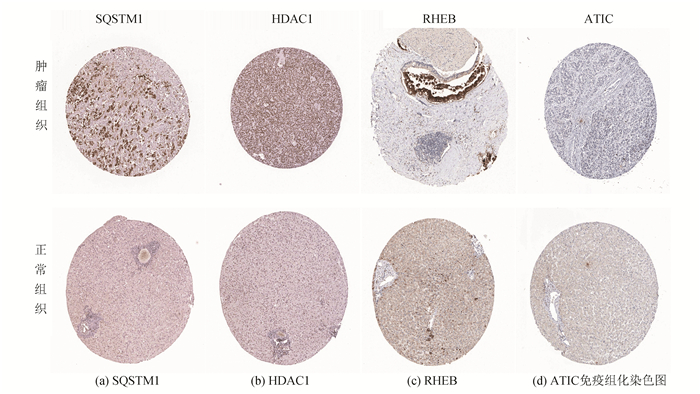
|
图 6 HPA数据库 Figure 6 HPA database |
自噬在肝细胞癌的发生发展中发挥重要作用,但其调控网络较为复杂,不同的自噬相关基因发挥促癌或抑癌作用。通过单因素与多因素Cox回归分析,将纳入预后模型的4个基因赋予不同的风险评分系数,以生存时间为判断标准,明确其对肿瘤起促进作用或抑制作用,这也为后续治疗靶点的选择提供了依据。
已知P62(即SQSTM1基因)是一个经典的自噬标记基因,在溶酶体降解途径中P62作为受体与泛素化蛋白结合并将其运送到自噬体参与选择性自噬的过程,细胞内自噬受损、P62蛋白累积使染色体突变从而促进细胞癌变、肿瘤形成。研究表明,P62和Caspase高表达的卵巢癌组织存活时间较低表达组织更长,P62和泛素化蛋白积累激活Caspase8,降低卵巢癌细胞对化疗的敏感性,促进卵巢癌的进展[13];在乳腺癌组织中,MYC的表达水平与P62增强乳腺癌细胞自我更新能力相关,高表达的P62通过抑制let-7a和let-7b的表达延缓MYC mRNA的降解,P62与MYC协同作用使乳腺癌恶化进展加速[14];激活Wnt/β-catenin通路诱导下游因子过量表达会促进黑色素瘤细胞侵袭转移,干扰P62表达后发现该通路及其下游因子的表达均受到抑制,表明P62可能通过调控该通路来促进癌细胞转移[15]。
HDAC1(即组蛋白去乙酰化酶)与HAT(组蛋白乙酰化酶)共同调节组蛋白的乙酰化水平,在真核细胞中组蛋白乙酰化水平降低会抑制部分抑癌基因的转录从而促进癌症发生。研究发现,HDAC1基因在多种肿瘤组织中高表达,且其高表达刺激癌细胞增殖;大肠癌细胞UVRAG突变可增强化疗敏感性,HDAC1基因上调抑制UVRAG表达,促进大肠癌的发展[16];抑制卵巢癌组织HDAC1基因表达将减弱卵巢癌细胞的增殖能力、逆转癌组织顺铂耐药性[17];HDAC1在乳腺癌组织的表达水平显著高于癌旁组织,其表达水平随淋巴结转移数、临床分期的增加而升高[18]。
细胞自噬受到RHEB基因(即脑Ras同源蛋白基因)的高度调控,该基因主要通过直接调节AMP活化蛋白激酶与控制mTOR信号通路进而调节其他ATGs两条途径来实现[19];mTOR信号通路与细胞生长、增殖与分化有关,RHEB基因是激活mTOR信号通路的关键基因。肝癌组织中RHEB表达水平显著高于正常组织,其表达水平随肝癌组织的临床分期而增加,敲除该基因发现HCC细胞生长速度和增殖能力明显降低[20],表明RHEB基因可能促进癌细胞生长增殖。
ATIC(氨基咪唑甲酰转移酶)是一种双功能蛋白酶,在生物合成过程中催化嘌呤合成的最后两步。肝癌组织中异常高表达的ATIC通过抑制AMPK的激活,活化mTOR-s6K1-S6信号,促进癌细胞的生长、增殖和迁移[21];研究表明,ATIC是一个有效的化疗放射增敏靶点,通过基因敲除或化学抑制可以使癌细胞转化到对辐射更敏感的细胞周期——G2/M期[22],从而提高治疗的有效性。
综上所述,本研究所筛选到的4个风险基因高表达与患者的不良预后显著相关,据此构建的预后风险评分模型有助于对HCC患者的预后情况进行评价,高风险组患者应接受更积极有效的治疗。本研究的特色之处在于,根据自噬与肿瘤发生发展具有密切联系这一前提出发,筛选到与患者生存预后密切相关的自噬基因,并成功构建预后模型,为HCC患者的精准预后评估提供新方法;本研究筛选到的风险基因,可作为HCC基础研究与治疗的可靠靶点;将风险基因的表达与生存情况在GEPIA2.0网页工具与HPA数据库中进行多维度的验证,具有一定新颖性。本研究的不足之处在于,没有在其他数据集中对所构建模型进行有效验证,从而提高模型的可靠性;没有针对风险基因在动物或细胞水平进行功能验证;所构建的预后模型仍需大规模多中心的临床试验进行检验;这些将在后续的研究中进一步探讨。
4 结论1) 本研究联合TCGA数据库与人类自噬数据库得到61个肿瘤组织中相较正常组织差异表达的自噬相关基因(DE-ATGs),对其进行GO富集和KEEG通路分析发现,DE-ATGs主要富集在自噬、利用自噬机制过程、细胞凋亡等过程中。
2) 单因素和多因素Cox回归分析筛选到4个与患者生存预后显著相关的基因,分别是SQSTM1、HDAC1、RHEB和ATIC,所构建预后风险评分模型为:风险值(Risk score)=SQSTM1表达量×0.185+HDAC1表达量×0.382+RHEB表达量×0.423+ATIC表达量×0.438。
3) 根据模型将HCC患者分为高风险组和低风险组,生存分析与临床相关性分析显示,4个风险基因与HCC患者的生存预后密切相关。
| [1] |
SHEN M Y, LIN L. Functional variants of autophagy-related genes are associated with the development of hepatocellular carcinoma[J]. Life Science, 2019, 235: 116675. DOI:10.1016/j.lfs.2019.116675 (  0) 0) |
| [2] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: A Cancer Journal for Clinicians, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 (  0) 0) |
| [3] |
DANIELA S, AUGUSTO V, SCOTT L F, et al. Liver cancer cell of origin, molecular class, and effects on patient prognosis[J]. Gastroenterology, 2017, 152(4): 745-761. DOI:10.1053/j.gastro.2016.11.048 (  0) 0) |
| [4] |
尹丽, 徐存拴. 自噬在大鼠肝再生中作用的初步探讨[J]. 生物信息学, 2017, 15(3): 156-163. YIN Li, XU Cunshuan. Preliminary research on the role of autophagy in rat liver regeneration[J]. Chinese Journal of Bioinformatics, 2017, 15(3): 156-163. DOI:10.3969/j.issn.1672-5565.20161222001 (  0) 0) |
| [5] |
MAES H, KUCHNIO A, PERIC A, et al. Tumor vessel normalization by chloroquine independent of autophagy[J]. Cancer Cell, 2014, 26(2): 190-206. DOI:10.1016/j.ccr.2014.06.025 (  0) 0) |
| [6] |
GUO J Y, WHITE E. Autophagy, metabolism, and cancer[J]. Cold Spring Harbor Symposia on Quantitative Biology, 2016, 81: 73-78. DOI:10.1101/sqb.2016.81.030981 (  0) 0) |
| [7] |
FAN Q, YANG L, ZHANG X D, et al. Autophagy promotes metastasis and glycolysis by upregulating MCT1 expression and Wnt/β-catenin signaling pathway activation in hepatocellular carcinoma cells[J]. Journal of Experimental & Clinical Cancer Research, 2018, 37(1): 9. DOI:10.1186/s13046-018-0673-y (  0) 0) |
| [8] |
CHEN D P, NING W R, LI X F, et al. Peritumoral monocytes induce cancer cell autophagy to facilitate the progression of human hepatocellular carcinoma[J]. Autophagy, 2018, 14(8): 1335-1346. DOI:10.1080/15548627.2018.1474994 (  0) 0) |
| [9] |
ZHENG W, XIE W W, YIN D Y, et al. ATG5 and ATG7 induced autophagy interplays with UPR via PERK signaling[J]. Cell Communication and Signaling, 2019, 17(1): 42. DOI:10.1186/s12964-019-0353-3 (  0) 0) |
| [10] |
LIU Y, WU L, AO H, et al. Prognostic implications of autophagy-associated gene signatures in non-small cell lung cancer[J]. Aging (Albany NY), 2019, 11(23): 11440-11462. DOI:10.18632/aging.102544 (  0) 0) |
| [11] |
WANG Z, GAO L, GUO X, et al. Development and validation of a nomogram with an autophagy-related gene signature for predicting survival in patients with glioblastoma[J]. Aging (Albany NY), 2019, 11(24): 12246-12269. DOI:10.18632/aging.102566 (  0) 0) |
| [12] |
CHEN Mei, ZHANG Shufang, NIE Zhenyu, et al. Identification of an autophagy-related prognostic signature for clear cell renal cell carcinoma[J]. Frontiers in Oncology, 2020, 29(10): 1-15. DOI:10.3389/fonc.2020.00873 (  0) 0) |
| [13] |
YAN Xiaoyu, ZHONG Xinru, YU Sihang, et al. p62 aggregates mediated caspase 8 activation is responsible for progression of ovarian cancer[J]. Journal of Cellular and Molecular Medicine, 2019, 23(6): 1-13. DOI:10.1111/jcmm.14288 (  0) 0) |
| [14] |
XU L Z, LI S S, ZHOU W, et al. p62/SQSTM1 enhances breast cancer stem-like properties by stabilizing MYC mRNA[J]. Oncogene, 2017, 36(3): 304-317. DOI:10.1038/onc.2016.202 (  0) 0) |
| [15] |
易娟娟, 招玉玲, 谢蒲辉, 等. P62基因对恶性黑色素瘤细胞侵袭的影响及机制研究[J]. 中国现代医学杂志, 2020, 30(10): 14-17. YI Juanjuan, ZHAO Yuling, XIE Puhui, et al. The effect and mechanism of p62 gene on invasion of malignant melanoma cells[J]. Chinese Journal of Modern Medicine, 2020, 30(10): 14-17. DOI:10.3969/j.issn.1005-8982.2020.10.003 (  0) 0) |
| [16] |
JO Y K, PARK N Y, SHIN J H, et al. Up-regulation of UVRAG by HDAC1 inhibition attenuates 5FU-induced cell death in HCT116 colorectal cancer cells[J]. Anticancer Research, 2018, 38(1): 271-277. DOI:10.21873/anticanres.12218 (  0) 0) |
| [17] |
吕腾. MiR-34a通过调控HDAC1抑制卵巢癌细胞增殖和顺铂耐药性机制的实验研究[D]. 济南: 山东大学, 2019. DOI: 10.27272/d.cnki.gshdu.2019.000697. LV Teng. The mechanism of miR-34a inhibiting ovarian cancer cell proliferation and cisplatin resistance by regulating HDAC1[D]. Jinan: Shandong University, 2019. DOI: 10.27272/d.cnki.gshdu.2019.000697. (  0) 0) |
| [18] |
刘鹏勇. HDAC1与E-cadherin在乳腺癌中的表达及其相关性的研究[D]. 佳木斯: 佳木斯大学, 2019. DOI: 10.27168/d.cnki.gjmsu.2019.000037. LIU Pengyong. Expression and correlation of HDAC1 and E-cadherin in breast cancer[D]. Jamusi: Jamusi University, 2019. DOI: 10.27168/d.cnki.gjmsu.2019.000037. (  0) 0) |
| [19] |
陈旭旭, 李丹妮, 陈鹏宇, 等. 脑Ras同源蛋白(Rheb)基因研究概况[J]. 绍兴文理学院学报(自然科学), 2020, 40(1): 53-58. CHEN Xuxu, LI Danni, CHEN Pengyu, et al. An ovweview of the research on Ras homolog enriched in brain (Rheb) gene[J]. Journal of Shaoxing University of Arts and Science (Natural Science), 2020, 40(1): 53-58. DOI:10.16169/j.issn.1008-293x.k.2020.02.009 (  0) 0) |
| [20] |
LIU Fuchen, PAN Zeya, ZHANG Jinmin, et al. Overexpression of RHEB is associated with metastasis and poor prognosis in hepatocellular carcinoma[J]. Oncology Letters, 2018, 15(3): 3838-3845. DOI:10.3892/ol.2018.7759 (  0) 0) |
| [21] |
LI Minjing, JIN Changzhu, XU Maolei, et al. Bifunctional enzyme ATIC promotes propagation of hepatocellular carcinoma by regulating AMPK-mTOR-S6 K1 signaling[J]. Cell Communication and Signaling, 2017, 15(1): 52. DOI:10.1186/s12964-017-0208-8 (  0) 0) |
| [22] |
LIU Xiangfei, PAILA U D, TERAOKA S N, et al. Identification of ATIC as a novel target for chemoradiosensitization[J]. International Journal of Radiation Oncology Biology Physics, 2018, 100(1): 162-173. DOI:10.1016/I,ijrobp.2017.08.033 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


