2. 石河子农业科学院 棉花研究所,新疆 石河子,832000
2. Cotton Research Institute, Shihezi Academy of Agricultural Sciences, Shihezi 832000, Xinjiang, China
活性氧(Reactive oxygen species,ROS)在植物不同器官各种代谢过程中产生[1]。植物正常的生长条件中,ROS可以作为信号分子激活逆境响应基因的表达,使逆境胁迫的植物做出相应的逆境应答反应,以适应环境变化[2-3]。但是,在遇到生物和非生物胁迫时,植物体内产生活性氧的速率迅速增加,导致ROS的积累[4]。ROS氧化蛋白质巯基而生成二硫键(P-S-S-P)和谷胱甘肽的二硫键(P-S-S-G),导致很多以巯基为催化中心的蛋白质功能丧失,从而造成细胞凋亡[5-6]。植物体内ROS的产生与清除处于动态平衡,因此其浓度保持在一定范围内不会对植物体产生损伤。谷氧还蛋白(Glutaredoxin,Grx)属于硫氧还蛋白超家族,广泛分布在各种生物体内。植物在逆境胁迫下体内ROS增加时,谷氧还蛋白会处于氧化状态从而导致植物体内ROS的功能活性降低。谷胱甘肽(Glutathione,GSH)的协助下可催化氧化状态的蛋白质二硫键或谷胱甘肽结合的二硫键还原为巯基,调节蛋白质氧化还原的状态从而调节其蛋白质的活性,避免植物氧化性损伤[7-8]。目前已从拟南芥[9]、番茄[1]、水稻[10]、文心兰“南茜”[11]等多种植物中分离到GRX基因,研究发现在植物抗逆和生长发育中GRX起着关键作用[12-13]。植物中的GRX根据其活性位点的保守残基可分为CPYC-、CGFS-和CC-三个亚型[14-15]。CPYC型含有的活性位点是Cxx[C/S],CGFS亚型的活性位点为CGFS,结构与CPYC型相似。CC型GRX的活性位点为CC(M/L)(C/S),主要存在于高等植物[10]。在对水稻和拟南芥GRX蛋白的研究中发现具有GRX功能特性的GRL(GRX-like)蛋白,因此GRX基因拓展为CPYC、CGFS、CC-type和GRL四个类型[16]。从各类型GRX中,CC型和GRL型所占比例最多,推测GRL基因在进化过程具有重要作用。GRL基因成员与典型的GRX基因同源性低,其编码蛋白质大多数具有八个保守的半胱氨酸,排列在C末端的两个CxxCx7CxxC簇中,但不涉及活性位点基序CxxC/S[17]。水稻的GRL型基因OsGRL2可以在抵抗砷胁迫的过程中发挥作用[18]。近期研究还发现,GRL型谷氧还蛋白基因相对于其他类型基因更广泛的参与非生物胁迫[19-20]。利用生物信息学方法对陆地棉GhGRL基因家族的进化关系、蛋白质序列、基因结构、保守基序和染色体分布进行了分析,并对该家族基因的表达模式进行了研究,为进一步揭示GhGRL基因的功能提供参考依据。
1 材料与方法 1.1 陆地棉类谷氧还蛋白家族基因的鉴定及理化特性分析根据拟南芥GRL蛋白序列,从棉花数据库(https://www.cottongen.org)和生物学数据库NCBI(http://www.ncbi.nlm.nih.gov/Blast)中检索,获得陆地棉中候选的GRL基因及蛋白序列(Gossypium hirsutum, NAU, Version 1.1),通过NCBI Conserved Domain Search (CDD, http://www.ncbi.nlm.nih.gov/Instruct-re/cdd/wrpsb.cgi)平台验证其保守结构域。染色体定位、相对分子质量、等电点、编码氨基酸长度等用在线软件Expasy (http://web.expasy.org/protparam)计算。GhGRL家族成员的亚细胞定位预测利用在线软件CELLO v2.5 (http://cello.life.nctu.edu.tw/)。
1.2 陆地棉类谷氧还蛋白家族基因进化树和基因结构图构建使用MEGA5.0进行进化树分析,进化树用邻接法,1000次重复。GhGRL基因结构图用在线工具GSDS(http://gsds.cbi.pku.edu.cn/)分析和绘制。
1.3 陆地棉类谷氧还蛋白家族基因多重序列比对及保守基序分析使用ClustalX和DNAMAN软件对GhGRL氨基酸序列进行多重序列比对。GhGRL蛋白序列的保守基序利用在线软件MEME(http://meme-suite.org/)进行预测,motif最大数目为10,其他参数为默认值。
1.4 陆地棉类谷氧还蛋白家族基因染色体定位及基因表达分析从基因组数据中提取基因在染色体上物理位置,用Mapinspect软件绘制染色体定位图。以新陆早33号位材料,分别提取其根、茎、雄蕊、雌蕊、子房、叶片和花,采用有参基因组的转录组分析方法进行基因表达分析,结果利用TBTOOLS软件进行处理。
2 结果与分析 2.1 GhGRL基因的鉴定与命名利用拟南芥已鉴定的13个GRL基因及氨基酸序列为查询序列,在陆地棉基因组数据库和NCBI中进行比对筛选,在陆地棉中最终获得32个GRL家族基因,依据各基因在染色体上的位置命名为GhGRL1至GhGRL32(见表 1),其基因描述为Uncharacterized protein At5g39865。蛋白质理化性质分析结果表明,GhGRL基因编码的蛋白质长度为230~390个氨基酸,相对分子质量为26.02~43.37kd,理论等电点为4.56~9.48。这些GRL蛋白质中12个基因显酸性,20个基因显碱性,表明其酸碱性不同。亚细胞定位预测的结果表明,陆地棉基因编码蛋白位于细胞核、叶绿体、质膜、线粒体、细胞质和胞外等细胞结构,其比例分别为36.4%、22.7%、18.2%、13.6%、6.8%、2.3%。
| 表 1 陆地棉GhGRL基因家族 Table 1 Information of GhGRL genes from Gossypium hirsutum |
利用陆地棉和拟南芥的GRL蛋白质序列构建系统进化树(见图 1)。由进化树显示两种植物中所有的GRL蛋白质被分为了三个亚组,分别是:GRLI、GRL II、GRL III。其中,GRL I亚组包含2个GhGRL基因,1个拟南芥GRL基因;GRL II亚组包含7个GhGRL基因,3个拟南芥GRL基因;GRL III亚组中包含23个GhGRL基因和9个拟南芥GRL基因。

|
图 1 陆地棉和拟南芥GRL进化树 Figure 1 Phylogenetic tree of GRL proteins in Gossypium hirsutum and Arabidopsis thaliana |
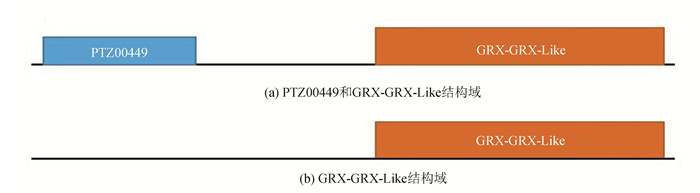
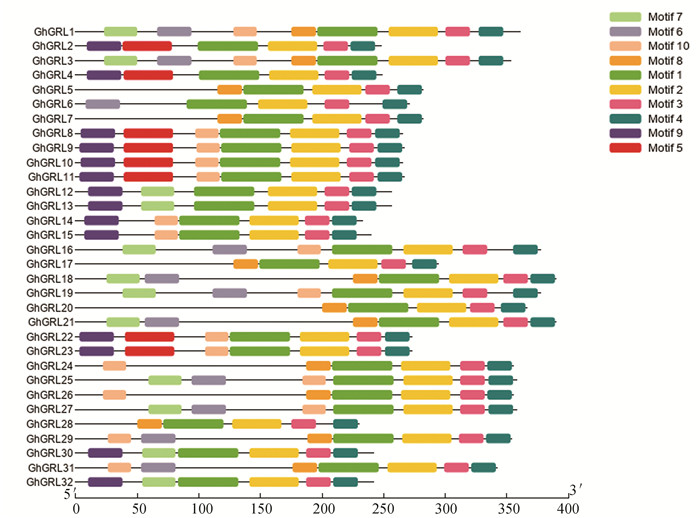
GhGRL蛋白二级结构预测发现,除GhGRL24、GhGRL26含有PTZ00449和GRX-GRX-Like两种结构域外(图 2(a)),其他基因只含有GRX-GRX-Like一种保守结构域(图 2(b));多重序列比对分析发现,32个基因氨基酸序列的相似性为31.31%(见图 3),并且C末端具有两个CXXCX7CXXC序列(见图 3-红色框)。蛋白保守基序的分析发现,陆地棉GRL蛋白有10个比较保守的基序,分别命名为Motif1-Motif 10(见图 4)。这类基因C末端均包含motif 1、motif 2、motif3、motif4四个保守基序,同时这四个基序与GRL蛋白的GRX-GRX-Like结构域(cd0301)相互重叠(见表 2)。32基因中,GhGRL1、GhGRL3包含的保守基序最多,具有8个保守基序。
|
图 2 GhGRL蛋白保守结构域预测 Figure 2 Predicted conserved domains of GhGRL proteins |

|
图 3 GhGRL氨基酸多重序列比对 Figure 3 Alignment of multiple GhGRL domain amino acid sequences |
|
图 4 陆地棉GRL蛋白基因基序 Figure 4 Motifs patterns of GRL protein sequences |
| 表 2 陆地棉GRL蛋白10个基序的信息 Table 2 Information of 10 motifs of GRL protein |
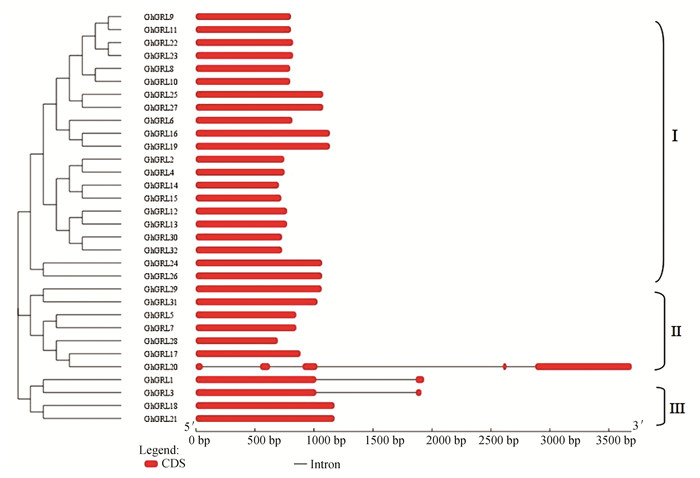
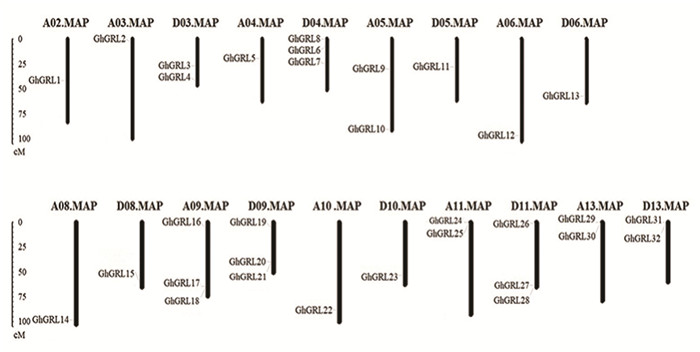
对GhGRL基因的结构分析发现,亚组Ⅰ的成员均没有内含子,亚组ⅡGhGRL20有4个内含子,GhGRL5、GhGRL7、GhGRL17、GhGRL28、GhGRL29、GhGRL31均没有内含子,亚组Ⅲ的GhGRL1和GhGRL3有1个内含子,GhGRL8和GhGRL21没有内含子(见图 5)。陆地棉GhGRL基因在基因组上分布不均匀,每条染色体上的该家族基因的数目差别很大,如D04、A09、D09、和D11染色体各含有3个GhGRL家族基因,D03、A05、A11、A13、D13染色体各有2个GhGRL家族基因,A02、A03、A04、D05、A06、D06、A08、D08、A10、D10染色体各含有1个GhGRL家族基因(见图 6)。
|
图 5 GhGRL基因结构 Figure 5 Gene structure of GhGRL |
|
图 6 GhGRL基因染色体分布 Figure 6 Chromosomal distribution of GhGRL genes in Gossypium hirsutum |
分别对GhGRL基因家族在棉花根、茎、雄蕊、雌蕊、子房、叶片和花中的表达模式进行分析,结果表明(见图 7),在根中,GhGRL8、GhGRL14两个基因不表达,GhGRL22表现出最大表达水平;在茎中,GhGRL5、GhGRL8两个基因不表达,GhGRL23表达水平最高;在雄蕊中,GhGRL12、GhGRL15、GhGRL29、GhGRL31四个基因不表达,GhGRL26表达水平最高;在雌蕊中,GhGRL12不表达,GhGRL26表达水平最高;在子房中,GhGRL5、GhGRL7、GhGRL8、GhGRL12四个基因不表达,GhGRL25表达水平最高;叶片中,GhGRL5、GhGRL7、GhGRL8三个基因不表达,GhGRL26表达水平最高;在花中,GhGRL5、GhGRL8、GhGRL12、GhGRL13、GhGRL29、GhGRL31七个基因不表达,GhGRL26表现出最大表达水平。相对于其他基因,GhGRL26在雄蕊、雌蕊、花和叶片四个组织中表达量都是最高。
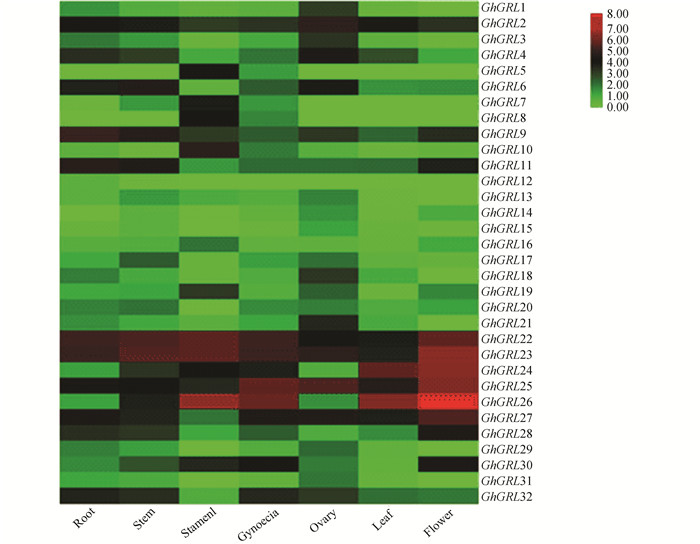
|
图 7 GhGRL基因表达模式 Figure 7 Expression patterns of GhGRL genes in Gossypium hirsutum |
目前对棉花中GRL基因在全基因组水平的生物信息学分析仍未见到报道。本研究通过生物信息学分析,在陆地棉中鉴定了32个具有GRX-GRX-Like结构域的GRL基因,并根据这些基因在染色体上的位置信息分别命名为GhGRL1-GhGRL32。与拟南芥[9]、水稻[18]、木薯[16]等植物相比陆地棉GRL基因数目明显较多,推测陆地棉谷氧还蛋白家族成员分化可能早于拟南芥、水稻、木薯的物种分化。本次实验对拟南芥和陆地棉谷氧还蛋白基因进行系统进化分析发现,陆地棉和拟南芥的GRL基因分为3个亚组,每一个亚组都有与拟南芥同源的基因,同时每组中陆地棉谷氧还蛋白基因数量多于拟南芥,由此推测陆地棉中谷氧还蛋白家族基因可能发生了物种特异性扩增[21]。
蛋白质处于特定的亚细胞位置上才能有效发挥其功能,因此了解亚细胞定位对研究蛋白质功能有一定的帮助,在本研究亚细胞预测的结果表明,陆地棉基因编码蛋白位于细胞核、叶绿体、质膜、线粒体、细胞质和胞外等细胞结构,这可能与基因功能分化的多样性有关。陆地棉GhGRL多重序列比对发现其氨基酸同源相似性为31.31%左右,通过保守基序和功能结构域分析发现,陆地棉GRL蛋白有10个比较保守的基序,这些保守基序又以不同的排序分布,这可能是造成GRL家族蛋白功能多样性的某一重要因素,而在这10个基序中,motif1、motif2、motif3、motif4四个保守基序编码功能结构域,由此推断,GhGRL基因编码的蛋白在C末端具有比较保守的结构域。
类谷氧还蛋白家族基因的结构存在相似性,大部分该家族的基因不存在内含子,无内含子的基因通常可变剪切的发生概率很低,因此陆地棉GRL家族基因的功能可能会相对保守一些[22]。基因中小片段DNA的插入可以改变基因的功能,也可能通过自然选择的方法使基因消失。而这些基因组中,只有GhGRL20有四个内含子和最长的基因长度,推测该基因在进化过程中,其结构或者功能发生了较大变化的缘故。染色体定位分析发现32个基因中,15个基因分布A基因组,17个基因分布D基因组,基因数量的不同可能与植物在进化过程中发生的基因复制、基因组重排、基因丢失都有关系,对此还有待进一步研究[23]。
拟南芥、木薯GRL家族基因在植物各个组织中广泛存在[24-25]。在本次研究也发现GhGRL基因在根、茎、雄蕊、雌蕊、子房、叶片和花中均有表达,表明它们可能在陆地棉生长发育中发挥了广泛的作用。相对于其他基因,GhGRL26在雄蕊、雌蕊、花和叶片四个组织中表达量都是最高,因此我们推测GhGRL26基因可能在棉花生长发育过程中起更重要的作用。
4 结论目前类谷氧还蛋白基因在植物中的生物功能逐渐受到重视。本研究根据拟南芥的AtGRL基因,在陆地棉中发现32个GhGRL基因,这些成员分为3个亚组,每个亚组成员具有相似的基因基序和基因结构,并且GhGRL基因具有GRL蛋白特有的结构域的和不同的染色体定位。大部分GhGRL基因在各个组织器官中均有表达。陆地棉GhGRL基因的鉴定及表达分析,为挖掘GhGRL基因的功能奠定了基础。
| [1] |
郭玉双. 番茄谷氧还蛋白基因SIGRX1的克隆与功能分析[D]. 杭州: 浙江大学, 2010. GUO Yushuang. Isolation and functional analysis of tomato glutaredoxin SIGRX1[D]. Hangzhou: Zhejiang University, 2010. (  0) 0) |
| [2] |
APEL K, HIRT H. Reactive oxygen species: metabolism, oxidative stress, and signal transduction[J]. Annual Review of Plant Biology, 2004, 55: 373-399. DOI:10.1146/annurev.arplant.55.031903.141701 (  0) 0) |
| [3] |
JIANG F, ZHANG Y, DUSTING G J, et al. NADPH oxidase-mediated redox signaling: roles in cellular stress response, stress tolerance, and tissue repair[J]. Pharmacological Reviews, 2011, 63(1): 218-242. DOI:10.1124/pr.110.002980 (  0) 0) |
| [4] |
孙晓梅. 小麦谷氧还蛋白和硫氧还蛋白的表达及其结构性质研究[D]. 广州: 华南理工大学, 2016. SUN Xiaomei. Expression, structure and characterization of wheat thioredoxin and glutaredoxin[D]. Guangzhou: South China University of Technology, 2016. (  0) 0) |
| [5] |
SCANDALIOS J G. The rise of ROS[J]. Trends in Biochemical Sciences, 2002, 27(9): 483-486. DOI:10.1016/S0968-0004(02)02170-9 (  0) 0) |
| [6] |
MILLER G, SUZUKI N, CIFTCI Y S, et al. Reactive oxygen species homeostasis and signaling during drought and salinity stresses[J]. Plant Cell and Environment, 2009, 33(4): 453-467. DOI:10.1111/j.1365-3040.2009.02041.x (  0) 0) |
| [7] |
SALMA A, JING J H, CEARY W, et al. Cysteines under ROS attack in plants: a proteomics view[J]. Journal of Experimental Botany, 2015, 66(10): 2935-2944. DOI:10.1093/jxb/erv044 (  0) 0) |
| [8] |
陈永坤, 赵启红, 刘海珍, 等. 美洲商陆谷氧还蛋白基因PaGRXC13的克隆与生物信息学分析[J]. 分子植物育种, 2016, 14(8): 2019-2024. CHEN Yongkun, ZHAO Qihong, LIU Haizhen, et al. Cloning and bioinformatics analysis of glutaredoxin gene PaGRXC13 in Phytolacca americana[J]. Molecular Plant Breeding, 2016, 14(8): 2019-2024. DOI:10.13271/j.mpb.014.002019 (  0) 0) |
| [9] |
NICOLAS N, ERIC G, JEAN P J, et al. Identification of a new family of plant proteins loosely related to glutaredoxins with four CxxC motives[J]. Photosynthesis Research, 2006, 89(2): 71-79. DOI:10.1007/s11120-006-9083-7 (  0) 0) |
| [10] |
孙瑶. 水稻谷氧还蛋白基因OsGRX20抗氧化功能的研究[D]. 金华: 浙江师范大学, 2016. SUN Yao. The antioxidative function of glutaredoxin gene OsGRX20 in rice[D]. Jinhua: Zhejiang Normal University, 2016. (  0) 0) |
| [11] |
高玉莹, 白玉, 时欢, 等. 文心兰'南茜'谷氧还蛋白基因克隆定位及表达[J]. 应用与环境生物学报, 2018, 24(3): 0533-0540. GAO Yuying, BAI Yu, SHI Huan, et al. Cloning, subcellular localization, and expression of a glutaredoxin (OnROXY1) in Oncidium'Gower Ramsey'[J]. Chinese Journal of Applied and Environmental Biology, 2018, 24(3): 0533-0540. DOI:10.19675/j.cnki.1006-687x.2017.08040 (  0) 0) |
| [12] |
KYUN O L, JUANG R L, JI Y Y, et al. GSH-dependent peroxidase activity of the rice (oryza sativa)glutaredoxin, a thioltransferase[J]. Biochemical and Biophysical Reasearch, 2002, 296(50): 1152-11556. DOI:10.1016/s0006-291x(02)02047-8 (  0) 0) |
| [13] |
ROUHIER N. Plant glutaredoxins: pivotal players in redox biology and iron-sulphur centre assembly[J]. New Phytologist, 2010, 186(2): 365-372. DOI:10.1002/ange.1971083221193 (  0) 0) |
| [14] |
STEPHANE D L. The glutaredoxin family in oxygenic photosynthetic organisms[J]. Photosynthesis Research, 2004, 79(3): 305-318. DOI:10.1023/B:PRES.0000017174.60951.74 (  0) 0) |
| [15] |
XING S, LAUR A, ZZCHGO S, et al. Redox regulation and flower development: a novel function for glutaredoxins[J]. Plant Biology, 2006, 8(5): 547-555. DOI:10.1055/s-2006-924278 (  0) 0) |
| [16] |
杨义伶. 木薯CC-type MeGRXs及MeDREB1D基因抗旱功能研究[D]. 武汉: 华中农业大学, 2016. YANG Yiling. Funtional analysis of CC-type MeGRXs and MeDREB1D genes from cassava in drought tolerance[D]. Wuhan: Huazhong Agriculture University, 2016. (  0) 0) |
| [17] |
黄淦, 王潇, 金学锋, 等. 拟南芥谷氧还蛋白GRXC9负调控叶片大小[J]. 植物学报, 2017, 52(5): 550-559. HUANG Gan, WANG Xiao, JIN Xuefeng, et al. GRXC9 negatively regulates leaf size in arabidopsis[J]. Chinese Bulletin of Botany, 2017, 52(5): 550-559. DOI:10.11983/CBB16208 (  0) 0) |
| [18] |
DUBEY A K, KUMAR N, SAHU N, et al. Response of Two rice cultivars differing intheirsensitivity towards arsenic, differs in their expresssion of glutaredoxin and glutathione S transferase genes and antioxidant usage[J]. Ecotoxicology and Environment Safety, 2016, 124: 393-405. DOI:10.1016/j.ecoenv.2015.10.017 (  0) 0) |
| [19] |
GAR R, JHANWAR S, TYAGI A K, et al. Genome-wide survey and expression analysis suggest diverse roles of glutaredoxin gene family members during development and response to various stimuli in rice[J]. DNA Research, 2010, 17: 353-367. DOI:10.1093/dnares/dsq023 (  0) 0) |
| [20] |
WAQAR A M, XIAO G W, XIN L W, et al. Genome-wide expression analysis suggests glutaredoxin genes response to various stresses in cotton[J]. International Journal of Biological Macromolecules, 2020, 153: 470-491. DOI:10.1016/j.ijbiomac.2020.03.021 (  0) 0) |
| [21] |
刘伟. 两个二倍体棉种CDPK和FAD基因家族的全基因组鉴定与基因结构功能分析[D]. 杭州: 浙江大学, 2015. LIU Wei. Genome-wide identification and structure-function analysis of CDPK and FAD gene families in two dipliod cotton species[D]. Hangzhou: Zhejiang University, 2015. (  0) 0) |
| [22] |
李锡花. 棉花SAUR基因家族分析与功能验证[D]. 杨凌: 西北农林科技大学, 2017. DOI: CNKI:CDMD:1.1018.038305. LI Xihua. Identification and functional analysis of the small auxin up RNA(SAUR) gene family in cotton[D]. Yangling: Northwest A & F University. DOI: CNKI:CDMD:1.1018.038305. (  0) 0) |
| [23] |
刘潮, 韩利红, 宋培兵, 等. 桑树WRKY转录因子的全基因组鉴定及生物信息学分析[J]. 南方农业学报, 2017, 48(9): 1691-1699. LIU Chao, HAN Lihong, SONG Peibing, et al. Genome-wide identification and bioinformatics analysis for mulberry WRKY transcription factors[J]. Journal of Southern Agriculture, 2017, 48(9): 1691-1699. DOI:10.3969/j.issn.2095-1191.2017.09.25 (  0) 0) |
| [24] |
ZIMMERMANN P, HIRSCH H M, HENNING L, et al. Arabidopsis microarray database and analysis toolbox[J]. Plant Physiology, 2004, 136(4): 2621-2632. DOI:10.1104/pp.104.046367 (  0) 0) |
| [25] |
王雪, 阮孟斌, 王斌, 等. 木薯干旱胁迫响应CC类谷氧还蛋白基因的筛选与体外表达分析[J]. 分子植物育种, 2017, 15(4): 1204-1211. WANG Xue, RUAN Mengbin, WANG Bin, et al. Screening and expression analysis in vitro of CC-type glutaredoxin genes of cassava in response to drought stress[J]. Molecular Plant Breeding, 2017, 15(4): 1204-1211. DOI:CNKI:SUN:FZZW.0.2017-04-004 (  0) 0) |
 2021, Vol. 19
2021, Vol. 19


