2002年,Sheehy等[1]通过消减克隆方法首次发现了宿主限制因子CEM15(即APOBEC3G,简称A3G),作为生命体重要的防御蛋白,APOBEC3(简称A3)胞嘧啶脱氨酶家族也就成为研究的热点,随后的研究中又陆续发现了位于人体22号染色体上的其他A3成员。哺乳动物A3可利用其胞嘧啶脱氨酶活性将慢病毒负链DNA上的C脱氨基成为U,从而导致正链DNA上的G→A超突变而抑制慢病毒复制,可通过引起病毒基因组中的突变和阻断逆转录来限制逆转录病毒感染,虽然这两种限制方法在体外都起作用,但在体内感染期间它们的作用知之甚少[2]。
哺乳动物A3基因在一个CBX6和CBX7两侧的染色体位点复制,A3基因的数量和A3复制过程的历史在每个哺乳动物谱系都是不同的[3]。小鼠只有一个A3(mA3)基因,mA3可诱发CTL免疫应答并产生针对Friend逆转录病毒感染的中和抗体[4]。人类有七个A3基因,而hA3G是A3家族中最明确的抗HIV-1蛋白[4]。Wang等[5]认为靶向hA3G可通过减弱DNA修复途径的激活而使癌细胞对辐射诱导的细胞死亡敏感。癌症基因组学的最新进展以及A3酶的生物化学表征现在已经暗示至少两个家族成员在肿瘤发展期间进行体细胞诱变[6]。Huff等[7]认为B16细胞中A3基因的敲除降低了体外抵抗水疱性口炎病毒(vesicular stomatitis virus,简称VSV)感染的能力,提高了体内VSV的治疗效果。
总之,A3家族是抗逆转录病毒、逆转录元件的抑制因子具有重要的免疫功能,最近A3在癌症发生中的作用也被发现[8]。因此,研究hA3G与mA3的结构有助于进一步了解其功能及其在生物进化中的作用。本文利用生物信息学方法对mA3和hA3G的结构进行分析,以进一步加深对其抗病毒等功能的了解,为研究其作用机制、设计疫苗、治疗癌症等提供思路。
1 材料与方法 1.1 材料本文在NCBI数据库收集hA3G及mA3蛋白的序列信息,选择hA3G(检索号:AAH24268.1)与mA3(检索号ACH89412.1)进行研究。
1.2 方法 1.2.1 蛋白质序列比对输入网址https://www.ebi.ac.uk/Tools/msa/,点击Launch Clustal Omega,分别将hA3G与mA3的蛋白序列fasta格式输入,对二者的蛋白质序列进行比对。
1.2.2 理化性质分析输入网址https://www.expasy.org/proteomics,在ExPASy数据分析系统选择ProtParam工具,分析hA3G和mA3的分子量、分子式、酸碱性和稳定性等理化性质。输入网址https://www.expasy.org/proteomics,点击Protscale,使用Protscale工具,点击输入hA3G和mA3蛋白序列,分析hA3G和mA3的亲疏水性。
1.2.3 亚细胞定位预测输入网址http://psort.hgc.jp,PSORTⅡPrediction,分别输入hA3G与mA3的蛋白序列,点击submit,进行亚细胞定位预测。
1.2.4 二级结构和高级结构分析输入网址http://www.cbs.dtu.dk/,选择Prediction Servers,利用TMHMM2.0工具,将hAG和mA3的蛋白序列分别输入,对hA3G和mA3的跨膜区域进行分析。输入网址https://npsa-prabi.ibcp.fr-/cgi-bin/secpred_sopma.pl,分别将A3G和mA3的序列输入,采用SOPMA工具对hA3G与mA3的二级结构及各成分所占比例进行预测。在NCBI数据库https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi,利用Consrved Domain数据库分析hA3G与mA3的结构域。
输入网址https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi,选择Phyre2工具,预测hA3G和mA3的蛋白质三维结构。
1.2.5 相互作用蛋白分析输入网址http://string-db.org/,利用STRING数据库构建与hA3G及mA3相互作用的蛋白网络。
2 结果与分析 2.1 蛋白序列比对分析hA3G编码384个氨基酸,mA3编码396个氨基酸,表 1显示了hA3G与mA3的cov为93%,pid为28.6%。
| 表 1 hA3G与mA3的Clustal Omega比对结果 Table 1 Sequence alignment results of hA3G and mA3 using Clustal Omega |
通过ExPASy数据库中的在线工具ProtParam,预测hA3G与mA3蛋白相应理化性质,见表 2。可以看出,hA3G蛋白与mA3蛋白均为碱性的不稳定蛋白质,二者含量最高的氨基酸也都是Leu。
| 表 2 hA3G与mA3的理化性质 Table 2 Physical and chemical properties of hA3G and mA3 |
利用ExPASy数据库中的ProtScale在线工具预测,结果见表 3,hA3G与mA3属于亲水性蛋白质(见图 1)。
| 表 3 hA3G和mA3蛋白的亲疏水性分析 Table 3 Analysis of hydrophilicity and hydrophobicity of hA3G and mA3 |
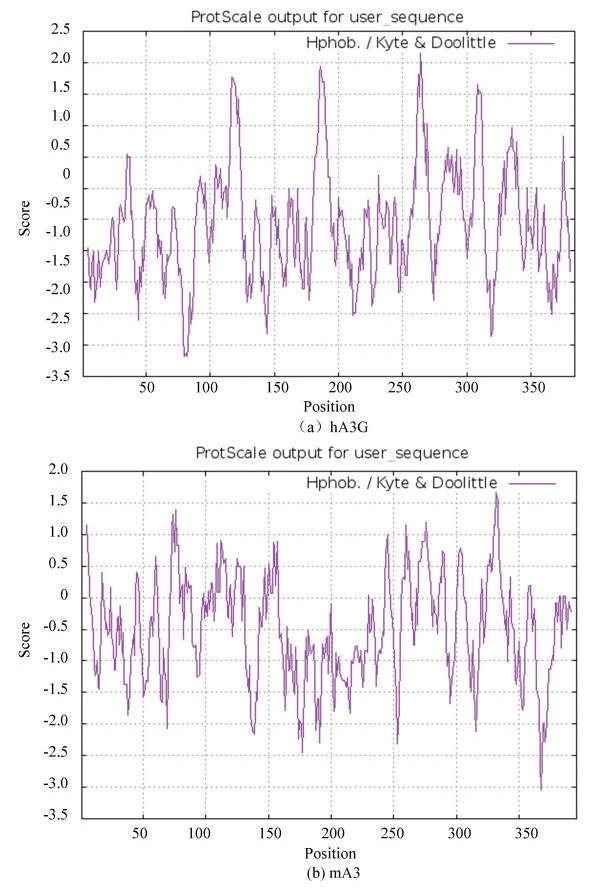
|
图 1 hA3G与mA3蛋白的亲疏水性分析 Figure 1 Analysis of hydrophilic and hydrophobic properties of hA3G and mA3 |
利用PSORTⅡ分析hA3G与mA3蛋白的亚细胞定位,结果见表 4,可看出hA3G和mA3蛋白均可能定位在细胞质、线粒体和细胞核,而最可能的细胞定位是在细胞质。
| 表 4 hA3G和mA3蛋白的亚细胞定位 Table 4 Subcellular localization of hA3G and mA3 |
经TMHMM2.0工具预测结果如图 2和表 5所示,hA3G和mA3均为不跨膜蛋白。红色细线表示跨膜区域hA3G和mA3位于膜外(粉色细线)的概率几乎为100%,位于膜内(蓝色细线)和跨膜区域(红色细线)的概率几乎为0,粉色粗线代表多肽链中跨膜区域所在位置,因没有跨膜区域,所以不显示相应标记。
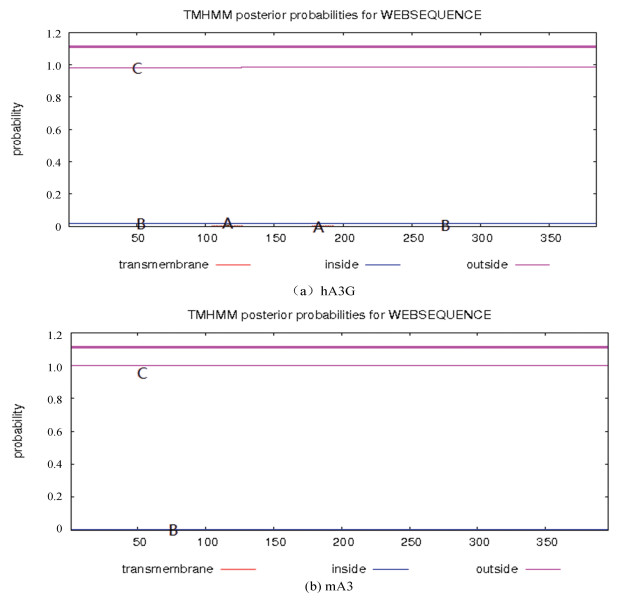
|
图 2 hA3G和mA3的跨膜结构分析 Figure 2 Analysis of transmembrane structure of hA3G and mA3 注:A代表跨膜部分;B代表膜内部分;C代表膜外部分. |
| 表 5 hA3G与mA3的TMHMM分析结果 Table 5 TMHMM analysis of hA3G and mA3 |
利用SOPMA工具分析hA3G和mA3蛋白的二级结构如图 3,与相应二级结构占比见表 6。由此可见,hA3G和mA3的二级结构主要为α螺旋和无规卷曲,二者的二级结构比例相差不大,结构非常相似。
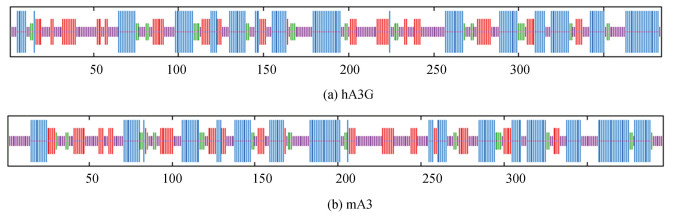
|
图 3 hA3G和mA3蛋白二级结构分析 Figure 3 Secondary structure analysis of hA3G and mA3 注:图 3中蓝色代表α helix;红色代表Extended strand;绿色代表β turn;紫色代表Random curl. |
| 表 6 hA3G和mA3二级结构类型及占比 Table 6 Secondary structure types and proportions of hA3G and mA3 |
通过NCBI的Conserved Domain数据库预测hA3G和mA3都属于APOBEC超家族,都具有两个保守的结构域。APOBEC超家族都包含串联的一个或两个胞嘧啶脱氨酶结构域。APOBEC的C末端结构域(CD2)具有催化活性,而N末端结构域(CD1)不具有使底物脱氨基的能力,即使它们维持核心胞苷脱氨酶折叠,CD1与ssDNA和RNA的结合可以上调或下调相邻CD2的催化活性[9]。
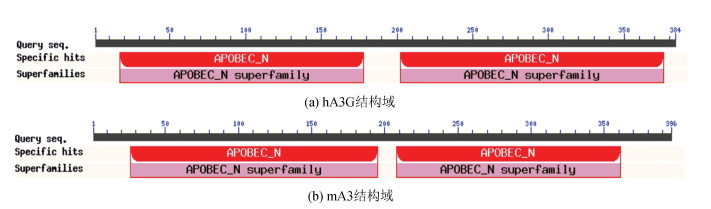
|
图 4 hA3G和mA3蛋白的结构域分析 Figure 4 Conserved domain analysis of hA3G and mA3 |
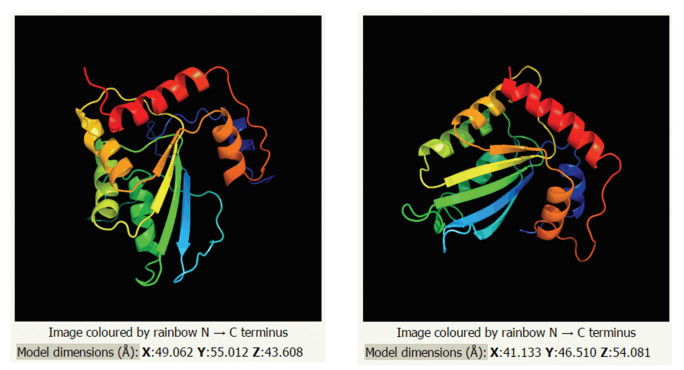
利用Phyre2工具,进行hA3G和mA3进行蛋白质三维结构预测,结果如图 5所示,三维结构中的二级结构预测结果如图 6。hA3G的模板为c2kboA,可信度100%,覆盖面为50%,一致性为99%;mA3的模板为c6bwyA,可信度100%,覆盖面为45%,一致性为36%。
|
图 5 hA3G和mA3蛋白的三维结构 Figure 5 Three-dimensional structure of hA3G and mA3 |

|
图 6 hA3G和mA3蛋白三维结构下的二级结构 Figure 6 Secondary structure of hA3G and mA3 in three-dimensional model |
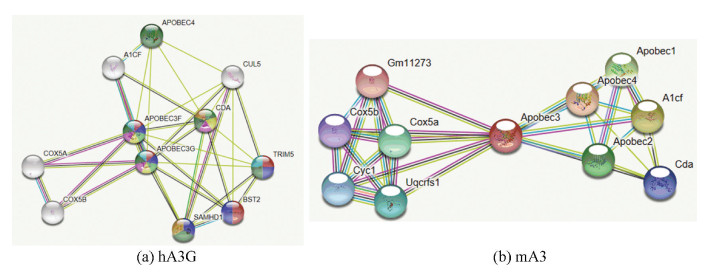
利用STRING数据库预测hA3G和mA3的蛋白相互作用网络,结果如图 7所示。与hA3G相互作用的蛋白质包括APOBEC3F(简称A3F)、SAMHD1(Sterile alpha motif and HD domain-containing protein 1,简称SAMHD1)、BST2(bone marrow stromal antigen 2)、A1CF(APOBEC1 complementation factor,简称A1CF)、COX5A(cytochrome c oxidase subunit 5A,简称COX5A)、COX5B(cytochrome c oxidase subunit 5B,简称COX5B)以及CDA(cytidine deaminase,简称CDA)等。hA3F和hA3G属于A3蛋白质,已被确认为有效的抗病毒因子,可限制Vif缺陷的HIV-1复制[10]。SAMHD1是人类细胞中唯一已知的dNTP酶,含有经典的核定位序列(NLS),抑制逆转录病毒和逆转录病毒的复制[11]。BST-2是一种Ⅰ型干扰素(IFN-I)诱导的细胞蛋白,最初被视为HIV-1释放的抑制剂,随后被证明可抑制多种包膜病毒的细胞释放,在先天抗病毒防御系统中起重要作用[12]。人们一直认为APOBEC1介导的编辑活性依赖于RNA结合蛋白A1CF,在成人肝脏、小肠或肾脏中正常的C→U编辑不需要A1CF[13]。COX5A是线粒体呼吸链复合物IV(细胞色素c氧化酶)的核编码亚基[14]。COX5B是电子传递链的末端酶,可将电子从还原的细胞色素c转移到氧气,并且在此过程中,在线粒体内膜上产生电化学梯度[15]。CDA是嘧啶补救途径的酶,可分别通过胞苷和2'-脱氧胞苷的水解脱氨作用催化尿苷和2'-脱氧尿苷的形成[16]。hA3G可能涉及到的生物过程包括病毒生命周期的负调控(GO: 1903901)、病毒防御应答(GO: 0051607)、胞嘧啶代谢过程(GO: 0046087)、碱基转换或替换编辑(GO: 0016553)以及胞嘧啶脱氨基(GO: 0009972)。hA3G可能具有水解酶活性(GO: 0016814)、胞嘧啶脱氨酶活性(GO: 0004126)、锌离子结合功能(GO: 0008270)、细胞色素c氧化酶活性(GO: 0004129)以及相同蛋白结合功能(GO: 0042802)。hA3G可能构成载脂蛋白BmRNA编辑酶复合物Ⅳ(GO: 0030895)、P-小体(GO: 0000932)及线粒体呼吸链复合物(GO: 0005751)。hA3G的KEGG通路主要是心肌收缩(hsa04260)、亨廷顿氏病(Huntington's disease,hsa05016)、帕金森病(Parkinson's disease,hsa05012)、阿尔茨海默病(Alzheimer's disease,hsa05010)及非酒精性脂肪性肝病(hsa04932)。hA3G的反应途径包括编辑体的形成(HSA-75094)、干扰素信号传递(HSA-913531)、TP53调节代谢基因(HSA-5628897)及核酸代谢(HSA-15869)等。
|
图 7 与hA3G和mA3相互作用的蛋白网络 Figure 7 Protein network interacting with hA3G and mA3 |
与mA3相互作用的蛋白质包括A1CF、COX5A、COX5B、APOBEC1、APOBEC2、Gm11273(一种细胞色素C氧化酶)、细胞色素C1(cytochrome c1,简称Cyc1)以及Uqcrfs1(ubiquinol-cytochrome c reductase, Rieske iron-sulfur polypeptide 1,简称Uqcrfs1)等。Uqcrfs1是线粒体呼吸链细胞色素bc1复合物的关键亚基,产生与ATP合成偶联的电化学势,以将电子从泛醇转移至细胞色素c[17]。mA3涉及到的生物过程包括C→U编辑(GO: 0016554)、胞嘧啶代谢过程(GO: 0046087)、胞嘧啶脱氨基(GO: 0009972)、含核碱基的小分子代谢过程(GO: 0055086)及mRNA修饰(GO: 0016556)。mA3可能具有水解酶活性(GO: 0016814)、脱氨酶活性(GO: 0019239)、胞嘧啶脱氨酶活性(GO: 0004126)、锌离子结合功能(GO: 0008270)、电子转移功能(GO: 0009055)以及结合金属离子的功能(GO: 0046872)。mA3可参与构成细胞色素复合物(GO: 0070069)、呼吸链复合体(GO: 0098803)、线粒体呼吸链复合物Ⅳ(GO: 0005751)及线粒体内膜蛋白复合物(GO: 0098800)。mA3的KEGG通路主要是心肌收缩(mmu04260)、帕金森病(Parkinson's disease,mmu05012)、非酒精性脂肪性肝病(mmu04932)、氧化磷酸化(mmu00190)以及阿尔茨海默病(Alzheimer's disease,mmu05010)。mA3参与的反应途径有编辑体的形成(MMU-75094)。
3 讨论mA3是胞苷脱氨酶,其可作用于逆转录病毒的单链DNA逆转录物,导致前病毒DNA的G→A超突变,其对Vif缺陷HIV-1的限制与hA3G一样强大[3, 18]。通过序列比对发现hA3G编码384个氨基酸,mA3编码396个氨基酸,hA3G与mA3的cov为93%,pid为28.6%。本文利用ExPASy数据库中的ProtParam和ProtScale在线工具预测hA3G和mA3均为碱性的不稳定亲水性蛋白质,二者含量最高的氨基酸也都是Leu。PSORTⅡ分析hA3G和mA3定位于细胞质。TMHMM2.0工具预测hA3G和mA3为不跨膜蛋白。
hA3G和mA3的二级结构主要为α螺旋和无规卷曲,属于APOBEC超家族,具有两个保守的结构域。已有研究显示hA3G C末端结构域具有脱氨酶活性,N-末端结构域是进入病毒粒子所必需的,mA3C末端结构域是进入病毒粒子所必需的,而N-末端结构域具有胞苷脱氨酶活性[3]。
利用STRING数据库发现与hA3G相互作用的蛋白质包括A3F、SAMHD1、BST2、A1CF、COX5A、COX5B。hA3G可能参与病毒生命周期的负调控、病毒防御应答、胞嘧啶代谢过程、碱基转换或替换编辑以及胞嘧啶脱氨基。与mA3相互作用的蛋白质包括A1CF、COX5A、COX5B、APOBEC1、APOBEC2、Gm11273、Cyc1以及Uqcrfs1等。mA3涉及到的生物过程包括C→U编辑、胞嘧啶代谢过程、胞嘧啶脱氨基、含核碱基的小分子代谢过程及mRNA修饰。hA3G和mA3具有水解酶活性和脱氨酶活性,具有锌离子结合功能。可以看出,与hA3G和mA3均相互作用的蛋白有A1CF、COX5A和COX5B;二者都可构成线粒体复合物,KEGG通路都是心肌收缩、帕金森病以及阿尔茨海默病。
哺乳动物APOBEC3蛋白是体内限制慢病毒的重要因子。为了克服APOBEC3介导的抗病毒作用,慢病毒在进化上获得了辅助蛋白——病毒感染因子Vif,而Vif通过泛素/蛋白酶体依赖性途径降解宿主APOBEC3蛋白。尽管Vif-APOBEC3相互作用及其进化意义已得到很好的研究,特别是灵长类动物慢病毒(包括HIV)和灵长类动物(包括人类)的相互作用,但对非灵长类慢病毒和非灵长类动物的研究很少。
啮齿动物仅编码一个APOBEC3家族成员,大鼠和小鼠均为APOBEC3Z2Z3基因融合并且缺失APOBEC3Z1基因[19-20]。mA3限制ΔVifHIV-1的能力与hA3G相似,并且在ΔVifHIV-1中诱导了非常高水平的G→A突变。mA3与hA3G的结构分析可能对进一步揭示哺乳动物APOBEC3的功能及其在治疗艾滋病和癌症等疾病中的作用有重要意义。
| [1] |
SHEEHY A M, GADDIS N C, CHOI J D, et al. Isolation of a human gene that inhibits HIV-1 infection and is suppressed by the viral Vif protein[J]. Nature, 2002, 418(6898): 646-650. DOI:10.1038/nature00939 (  0) 0) |
| [2] |
OKEOMA C M, LOVSIN N, PETERLIN B M, et al. APOBEC3 inhibits mouse mammary tumour virus replication in vivo[J]. Nature, 2007, 445(7130): 927-930. DOI:10.1038/nature05540 (  0) 0) |
| [3] |
STAVROU S, ZHAO W, BLOUCH K, et al. Deaminase-dead mouse APOBEC3 is an in vivo retroviral restriction factor[J]. Journal of Virology, 2018, 92(11): e00168-18. DOI:10.1128/JVI.00168-18 (  0) 0) |
| [4] |
COLOMER-LLUCH M, RUIZ A, MORIS A. et al. Restriction factors: From intrinsic viral restriction to shaping cellular immunity against HIV-1[J/OL]. Frontiers of Immunology, 2018, 9: 2876. DOI: 10.3389/fimmu.2018.02876.
(  0) 0) |
| [5] |
WANG Y, WU S F, ZHENG S Y, et al. APOBEC3G acts as a therapeutic target in mesenchymal gliomas by sensitizing cells to radiation-induced cell death[J]. Oncotarget, 2017, 8(33): 54285-54296. DOI:10.18632/oncotarget.17348 (  0) 0) |
| [6] |
HENDERSON S, FENTON T. APOBEC3 genes: Retroviral restriction factors to cancer drivers[J]. Trends in Molecular Medicine, 2015, 21(5): 274-284. DOI:10.1016/j.molmed.2015.02.007 (  0) 0) |
| [7] |
HUFF A L, WONGTHIDA P, KOTTKE T, et al. APOBEC3 mediates resistance to oncolytic viral therapy[J/OL]. Molecular Therapy Oncolytics, 2018, 11: 1-13. DOI: 10.1016/j.omto.2018.08.003.
(  0) 0) |
| [8] |
KING J J, LARIJANI M. A novel regulator of activation-induced cytidine deaminase/APOBECs in immunity and cancer: Schrödinger's CATalytic pocket[J/OL]. Frontiers of Immunology, 2017, 8: 351. DOI: 10.3389/fimmu.2017.00351.
(  0) 0) |
| [9] |
SALTER J D, SMITH H C. Modeling the embrace of a mutator: APOBEC selection of nucleic acid ligands[J]. Trends in Biochemical Sciences, 2018, 43(8): 606-622. DOI:10.1016/j.tibs.2018.04.013 (  0) 0) |
| [10] |
FANG Y, XIAO X, LI S X, et al. Molecular interactions of APOBEC3F catalytic domain with a single-stranded DNA[J]. Journal of Molecular Biology, 2018, 430(1): 87-101. DOI:10.1016/j.jmb.2017.11.007 (  0) 0) |
| [11] |
DU J, PENG Y, WANG S, et al. Nucleocytoplasmic shuttling of SAMHD1 is important for LINE-1 suppression[J]. Biochemical and Biophysical Research Communications, 2019, 510(4): 551-557. DOI:10.1016/j.bbrc.2019.02.009 (  0) 0) |
| [12] |
URATA S, KENYON E, NAYAK D, et al. BST-2 controls T cell proliferation and exhaustion by shaping the early distribution of a persistent viral infection[J]. PLoS Pathogens, 2018, 14(7): e1007172. DOI:10.1371/journal.ppat.1007172 (  0) 0) |
| [13] |
SNYDER E M, MCCARTY C, MEHALOW A, et al. APOBEC1 complementation factor (A1CF) is dispensable for C-to-U RNA editing in vivo[J]. RNA-A Publication of the RNA Society, 2017, 23(4): 457-465. DOI:10.1261/rna.058818.116 (  0) 0) |
| [14] |
BAERTLING F, AL-MURSHEDI F, SÁNCHEZ-CABALLERO L, et al. Mutation in mitochondrial complex IV subunit COX5A causes pulmonary arterial hypertension, lactic acidemia, and failure to thrive[J]. Human Mutation, 2017, 38(6): 692-703. DOI:10.1002/humu.23210 (  0) 0) |
| [15] |
HU T, XI J. Identification of COX5B as a novel biomarker in high-grade glioma patients[J]. Onco Targets & Theropy, 2017, 10: 5463-5470. DOI:10.2147/OTT.S139243 (  0) 0) |
| [16] |
MICOZZI D, CARPI F M, PUCCIARELLI S, et al. Human cytidine deaminase: A biochemical characterization of its naturally occurring variants[J]. International Journal of Biological Macromolecules, 2014, 63: 64-74. DOI:10.1016/j.ijbiomac.2013.10.029 (  0) 0) |
| [17] |
JUN K H, KIM S Y, YOON J H, et al. Amplification of the UQCRFS1 gene in gastric cancers[J]. Journal of Gastric Cancer, 2012, 12(2): 73-80. DOI:10.5230/jgc.2012.12.2.73 (  0) 0) |
| [18] |
BOI S, FERRELL M E, ZHAO M, et al. Mouse APOBEC3 expression in NIH 3T3 cells mediates hypermutation of AKV murine leukemia virus[J]. Virology, 2018, 518: 377-384. DOI:10.1016/j.virol.2018.03.014 (  0) 0) |
| [19] |
NAIR S, SANCHEZ-MARTINEZ S, JI X, et al. Biochemical and biological studies of mouse APOBEC3[J]. Journal of Virology, 2014, 88(7): 3850-3860. DOI:10.1128/JVI.03456-13 (  0) 0) |
| [20] |
MVNK C, WILLEMSEN A, BRAVO I G. An ancient history of gene duplications, fusions and losses in the evolution of APOBEC3 mutators in mammals[J/OL]. BMC Evolutionary Biology, 2012, 12: 71. DOI: 10.1186/1471-2148-12-71.
(  0) 0) |
 2021, Vol. 19
2021, Vol. 19

