2. 浙江大学 生命科学学院,杭州 310058
2. College of Life Sciences, Zhejiang University, Hangzhou 310058, China
生物信息学是一门融合了生命科学、计算机科学和数学等多学科的新兴交叉学科,它通过对生物学实验数据的获取、加工、存储、检索与分析,揭示数据所蕴含的生物学意义,它既是一门科学学科,更是一种重要的研究平台与工具,参与着生命科学各领域的研究[1]。近年来,随着生命科学技术的不断发展和革新,生物学相关的数据规模正指数级地快速增长。正确运用生物信息学方法从海量数据中挖掘并获得信息和知识,是生物学、生态学、医学、农学、环境科学、生物工程、食品科学等学科领域人才的必备实践能力。因此,生物信息学实验教学在提升和体现生物学相关专业人才培养质量和培养水平中发挥重要作用[2]。
生物信息学实验涵盖内容广泛,如何使学生将抽象的理论知识转化为实用的分析技能,使学生能够针对具体问题运用恰当方法正确处理数据并得出准确结论,是生物信息学实验教学的重点。而传统实验教学内容和模式存在的一些问题使得教学效果不够理想,如教学案例和实验材料抽象导致学生学习积极性不高,学生未能充分认识研究背景和数据来源导致学习效果不显著,不同实验间缺乏联系导致学生的实际应用能力和创新能力提升不足等。根据“双一流建设”要求和创新型人才培养需要,浙江大学生命科学学院对生物信息学相关课程开展了系列改革,积极更新传统教学内容并探索新的教学模式。结合实际科研问题和成果,设计问题导向式的综合实验,充分结合理论知识和实践体系,提升学生的实验操作技能和综合创新能力,提高实验教学效果和教学质量[3-4]。
1 综合实验的设计思路和特色立足于生物信息学实验教学需求,依托实际科研问题和成果,以提高学生学习兴趣和课堂教学效率为目标,进行综合实验教学设计[5]。综合实验以环境样品中基因资源挖掘以及蛋白质结构和功能分析为主线,以海洋来源的两个同源宏基因组片段及片段上的两个同源类水解酶基因为实验材料,通过问题导向法引导学生深入探索,从组学角度注释分析宏基因组片段并获得同源类水解酶基因,从基因角度开展序列比对与系统发育分析,从蛋白质角度比较分析同源酶的序列和结构特征从而回答其底物特异性和偏好性机制的科学问题[6]。
实验设计结合了生物信息学、宏基因组学、生物化学、分子生物学、结构生物学等多个学科领域,贯穿了文献检索、数据库检索、开放阅读框预测、序列相似性搜索、基因功能注释、序列比对、系统发育分析、蛋白质结构分析与预测、分子对接等知识点,综合了多种生物信息学数据库和分析工具的使用。实验通过实际科研问题的引导,充分激发学生自主学习和探索的兴趣;通过对理论知识和实验技能的融会贯通,全面提升学生对专业知识的掌握及运用能力;通过以学生为中心的探索实践,培养学生科研实践能力和综合创新能力,为学生后续开展科研训练和创新创业奠定坚实基础。
2 实验内容 2.1 序列下载在充分了解科学背景的基础上,登陆NCBI数据库(https://www.ncbi.nlm.nih.gov/),根据给定的GenBank登陆号下载两个宏基因组片段的核苷酸序列FASTA格式序列,同时熟悉GenBank页面信息。
2.2 宏基因组注释与分析利用MetaGeneMark在线工具(http://exon.gatech.edu/meta_gmhmmp.cgi)[7]识别基因阅读框,获得基因阅读框的核苷酸和氨基酸序列;利用NCBI的BLASTN和BLASTP工具(https://blast.ncbi.nlm.nih.gov/Blast.cgi)[8]针对NR和UniProtKB/Swiss-Prot等数据库进行核苷酸和氨基酸序列数据到数据库的比对,查看并分析比对结果;利用eggNOG-mapper(http://eggnog-mapper.embl.de/)[9-10]进行GO、KEGG、COG等功能注释。整理、分析并比较两个宏基因组片段编码的基因及其功能。
2.3 序列比对与系统发育分析在BLAST比对结果中筛选或根据文献资料和数据库检索下载同源氨基酸序列,利用Clustal X[11]和MEGA[12]软件进行序列比对并构建系统发育树,分析目标蛋白质的家族归属、演化地位及与同源蛋白质的演化关系。
2.4 蛋白质一级结构分析利用ProtParam工具(https://web.expasy.org/protparam/)[13]计算目标蛋白质的理化性质参数,包括蛋白质的分子量、等电点、氨基酸组成、消光系数、不稳定系数等;利用ProtScale工具(https://web.expasy.org/protscale/)[13]分析蛋白质亲疏水性和极性的分布;利用SignalP工具(http://www.cbs.dtu.dk/services/SignalP/)[14]预测目标蛋白质是否有信号肽。
2.5 蛋白质二级结构和结构域分析利用基于人工神经网络算法的PredictProtein工具(https://www.predictprotein.org/)[15]和PSIPRED工具(http://bioinf.cs.ucl.ac.uk/psipred/)[16]以及整合多种算法的SOPMA工具(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)[17]预测二级结构,并比较不同算法的预测结果差异。利用InterPro工具(http://www.ebi.ac.uk/interpro/search/sequence/)[18]分析目标蛋白质的结构域、蛋白质家族和功能位点信息等。
2.6 蛋白质三级结构模拟与分析利用蛋白质结构预测工具Swiss-Model(https://swissmodel.expasy.org/)[19]进行同源建模,获得目标蛋白质的三维结构模型;在PDB数据库[20]中查看相关结构模板信息,熟悉PDB数据库的数据格式;比较基于不同模板构建的模型差异和评估结果差异;使用PyMOL软件(http://pymol.sourceforge.net)可视化分析两个目标蛋白质结构模型,通过叠合比较结构差异,特别是底物结合口袋差异;利用DALI服务器(http://ekhidna2.biocenter.helsinki.fi/dali/)[21]通过比对蛋白质结构比较结构差异。
2.7 酶与底物分子对接以两个目标蛋白质三维结构模型为受体,以不同链长的对硝基苯酚酯为配体,使用AutoDock软件[22]进行分子对接,观察并比较不同构象的配体与受体的相互作用情况,分析两个目标蛋白质的底物结合差异。
3 实施方案基于科研实际科学问题,建立一套针对宏基因组、基因和蛋白质分析的生物信息学综合实验。本文中实施方案以16学时为例阐述(见图 1)。在实际教学实践中,根据学情和教学安排不同,可做具体调整。例如,针对生物学基础较为薄弱的学生或学时数较少的课程,可以安排学生只开展教师设计完善的实验,而不开展自主实验设计,使学生掌握生物信息学的基本理论知识和技术方法;针对具备完善生物化学和分子生物学等学科知识背景的生物专业相关学生,可以增加学时数,拓展数据库利用、高通量测序与分析、组学和系统生物学等相关内容,进一步提高学生的科研实践能力和综合创新能力。
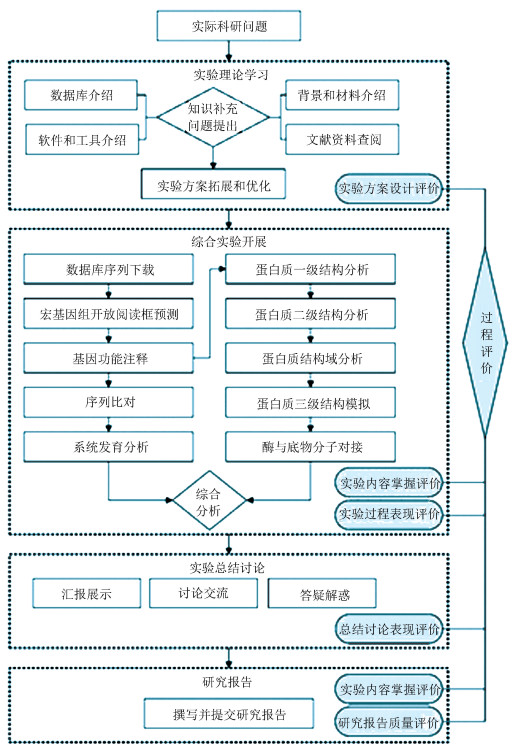
|
图 1 生物信息学综合实验教学实施方案 Figure 1 Implementation scheme of comprehensive practice of bioinformatics |
介绍综合实验的教学目的和教学内容;在理论课的基础上,补充介绍综合实验相关的数据库、工具和软件及其基本算法原理;介绍综合实验的科学背景和实验材料,引出科学问题;指导学生查阅相关文献和资料;在已有实验方案基础上,学生根据自己兴趣,提出创新性实验方案;或学生根据自己科研项目内容,提出自主综合实验内容和实验方案。
3.2 综合实验开展(8学时)学生在教师指导下完善实验方案,根据方案和要求自主开展实验,对实验过程中获得的原始数据和分析结果进行详实记录,并对实验结果进行整理、归纳、统计、比较和分析,结合已知的科学背景及生物学现象,从生物信息学角度分析阐释其发生机制。
3.3 实验总结讨论(4学时)采取实验总结交流会的形式,学生总结并汇报实验结果(见图 2),教师组织学生开展讨论交流。学生分享经验教训,锻炼表达和答辩能力;学生之间相互讨论学习,拓宽思路;师生共同解决实验过程和实验结果解析的疑问,使学生进一步巩固理论知识和实验技能。
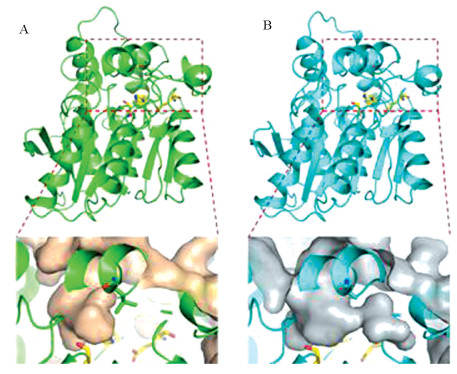
|
图 2 两种酶的三维结构模型和底物结合口袋对比图(实验总结交流会上学生展示结果) Figure 2 Comparison of three-dimensional structure model and substrate-binding pocket of two enzymes |
学生于课后独立撰写并提交研究报告,研究报告格式要求参照一般科研论文,需包括题目、摘要、前言、方法、结果、讨论和参考文献等。
3.5 考核评价方式课程考核评价不仅仅考虑实验结果,而是采取多元化的过程考核评价方法[23],考核内容包括对实验方案设计、实验过程表现、实验内容掌握、总结讨论表现、研究报告质量的多方面评价(见表 1)。该评价体系的建立,有助于提升和评价学生实验态度和兴趣、专业知识基础、实验设计能力、实验操作能力、表达展示能力、分析解决问题能力等多方面综合素质。
| 表 1 综合实验考核评价体系 Table 1 Comprehensive practice assessment system |
综合性实验的设计和实施改善了以往生物信息学实验抽象化、任务化和片段化的问题,以实际科学问题为导向,将多种学科背景、多个知识点、多类分析技术交叉融合为围绕科学问题开展的综合实验。实验充分发挥学生的主观能动性,使学生占据教学中心,通过学生自主的文献阅读、独立思考、实验设计、探索研究、互动讨论、归纳总结等环节,使学生系统地掌握从实验设计、数据分析到报告撰写的全过程,体验和学习科学研究的思路、过程和方法,激发学生的学习积极性,强化学生对专业知识的理解和掌握,扩展学生视野和实验教学内容,提升学生生物信息学实验操作技能,锻炼学生通过独立思考提出问题、分析问题和解决问题的能力,提高学生综合素质和创新能力。
本年度由于受新冠肺炎疫情的影响,响应教育部对高校在疫情防控期间“停课不停学”的要求,教学团队积极开展线上教学工作,利用生物信息学在计算机上开展实验的优势,利用“学在浙大”和“浙大钉”等平台,构建直播课程,实现线上教、学、测、评的全流程教学活动,保证了课程的顺利开展,也积累了宝贵的线上教学经验。后续教学团队将积极探索构建线上课程,共享线上教学资源,同时也促进学生自主学习,推动线上线下混合式课程建设。
| [1] |
陈铭. 生物信息学[M]. 第三版. 北京: 科学出版社, 2018. CHEN Ming. Bioinformatics[M]. 3rd ed. Beijing: Science Press, 2018. (  0) 0) |
| [2] |
刘艳, 朱瑞新, 钱洁, 等. 数字化生物信息实践教学平台的构建和实践[J]. 实验室研究与探索, 2014, 33(5): 209-212. LIU Yan, ZHU Ruixin, QIAN Jie, et al. Construction and practice of a digital bioinformatics practical teaching platform[J]. Research and Exploration in Laboratory, 2014, 33(5): 209-212. DOI:10.3969/j.issn.1006-7167.2014.05.052 (  0) 0) |
| [3] |
宋小飞, 刘惟飞, 谭锦才, 等. 问题导向学习法在环境分析技术实践教学中的应用[J]. 实验技术与管理, 2019, 36(7): 222-224. SONG Xiaofei, LIU Weifei, TAN Jincai, et al. Application of problem-oriented learning method in practical teaching of environmental analysis technology[J]. Experimental Technology and Management, 2019, 36(7): 222-224. DOI:10.16791/j.cnki.sjg.2019.07.054 (  0) 0) |
| [4] |
魏哲铭, 卢荣. 问题导向教学在大学基础教学中的应用[J]. 高等理科教育, 2007, 3: 116-118. WEI Zheming, LU Rong. Application of problem-based teaching in basic university education[J]. Higher Education of Sciences, 2007, 3: 116-118. (  0) 0) |
| [5] |
吴根福, 吴科杰, 高海春. 优化设计整合资源提高实验教学质量[J]. 实验室研究与探索, 2016, 35(8): 203-206. WU Genfu, WU Kejie, GAO Haichun. Improving experimental education quality by optimizing experimental design and integration of resources[J]. Research and Exploration in Laboratory, 2016, 35(8): 203-206. DOI:10.3969/j.issn.1006-7167.2016.08.051 (  0) 0) |
| [6] |
HUO Yingyi, JIAN Shuling, CHENG Hong, et al. Two novel deep-sea sediment metagenome-derived esterases:Residue 199 is the determinant of substrate specificity and preference[J]. Microbial Cell Factories, 2018, 17(1): 16. DOI:10.1186/s12934-018-0864-4 (  0) 0) |
| [7] |
ZHU Wenhan, LOMSADZE A, BORODOVSKY M. Ab initio gene identification in metagenomic sequences[J]. Nucleic Acids Research, 2010, 38(12): e132. DOI:10.1093/nar/gkq275 (  0) 0) |
| [8] |
COCK P, CHILTON J, GRVNING B, et al. NCBI BLAST+integrated into Galaxy[J]. GigaScience, 2015, 4(1): 39. DOI:10.1186/s13742-015-0080-7 (  0) 0) |
| [9] |
HUERTA-CEPAS J, SZKLARCZYK D, HELLER D, et al. eggNOG 5.0:A hierarchical, functionally and phylogenetically annotated orthology resource based on 5090 organisms and 2502 viruses[J]. Nucleic Acids Research, 2019, 47(D1): D309-D314. DOI:10.1093/nar/gky1085 (  0) 0) |
| [10] |
HUERTA-CEPAS J, FORSLUND K, COELHO L P, et al. Fast genome-wide functional annotation through orthology assignment by eggnog-mapper[J]. Molecular Biology & Evolution, 2017, 34(8): 2115-2122. DOI:10.1093/molbev/msx148 (  0) 0) |
| [11] |
LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948. DOI:10.1093/bioinformatics/btm404 (  0) 0) |
| [12] |
KUMAR S, STECHER G, LI M, et al. MEGA X:Molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology & Evolution, 2018, 35(6): 1547-1549. DOI:10.1093/molbev/msy096 (  0) 0) |
| [13] |
GASTEIGER E, HOOGLAND C, GATTIKER A, et al.Protein identification and analysis tools on the ExPASy server[M]//.WALKER J M.The Proteomics Protocols Handbook, Totowa, NJ: Humana Press, 2005, 571-607.
(  0) 0) |
| [14] |
ALMAGRO A J, TSIRIGOS K D, SONDERBY C K, et al. SignalP 5.0 improves signal peptide predictions using deep neural networks[J]. Nature Biotechnology, 2019, 37(4): 420-423. DOI:10.1038/s41587-019-0036-z (  0) 0) |
| [15] |
YACHDAV G, KLOPPMANN E, KAJAN L, et al. PredictProtein-an open resource for online prediction of protein structural and functional features[J]. Nucleic Acids Research, 2014, 42(W1): W337-W343. DOI:10.1093/nar/gku366 (  0) 0) |
| [16] |
BUCHAN D, JONES D T. The PSIPRED protein analysis workbench:20 years on[J]. Nucleic Acids Research, 2019, 47(W1): W402-W407. DOI:10.1093/nar/gkz297 (  0) 0) |
| [17] |
GEOURJON C, DELÉAGE G. SOPMA:Significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J]. Computer Applications in the Biosciences, 1995, 11(6): 681-684. DOI:10.1093/bioinformatics/11.6.681 (  0) 0) |
| [18] |
MITCHELL A L, ATTWOOD T K, BABBITT P C, et al. InterPro in 2019:Improving coverage, classification and access to protein sequence annotations[J]. Nucleic Acids Research, 2019, 47(D1): D351-D360. DOI:10.1093/nar/gky1100 (  0) 0) |
| [19] |
WATERHOUSE A, BERTONI M, BIENERT S, et al. SWISS-MODEL:Homology modelling of protein structures and complexes[J]. Nucleic Acids Research, 2018, 46(W1): W296-W303. DOI:10.1093/nar/gky427 (  0) 0) |
| [20] |
BURLEY S K, BERMAN H M, BHIKADIYA C, et al. RCSB Protein Data Bank:Biological macromolecular structures enabling research and education in fundamental biology, biomedicine, biotechnology and energy[J]. Nucleic Acids Research, 2019, 47(D1): D464-D474. DOI:10.1093/nar/gky1004 (  0) 0) |
| [21] |
HOLM L. Benchmarking fold detection by DaliLite v.5[J]. Bioinformatics, 2019, 35(24): 5326-5327. DOI:10.1093/bioinformatics/btz536 (  0) 0) |
| [22] |
TROTT O, OLSON A J. AutoDock Vina:Improving the speed and accuracy of docking with a new scoring function, efficient optimization, and multithreading[J]. Journal of Computational Chemistry, 2010, 31(2): 455-461. DOI:10.1002/jcc.21334 (  0) 0) |
| [23] |
张琇, 倪志婧, 王薇, 等. 综合研究性实验项目的教学设计与过程管理[J]. 实验技术与管理, 2012, 29(2): 159-161. ZHANG Xiu, NI Zhijing, WANG Wei, et al. Instructional design and process management of comprehensive research-type experiment in microbiology course[J]. Experimental Technology and Management, 2012, 29(2): 159-161. DOI:10.3969/j.issn.1002-4956.2012.02.049 (  0) 0) |
 2020, Vol. 18
2020, Vol. 18
