革兰氏阳性细菌是指在革兰氏染色剂染色后仍保持原来的蓝紫色结晶的细菌,它广泛存在于人体中,对人体的健康危害极大,因此,研究革兰氏阳性细菌的蛋白质亚细胞定位具有重要的生物学和医学意义。到目前为止革兰氏阳性菌蛋白亚细胞定位的研究相对较少,2007年Shen和Chou[1]建立了一个分为五类的革兰氏阳性菌数据集,利用Gene Ontology-Pseudo Amino Acid离散模型和Optimized evidence theoretic-K-nearest neighbors方法,然后通过Jackknife测试和独立数据集测试,成功率分别为82.7%和84.1%。2009年Shen和Chou[2]重新建立了四类革兰氏阳性菌亚细胞定位数据集,结合基因本体等特征信息,最后总的成功率达到了82.2%。2012年Hu等人[3]构建了GP25革兰氏阳性菌亚细胞定位数据集,利用Localization motif算法对革兰氏阳性菌蛋白质亚细胞定位进行预测,总成功率达到了85.9%。2016年在第九届电气与计算机工程国际会议上,Julia Rahman等人[4]分别利用Amino Acid Composition和Pseudo Amino Acid Composition特征信息与Physiochemical Property Model特征信息融合对革兰氏阳性菌蛋白质亚细胞定位进行预测,预测成功率均为73.2%。2017年XiaoXuan等人[5]采用新的算法对四类革兰氏阳性菌亚细胞定位进行预测,取得了较好的预测结果。在这些预测研究中,提取的特征信息没有考虑蛋白质的结构域(Domain)特征,结构域通常是由50~400个氨基酸残基构成的球状实体,它在蛋白质中可以独立稳定的存在,并且和完成蛋白质功能有着密切的关系[6]。本文基于Swiss-Prot数据库构建了4类共700条革兰氏阳性细菌蛋白质的数据集,对其结构域进行搜索和功能分析,找到了细胞壁区域蛋白质的结构域、细胞外区域蛋白质的结构域、细胞质区域蛋白质的结构域以及细胞膜区域蛋白质的结构域。在PDBsum数据库中进一步搜索找到了部分结构域所对应的二级和三级结构图,观察这些结构图找到结构域的特征,这些结构域的特征有助于革兰氏阳性细菌蛋白质亚细胞定位预测,可以进一步了解革兰氏阳性细菌的发病原理,有望对相应的疾病进行针对性治疗。
1 数据集数据集严格按照以下标准构建:(1)从UniProtKB/Swiss-Prot (2018-09)数据库中检索关键字“Firmicutes”和“Actinobacteria”的所有蛋白序列;(2)在“Subcellular Location”注释中选择该蛋白的不同位置,去除“By similarity”、“Probably”等含糊或不确定的术语;(3)在“Sequence”信息中选择50-3 000 aa蛋白序列;(4)剔除片段“B”、“X”、“Z”注释序列;(5)为避免同源性偏差,采用软件CD-HIT对蛋白序列进行相似比对,序列相似性为25%。完成上述步骤后,得到700条革兰氏阳性菌蛋白质序列,它们分成4个区域分别是:细胞壁(Cell wall)22条、细胞外(Extracell)214条、细胞质(Cytoplasm)252条、细胞膜(Cell membrane)212条。
在Swiss-Prot数据集中的Family & Domains条目下提取每条革兰氏阳性细菌蛋白质的结构域信息,最后发现细胞壁区域含有结构域信息的革兰氏阳性细菌蛋白质有10条,细胞外区域有67条蛋白质有结构域信息,在细胞质区域中有78条,而细胞膜区域则有61条。进一步在PDBsum数据库找到了这些结构域所对应的二级和三级结构图。表 1~表 4分别为革兰氏阳性菌4个位置蛋白质所对应的结构域的名称和数量,其中小于2条结构域的蛋白质区域没有列出。
| 表 1 细胞壁蛋白质结构域个数 Table 1 Number of cell wall protein domain |
| 表 2 细胞外蛋白质结构域个数 Table 2 Number of extracellular protein domain |
| 表 3 细胞质蛋白质结构域个数 Table 3 Number of cytoplasm protein domain |
| 表 4 细胞膜蛋白质结构域个数 Table 4 Number of cell membrane protein domain |
由于细胞壁的蛋白质数量只有22条,所以细胞壁蛋白质的结构域数量也不是很多,其中出现次数不小于2次的结构域只有PE结构域和NEAT结构域,在这里分别介绍下这两个结构域。PE结构域是高度保守的N端亲水性结构域[7]。PE结构域是对蛋白质的翻译和定位起着重要作用的功能结构域,PE结构域大约有110个氨基酸残基。在细胞壁区域中一共有5条蛋白质含有PE结构域,它们都属于PE家族。在PDBsum库中没有找到PE结构域相对应的二级结构图和三级结构图。
NEAT结构域是调控铁元素表面蛋白的特异性保守结构域,大约由125个氨基酸残基构成,它的二级结构由1个螺旋束和C端的β发夹组成,其中包含9个平行的β折叠片。革兰氏阳性细菌中摄取血红素的最佳系统是lsd系统,在lsd系统中细胞壁作为受体将与血红素结合,在细胞壁中发挥主要作用的就是高度保守的NEAT结构域[8]。图 1给出的是NEAT结构域的二级结构图和三级结构图。
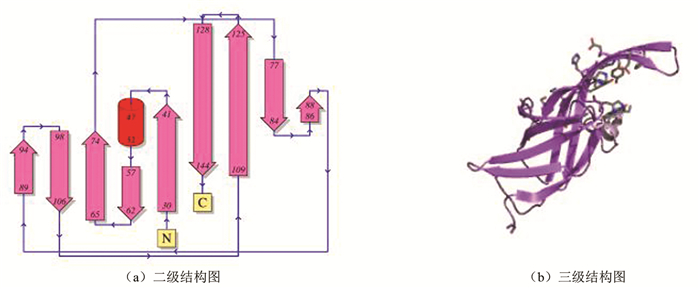
|
图 1 NEAT结构域对应的结构图 Figure 1 Diagarm of NEAT domain |
从表 2可以看到细胞外区域出现次数不少于5次的结构域有PINc结构域、N-acetylmuramoyl-L-alanine amidas结构域、PE结构域和Peptidase S8结构域。其中PE结构域是细胞壁和细胞膜共有的结构域。在这里我们主要介绍出现次数最多的PINc结构域,PINc结构域属于PIN家族,它具有高度的保守性,通常由150个氨基酸残基构成。在真核生物中,PINc结构域参与mRNA衰变的过程,而且具有核糖核酸酶的功能。在原核生物中,PINc结构域是抗毒素(TA)系统的毒性成分,它们的毒性是由于核糖核酸酶活性产生的。PINc结构域蛋白的活性位点由四种保守的酸性氨基酸组成,这些氨基酸参与二价金属离子结合和酶的活性过程[9]。在PDBsum库中并没有找到PINc结构域相对应的二级结构图和三级结构图。
2.3 细胞质区域的结构域Response regulatory结构域、Histidine kinase结构域和S1 motif结构域是出现在细胞质区域不少于5次的结构域。由于在PDBsum库中没有Response regulatory结构域的二级结构图和三级结构图,在这里主要介绍Histidine kinase结构域和S1 motif结构域。Histidine kinase结构域属于HWE家族,Histidine kinase结构域是由二聚组氨酸磷酸受体子域(DPH)和与催化ATP结合的子域(CA)构成,其中DPH是由两个螺旋束和一个发夹环连接,形成了4个螺旋体束,而CA是一个高度保守的αβ三明治结构,由3个α螺旋和5个β链构成。大多数原核生物信号转导系统和少数真核细胞转导途径都和Histidine kinase结构域有关。在革兰氏阳性细菌的细胞质当中,Histidine kinase结构域起到了同源反应调节剂的作用[10]。在图 2中给出Histidine kinase结构域的二级结构图和三级结构图。
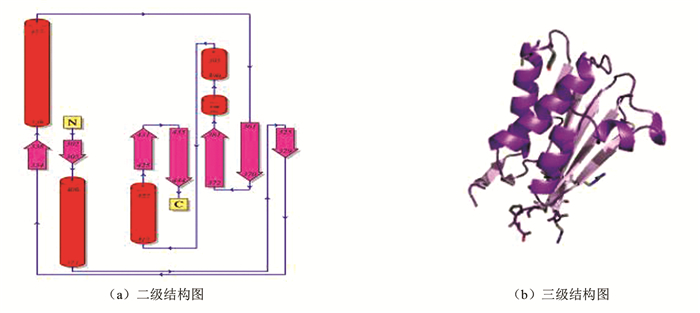
|
图 2 Histidine kinase结构域对应的结构图 Figure 2 Diagarm of Histidine kinase domain |
S1 motif结构域大量存在于RNA结合蛋白质中,该结构域使用特殊的方式和RNA结合,在细胞质区域当中和mRNA、核糖体相互作用,它对蛋白质的翻译起到重要的作用。S1 motif结构域和shock蛋白的结构相似,S1 motif结构域一般由70~90个氨基酸残基构成,由5个反向平行β的折叠片构成[11]。图 3是S1 motif结构域在PDBsum数据库的二级结构图和三级结构图。
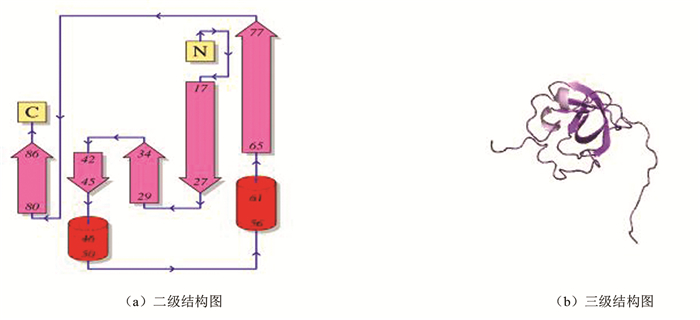
|
图 3 S1 motif结构域对应的结构图 Figure 3 Diagarm of S1 motif domain |
细胞膜区域出现的次数不小于5次的结构域有ABC transmembrane type-1结构域、Histidine kinase结构域、HAMP结构域,Protein kinase结构域和ABC transporter结构域。其中Histidine kinase结构域是细胞膜和细胞质区域共有的结构域,在细胞质区域的结构域中已经介绍过Histidine kinase结构域,在这主要介绍HAMP结构域和Protein kinase结构域。HAMP结构域是原核生物中跨核膜受体中最后一个没有被人类发现结构但已经被人广泛熟知存在的结构域。原核生物通过跨膜受体来感应环境的变化,其胞内和胞外通常由HAMP结构域连接,而且连接点始终位于HAMP结构域的C端,根据以上特征人们猜想HAMP结构域的结构是由两个具有卷曲线圈特性的螺旋体构成[12]。
从革兰氏阳性菌到人类,Protein kinase结构域的功能在进化上是高度保守的。Protein kinase结构域是由6个β发夹,7个β凸起和13个螺旋体构成的。Protein kinase结构域在多种细胞过程中发挥作用,包括细胞的分裂、增殖、凋亡和分化。在真核生物中Protein kinase结构域蛋白酶是一类非常广泛的蛋白家族中的酶,与丝氨酸、苏氨酸和酪氨酸蛋白酶一样都具有保守的催化功能。Protein kinase结构域蛋白酶催化ATP转移到蛋白底物侧链上的一个或多个氨基酸残基,导致其结构改变从而影响蛋白功能[13]。图 4给出的是Protein kinase结构域在PDBsum数据库的二级结构图和三级结构图。
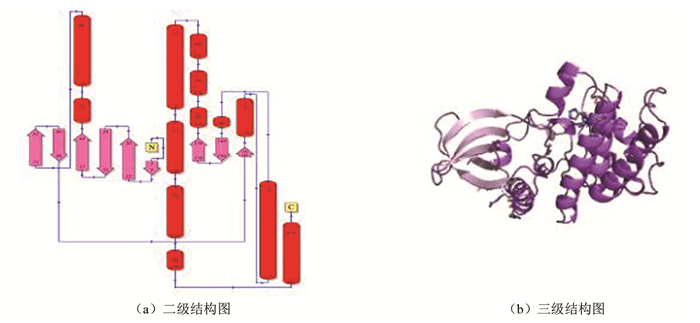
|
图 4 Protein kinase结构域对应的结构图 Figure 4 Diagarm of Protein kinase domain |
对革兰氏阳性细菌蛋白质4个亚细胞位置上的结构域种类和个数分别进行了统计,发现PE结构域是革兰氏阳性菌细胞壁和细胞外两个区域共同含有的结构域,其对蛋白质的翻译起着至关重要的作用。而Histidine kinase结构域是细胞质区域和细胞膜区域共同含有的结构域,Histidine kinase结构域在同源中反应起到了调节剂的作用。NEAT结构域是细胞壁区域独有的结构域,它的主要作用作为细胞壁受体与血红素结合。PINc结构域是细胞外区域独有的结构域,PINc结构域参与核糖体RNA的过程同时也具有核糖核酸酶的功能。细胞质区域独有的结构域是S1 motif结构域,它对蛋白质的翻译起着重要的作用。Protein kinase结构域是细胞膜区域特有的结构域,它影响着蛋白质的功能。随着数据库的不断完善,会找到更多的结构域特征,利用这些特征信息可以更深入的了解革兰氏阳性菌蛋白质的结构和功能。
| [1] |
SHEN Hongbin, CHOU Kuochen. Gpos-PLoc: An ensemble classifier for predicting subcellular localization of Gram-positive bacterial proteins[J]. Protein Engineering Design and Selection, 2007, 20(1): 39-46. DOI:10.1093/protein/gzl053 (  0) 0) |
| [2] |
SHEN Hongbin, CHOU Kuochen. Gpos-mPLoc: A top-down approach to improve the quality of predicting subcellular localization of gram-positive bacterial proteins[J]. Protein & Peptide Letters, 2009, 16(12): 1478-1484. DOI:10.2174/092986609789839322 (  0) 0) |
| [3] |
HU Yinxia, LI Tonghua, SUN Jiangming, et al. Predicting Gram-positive bacterial protein subcellular localization based on localization motifs[J]. Journal of Theoretical Biology, 2012, 308: 135-140. DOI:10.1016/j.jtbi.2012.05.031 (  0) 0) |
| [4] |
RAHMAN J, MONDAL M N I, ISLAM M K B, et al. Gram-positive bacterial protein subcellular localization prediction using features fusion strategy[C]. International Conference on Electrical & Computer Engineering, IEEE, 2017. DOI: 10.1109/ICECE.2016.7853913.
(  0) 0) |
| [5] |
XIAO Xuan, CHENG Xiang, SU Shengchao, et al. pLoc-mGpos: Incorporate key gene ontology information into general PseAAC for predicting subcellular localization of gram-positive bacterial proteins[J]. Natural Science, 2017, 9: 330-349. DOI:10.4236/ns.2017.99032 (  0) 0) |
| [6] |
王文娟, 李凤敏. 核定位蛋白的结构域特征分析[J]. 内蒙古大学学报:自然科学版, 2018, 49(1): 70-76. WANG Wenjuan, LI Fengmin. Analysis of nucleoprotein domain feature[J]. Journal of Inner Mongolia University (Natural Science Edition), 2018, 49(1): 70-76. DOI:10.13484/j.nmgdxxbzk.20180112 (  0) 0) |
| [7] |
CASCIOFERRO A, DELOGU G, COLONE M, et al. PE is a functional domain responsible for protein translocation and localization on mycobacterial cell wall[J]. Molecular Microbiology, 2008, 66(6): 1536-1547. DOI:10.1111/j.1365-2958.2007.06023.x (  0) 0) |
| [8] |
CHOO J M, CHEUNG J K, WISNIEWSKI J A, et al. The NEAT domain-containing proteins of clostridium perfringens bind heme[J]. PLoS One, 2016, 11(9): e0162981. DOI:10.1371/journal.pone.0162981 (  0) 0) |
| [9] |
BLEICHERT F, GRANNEMAN S, OSHEIM Y N, et al. The PINc domain protein Utp24, a putative nuclease, is required for the early cleavage steps in 18S rRNA maturation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(25): 9464-9469. DOI:10.1073/pnas.0603673103 (  0) 0) |
| [10] |
RINALDI J, ARRAR M, SYCZ G, et al. Structural insights into the HWE histidine kinase family: The Brucella blue light-activated histidine kinase domain[J]. Journal of Molecular Biology, 2016, 428(6): 1165-1179. DOI:10.1016/j.jmb.2016.01.026 (  0) 0) |
| [11] |
BYCROFT M, HUBBARD T J P, PROCTOR M, et al. The solution structure of the S1 RNA binding domain: A member of an ancient nucleic acid-binding fold[J]. Cell, 1997, 88(2): 235-242. DOI:10.1016/S0092-8674(00)81844-9 (  0) 0) |
| [12] |
FLACK C E, PARKINSON J S. A zipped-helix cap potentiates HAMP domain control of chemoreceptor signaling[J]. Proceedings of the National Academy of Sciences, 2018, 115(15): 201721554. DOI:10.1073/pnas.1721554115 (  0) 0) |
| [13] |
DEBANT A, CARLES SERRA-PAGÈS, SEIPEL K, et al. The multidomain protein trio binds the lar transmembrane tyrosine phosphatase, contains a protein kinase domain, and has separate rac-specific and rho-specific guanine nucleotide exchange factor domains[J]. Proceedings of the National Academy of Sciences of the United States of America, 1996, 93(11): 5466-5471. DOI:10.2307/39455 (  0) 0) |
 2020, Vol. 18
2020, Vol. 18


