罗汉果(Siraitia grosvenorii)属葫芦科藤本落叶植物,具有一年种多年生的块根状宿根性,其药食两用的果实被誉为“东方神果”,全球90%以上的罗汉果产于广西桂林地区[1-2]。根结线虫病是一类危害大、分布广、防治难的植物病虫害,也是罗汉果生产中的主要病害之一。根结线虫不仅危害罗汉果植株根部,破坏根组织的分化和生理活动,导致地上部分生长发育迟缓、推迟开花结果或不开花结果,同时还会加重枯萎病、根腐病等土传性真菌病害和部分细菌病害的发生,有的果园未到结果盛期就因严重感染根结线虫病而被迫淘汰,损失极为严重[3-4]。因此,如何防治根结线虫病是目前罗汉果种植中迫于要解决的主要问题之一,而利用生物技术进行抗性育种被认为是在植物生物防治中最有前景的策略[5]。
几丁质酶(EC 3. 2. 1. 14)是一种能够将几丁质水解成N-乙酰葡萄糖胺的糖苷酶[6]。因真菌细胞壁均含有几丁质,线虫的卵壳和表皮中也含有几丁质物质,几丁质酶作为病原物和寄主植物相互作用中的一种重要蛋白质,已成为在植物抗病虫害研究中的热点[7]。植物虽然不含几丁质,但也存在着几丁质酶基因,植物几丁质酶可分布于植物的茎、叶、种子及愈伤组织中,分子量为25~55 kDa,多为单体,呈酸性或碱性,主要是内切几丁质酶,它随机水解几丁质内部β-1,4-糖苷键,释放出寡糖[8-12],植物几丁质酶部分家族成员还具有溶菌酶同源性[13-14],相关研究也表明植物几丁质酶的高水平表达能够增强植物对害虫、病原菌和环境胁迫的抵抗能力[10-12, 15-16]。
随着高通量测序技术和生物信息学技术的发展,转录组测序技术(RNA sequencing,RNA-Seq)目前被广泛应用于植物抗病性等相关基因的鉴定与分析[16-20],本研究基于RNA-Seq技术获得罗汉果根系转录组测序数据的基础上,对其中的几丁质酶基因家族成员进行生物信息学分析,期望为今后研究几丁质酶对罗汉果植株根结线虫病抗性分子机理提供参考,为罗汉果抗性育种提供基因来源。
1 材料与方法 1.1 材料罗汉果为当地栽培品种青皮果,属于南方根结线虫易感品种,以罗汉果幼苗根系为植物材料进行转录组测序,委托上海派森诺生物科技有限公司Illumina HiSeq 2500平台进行。
1.2 方法 1.2.1 罗汉果几丁质酶基因鉴定及理化特性分析基于罗汉果幼苗根系的转录组测序数据及其注释结果,将搜索到的注释为几丁质酶的Unigene通过开放阅读框ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder)和预测保守域CD Search(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)在线软件检测确认,筛选出含GH18或GH19基因家族成员结构域特征且具有完整ORF的Unigene序列。将鉴定到的罗汉果几丁质酶基因编码的氨基酸序列使用ExPAsy (http//www.expasy.org/)进行在线预测其理化特性和跨膜结构域预测(http://www.cbs.dtu.dk/services/TMHMM)。
1.2.2 信号肽预测及亚细胞定位利用TargetP[21] (http://www.cbs.dtu.dk/services/TargetP/)和在线软件CELLO v2.5[22]对15条罗汉果几丁质酶序列进行信号肽预测和亚细胞定位。
1.2.3 罗汉果几丁质酶系统进化树构建及氨基酸序列结构分析基于几丁质酶的氨基酸序列,采用MEGA6.0邻接法(Neighbor-Joining,NJ)缺失序列删除的方式,构建罗汉果几丁质酶系统进化树(bootstrap=1 000)。并以NCBI数据库(https://www.ncbi.nlm.nih.gov/)下载的19条拟南芥几丁质酶序列(本研究中将其编号AtCht1到AtCht19,序列相关信息见表 1)为参考,对罗汉果几丁质酶进行分组。利用DNAMAN软件、MEME和Phyre2[23]在线工具对罗汉果几丁质酶基因编码的氨基酸序列结构域进行分析。
| 表 1 拟南芥几丁质酶序列信息 Table 1 Chitinases in Arabidopsis thaliana downloaded from NCBI |
基于南方根结线虫侵染早期罗汉果根系转录组测序结果,搜索到注释为几丁质酶基因的Unigene共有28条,然后分别经ORF finder和CD-Search确认具有完整开放阅读框ORF和GH18或GH19基因家族成员结构域特征的Unigene,最后共鉴定到15条罗汉果几丁质酶Unigene,命名为SgCht1到SgCht15(见表 2),ORF finder在线预测其编码的几丁质酶前体氨基酸数目最少的为SgCht6 (260个),最大的为SgCht12和SgCht15(均是343个)。利用ExPASy在线软件对罗汉果几丁质酶基因编码的蛋白质进行理化性质预测结果见表 2,罗汉果几丁质酶的分子量在27 kDa到37 kDa之间,多数为酸性蛋白质,只有SgCht1、SgCht4和SgCht9的理论等电点大于8.0;不稳定系数显示SgCht1、2、4、6、7、10、11和14为稳定蛋白,其余为不稳定蛋白;总平均亲水性显示SgCht1、2、9、12、13为疏水性蛋白,其余均属于亲水性蛋白。
| 表 2 罗汉果几丁质酶特征 Table 2 Characteristics of chitinases in Siraitia grosvenorii |
信号肽预测结果显示,本研究鉴定到的15个罗汉果几丁质酶前体其N端均有一段长度范围为19-30个氨基酸残基的信号肽(见表 2),亚细胞定位结果显示这些几丁质酶均定位在胞外。
2.3 罗汉果几丁质酶系统发育分析及分类植物中的几丁质酶主要为糖苷水解酶18家族和19家族成员(GH18和GH19),根据氨基酸序列特征,植物的内切几丁质酶分为5类,GH18包含Ⅲ类和Ⅴ类植物几丁质酶,GH19包含Ⅰ、Ⅱ、Ⅳ类[13-14]。本研究以拟南芥几丁质酶的分类为参考,基于几丁质酶氨基酸序列的NJ系统发育树结果如图 1所示,15条罗汉果几丁质酶序列(图中星号所示)归为3类即SgCht11属于Ⅰ类;SgCht6、SgCht7和SgCht10属于Ⅳ类,其余为Ⅲ类。
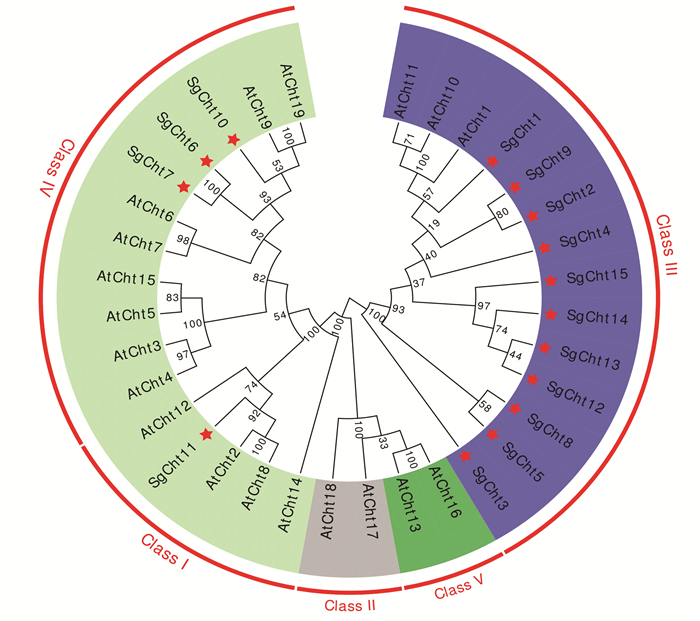
|
图 1 罗汉果与拟南芥几丁质酶的系统发育树 Figure 1 Phylogenetic tree of chitinases from Siraitia grosvenorii and Arabidopsis thaliana |
DNAMAN9多序列比对结果显示,Ⅰ类和Ⅳ类具有两个高度保守区,这两个保守区由一段富含脯氨酸、甘氨酸和精氨酸的可变交联区(见图 2,虚线下划线所示)相隔开,N端高度保守区富含半胱氨酸(见图 2,虚线框所示),其作用是与几丁质结合,简称CBD(Chitin-binding domain);C端是高度保守的催化区,有三个活性位点(见图 2实线方框所示)。Ⅳ类几丁质酶(SgCht6、SgCht7和SgCht10)与Ⅰ类(SgCht11)相比有4处缺失(见图 2),氨基酸数目(见表 1)明显比Ⅰ类少。

|
图 2 GH19基因家族成员保守结构域氨基酸序列比对 Figure 2 Alignment of conserved regions of GH19 amino acid sequences |
罗汉果GH18家族成员全部是Ⅲ类几丁质酶,它们不含与几丁质结合的富半胱氨酸功能域,而且仅有一个催化结构域,根据活性中心是否是谷氨酸残基分为几丁质酶和类几丁质酶(见图 3,虚线框所示),SgCht1、2、4、8、9、12和SgCht15均有一个谷氨酸活性中心,SgCht5、SgCht13缺失了谷氨酸活性中心,而SgCht3和SgCht14的活性中性则分别是天冬氨酸和丙氨酸残基。
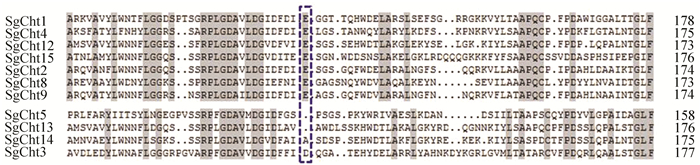
|
图 3 GH18基因家族成员保守结构域氨基酸序列比对 Figure 3 Alignment of conserved regions of GH18 amino acid sequences |
利用MEME 5.0.4软件对15个罗汉果几丁质酶的氨基酸序列进行motif查找,设置查找motif数目为6个,其余参数为默认值,结果如图 4所示,GH18家族成员均具有motif1、motif2和motif4;而GH19家族成员仅有motif6。

|
图 4 罗汉果几丁质酶蛋白基序 Figure 4 Motifs of chitinase in Siraitia grosvenorii |
GH18基因家族成员编码的蛋白质三级结构具有典型的(α/β)8桶状结构,即外部为8个α螺旋域,内部为8个平行的β折叠域[24],而GH19家族成员仅有α螺旋结构域。图 5显示了罗汉果几丁质酶GH18基因家族成员SgCht12(见图 5a)和GH19家族成员SgCht6(见图 5b)由Phyer2软件预测的三级结构模型。其中,SgCht12以c6idnA结构为模板,有268个氨基酸残基与模板达到100 %的可信度;SgCht6以c4mckA结构为模板,有199个氨基酸残基与模板达到100%的可信度。
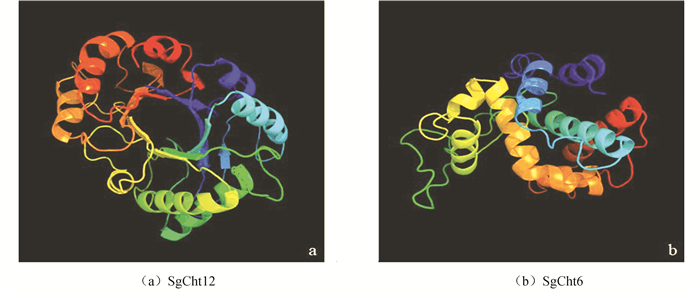
|
图 5 三级结构模型 Figure 5 Tertiary structure of SgCht12(a)and SgCht6(b) |
罗汉果为广西特有经济植物,但罗汉果植株易感根结线虫病[3-4],被根结线虫感染后的罗汉果植株更容易被其他病菌的侵害,其中原因未见报道。几丁质是N-乙酰葡聚胺以β-1,4-糖苷键连接起来的直链多聚物,是自然界中含量仅次于纤维素的一大类可再生聚合物,广泛存在于虾、蟹、昆虫、真菌等生物[25-26]。线虫卵壳及表皮的主要组成成分是几丁质,与真菌病原物一样,植物被线虫侵染时,可诱导产生几丁质酶[7]。几丁质酶作为病原物和寄主植物相互作用中的一种重要蛋白质,已成为在植物抗病虫害研究中的热点[5-7]。在植物抗病基因工程中,通过过量表达几丁质酶基因来提高植物抗病性相关研究已有报道[12, 15, 27-28]。郭悦等[12]将克隆到的花生几丁质酶基因AhChi进行遗传转化实验验证其生物学功能,结果表明转植株在黑斑病菌接种7d时其叶片褐化及坏死程度明显较非转基因植株要轻。谷天燕等[15]通过两端融合表达几丁质结合结构域的方法不但提高了几丁质酶对ɑ-几丁质和黑曲霉几丁质的结合能力和活性,同时也增强了其对病原真菌长枝木霉的抑制作用;南相日[27]的研究结果表明,转菜豆几丁质酶基因的马铃薯抗病性比对照提高了30%;王果萍等[28]也发现几丁质酶对镰刀菌引起的西瓜枯萎病有一定的抑制作用。随着对几丁质酶作用机理研究的深入[29-31],通过现代生物技术手段提高植物对真菌、昆虫、线虫等病害抗性将为有效生物防治提供新途径。本研究采用生物信息学方法从罗汉果根系转录组测序结果中鉴定到15个罗汉果几丁质酶基因,并对其编码的蛋白质进行了理化特性预测及系统发育分析,可为罗汉果几丁质酶基因生物学功能的鉴定提供理论基础,为罗汉果的抗病育种提供基因来源参考。本研究对转录组测序结果中的罗汉果几丁质酶基因的表达进行差异分析,结果发现接种根结线虫7 d,SgCht12基因和SgCht14出现显著性下调表达,分别是未接种根结线虫的0.17倍和0.21倍,这是否意味着已经被入侵的根结线虫建立了取食位点的罗汉果植株,很有可能由于几丁质酶的表达受到了抑制而导致对其他病菌的侵染更为敏感?这有待今后对罗汉果几丁质酶的生物学功能进行更深入的研究来进一步确定。
| [1] |
范建华, 梁敏, 刘布鸣. 基于双聚类的近10年罗汉果研究热点可视化分析[J]. 中国中医药信息杂志, 2017, 24(11): 91-95. FAN Jianhua, LIANG Min, LIU Buming. Visualization study on hot spots of research on Siraitiae fructus in recent ten years based on double clustering analysis[J]. Chinese Journal of Information on TCM, 2017, 24(11): 91-95. DOI:10.3969/j.issn.1005-5304.2017.11.022 (  0) 0) |
| [2] |
万艳娟, 吴军林, 吴清平. 功能性甜味剂罗汉果甜苷的生理功能及食品应用研究进展[J]. 食品与发酵科技, 2015, 51(5): 51-56. WAN Yanjuan, WU Junlin, WU Qingping. Advances of physiological functions and application of mogroside[J]. Food and Fermentation Sciences & Technology, 2015, 51(5): 51-56. DOI:10.3969/j.issn.1674-506X.2015.05-013 (  0) 0) |
| [3] |
刘纪霜, 黄金玲, 张正淳, 等. 罗汉果根结线虫病田间发病情况调查及病原鉴定[J]. 广东农业科学, 2011, 38(24): 66-68. LIU Jishuang, HUANG Jinling, ZHANG Zhengchun, et al. Investigation in different sites and identified of Siraitia grosvenorii root-knot nematode[J]. Guangdong Agricultural Sciences, 2011, 38(24): 66-68. DOI:10.3969/j.issn.1004-874X.2011.24.023 (  0) 0) |
| [4] |
尹庆璋, 梁秀荣, 游玉明, 等. 罗汉果根结线虫病的发生与防治[J]. 广西农学报, 2010, 25(5): 24-25. YIN Qingzhang, LIANG Xiurong, YOU Yuming, et al. Occurrence and control of root-knot nematode disease on momordica grosvenori swingle[J]. Journal of Guangxi Agriculture, 2010, 25(5): 24-25. DOI:10.3969/j.issn.1003-8779.2005.04.014 (  0) 0) |
| [5] |
巩凯玲, 陈双慧, 纪晓晨, 等.植物几丁质酶的研究进展[J/OL].分子植物育种, 2018, http://kns.cnki.net/kcms/detail/46.1068.S.20181121.1005.004.html. GONG Kailing, CHEN Shuanghui, JI Xiaochen, et al. The research progress of plant chitinases[J/OL]. Molecular Plant Breeding, 2018, http://kns.cnki.net/kcms/detail/46.1068.S.20181121.1005.004.html. (  0) 0) |
| [6] |
ASHISH M, PREET Y. Purification and properties of plant chitinases: A review[J]. Journal of Food Biochemistry, 2019, 43(3): e12762. DOI:10.1111/jfbc.12762 (  0) 0) |
| [7] |
吴海燕, 段玉玺. 几丁质酶与大豆抗胞囊线虫关系初步研究[J]. 植物病理学报, 2004, 344(6): 555-557. WU Haiyan, DUAN Yuxi. Interaction of chitinase isozyme and soybean resistant to Heterodera glyeines[J]. Acta Phytopathologica Sinica, 2004, 344(6): 555-557. DOI:10.3321/j.issn:0412-0914.2004.06.013 (  0) 0) |
| [8] |
贺哲, 王园秀, 张圣洁, 等. 猕猴桃几丁质酶基因克隆及生物信息学分析[J]. 江西农业大学学报(自然科学版), 2018, 40(6): 1241-1247. HE Zhe, WANG Yuanxiu, ZHANG Shengjie, et al. An analysis of cloning and bioinformatics of chitinase gene from kiwi fruit[J]. Acta Agriculturae Universitis Jiangxiensis(Natural Sciences Edition), 2018, 40(6): 1241-1247. DOI:10.13836/j.jjau.2018156 (  0) 0) |
| [9] |
LI Chen, LI Xiaoping, BAI Chengzhi, et al. A chitinase with antifungal activity from naked oat (Avena chinensis) seeds[J]. Journal of Food Biochemistry, 2019, 43(2): e12713. DOI:10.1111/jfbc.12713 (  0) 0) |
| [10] |
李伟阳, 曾斌, 李疆, 等. 梨几丁质酶基因克隆及其生物信息学分析[J]. 中国农学通报, 2015, 31(9): 184-193. LI Weiyang, ZENG Bin, LI Jiang, et al. Cloning of pear chitinase gene and bioinformatics analysis[J]. Chinese Agricultural Science Bulletin, 2015, 31(9): 184-193. DOI:10.11924/j.issn.1000-6850.casb14120083 (  0) 0) |
| [11] |
朱晨, 张舒婷, 常笑君, 等. 茶树几丁质酶基因的克隆及其在干旱胁迫下的表达分析[J]. 热带作物学报, 2017, 38(5): 894-905. ZHU Chen, ZHANG Shuting, CHANG Xiaojun, et al. Cloning and its expression analysis of chitinase under drought stress in Camellia sinensis[J]. Journal of Tropical Crops, 2017, 38(5): 894-605. DOI:10.3969/j.issn.1000-2561.2017.05.018 (  0) 0) |
| [12] |
郭悦, 姜平平, 潘雷雷, 等. 花生几丁质酶基因的克隆及抗病性验证[J]. 华北农学报, 2018, 33(6): 42-48. GUO Yue, JIANG Pingping, PAN Leilei, et al. Cloning and disease resistance verification of chitinase gene in peanut[J]. Acta Agriculturae Boreali-Sinica, 2018, 33(6): 42-48. DOI:10.7668/hbnxb.2018.06.006 (  0) 0) |
| [13] |
NEUHAUS J M, FRITIG B, LINTHORST H J M, et al. A revised nomenclature for chitinase genes[J]. Plant Molecular Biology Reporter, 1996, 141(2): 102-104. (  0) 0) |
| [14] |
UBHAYASEKERA W, RAWAT R, HO S W T, et al. The first crystal structures of a family 19 class Ⅳ chitinase: The enzyme from Norway spruce[J]. Plant Molecular Biology, 2009, 71(3): 277-289. DOI:10.1007/s11103-009-9523-9 (  0) 0) |
| [15] |
谷天燕, 刘晓楠, 李玲聪, 等. 融合表达几丁质结合结构域提高几丁质酶抗真菌活性[J]. 微生物学报, 2018, 58(12): 1-10. GU Tianyan, LIU Xiaonan, LI Lingcong, et al. Enhance antifungal activity of chitinase by fusion of chitin binding domain at both terminus[J]. Acta Microbiologica Sinica, 2018, 58(12): 1-10. DOI:10.13343/j.cnki.wsxb.20180418 (  0) 0) |
| [16] |
ZHAO Xiaobo, LI Chunjuan, YAN Caixia, et al. Transcriptome and proteome analyses of resistant preharvest peanut seedcoat in response to Aspergillus flavus infection[J]. Electronic Journal of Biotechnology, 2019, 39(5): 82-90. DOI:10.1016/j.ejbt.2019.03.003 (  0) 0) |
| [17] |
GOYER A, HAMLIN L, CROSSLIN J M, et al. RNA-Seq analysis of resistant and susceptible potato varieties during the early stages of potato virus Y infection[J]. BMC Genomics, 2015, 16(1): 472. DOI:10.1186/s12864-015-1666-2 (  0) 0) |
| [18] |
AN Jing, SHEN Xuefeng, MA Qibin, et al. Transcriptome profiling to discover putative genes associated with paraquat resistance in goosegrass (Eleusine indica L.)[J]. PloS One, 2014, 9(6): e99940. DOI:10.1371/journal.pone.0099940 (  0) 0) |
| [19] |
JAIN S, CHITTEM K, BRUEGGENMAN R, et al. Comparative transcriptome analysis of resistant and susceptible common bean genotypes in response to soybean cyst nematode infection[J]. PloS One, 2016, 11(7): e0159338. DOI:10.1371/journal.pone.0159338 (  0) 0) |
| [20] |
NEJAT N, CAHILL D, VADAMALAI G, et al. Transcriptomics-based analysis using RNA-Seq of the coconut (Cocos nucifera) leaf in response to yellow decline phytoplasma infection[J]. Molecular Genetics and Genomics, 2015, 290(5): 1899-1910. DOI:10.1007/s00438-015-1046-2 (  0) 0) |
| [21] |
EMANUELSSON O, NIELSEN H, BRUNAK S, et al. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence[J]. Journal of Molecular Biology, 2000, 300(4): 1005-1016. DOI:10.1006/jmbi.2000.3903 (  0) 0) |
| [22] |
YU Chinsheng, CHEN Yuching, LU Chihhao, et al. Prediction of protein subcellular localization[J]. Proteins, 2006, 64(3): 643-651. DOI:10.1002/prot.21018 (  0) 0) |
| [23] |
KELLY L A, MEZULIS S, YATES C M, et al. The Phyre2 web portal for protein modeling, prediction and analysis[J]. Nature Protocol, 2015, 10(6): 845-858. DOI:10.1038/nprot.2015.053 (  0) 0) |
| [24] |
HAI L, GREENE L H, HAIBING L. Sequence and structural analysis of the chitinase insertion domain reveals two conserved motifs involved in chitin-binding[J]. PLoS One, 2010, 5(1): e86542010. DOI:10.1371/journal.pone.0008654 (  0) 0) |
| [25] |
谢树章, 刘亚娟, 秦平伟, 等. 植物几丁质酶及应用研究进展[J]. 安徽农学通报, 2009, 15(8): 58-61. XIE Shuzhang, LIU Yajuan, QIN Pingwei, et al. Progress on research of plant chitinase and its application[J]. Anhui Agricultural Science Bulletin, 2009, 15(8): 58-61. DOI:10.3969/j.issn.1007-7731.2009.08.031 (  0) 0) |
| [26] |
赵和. 几丁质酶与植物抗病[J]. 河北农业科学, 2004, 8(2): 78-84. ZHAO He. Chitinases and plant protection[J]. Journal of Hebei Agricultural Sciences, 2004, 8(2): 78-84. DOI:10.3969/j.issn.1088-1631.2004.02.019 (  0) 0) |
| [27] |
南相日. 菜豆几丁质酶基因转化马铃薯及后代表达[J]. 中国农学通报, 2006, 22(2): 75-77. NAN Xiangri. Transformation and expression of chitinase in potato[J]. Chinese Agricultural Science Bulletin, 2006, 22(2): 75-77. DOI:10.3969/j.issn.1000-6850.2006.02.020 (  0) 0) |
| [28] |
王果萍, 王景雪, 孙毅, 等. 几丁质酶基因导入西瓜植株及其抗病性鉴定研究[J]. 植物遗传资源学报, 2003, 4(2): 104-109. WANG Guoping, WANG Jingxue, SUN Yi, et al. Transformation of watermelon plants with chitinase gene and evaluation for fusarium wilt resistance[J]. Journal of Plant Genetic Resources, 2003, 4(2): 104-109. DOI:10.3969/j.issn.1672-1810.2003.02.004 (  0) 0) |
| [29] |
王曦茁, 汪来发, 孟繁丽, 等. 淡紫拟青霉T-DNA插入突变体几丁质酶和蛋白酶活性及致病力的变化[J]. 四川农业大学学报, 2015, 34(1): 78-84. WANG Xizhuo, WANG Laifa, MENG Fanli, et al. Changes in protease and chitinase of T-DNA insertional mutants of Paecilomyces lilacinus and their pathogenicity against Meloidogyne incognita[J]. Journal of Sichuan Agricultural University, 2015, 34(1): 78-84. DOI:10.16036/j.issn.1000-2650.2016.01.014 (  0) 0) |
| [30] |
罗晶晶, 张仁英, 齐晓花, 等. 黄瓜几丁质酶基因克隆及与白粉病抗性关系的初步研究[J]. 分子植物育种, 2015, 13(7): 1584-1591. LUO Jingjing, ZHANG Renying, QI Xiaohua, et al. A preliminary study on cloning of chitinase gene and relationships to resistance of powdery mildew in cucumber (Cucumis Sativus L.)[J]. Molecular Plant Breeding, 2015, 13(7): 1584-1591. DOI:10.13271/j.mpb.013.001584 (  0) 0) |
| [31] |
李雪艳, 张涛, 杨红梅, 等. 棉花黄萎病拮抗细菌胞外酶检测及其酶活测定[J]. 新疆农业科学, 2018, 55(9): 1663-1673. LI Xueyan, ZHANG Tao, YANG Hongmei, et al. Detection of extracellular enzymes of antagonizing cotton verticillium wilt bacteria and determination of enzyme activity[J]. Xinjiang Agricultural Sciences, 2018, 55(9): 1663-1673. DOI:10.6048/j.issn.1001-4330.2018.09.012 (  0) 0) |
 2019, Vol. 17
2019, Vol. 17


