2018年全球癌症报告结果显示,结肠癌的发病率仍高居前五位。近几年来我国患上结肠癌的人数每年都在增加,结肠癌是我国常见的消化道肿瘤之一,具有发病率高、早期治愈率高的特点。尽管在众多研究人员与临床医生的共同努力下结肠癌的诊治技术及疗效已经有很大的提高,但是结肠癌的患者仍逐年增加[1]。目前,众多研究主要是针对结肠癌发病机制的众多研究仍不够深入,对疾病本身的发生、发展机制尚不够深入,因而没有一个高效的诊疗测序对结肠癌的发生进行有效的预防和预警。
迄今为止的miRNA和基因表达之间的关系研究主要集中在miRNA的3’非编码区域的负向调控基因的翻译过程。但随着miRNA作用机制研究的深入,有研究发现miRNA在细胞核内也存在通过增强子介导而激活基因表达的调控机制[2]。那么利用miRNA细胞核内调控机制,我们利用生物信息的多种分析方法对结肠癌的miRNA和基因进行补充性分析,从而为结肠癌发病的研究提供新的视角。
通过整合多个数据库的miRNA、基因表达谱数据,结肠特异的增强子数据,通知利用miRNA增强子预测算法对上述数据进行系统的整合分析,并肩miRNA-增强子-靶基因调控网络,并对网络进行拓扑学分析,最后通过分析网络中差异表达基因的功能,发现了多个与结肠癌发生密切相关的生物学功能。
1 材料与方法 1.1 miRNA表达数据从HMDD V3.0数据库[3](http://www.cuilab.cn/hmdd)筛选得到了被多篇文献证实结肠癌中差异表达的miRNA信息。
1.2 基因表达数据从CoRECG数据库[4](http://lms.snu.edu.in/corecg/)下载至少被一篇文献报道过在结肠癌样本中差异表达的基因数据。
1.3 增强子及其调控靶基因的关系数据从EnhancerAtlas数据库(http://enhanceratlas.org/download.php)下载了结肠特异的增强子和靶基因关系数据。
1.4 增强子区域的序列处理利用从EnhancerAtlas数据库[5]中得到的结肠特异的增强子区域信息,首先将位置信息转化为每个增强子基因组序列信息,即得到对应增强子的fastq格式的文件。
1.5 miRNA的增强子靶基因关系预测分析利用RNA biology文章中提供的miRNA与增强子靶向关系预测算法[2],计算并筛选miRNA靶向的增强子区域。
1.6 miRNA-增强子-差异表达靶基因网络构建及分析利用上述方法得到miRNA-增强子-靶基因关系网络,并结合结肠癌差异表达靶基因的信息,最终得到差异表达miRNA-结肠特异增强子-结肠癌差异表达靶基因网络。并使用Cytoscape软件对网路进行相关的网络参数分析。
1.7 miRNA激活调控差异表达基因的GO分析基因本体论(Gene Ontology,GO)[6]是一种常用的基因相关功能注释软件,该数据库为树状结构。该数据库主要包含生物学过程、细胞组分、分子功能三个主要的功能分支。我们将网络中的差异表达靶基因进行功能注释(P < 0.01,FDR < 0.05)。
2 结果分析 2.1 差异表达miRNA筛选将下载的数据进行筛选整理,最终得到的结肠癌差异表达miRNA共计24个,其中7个下调的miRNA,35个上调的miRNA.
2.2 差异表达基因筛选结果经过筛选分析得到在正常和结肠癌样本中差异表达基因1 551个,其中上调基因1 144个,下调基因407个。
2.3 增强子介导的miRNA向差异表达基因关系筛选结果将24个差异表达miRNA分别利用NamiRNA增强子预测分析算法在842个结肠特异性增强子区域进行靶向位点预测,共得到miRNA靶向增强子关系对76 274对。整合结肠癌差异表达基因数据,最终得到上调miRNA-上调靶基因关系对3 839个,下调miRNA-下调靶基因关系对1 042个。miRNA激活调控的差异表达基因共计249个,上调基因201个,下调基因48个。
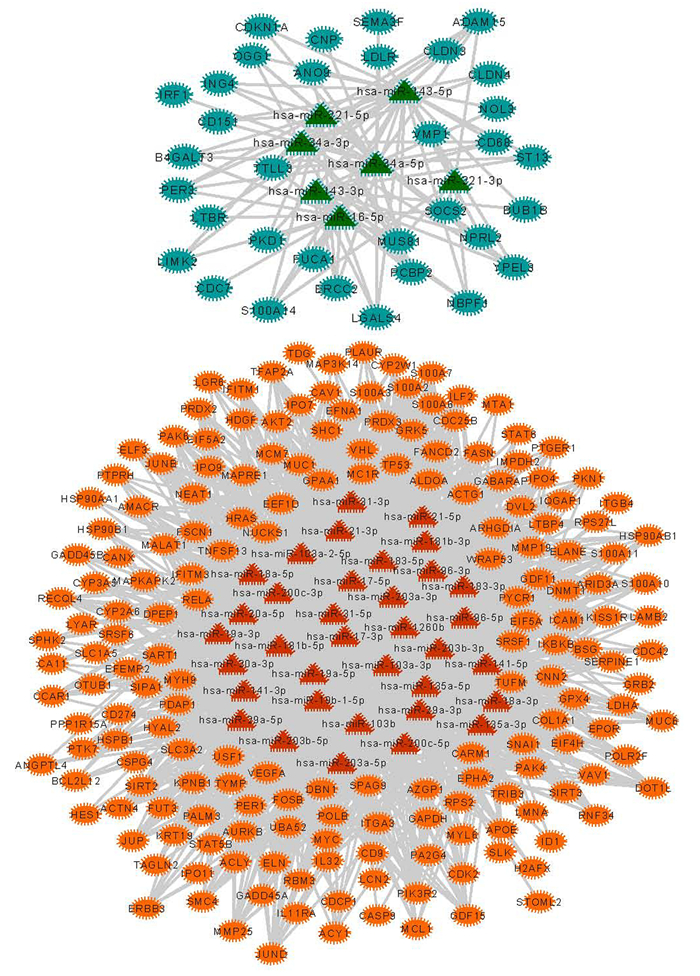
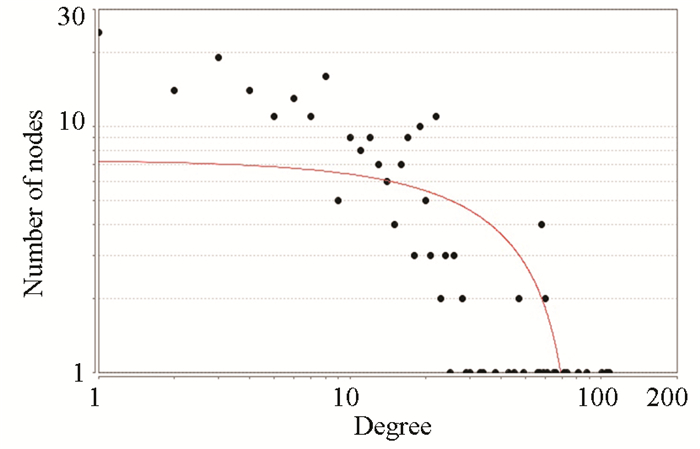
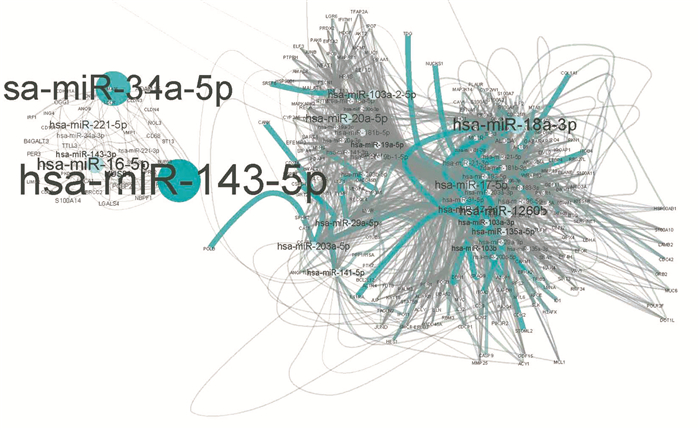
2.4 miRNA-增强子-差异表达靶基因网络构建及分析利用上述步骤建立的miRNA-增强子,增强子靶向的差异表达基因关系对,最终得到miRNA-靶基因关系对2 121个,259个节点,其中包含34个下调基因、183个上调的基因,7个下调的miRNA,35个上调的miRNA(见图 1)。网络中的节点的度分布如图 2所示,网络中的失调的miRNA可以通过细胞核内激活调控的方式同时调控217个基因的差异表达。其次,对网络中所有基因和miRNA的度进行分析,筛选度比较高的基因节点,认为该部分的基因是潜在的结肠癌诊疗靶点,如图 3所示。利用网络的度分析,筛选到TDG、NUCKS1、IL11RA、POLB、COL1A1,ITGB4等关键基因。
|
图 1 miRNA-激活调控靶基因网络 Figure 1 miRNA positive regulated genes network 注:图中的桔色圆点代表上调靶基因;红色三角形代表上调的miRNA;绿色三角形代表下调miRNA;绿色圆形代表下调靶基因.彩图见电子版http://swxxx.alljournals.cn/ch/index.aspx.2019年第2期. |
|
图 2 网络中度的分布图 Figure 2 Distribution of degree in the network |
|
图 3 网路度分布连通图 Figure 3 Network overview by node degree 注:网络是节点的大小显示该节点的度,网络中绿色边为网路中度较高节点的边. |
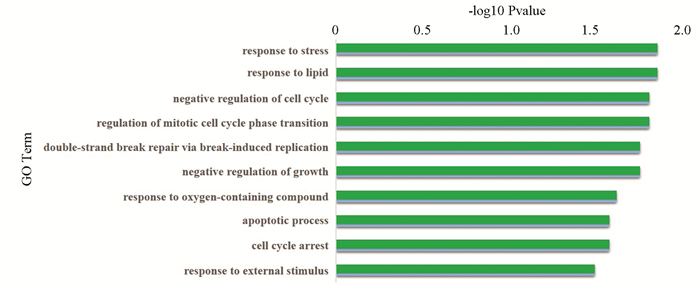
差异表达靶基因GO功能富集分析,结果显示,结肠癌中被下调miRNA正向调控的下调靶基因主要富集在应激反应[7]、对脂质体的反应、细胞周期负向调控、细胞生长的负调控、细胞凋亡[8]、双链断裂修复、对生物刺激的反应等生物学功能均被抑制因此出现细胞的异常增生和分裂的异常。功能中排在前十位被下调的功能如下图(见图 4)。
|
图 4 下调基因GO功能分析 Figure 4 GO analysis on down-regulated genes |
结肠癌中被上调miRNA正向调控的上调靶基因主要富集细胞对外界刺激的反应、对于应激反应和化学刺激的反应、细胞代谢和细胞过程和巨噬细胞代谢的调控等生物学过程[9-10],如图 5所示。

|
图 5 上调基因GO功能分析 Figure 5 GO analysis on up-regulated genes |
通过整合结肠癌差异表达的miRNA、结肠特异性增强子数据和结肠癌差异表达基因数据,同时,采用NamiRNA靶向增强子预测算法筛选miRNA和靶向增强子区域的靶向关系,从而构建结肠癌特异的miRNA-增强子-靶基因激活调控网络。该网络包含miRNA-靶基因关系对2 121个,259个节点,其中包含34个下调基因、183个上调的基因,7个下调的miRNA,35个上调的miRNA。而后我们分析了网络进行的节点度的整体分布情况,发现网络中大部分的节点的度都是小于10的,仅有少量miRNA结合和部分的差异表达基因节点的度大于10。
其次,利用网络中节点的度对网络中关键节点进行分析,发现除了筛选到的42个miRNA外,还有部分差异表达基因受到多个miRNA的调控,例如TDG、NUCKS1、IL11RA、POLB、COL1A1、ITGB4等关键基因。筛选的关键基因中如IL11RA于结肠癌中高表达,并且与结肠癌的临床病理因素密切相关[11]。TDG(胸腺嘧啶糖基化酶)等基因也被多篇文章报道可以作为结肠癌的标记。
通过分别对miRNA-增强子-靶基因网络中上调和下调靶基因的GO功能分析,发现结肠癌中激活表达的基因主要参与了一些应激反应相关的功能和,同时,抑制了细胞周期、细胞凋亡、细胞死亡巨噬细胞代谢等相关功能,通过激活和抑制相关功能诱发结肠癌的发生。
| [1] |
SMITH R A, ANDREWS K S, BROOKS D, et al. Cancer screening in the United States, 2018: A review of current American Cancer Society guidelines and current issues in cancer screening[J]. A Cancer Journal for Clinicans, 2018, 68(4): 297-316. DOI:10.3322/caac.21446 (  0) 0) |
| [2] |
XIAO M, LI J, LI W, et al. MicroRNAs activate gene transcription epigenetically as an enhancer trigger[J]. RNA Biology, 2017, 14(10): 1326-1334. DOI:10.1080/15476286.2015.1112487 (  0) 0) |
| [3] |
HUANG Z, SHI J, GAO Y, et al. HMDD v3.0: A database for experimentally supported human microRNA-disease associations[J]. Nucleic Acids Research, 2019, 47(D1): 1013-1017. DOI:10.1093/nar/gky1010 (  0) 0) |
| [4] |
AGARWAL R, KUMAR B, JAYADEV M, et al. CoReCG: A comprehensive database of genes associated with colon-rectal cancer[J]. Database, 2016, 2016: baw059. DOI:10.1093/database/baw059 (  0) 0) |
| [5] |
GAO T, HE B, LIU S, et al. EnhancerAtlas: A resource for enhancer annotation and analysis in 105 human cell/tissue types[J]. Bioinformatics, 2016, 32(23): 3543-3551. DOI:10.1093/bioinformatics/btw495 (  0) 0) |
| [6] |
SZKLARCZYK D, GABLE A L, LYON D, et al. STRING v11: Protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets[J]. Nucleic Acids Research, 2019, 47(D1): 607-613. DOI:10.1093/nar/gky1131 (  0) 0) |
| [7] |
PETTERSEN K, MONSEN V T, HAKVAG PETTERSEN C H, et al. DHA-induced stress response in human colon cancer cells-Focus on oxidative stress and autophagy[J]. Free Radic Biology Medcine, 2016, 90(1): 158-172. DOI:10.1016/j.freeradbiomed.2015.11.018 (  0) 0) |
| [8] |
LI X, HAN F, LIU W, et al. PTBP1 promotes tumorigenesis by regulating apoptosis and cell cycle in colon cancer[J]. Bull Cancer, 2018, 105(12): 1193-1201. DOI:10.1016/j.bulcan.2018.08.013 (  0) 0) |
| [9] |
PODGÓRSKA M, PIETRASZEK-GREMPLEWICZ K, NOWAK D. Apelin effects migration and invasion abilities of colon cancer cells[J]. Cells, 2018, 7(8): 113-130. DOI:10.3390/cells7080113 (  0) 0) |
| [10] |
CRUZ A L S, CARROSSINI N, TEIXEIRA L K, et al. Cell cycle progression regulates biogenesis and cellular localization of lipid droplets[J]. Molecular and Cellular Biology, 2019, 39(9): e00374-18. DOI:10.1128/MCB.00374-18 (  0) 0) |
| [11] |
YAMAZUMI K, NAKAYAMA T, KUSABA T, et al. Expression of interleukin-11 and interleukin-11 receptor alpha in human colorectal adenocarcinoma; immunohistochemical analyses and correlation with clinicopathological factors[J]. World Journal of Gastroenterol, 2006, 12(2): 317-321. DOI:10.3748/wjg.v12.i2.317 (  0) 0) |
 2019, Vol. 17
2019, Vol. 17


