2. 上海大学 量子与分子结构中心, 上海 200444
2. International Center for Quantum and Molecular Structure, Shanghai University, Shanghai 200444, China
在生物的表观遗传学中,Bromodomains(BRDs)识别组蛋白上配体(乙酰化赖氨酸)并与之结合的机制是基因转录和翻译过程中的重要一环,而与该机制相应的药物研究在癌症、白血病和HIV等重大免疫性疾病治疗领域具有巨大价值[1-2]。近年来,在众多BRDs识别配体机制的研究中,广大研究者为了寻找新的BRDs结构或开发BRDs小分子抑制剂,大多以BRDs的自身结构作为出发点,虽然新的BRDs结构和小分子抑制剂层出不穷,但是其结构稳定性或药物分子与目标氨基酸分子结合之后的稳定性却少有观察和研究[3]。本研究将以BRDs与配体的结合体为研究对象,首先对BRDs结合位点的自身几何结构采用2015年新开发的结合位点拓扑学方法进行分析,进而通过分子动力学模拟对结合体进行研究,两种方法的结果互相印证结合体的稳定性,最终结果将有助于BRDs小分子药物稳定性提高的研究。
1 材料与方法 1.1 材料在酿酒酵母体内的酵母重组蛋白-4亚基(Rsc4)中存在着一种Bromodomain,是为数不多的Bromo家族的一员,在蛋白质晶体库中其蛋白晶体结构名称为2r0v,它是Bromodomain与赖氨酸的稳定结合体。为了提高研究效率,我们直接将结合体中结合位点所在的蛋白链A作为主要的研究对象, 蛋白链A的氨基酸总数为293,有四个螺旋结构和两个疏水性绊环,这些结构形成了一个疏水性的口袋。为了探究Bromodomains识别并结合配体(ACK:乙酰化赖氨酸)的机理,通过FCTM和分子动力学方法分别对蛋白链A进行研究。
1.2 结合位点拓扑学方法(FCTM)FCTM(Fragment-centric topographical mapping)是Yingkai Zhang新开发的结合位点中心拓扑学映射方法, 是基于结合位点的局域几何特征的蛋白质结合位点分析方法[4],其通过紧邻原子中心坐标得到与这些中心点相切的alpha球,其中过大的alpha球意味着这些紧邻原子分布过于平坦,它们不能提供很好的凹性,过小的alpha球能提供足够的空隙以结合配体。因此该方法先滤除过大或过小的alpha球,并通过对alpha球聚类,得到alpha球形成的数个团簇,这样的团簇意味着它们镶嵌在真实原子所形成的子结合位点(Subpocket)之中,因此能够对蛋白质表面进行划分得到蛋白质表面的子结合位点,并用不同颜色给蛋白质表面染色以区分这些子结合位点。为定量描述这些子结合位点的性质,FCTM设计了几个定量的特性参数,包括凹性得分(Score)、配体在该子结合位点上的占有率(Occupied)和非极性程度(Non-ploar)等,其中凹性得分越高说明结合位点识别能力越强,反之则弱;占有率越高说明配体处在pocket的位置越深,结合也就越紧密,反之则疏松。
通过FCTM方法对初始2r0v模型结构进行分析,可看到在结合位点的凹性方面score=60,同时其alphaspace=131,这两项结果表明2r0v结合位点的几何性质有非常高的可识别性[5],结果如表 1所示。
| 表 1 经FCTM分析结果 Table 1 Results of FCTM analysis |
分子动力学方法(MD:Molecular-dynamics simulation)是研究大分子体系时被广泛采取的方法。MD方法主要应用量子力学、牛顿力学以及统计力学等物理理论,其理论具有物理理论依据全面和精确度高的优势[6]。
1.3.1 模拟模型的建立分子动力学模拟最关键的工作是模型的建立,为了提高分子模拟的效率,我们直接将分子模拟的建模工作集中在结合位点的修饰上,具体步骤为:1.将晶体模型中的赖氨酸残基人工改为乙酰化赖氨酸;2.补足数据库中由于实验误差和解谱精确度导致缺失的原子,改正数据库中指认错误的原子;3.构建电中性且溶解于水的蛋白质模型;4.应用适当的力场。我们采用最流行的AMBER14SB力场对BRD建模[7],对于水,则采用TIP3P模型。整个分子动力学模拟分3个步骤:
(1) 在NVT系综将模型优化,通过可视化软件VMD观察到整个“水盒子”里的水分子和蛋白质都均匀地舒展开来,优化过程需10 ps;
(2) 约束蛋白质,让水溶液升温,升至合理的体系温度300 K,整个过程持续的时间为100 ps;
(3) 模拟蛋白质在生物环境中的运动,通过逐步减小约束力常数来弛豫蛋白质,而此时整个模拟的环境是NPT系综,具体的模拟过程总结如表 2所示。
| 表 2 整个分子动力模拟过程的参数设置 Table 2 Parameter setting of the overall molecular dynamics simulation |
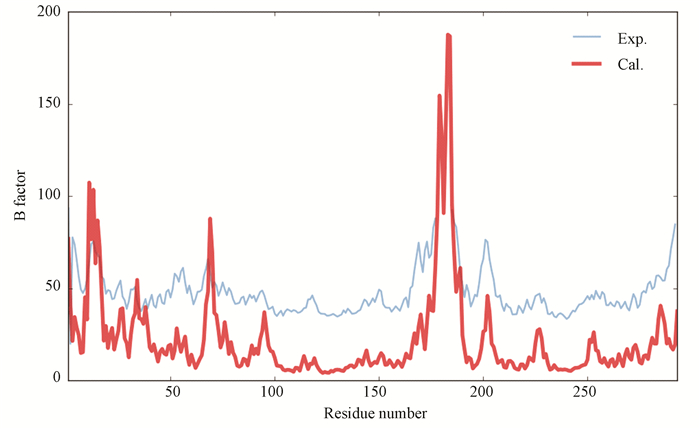
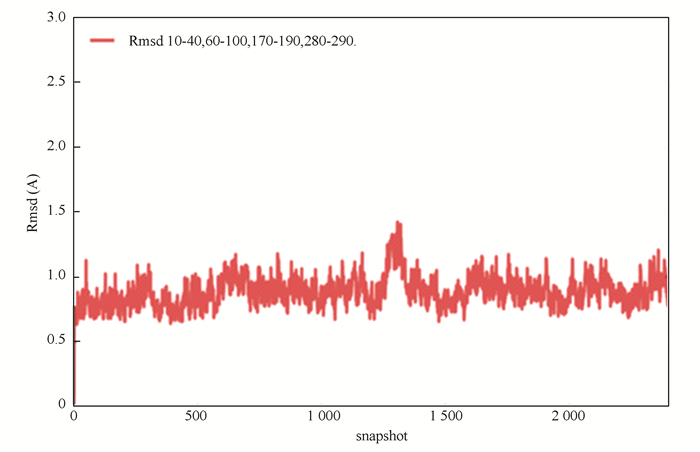
MD模拟之后,模拟过程中蛋白链的B-factor(温度系数)[6]和RMSD(蛋白链柔软度)[8]如图 1和图 2所示。
|
图 1 A蛋白链B-factor值 Figure 1 B-factor value of the A protein chain |
|
图 2 A蛋白链在模拟时RMSD值 Figure 2 RMSD value of the A protein chain in simulation |
从B-factor和RMSD图清晰看到整条蛋白链A保持在一个稳定的柔性状态,图 1中B-factor值在实验与模拟之间存在着较大数值上的差距,主要是因为在约束立场参数设置下模拟比实验更具稳定性,所以模拟的整体结果数值更小[6],而在残基序列170-190的位置分子模拟因为蛋白链的柔韧性存在一个巨大的峰值,但该值和蛋白晶体数据库中的B-factor值(实验值)是一致的,且实验值与模拟值之间的涨落趋势是大致相同的,这足以说明模拟过程中蛋白链的稳定性是准确的。结合图 2中RMSD的变化就能充分说明蛋白链保持着相应的柔韧度,而在整条链的前段、尾端、突出处和少量的折叠部分存在着较大的构象变化,具体位置在残基序列10-40, 60-100, 170-190, 280-290,蛋白链A的平均构象变化小于1.7 Å。上述结果表明该蛋白结构域与配体的结合体是稳定的。除了上述结果也可通过常规的氢键网络来分析其结合的状态,具体如图 3所示:
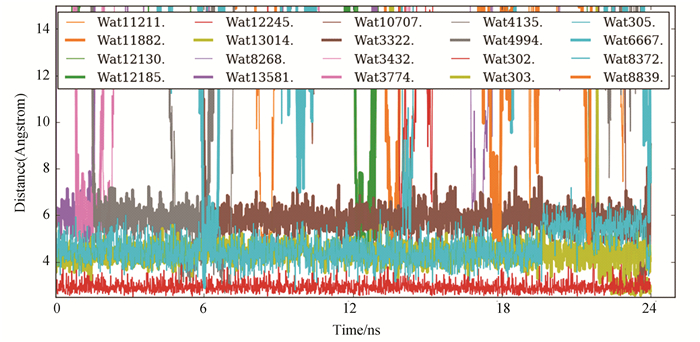
|
图 3 模拟过程中水分子对配体的氢键网络 Figure 3 Hydrogen bonding network of water molecules to ligands during simulation |
在生物化学作用中氢键占据重要的地位,在模拟的水环境中,电负性原子与另一个电负性原子之间以共价方式结合形成弱相互作用的氢键,两电负性原子不等分配,一个为氢供体,另一个为氢受体。氢键的强弱与原子的电负性或半径大小有关,电负性越大,半径越小则氢键作用越强。在实际水环境中氢键的种类会因为原子种类的不同而不同,最多的是水分子之间的(O-H…O-)氢键,键能为18.8 kJ/mol。图 3反映了模拟过程中从水分子到配体分子的距离来间接表示氢键的作用域[9],该图清晰地表明在整个分子动力学模拟期间,陆续会有不同水分子(其中包含晶体结晶水)与配体匹配,形成稳定的氢键作用,当然在约1 510-1 520(10 ps)之间氢键网络有稍微缺失,即存在一个较弱的氢键作用,而此时配体与pocket的构象有较高模糊度[10]。
在Bromodomains的结构特征中,由于有两个高度保守的袢环结构(ZA、BC环),同时还有一定数量的结晶水分子,这些水分子与配体之间的相互作用,促进Bromodomain识别配体并与其结合。在分子动力学模拟过程中,以配体N末端(O1-C1-NE-CE)四个原子组成的二面角构象随着蛋白链的摆动而发生变化,但是构象整体处于稳定状态。通过可视化软件VMD,可清晰看到有1个高度保守的氨基酸分子:Pro66与配体ACK之间形成紧密的非键相互作用,同时在整个分子动力学模拟期间持续有不同的氨基酸分子与配体保持较小距离,并持续发生分子间作用[11],具体情形如下图 4所示。图 4是分子动力学模拟前后结合位点的结构图,图中脯氨酸分子Pro66在配体(黄色ACK)右下方稳定地存在着,且两分子之间的距离小于5 Å。
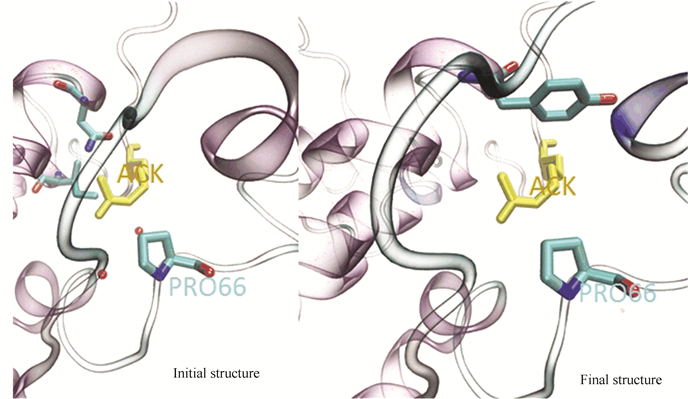
|
图 4 结合位点表面氨基酸分子对配体的作用力 Figure 4 Force from amino acid molecules on the binding site to ligand |
在分析蛋白识别氨基酸机制时,研究者通常以蛋白-蛋白反应机理为基础,而对蛋白或氨基酸自身几何结构的分析也是研究的重点[12]。对Bromodomain结合位点的几何结构进行FCTM分析,pocket的凹凸性分数为60,该结果说明该Bromodomain的结合位点具有高凹性,这就使得其识别配体时表现敏感,同时其alphaspace值为131也表明结合位点内部容积较大,这些都是识别配体的有利条件, 也间接地表示该保守结构域稳定结合配体的较高可能性。分子动力学模拟结果显示:模拟过程中配体受到的来自结合位点表面脯氨酸(Pro66)分子的持续分子间作用,同时pocket内的水分子分布在配体周围并对其给予氢键网络的作用,这些都是配体能被Bromodomain识别并且无法逃离的重要原因。
4 结论综上两种方法得到的结果,该Bromodomain识别乙酰化赖氨酸并与之稳定结合的关键因素有两个:
1) 蛋白结构域自身结合位点的几何结构提供了结构基础。
2) 结合位点表面氨基酸分子和内部水分子对配体的持续分子间作用对配体有着重要影响。
| [1] |
LU P, SHEN Y, YANG H, et al. BET inhibitors RVX-208 and PFI-1 reactivate HIV-1 from latency[J]. Scientific Reports, 2017, 7(12): 1-12. DOI:10.1038/s41598-017-16816-1 (  0) 0) |
| [2] |
HARDER E, DAMM W, MAPLE J, et al. OPLS3:A force field providing broad coverage of drug-like small molecules and proteins[J]. Journal of Chemical Theory & Computation, 2016, 12(1): 281-296. DOI:10.1021/acs.jctc.5b00864 (  0) 0) |
| [3] |
BRAND M, MEASURES A M, WILSON B G, et al. Small molecule inhibitors of bromodomain acetyl-lysine interactions[J]. American Chemical Society, 2015, 10(17): 22-39. DOI:10.1021/cb500996u (  0) 0) |
| [4] |
ROOKLIN D, WANG C, KATIGBAK J, et al. AlphaSpace:Fragment-centric topographical mapping to target protein-protein interaction interfaces[J]. Chemical Information and Modeling, 2015, 55(15): 1585-1599. DOI:10.1021/acs.jcim.5b00103 (  0) 0) |
| [5] |
FILIPPAKOPOULOS P, PICAUD S, MANGOS M, et al. Histone recognition and large-scale structural analysis of the human bromodomain family[J]. Cell, 2012, 149(18): 214-231. DOI:10.1016/j.cell.2012.02.013 (  0) 0) |
| [6] |
WANG J. Molecular dynamics simulations of a protein crystal[J]. Bioenergetics:Open Access, 2014, 02(3): 1-3. DOI:10.4172/2167-7662.1000e117 (  0) 0) |
| [7] |
MCDOWELL S E, SPACKOVA N, SPONER J, et al. Molecular dynamics simulations of RNA:an in silico single molecule approach[J]. Biopolymers, 2007, 85(27): 1-27. DOI:10.1002/bip.20620 (  0) 0) |
| [8] |
NEIRA J L. NMR as a tool to identify and characterize protein folding intermediates[J]. Archives of Biochemistry and Biophysics, 2013, 531(10): 90-99. DOI:10.1016/j.abb.2012.09.003 (  0) 0) |
| [9] |
WANG H, CHEN Y, HUANG C, et al. Insight into the function of the key residues in the binding clefts of the substrate with CBM4-2 of xylanase Xyn10A by molecular modeling and free energy calculation[J]. Computational and Theoretical Chemistry, 2017, 1111(6): 14-19. DOI:10.1016/j.comptc.2017.03.024 (  0) 0) |
| [10] |
KOVERMANN M, ROGNE P, WOLF-WATZ M. Protein dynamics and function from solution state NMR spectroscopy[J]. Quarterly Reviews of Biophysics, 2016, 49(43): 1-43. DOI:10.1017/S0033583516000019 (  0) 0) |
| [11] |
WEN B, ZHANG L, WANG D, et al. The distribution of excess carriers and their effects on water dissociation on rutile (110) surface[J]. Computational Materials Science, 2017, 136(7): 150-156. DOI:10.1016/j.commatsci.2017.04.037 (  0) 0) |
| [12] |
刘畅, 刘安. 生物信息学分析RIPK4的分子结构与功能[J]. 生物信息学, 2018, 16(8): 105-113. LIU Chang, LIU An. Bioinformatics analysis of the structure and function of RIPK4[J]. Chinese Journal of Bioinformatics, 2018, 16(8): 105-113. DOI:10.3969/j.issn.1672-5565.201708006 (  0) 0) |
 2019, Vol. 17
2019, Vol. 17


