众所周知,癌症是当前世界上引发人类死亡的主要疾病之一。它是一种非常复杂的由多基因协同作用而引发的疾病,可以发生在人体内一个系统的多个器官,也可以发生在一个器官的多个系统,也有可能是多个系统多个器官。由于严重程度、持续时间、发病位置、对药物的敏感性和耐受程度、细胞分化和发生以及对发病机理了解的不同,该病的诊断、预后和治疗效果都不尽如意。根据传统的指标如肿瘤大小、临床分期、病理分级、淋巴结转移数目等对癌患者进行诊断,即便是处于同一分期的患者经过正规治疗的预后也会有很大的不同。因此,从基因水平出发,寻找与癌症密切相关的差异表达基因或者基因突变对于癌症的精准化治疗便显得尤为重要。
基因芯片和测序技术是基因水平研究的两大主要手段。基因芯片基于已知序列信息进行探针设计,通过碱基互补杂交来识别基因并鉴定其表达,检测技术和分析方法都相对成熟。高通量测序从sanger测序到现在新兴的纳米孔测序技术已经有三十多年历史,目前已经被成功应用在约2 500多种疾病的检测上。其中靶向基因测序虽然成本较低,但是可以覆盖几乎所有的癌基因而被许多实验室当作常规检测手段来使用。近年来,虽然高通量测序的势头越来越高,但是基因芯片以其经济快速准确等特点在临床应用上也同样备受欢迎。由于基因芯片不能发现新的序列突变以及转录本等缺陷,而高通量测序则可以对其进行补充。因此,二者在临床研究中都有不可替代的研究作用。比如,基因表达谱芯片和RNAseq都是对RNA样本进行制备和分析,他们二者的优缺点见表 1,需要根据研究目的和经费预算而进行选择。接下来,我们将分别介绍基因表达谱芯片以及核酸测序技术在癌症基因组学中的应用,加强我们对癌症发病机制的理解从而加速癌症的个体化治疗进程。
| 表 1 基因表达谱芯片与RNAseq的比较 Table 1 Comparison of gene microarray and RNAseq |
基因芯片技术是将许多特定的寡核苷酸片段或基因片段有规律地排列固定于支持物(如膜、硅片、陶瓷片及玻片)上,然后通过类似于Northern,Southern的方法与待测的标记样品按碱基配对原理进行杂交,再通过检测系统对其进行扫描,并用相应软件对信号进行比较和检测,得到所需的大量信息,进行基因的高通量、大规模、平行化、集约化的信息处理和功能研究。基因芯片技术已成为功能基因组学研究中一项非常重要和关键的实用技术,可自动、快速地检测出上万个基因的表达情况,从而对遗传信息进行快速准确的分析,可用于遗传病相关基因的定位、肿瘤诊断、耐药菌株和药敏检测等。
目前的芯片主要来自于三个生产厂家:Affymetrix GeneChips, Illumina BeadArrays, and Agilent 2-channel arrays[1]。这些芯片基本上可以满足当下人类组基因表达的所有需求,如果需要检测特殊的基因表达,还可以通过定制基因芯片来实现。
1.1 数据分析目前,常用的基因表达谱芯片数据库有GEO(https://www.ncbi.nlm.nih.gov/geo/)和Arrayexpress(https://www.ebi.ac.uk/arrayexpress/)。得到数据以后,首先,需要对芯片的原始数据进行质控分析,可以直接通过ArrayQualityMetricx等软件包。通常芯片自身的质控合格需要达到以下几点:背景信号在150以下,Corner角落信号和Central-信号一般在15 000~20 000以下,看家基因GAPDH和βactin的3'/5'值小于3。杂交对照包括bioB、bioC、bioD和cre应该被检测到。ploy-A对照包括dap,lys,phe以及thr应被检测到,同时信号逐级升高。为了使得数据之间可进行比较,还需采用R语言中的affy、affycoretools以及simpleaffy等软件包,绘制芯片的箱线图、直方图、RNA降解图和主成份图。由于芯片的类型和物种的不同,这些质控图都没有固定的形状,一般来讲,与其他样本偏差较大的样本可能会存在一些问题,需要排除掉。剩下的芯片可以用rma进行标准化处理。对于有多个探针的基因,计算平均值作为该基因的表达值。而对于miRNA一般使用miRNA_QC_tool来对其进行质控分析。包括箱线图、直方图、相关系数、质控探针的表达值以及杂交对照bioB、bioC、bioD和cre的信号图。随后使用rma方法获得芯片的标准化数据。limma统计方法通常用于获取差异表达基因,随后可以进一步将这些基因进行富集分析或者建立模型等[2]。
1.2 在癌症上的临床应用实例 1.2.1 MammaPrint的应用MammaPrint是由70个基因组成的检测芯片,已经被多个研究证实其对早期乳腺癌患者预后的预测作用[3-4]。该检测芯片是从5 000个基因中选取出来用于预测淋巴结阴性乳腺癌患者的无疾病生存和整体生存。而且该芯片检测也被证实对于淋巴结阳性的肿瘤也有显著的预测效果[5]。因其成本不高而且可以改善患者的生存质量,通过该方法预测出来的低风险组,其5年生存率可以高达90%,从而使患者避免不必要的化疗过程。
1.2.2 在癌症药物的研究和模型开发上的应用Chen等人[2]在近年开发的ER阴性单药物模型就是基于DNA芯片数据。作者从DNA芯片数据中分别针对紫杉醇,5-氟尿嘧啶,阿霉素以及环磷酰胺开发了单药物模型。作者从细胞系芯片数据出发,采用pearson、spearman、t-test、ancova以及rank based ancova多种统计学方法提取与四种药物的GI50有关的基因,随后使用COXEN的方法[6]从细胞系中筛选出可以用到人类组织中的基因标志物。最后用独立样本集来验证模型对ER阴性乳腺癌患者在各种化疗组合方案的反应和生存,取得了不错的预测精度。He等人[7]通过对大肠癌细胞系的基因芯片进行分析,发现Wnt通路与5-FU的抗性有关,同时他们还发现在5-FU抗性细胞系中,CHK1通路被Wnt通路抑制,揭示了这两条通路之间的相互作用。此外,还可以将非编码RNA制作成芯片进行研究,比如Tian等人[8]在肺癌对紫杉醇抗药性的研究中就使用了这一技术。
1.2.3 在癌症的免疫治疗研究中的应用比如,评估ZAP-70在慢性淋巴白血病中白血病细胞中的表达[5]。研究表明,ZAP-70如果在T细胞中的表达水平高,那么就可以将患者分配到正确的IgVH突变亚型,从而决定患者接受何种治疗方案[9]。除此之外,还可以通过基因芯片研究不同时间段的T细胞基因表达,以及T辅助细胞亚群在免疫应答过程中的分化机制。
1.2.4 在癌症分型上的应用由于癌症是一个多基因主导的复杂性疾病,所以如果能够将其精确划分成不同的亚型,那么将有助于对癌症的诊断和治疗。基因芯片在这一方面有着突出的贡献,miRNA芯片、mRNA芯片以及IncRNA都曾用于此研究[10-12]。
2 高通量测序在癌症基因突变检测上的应用 2.1 文库制备目前常用的高通量测序技术主要是第二代测序,文库构建根据所测序列分为DNA类文库以及RNA类文库。整体来讲,构建文库主要分为以下四步[13]:(1)将目标序列打断,如果是RNA序列则需要现反转录为DNA在进行打碎,一般常用的是物理的方法(超声波)和酶反应的方法,此外还有化学的方法,从而获得实验所需长度的片段;(2)将末端补平,并在3’端连接碱基A,随后将可以与测序平台或者磁珠相结合的接头加上;(3)文库扩增,对于Illumina是通过生成DNA簇,而半导体测序则是通过microemulsion PCR;(4)转到测序芯片或者磁珠上,根据长度进行片段的选择和纯化,从而完成文库构建(见图 1)。
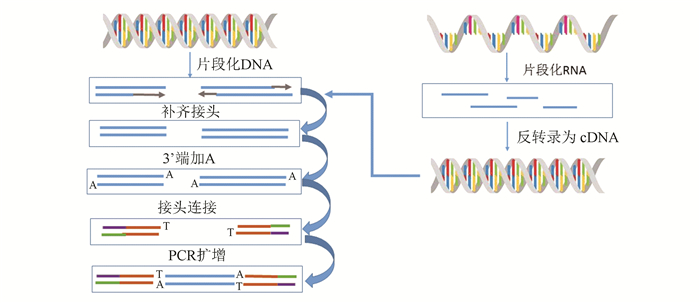
|
图 1 文库构建基本流程图 Figure 1 Flow chart of library preparation |
对于文库的构建来讲,从DNA样本出发,针对全基因组、外显子基因组以及chip-seq和PCR扩增子的测序文库构建基本都遵循一样的流程,最终的目的都是尽可能的提高文库的复杂度。目前,也有很多成熟的商业化的试剂盒,用户可以根据需要来进行选择。
2.2 数据分析二代测序常用的数据库有TCGA(https://cancergenome.nih.gov/)以及NCBI中的SRA数据库(https://www.ncbi.nlm.nih.gov/sra/)。二代测序数据比芯片数据分析起来稍微复杂一些,基于RNA[14]和DNA序列,其分析流程也有所不同。但是第一步都需要对数据进行质控,一般使用fastqc对数据的质量进行基本的评估,然后使用cutadptor等软件将接头以及低质量数据去除。RNA序列分析根据是否拼接选取相应的软件进行比对,随后根据是否需要参考基因组,选用不同的软件进行序列的组装,在根据基因水平还是isoform水平,选取对应的软件进行定量标准化分析和差异表法分析(见图 2)。其中每一步骤都列出很多种不同的软件,这些软件基于不同的算法和语言开发,精度略有不同,比如BitSeq在差异表法分析上要优于Cuffdiff[15]。但是大多数算法的精度都差不多,用户可以根据所使用的平台和语言对其进行选择。而对于DNA序列,其序列比对软件主要有Bowtie和BWA,其中Bowtie速度很快,主要用于局部序列比对;BWA经常用于全基因组和外显子组的重序列比对。值得注意的是,STAR是专门针对RNA序列比对所涉及的比对软件,不被用在DNA序列比对上,其速度是目前比对软件中最快的,但是需要至少30G的内存来运行。DNA比对完以后用GATK toolbox来对数据进行处理,进而检测突变。可以使用GATK中自带的突变检测程序,也可以使用Mutect2以及Strelka2等软件(见图 3)。

|
图 2 RNA序列分析流程及所用工具 Figure 2 Flow chart of RNAseq analysis and related tools |
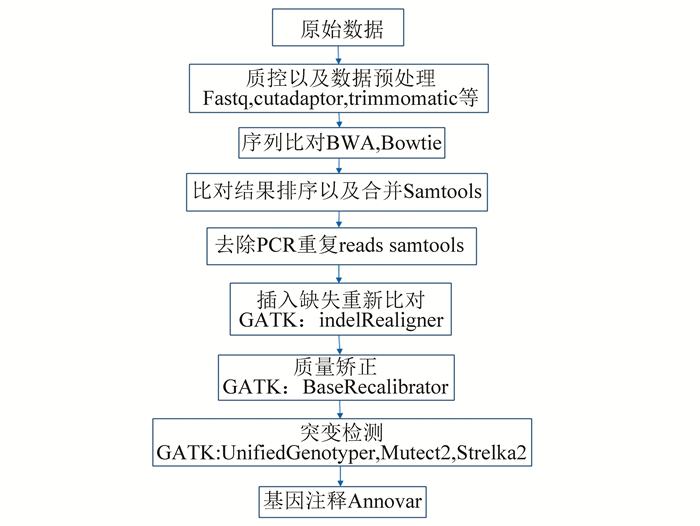
|
图 3 DNA序列分析流程及所用工具 Figure 3 Flow chart of DNAseq analysis and related tools |
测序包括mRNA、小RNA以及长链非编码RNA。一般mRNA经常用于比较不同组织之间的差异表达,从而发现与癌症相关的变异,比如基因融合[16]以及可变剪切等[17]。小RNA测序则通过分析表达差异,预测其靶基因,筛选用于疾病诊断的分子标记。长链非编码RNA一般与样品中的mRNA共表达分析,挖掘其功能和作用机制,从而发现与癌症之间的关系。比如Gradia等人从长链非编码RNA和mRNA的数据出发,分析TUG1的表达与乳腺癌不同亚型之间的关系[18]。
DNA测序一般用于研究癌症基因组的单核苷酸变异、插入以及缺失与癌症的相关性。比如,TP53, PTEN, RUNX1, CCND3,BRCA1、EGFR以及PTPN22等已知的癌基因的变异在肺癌[19]、卵巢癌[20]以及乳腺癌[21]中的作用。此外,还可以通过DNA测序对免疫组学进行研究,比如检测T细胞受体库的多样性,从而了解机体免疫应答状态。
3 总结综上可见,芯片技术和高通量测序技术都已经被广泛的应用在癌症的研究当中,每种方法都有自己的优点和不足。总体来讲,基因芯片技术无论是在制备还是分析上都很成熟,而测序技术仍处于飞速发展阶段。比如最近的纳米孔测序,虽然读长很长,但是准确度还不尽人意。希望不久的将来,可以出现高通量、高精度、低成本的测序技术,那么将极大的加快对癌症的研究。
| [1] |
COORDINATORS N R. Database resources of the national center for biotechnology information[J]. Nucleic Acids Research, 2017, 45(D12): D17. DOI:10.1093/nar/gkw1071 (  0) 0) |
| [2] |
CHEN Y Z, KIM Y, SOLIMAN H H, et al. Single drug biomarker prediction for ER-breast cancer outcome from chemotherapy[J]. Endocr Relat Cancer, 2018, 25(6): 595-605. DOI:10.1530/ERC-17-0495 (  0) 0) |
| [3] |
GROENENDIJK F H, JAGER A, CARDOSO F, et al. A nationwide registry-based cohort study of the MammaPrint genomic risk classifier in invasive breast cancer[J]. Breast, 2018, 38: 125-131. DOI:10.1016/j.breast.2017.12.015 (  0) 0) |
| [4] |
XIN L, LIU Y H, MARTIN T A, et al. The era of multigene panels comes?The clinical utility of oncotype dx and mammaprint[J]. World Journal of Oncology, 2017, 8(2): 34-40. DOI:10.14740/wjon1019w (  0) 0) |
| [5] |
NARRANDES S, XU W. Gene expression detection assay for cancer clinical use[J]. Journal of Cancer, 2018, 9(13): 2249-2265. DOI:10.7150/jca.24744 (  0) 0) |
| [6] |
LEE J K, HAVALESHKO D M, CHO H, et al. A strategy for predicting the chemosensitivity of human cancers and its application to drug discovery[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(32): 13086-13091. DOI:10.1073/pnas.0610292104 (  0) 0) |
| [7] |
HE Lingfeng, ZHU Hong, ZHOU Shiying, et al. Wnt pathway is involved in 5-FU drug resistance of colorectal cancer cells[J]. Experimental Molecular Medicine, 2018, 50(8): 101. DOI:10.1038/s12276-018-0128-8 (  0) 0) |
| [8] |
TIAN X, ZHANG H, ZHANG B, et al. Microarray expression profile of long non-coding RNAs in paclitaxel-resistant human lung adenocarcinoma cells[J]. Oncology Reports, 2017, 38(1): 293-300. DOI:10.3892/or.2017.5691 (  0) 0) |
| [9] |
WIESTNER A, ROSENWALD A, BARRY T S, et al. ZAP-70 expression identifies a chronic lymphocytic leukemia subtype with unmutated immunoglobulin genes, inferior clinical outcome, and distinct gene expression profile[J]. Blood, 2003, 101(12): 4944-4951. DOI:10.1182/blood-2002-10-3306 (  0) 0) |
| [10] |
SONG J, ZHANG W, WANG S, et al. A panel of 7 prognosis-related long non-coding RNAs to improve platinum-based chemoresistance prediction in ovarian cancer[J]. International Journal of Oncology, 2018, 53(2): 866-876. DOI:10.3892/ijo.2018.4403 (  0) 0) |
| [11] |
REN Z, WANG W, LI J. Identifying molecular subtypes in human colon cancer using gene expression and DNA methylation microarray data[J]. International Journal of Oncology, 2016, 48(2): 690-702. DOI:10.3892/ijo.2015.3263 (  0) 0) |
| [12] |
SUN E H, ZHOU Q, LIU K S, et al. Screening miRNAs related to different subtypes of breast cancer with miRNAs microarray[J]. European review for medical and pharmacological sciences, 2014, 18(19): 2783-2788. (  0) 0) |
| [13] |
HEAD S R, KOMORI H K, LAMERE S A, et al. Library construction for next-generation sequencing:overviews and challenges[J]. Biotechniques, 2014, 56(2): 61-64, 66, 68. DOI:10.2144/000114133 (  0) 0) |
| [14] |
YANG I S, KIM S. Analysis of whole transcriptome sequencing data:workflow and software[J]. Genomics Informmation, 2015, 13(4): 119-125. DOI:10.5808/GI.2015.13.4.119 (  0) 0) |
| [15] |
CONSORTIUM SM-I. A comprehensive assessment of RNA-seq accuracy, reproducibility and information content by the sequencing quality control consortium[J]. Nature Biotechnology, 2014, 32(9): 903-914. DOI:10.1038/nbt.2957 (  0) 0) |
| [16] |
MA Y, MIAO Y, PENG Z, et al. Identification of mutations, gene expression changes and fusion transcripts by whole transcriptome RNAseq in docetaxel resistant prostate cancer cells[J]. Springerplus, 2016, 5(1): 1861. DOI:10.1186/s40064-016-3543-0 (  0) 0) |
| [17] |
MA Y, MIAO Y, PENG Z, et al. Erratum to:Identification of mutations, gene expression changes and fusion transcripts by whole transcriptome RNAseq in docetaxel resistant prostate cancer cells[J]. Springerplus, 2016, 5(1): 2084. DOI:10.1186/s40064-016-3759-z (  0) 0) |
| [18] |
GRADIA D F, MATHIAS C, COUTINHO R, et al. Long non-coding rna tug1 expression is associated with different subtypes in human breast cancer[J]. Noncoding RNA, 2017, 3(4): 26. DOI:10.3390/ncrna3040026 (  0) 0) |
| [19] |
SUNG J S, CHONG H Y, KWON N J, et al. Detection of somatic variants and EGFR mutations in cell-free DNA from non-small cell lung cancer patients by ultra-deep sequencing using the ion ampliseq cancer hotspot panel and droplet digital polymerase chain reaction[J]. Oncotarget, 2017, 8(63): 106901-106912. DOI:10.18632/oncotarget.22456 (  0) 0) |
| [20] |
SUKHBAATAR N, BACHMAYR-HEYDA A, AUER K, et al. Two different, mutually exclusively distributed, TP53 mutations in ovarian and peritoneal tumor tissues of a serous ovarian cancer patient:indicative for tumor origin?[J]. Molecular Case Studies, 2017, 3(4): a001461. DOI:10.1101/mcs.a001461 (  0) 0) |
| [21] |
BRIANESE R C, NAKAMURA K D D M, ALMEIDA F, et al. BRCA1 deficiency is a recurrent event in early-onset triple-negative breast cancer:a comprehensive analysis of germline mutations and somatic promoter methylation[J]. Breast Cancer Research and Treat, 2018, 167(3): 803-814. DOI:10.1007/s10549-017-4552-6 (  0) 0) |
 2019, Vol. 17
2019, Vol. 17

