乳腺癌是我国常见多发疾病,在妇科疾病中其发病率和死亡率已高居各种恶性肿瘤的首位,大多数患者被临床诊断发现后已是肿瘤生长的晚期阶段,失去了临床早期治疗的宝贵机会。因此该疾病对患者生命健康具有严重的威胁,严重影响患者的生活质量。
缺乏早期诊断技术、没有持久有效的治疗方案仍然是乳腺癌诊治中存在的棘手问题。目前, 常用的实时定量qRT-PCR法存在非特异性杂交以及错误扩增等问题[1],而传统的检测方法Northern杂交法,对miRNA检测灵敏度低[2],,而其他的方法则大多需要对目标分子进行化学修饰或特殊标记。由于早期诊断有助于改善乳腺癌的治疗和预后效果,因此寻找新的、有效的乳腺癌早期标志分子成为目前国际上肿瘤研究的一个焦点。由于纳米孔传感器具有高灵敏度、高选择性、高通量、无标记和无扩增的检测优势,可简单快捷的实现乳腺癌的早期检测。1996年,Kasianowicz和他的同事们在NIST上第一次报道了关于α-溶血素纳米通道在核酸检测中的应用[3],迄今为止,研发人员也相继研发出各种新型生物纳米孔道[4-7]。纳米孔单通道检测技术也随之被应用到DNA测序[4, 6]、多肽检测[8-10]、及RNA检测[11-14]等研究领域。由于生物分子的物理占位效应,当生物分子通过纳米通道时,堵塞会引起方波信号,当分子长度较长时,引起的调制电流持续时间变长。所以不同待测物分子通过纳米孔通道时离子流的改变不同,特征信号的堵塞程度和持续时间不同[15-16],因此,纳米孔单通道可视为一种天然的探测单分子的传感器,实现单分子水平高灵敏的特异性检测。
人的血浆中含有核糖核酸(RNA)[17],从开始发现循环RNA,人们就一直研究它在临床疾病的诊断、预警等方面的应用。循环RNA的表达与多种癌症相关,得到注解的大约50%循环RNA在基因组上定位于与肿瘤相关的脆性位点,可通过检测癌症病人的循环RNA中的特异信使RNA(mRNA)来进行癌症的诊断治疗,因此血浆中的RNA作为生物标志物在癌症诊断中具有良好的应用前景[18-19]。microRNA(miRNAs)是一类非编码的小RNA分子,它是重要的基因表达调控因子,许多研究已经证明异常的miRNA表达与癌症发生、发展有关,且miRNA表达不同则癌症的类型不同[20],因此对癌症的早期诊断及预后判断具有重要的临床指导意义。研究结果发现,miRNA在乳腺癌患者组织中异常表达[21],而且另一项研究表明在血浆中miRNA具有高度的稳定性[22],由于其特殊的调控功能,miRNA不仅在细胞分化和生物发育过程中起重要作用,而且还与各种疾病的发生发展中发挥巨大作用,比如医学检验、肿瘤和免疫方面等[23-25]。因此miRNA的检测,在分子生物学及医学领域都有着非常重要的指导意义。研究表明miRNA是乳腺癌的新一代的肿瘤标志物[26]。经文献报道乳腺癌肿瘤大小与乳腺癌患者血浆中的miR-31、miRNA 195及miRNA 892b的表达水平密切相关[27]。
本研究设计了能够特异性识别目标miRNA的DNA探针(Probe),probe与目标miRNA中间识别区完全互补配对,可用于miRNA的特异性检测。以miRNA两端为引导链中间为probe的识别区可以使αHL对miRNA·probe分子迅速捕获。本研究主要目的是利用αHL纳米孔单通道对miRNA·probe复合分子的高灵敏度分辨,实现乳腺癌miRNAs的高效检测。实验结果表明,由于probes序列的差异,miRNA 31·probe 31、miRNA195·probe 195、miRNA 892b·probe 892b三种复合物分子形成的不同形状、不同阻塞时间的特征电流信号,因此该特征信号可用来有效区分miRNA 31、miRNA 195、miRNA 892b。
1 材料与方法 1.1 试剂与仪器试剂:miRNA,α-Hemolysin(Sigma Aldrich),卵磷脂(Sigma Aldrich),正戊烷,十六烷,Teflon膜(厚度25 μm,Goodfellow (Malvern, PA)),参考之前文献[12]实验中所使用的缓冲液由1 mol/ L KCl、10 mmol/ L Tris配制,用HCl调节pH为7.5;H液由1 mL戊烷(pentane)与100 μL十六烷(hexadecane)配置;L液由25 mg磷脂(phospholipid)与2.5 mL戊烷(pentane)配置。所有试剂均为分析纯,实验中所用水均为超纯水(18.2 MΩ·cm, 25 ℃)。
仪器:Integrating patch clamp (Axon Instruments, Forest City, CA, USA),Digidata 1 550 series (Axon Instruments, Forest City, CA, USA),Arbitrary Function Generator (Axon Instruments, Forest City, CA, USA),高速离心机,冰箱,电子天平,数值式水浴温度控制仪,自动双重纯水蒸馆器,可调式移液器,pH计。
1.2 样品的制备本实验所用miRNAs和probes均由上海生工生物工程有限公司合成并HPLC纯化。本实验将miRNA和probe等量混合,先在95 ℃水浴条件下孵育3 min,然后立刻放在在35 ℃培养箱中培养1 min,高低温反复3次,制得miRNA·probe复合物。
1.3 实验方法纳米孔道单分子检测方法为首先在Toflon薄膜两边靠近微米孔处分别滴上一滴约10 μL戊烷与十六烷混合液,并迅速吹开,使其在膜上延展散开;其次在检测池的两个腔室中分别加入2 mL 1.0 mol/L KCl,10 mmol/L Tris,pH 8.0的缓冲液;然后在两个腔室中加入20 μL磷脂戊烷溶液,形成磷脂双分子层;接着系统施加+180 mV跨膜电压,在检测池的cis 端加入一定量的αHL,自组装成稳定的单个纳米孔通道;最后在cis端加入待测物,记录信号。电流信号使用的是5 kHz低通滤波,采样频率是50 kHz。所得数据使用ClampFit 10.4(Molecular Devices, ForestCity, CA, USA)和OriginLab 8.5进行分析。
2 实验结果与分析 2.1 纳米单通道检测miRNA的检测原理纳米单通道检测miRNA是基于DNA杂交的原理(见图 1)。在一定的电压下,不同DNA或RNA分子穿过纳米通道时产生的下降脉冲电流是不一样的,脉冲的宽度和阻塞时间与碱基的个数有关,脉冲的数量与浓度有关。当Probe与Target单独通过通道时由于其分子比纳米孔小,所以很快的通过通道,信号也就相对较短;当Probe与Target配对后通过通道时,由于其分子比纳米孔大,所以通过通道时所需时间较长,信号也长;当Probe与Target配对,但有一个碱基错配后通过通道时的阻塞时间也比完全配对的时间要短,信号也相对较短。
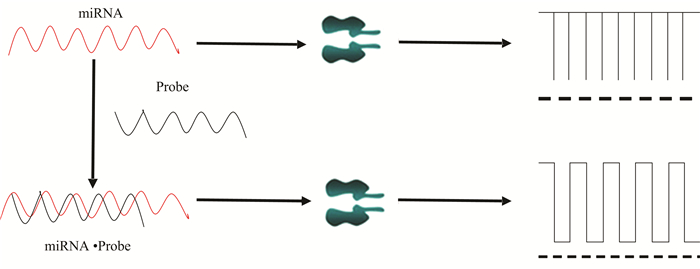
|
图 1 纳米通道检测miRNA Figure 1 Detection of miRNA in a nanopore |
图 2是Probe、miRNA 31及miRNA 31·probe 31通过纳米单通道时的信号图与其对应的散点分析图,图 3是上面所提到的几种核酸的平均阻塞时间值。其中Amplitude是脉冲电流的大小,Dwell Time是阻塞时间,结果可以看出不同核酸在穿过纳米孔通道时脉冲电流大小不同,阻塞时间也不同,probe、miRNA 31及miRNA 31·probe 31的脉冲电流与阻塞时间依次增大,实验结果表明miRNA在穿过纳米孔通道时其脉冲电流及阻塞时间的大小与链的长短和碱基的数量有关。
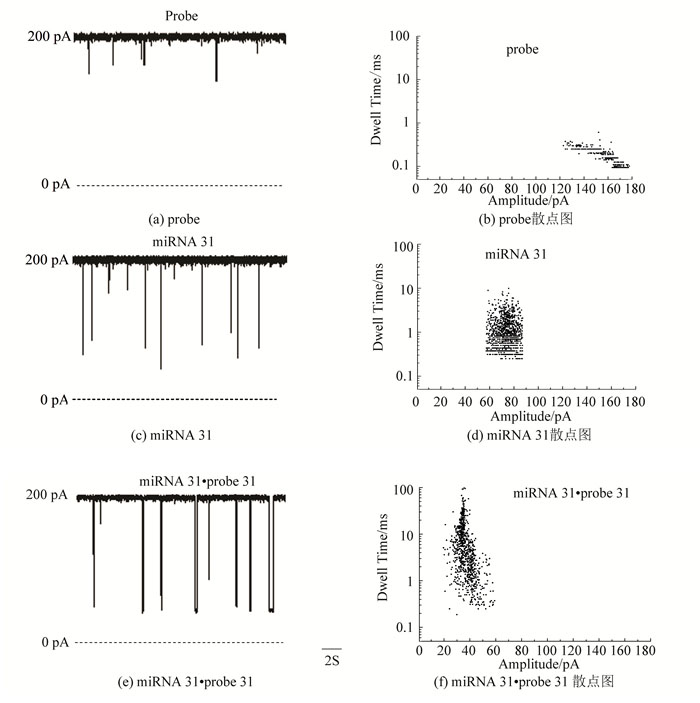
|
图 2 纳米通道检测不同核酸样品典型的单通道信号及其相对的散点图 Figure 2 Nanopore detection of various nucleic acid samples and corresponding scatter diagrams |
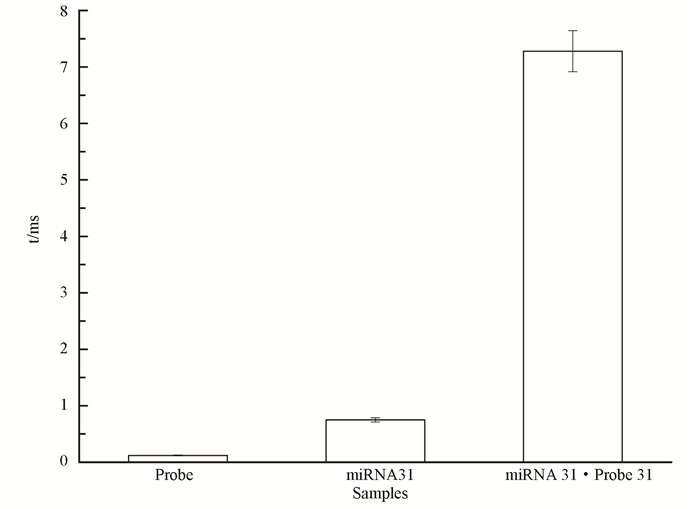
|
图 3 不同核酸样品的平均阻塞时间 Figure 3 Mean residence time values of various nucleic acid samples |
实验2.2设计的Probe与miRNA 31中间配对的miRNA 31·probe 31,为了更好地验证该设计方法更有利于后期结果的分析,做了与miRNA 31不同位置配对位置的对比实验。图 4分别是Probe与miRNA 31后端配对和前端配对的单通道信号与散点分析图。从结果可以看出无论后配还是前配该miRNA都有两种信号,这是因为miRNA通过通道时的位置顺序不一样。尾端配对的miRNA进入通道时若是前端先通过,由于前端部分是单链则通过的快,阻塞时间短,脉冲电流小,信号就短,则图中的短信号就有可能是尾端配对miRNA前端通过通道时的信号但也有可能是未配对的Target与Probe信号;若是尾端先进入通道,由于尾端是双链则阻塞时间长,脉冲电流大,信号长,则图中的长信号就是尾端配对miRNA尾端先进入通道时的信号。反之前端配对的miRNA也同样理论。结果证明无论哪端配对通过纳米通道都有两种信号结果而且增加了结果分析的难度,所以我们选取与miRNA 31中间位置完全匹配的Probe。
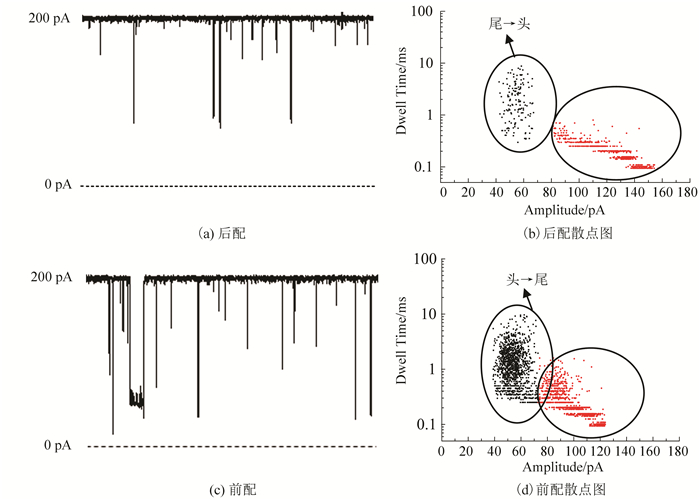
|
图 4 纳米单通道检测miRNA 31后配与前配典型的单通道记录及对应的散点图 Figure 4 Nanopore detection of miRNA 31 end match and front match and corresponding scatter diagrams |
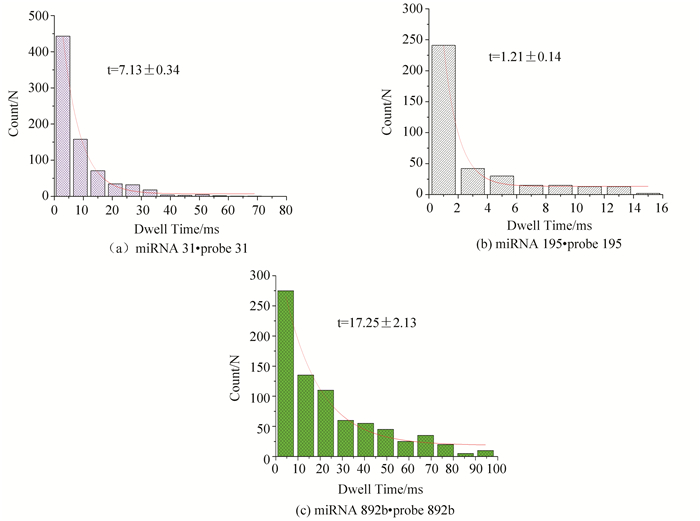
由于探针probe只能与目标miRNA的识别区完全互补配对,所以不同的miRNA·probe经过纳米孔通道时产生的特征信号不同,因此就可以有效区分多种miRNAs。基于此,设计了probe 195来检测miRNA 195,设计了probe 892b来检测miRNA 892b。miRNA 195·probe 195的阻塞时间t=1.21±0.14 ms(见图 5(b)),约为miRNA 31·probe 31的阻塞时间7.13±0.34 ms的1/6(见图 5(a))。miRNA 892b·probe 892b的阻塞时间t=17.25±2.13 ms(见图 5(c)),大于miRNA 31·probe 31的阻塞时间。因此,通过调控probe序列,可以使miRNA 31·probe31、miRNA195·probe 195、miRNA 892b·probe 892b三种复合物分子具有不同特征信号和阻塞时间,因此可以有效区分miRNA 31、miRNA 195、miRNA 892b三种miRNAs。
|
图 5 不同miRNAs·probe过孔的特征电流信号的阻塞时间柱状统计图 Figure 5 Residence time histogram of typical current for different miRNAs·probes |
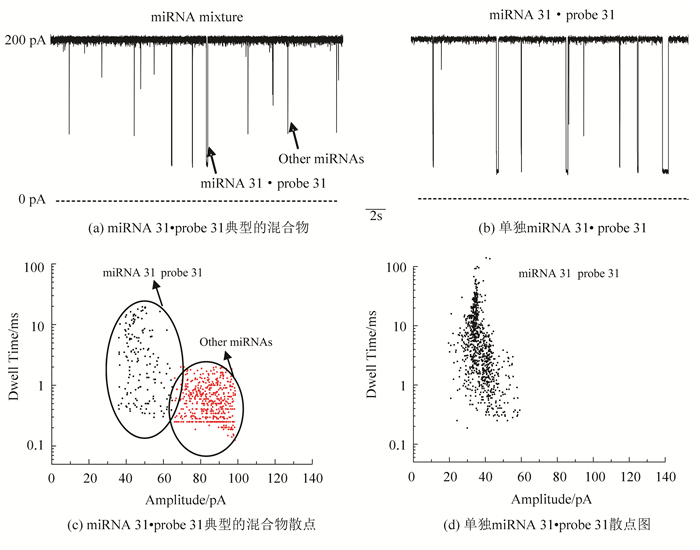
查找其他5种癌症miRNA:肺癌miRNA 25,胰腺癌miRNA 221,卵巢癌miRNA 215,胃癌miRNA 106a,前列腺癌miRNA 141。将这五种癌症的miRNA与miRNA 31·probe 31混合,然后通过纳米通道检测miRNA 31·probe 31(见图 6),实验结果分析表明混合物中有两类信号:短信号是其他5种癌症的miRNA,长信号是miRNA 31·probe 31的信号,该方法可以有效地将miRNA 31·probe 31成功检测出来且与单独检测miRNA 31·probe 31的信号一致,说明该方法可以从复杂的样品中快速有效的检测miRNA 31,同样也说明如果设计其他五种癌症的Probe,该方法也能将各自的miRNA检测出来。
|
图 6 纳米孔检测混合miRNAs中的miRNA 31·probe 31典型的混合物及单独miRNA 31·probe 31单通道记录及对应的散点图 Figure 6 Nanopore detection of miRNA 31·probe 31 in the presence of other miRNAs and the corresponding scatter diagrams |
1) 本文采用纳米通道技术对乳腺癌的早期检测进行研究。实验中设计了特殊序列的probe与目标miRNA结合,通过生物纳米孔单通道单技术对miRNA·probe复合物分子进行高灵敏度、快速检测,实现了对乳腺癌miRNAs的高效检测。
2) miRNA 31·probe 31、miRNA195·probe 195、miRNA 892b·probe 892b三种复合物分子在通过纳米孔通道时,由于不同的probe序列,形成了不同的信号形态、不同堵塞时间的特征信号,最终实现此三种miRNAs的有效区分。
3) 纳米孔单通道技术以其无需标记、无需扩增、高分辨、高灵敏等特点为miRNA的检测提供了的有效的平台,也在癌症的早期诊断、治疗方面有广阔的发展前景。
| [1] |
PARK J L, PARK S M, KWON O H, et al. Microarray screening and qRT-PCR evaluation of microRNA markers for forensic body fluid identification[J]. Electrophoresis, 2014, 35(21-22): 3062-3068. DOI:10.1002/elps.201400075 (  0) 0) |
| [2] |
VARALLYAY E, BURGYAN J, HAVELDA Z. MicroRNA detection by northern blotting using locked nucleic acid probes[J]. Nature Protocols, 2008, 3(2): 190-196. DOI:10.1038/nprot.2007.528 (  0) 0) |
| [3] |
KASIANOWICZ J J, BRANDIN E, BRANTON D, et al. Characterization of individual polynucleotide molecules using a membrane channel[J]. Proceedings of the National Academy of Science of the United States of America, 1996, 93(24): 13770-13773. DOI:10.1073/pnas.93.24.13770 (  0) 0) |
| [4] |
CAO C, LIAO D F, YING Y L, et al. Detection of single oligonucleotide by an aerolysin nanopore[J]. Acta Chimica Sinica, 2016, 74(9): 734-737. DOI:10.6023/A16070352 (  0) 0) |
| [5] |
WANG S, ZHAO Z, HAQUE F, et al. Engineering of protein nanopores for sequencing, chemical or protein sensing and disease diagnosis[J]. Current Opinion in Biotechnology, 2018, 51: 80-89. DOI:10.1016/j.copbio.2017.11.006 (  0) 0) |
| [6] |
MANRAO E A, DERRINGTON I M, LASZLO A H, et al. Reading DNA at single-nucleotide resolution with a mutant MspA nanopore and phi29 DNA polymerase[J]. Nature Biotechnology, 2012, 30(4): 349-353. DOI:10.1038/nbt.2171 (  0) 0) |
| [7] |
NICOLE R. Disruptive nanopores[J]. Nature Methods, 2013, 10(1): 35. DOI:10.1038/nmeth.2292 (  0) 0) |
| [8] |
STEFUREAC R I, LONG Y T, KRAATZ H B, et al. Transport of α-Helical peptides through α-Hemolysin and aerolysin pores[J]. Biochemistry, 2006, 45(30): 9172-9179. DOI:10.1021/bi0604835 (  0) 0) |
| [9] |
SUTHERLAND T C, LONG Y T, STEFUREAC R I, et al. Structure of peptides investigated by nanopore analysis[J]. Nano Letters, 2004, 4(7): 1273-1277. DOI:10.1021/nl049413e (  0) 0) |
| [10] |
WANG H Y, YING Y L, LI Y, et al. Nanopore analysis of β-amyloid peptide aggregation transition induced by small molecules[J]. Analytical Chemistry, 2011, 83(5): 1746-1752. DOI:10.1021/ac1029874 (  0) 0) |
| [11] |
HU Z L, DU J H, YING Y L, et al. Single-molecule analysis of colorectal cancer-associated MicroRNAs via a biological nanopore[J]. Acta Chimica Sinica, 2017, 75: 1087-1090. DOI:10.6023/A17090433 (  0) 0) |
| [12] |
JASINSKI D, HAQUE F, BINZEL D W, et al. Advancement of the emerging field of RNA nanotechnology[J]. ACS Nano, 2017, 11(2): 1142-1164. DOI:10.1021/acsnano.6b05737 (  0) 0) |
| [13] |
SMITH J A, BRAGA A, VERHEYEN J, et al. RNA nanotherapeutics for the amelioration of astroglial reactivity[J]. Molecular Therapy Nucleic Acids, 2018, 10: 103-121. DOI:10.1016/j.omtn.2017.11.008 (  0) 0) |
| [14] |
HAQUE F, PI F, ZHAO Z, et al. RNA versatility, flexibility, and thermostability for practice in RNA nanotechnology and biomedical applications[J]. Wiley Interdisciplinary Reviews:RNA, 2018, 9(1): e1452. DOI:10.1002/wrna.1452 (  0) 0) |
| [15] |
WANG G, ZHAO Q, KANG X, et al. Probing mercury(Ⅱ)-DNA interactions by nanopore stochastic sensing[J]. Journal of Physical Chemistry B, 2013, 117: 4763-4769. DOI:10.1021/jp309541h (  0) 0) |
| [16] |
WANG L, HAN Y, ZHOU S, et al. Nanopore biosensor for label-free and real-time detection of anthrax lethal factor[J]. ACS Applied Materials & Interfaces, 2014, 6(10): 7334-7339. (  0) 0) |
| [17] |
KAMM R C, SMITH A G. Nucleic acid concentrations in normal human plasma[J]. Clinical Chemistry, 1972, 18(6): 519-522. (  0) 0) |
| [18] |
XU C, HAQUE F, JASINSKI D L, et al. Favorable biodistribution, specific targeting and conditional endosomal escape of RNA nanoparticles in cancer therapy[J]. Cancer Letters, 2017, 5, 414: 57-70. DOI:10.1016/j.canlet.2017.09.04 (  0) 0) |
| [19] |
PI F, ZHANG H, LI H, et al. RNA nanoparticles harboring annexin A2 aptamer can target ovarian cancer for tumor-specific doxorubicin delivery[J]. Nanomedicine, 2017, 13(3): 1183-1193. DOI:10.1016/j.nano.2016.11.015 (  0) 0) |
| [20] |
LU J, GETZ G, MISKA E A, et al. MicroRNA expression profiles classify human cancers[J]. Nature, 2005, 435(7043): 834-838. DOI:10.1038/nature03702 (  0) 0) |
| [21] |
LORIO M V, FERRACIN M, LIU C G. MicroRNA gene expression deregulation in human breast cancer[J]. Cancer Research, 2005, 65(16): 7065-7070. DOI:10.1158/0008-5472.CAN-05-1783 (  0) 0) |
| [22] |
CHEN X, BA Y, MA L J. Characterization of microRNAs in serum: A novel class of biomarkers for diagnosis of cancer and other diseases[J]. Cell Research, 2008, 18(10): 997-1006. DOI:10.1038/cr.2008.282 (  0) 0) |
| [23] |
元艳, 汝琦. miRNA检测技术进展及其在医学检验中的应用[J]. 医学信息, 2011, 8: 3809-3809. YUAN Yan, RU Qi. Progress of microRNA detection technology and its application in laboratory medicine[J]. Medical Information, 2011, 8: 3809-3809. DOI:10.3969/j.issn.1006-1959.2011.08.338 (  0) 0) |
| [24] |
石明, 仇雪梅, 樊红. 血浆miRNA检测在肿瘤临床应用的研究进展[J]. 东南大学学报(医学版), 2012, 31(1): 122-125. SHI Ming, QIU Xuemei, FAN Hong. Advances in plasma miRNA detection in tumor clinical applications[J]. Journal of Southeast University (Med Sci Edi), 2012, 31(1): 122-125. DOI:10.3969/j.issn.1671-6264.2012.01.030.j.issn.1671-6264.2012.01.030 (  0) 0) |
| [25] |
XIAO C C, RAJEWSKY K. MicroRNA control in the immune system: Basic principles[J]. Cell, 2009, 136(1): 26-36. DOI:10.1016/j.cell.2008.12.027 (  0) 0) |
| [26] |
ZHAO H, SHEN J, MEDICO L, et al. A pilot study of circulating miRNAs as potential biomarkers of early stage breast cancer[J]. PLoS ONE, 2010, 5(10): e13735. DOI:10.1371/journal.pone.0013735 (  0) 0) |
| [27] |
张学营, 甄林林, 韩学东, 等. 血浆中miR-31水平与乳腺癌关系的研究[J]. 现代医学, 2012, 40(2): 133-137. ZHANG Xueying, ZHEN Linlin, HAN Xuedong, et al. Circulating miR-31 in plasm of patient with breast cancer[J]. Modern Medical Journal, 2012, 40(2): 133-137. DOI:10.3969/j.issn.1671-7562.2012.02.001. (  0) 0) |
 2018, Vol. 16
2018, Vol. 16


