植物激素脱落酸(Abscisic acid,ABA)属于含有15个碳原子的萜类化合物,其作用贯穿于从种子萌发到生殖发育的植物生命周期的多种生理过程[1]:包括种子休眠的建立、胚胎成熟、种子萌发、根发育和叶片的衰老等[2]。ABA在干旱、冷、盐和渗透等非生物胁迫[2],以及抵御病原菌侵害等生物胁迫生理过程中也发挥作用[3]。环境胁迫通常导致植物ABA水平的提高,进而引起生理学水平的适应性改变,包括气孔关闭、生长抑制和基因响应等[4-5]。
泛素(Ubiquitin)的主要功能是标记需要分解的蛋白使其被水解。泛素介导的蛋白质降解途径是真核生物体内最主要的蛋白水解途径[6]。泛素-蛋白酶体系统(Ubiquitin-proteasome system,UPS)的组成主要包括,泛素活化酶E1、泛素耦联酶E2、泛素连接酶E3和26S蛋白酶体[7]。泛素连接酶E3可归为HECT、RING/U-box、SCF (Skp, Cullin, F-box)复合体和APC (Anaphase promoting complex)复合体4种类型,其中SCF复合物是最大的一个家族[8]。SCF复合体由四个亚基组成,包括SKP1(拟南芥中为Arabidopsis SKP1-like[ASK])、CUL1、RBX1(RING-BOX1)和F-box蛋白[8]。F-box蛋白作为SCF复合体的组成部分,通过特异结合和识别而介导靶蛋白的降解[9]。
F-box蛋白在植物生长发育的多种信号途径中起重要作用,包括植物激素ABA、赤霉素、乙烯和生长素信号途径[10-11]。例如:F-box蛋白FBS1[12]、EDL3[13]和FOA2[14]被证明在ABA信号通路、拟南芥开花转换过程中具有重要调控作用。拟南芥基因组中约含有700个F-box基因[15]。为了进一步发掘ABA信号通路相关的F-box基因,本研究采用生物信息学方法,筛选了受ABA上调或下调的拟南芥候选F-box基因。在此基础上,进一步分析了候选基因启动子序列的顺式作用元件、候选基因的表达谱,以及其互作蛋白,为深入研究ABA调节植物生长发育和抗逆的分子机制提供线索。
1 材料和方法 1.1 芯片数据的获取以“Abscisic acid”、“Arabidopsis”和“Wildtype”为关键词,在NCBI(National Center for Biotechnology Information)网站提供的GEO数据库(https://www.ncbi.nlm.nih.gov/gds/?term=)中进行检索,筛选了5组芯片[16]。其登录号(Accession)分别是GSE3454、GSE7112、GSE8955、GSE19520和GSE28800。在挑选芯片数据时,仅选取以野生型拟南芥为材料经过ABA处理的数据。其中GSE19520包括生长5周的Col-0成熟叶片的保卫细胞组织数据集(野生型vs ABA处理),以及生长5周的Col-0叶片组织数据集(野生型vs ABA处理)。GSE3454、GSE7112、GSE8955和GSE28800每组仅有一组数据集(野生型vs ABA处理)。因此,5组芯片共获取6组数据集。
1.2 芯片数据分析在6组数据集中初步筛选受ABA上调或者下调的候选F-box基因,具体步骤如下:
(1) 利用Excel提供的QUARTILE函数分别计算6组数据集中基因表达量的25分位值,排除小于25分位值的基因。
(2) 利用GEO数据库自带的在线分析程序GEO2R(https://www.ncbi.nlm.nih.gov/geo/geo2r/)分别求出6组数据集的P值和LogFC,筛选P < 0.05的差异表达基因。
(3) 分别将6组数据集(1)和(2)的筛选结果取交集;将各交集与所有F-box基因对比,再次取交集。获得6组差异表达F-box基因初筛结果。
(4) 对6组F-box基因粗筛结果两两取交集,获得差异表达候选基因。
1.3 启动子分析在Tair网站(https://www.arabidopsis.org/)检索差异表达候选基因的启动子序列(起始密码子上游1500bp),使用PlantCARE网站(http://bioinformatics.psd.ugent.be/webtools/plantcare/html/)提供的在线分析工具对启动子序列进行分析,并统计候选基因中存在的相关顺式作用元件,包括ABA、干旱、胁迫、分生组织表达和光照相关的顺式作用元件[17]。
1.4 差异表达候选F-box基因的表达谱和蛋白互作因子分析利用Genevestigator(https://genevestigator.com/gv/)分别分析候选F-box基因在不同组织器官中的表达情况,在激素(ABA、IAA)、冷、热、干旱、盐和不同光照处理条件下的表达情况,以及在种子萌发过程中的表达情况[18]。通过BioGrid数据库(https://thebiogrid.org)搜索候选F-box基因的蛋白互作因子[19]。
1.5 ABA处理将野生型拟南芥Col-0的种子在无菌条件下进行表面消毒,即:用70%的乙醇消毒30 s,然后加入15% bleach消毒3 min。用无菌水清洗5~6次后,播种于1/2MS培养基上,在4 ℃黑暗下低温处理2 d,然后转到持续白光下培养14 d。将幼苗取出,在100 μmol ABA溶液中分别浸泡0、1、2、3 h后收集材料(ABA处理前收集样品的时间定为0 h)。将材料置于液氮中冷冻,然后保存于-80 ℃冰箱,用于RNA分析。
1.6 实时荧光定量PCR分析采用TRIGene试剂(Takara,Japan)抽提总RNA,然后用PrimeScript RT Reagent Kit with gDNA Eraser试剂盒(Takara,Japan)将RNA逆转录,获得cDNA。随后,用SYBR Premix Ex TaqTM试剂盒(Takara,Japan)进行实时荧光定量PCR实验。实时荧光定量PCR实验在Mx3000 Real-time PCR系统(Stratagene,The Netherlands)进行,反应程序为:95 ℃ 5 min,95 ℃ 30 s,58 ℃ 20 s,72 ℃ 30 s,循环数为45个。
PCR的引物为:At1g26930(5’-ATTAATTGTATAATT- TTTTTGTCAGAG-3’和5’-TTACTAGGCAAGAAACA-GTCGGT-3’);At1g80110(5’-CAAAAAGCTGATCA-TATAAAAACAT-3’和5’-CCCTAATTGTATCCTACT-CCTAAA-3’);At2g18910(5’-TACAACAAACCAA-ATGACCAATG-3’和5’-CTTGAGGAAAGTTCTCTT-CAGATG-3’);At3g23880(5’-ATCTTGGATAGAAA-CATTAGGAACA-3’和5’-TACCCAGTTTCAAACAG-CATAAGT-3’);At4g14090(5’-AAGGCTGCTTTGTA-ATGTTAAT-3’和5’-ACGAGGATGAGTAGTGGGT-TT-3’);At5g04010(5’-TCAAGACATCGTCAACGA-ACAG-3’和5’-TGGTGGGAGAAGTCAAGCC-3’);Actin2(5’-CACTGTGCCAATCTACGAGGGT-3’和5’-ACAAACGAGGGCTGGAACAAG-3’)。其中,Actin2为拟南芥持家基因,其表达水平作为内参,用来衡量待测基因的相对表达水平。每个实验重复3次。
2 结果与分析 2.1 筛选受ABA上调或下调的拟南芥F-box基因并分析其启动子拟南芥基因组中存在约700个F-box基因[15]。根据1.2芯片数据分析的方法,对GEO数据库下载的5组基因芯片中的6组数据集进行分析,初步筛选获得38个受ABA上调或者下调的拟南芥候选F-box基因(见表 1)。
| 表 1 候选拟南芥F-box基因的统计 Table 1 Statistics of selected Arabidopsis F-box genes |
利用PlantCARE分析候选基因的启动子序列,结果显示,38个候选F-box基因的顺式作用元件主要为ABA、干旱、胁迫、分生组织表达和光照相关的作用元件(见表 2)。ABRE是响应ABA的顺式作用元件[20]。38个候选F-box基因中,具有不同数量ABRE元件的基因有13个,其中At3g53000(PP2-A15)基因的启动子中含有5个ABRE元件。存在干旱响应元件的基因有24个;存在胁迫响应元件的基因有19个。这进一步说明了这38个候选基因与ABA的相关性。此外,存在分生组织表达响应元件的基因有13个;所有38个候选基因都带有不同数量的光响应元件。
| 表 2 候选拟南芥F-box基因的响应元件 Table 2 Motifs of selected Arabidopsis F-box genes |
实时荧光定量PCR可用于鉴定基因的mRNA表达水平。从筛选到的38个候选基因中,选择表达量改变倍数最大的6个F-box基因。在筛选这6个基因时,选择5组芯片(包含6组数据)中改变倍数大于2的所有F-box基因,并在所有符合标准的基因中,选择至少在一组芯片中改变倍数大于5,且在至少两组芯片中上调或下调表现一致的基因。这6个基因分别是At1g26930,At1g80110,At2g18910,At3g23880,At4g14090和At5g04010。为了更直观的展示这6个基因在各个数据集中的情况,通过折线图列出了各数据集中基因的LogFC(见图 1)。
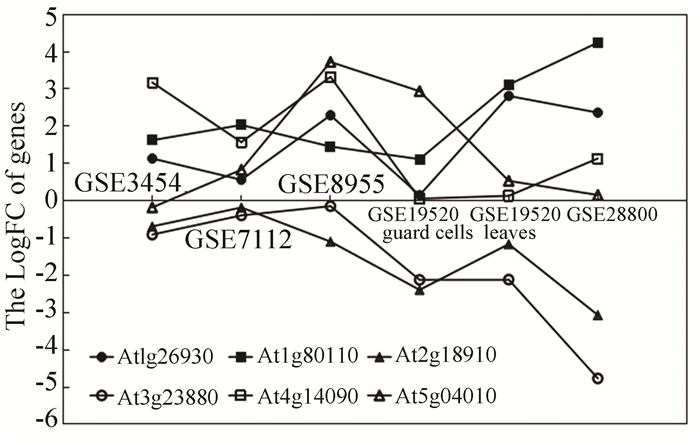
|
图 1 筛选到的6个基因在各数据集中的LogFC Figure 1 LogFC of 6 selected genes in each dataset |
为进一步确定生物信息学分析结果的可靠性,我们采用实时荧光定量PCR鉴定了这6个F-box基因在ABA处理拟南芥中的表达量。结果显示,这6个F-box基因对外源ABA处理均有响应(见图 2),且表达量变化趋势与芯片结果大体一致。例如,随ABA处理时间延长,At1g26930、At1g80110和At5g04010基因的表达量增加(见图 2(a)、2(b)和2(f)),符合芯片的上调结果。在ABA处理0、1、2 h时,At2g18910和At3g23880基因的表达量表现出下降的趋势(见图 2(c)、2(d)),符合芯片的下调结果。At4g14090在ABA处理后,表达量下降,但在1、2、3 h表现出表达量增加的趋势(图 2 (e)),同样可以解释芯片结果。其中,At2g18910和At3g23880基因在ABA处理2 h时达到最低,然后再升高,而At4g14090基因则在ABA处理1 h时达到最低,然后再升高。这可能是由于不同的基因受ABA调控的模式不完全一致。上述这些基因表达受ABA调节的分析结果,进一步证实了筛选结果的可靠性。
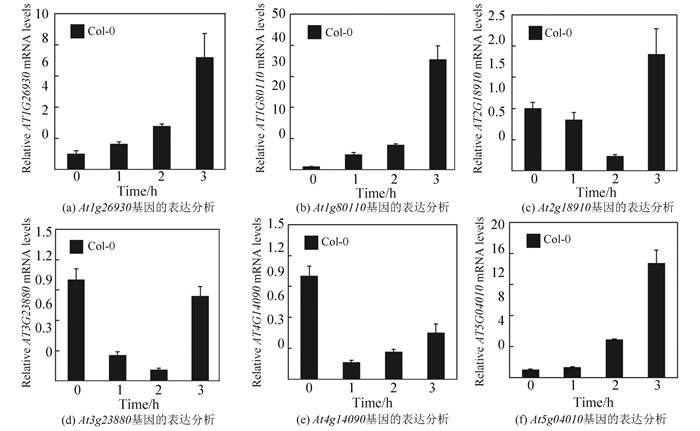
|
图 2 外源ABA处理对6个候选F-box基因表达的影响 Figure 2 Effect of exogenous ABA treatment on the expression of 6 candidate F-box genes |
利用BioGrid数据库检索38个候选F-box蛋白的蛋白互作因子,发现25个蛋白存在蛋白互作因子(见表 3)。其中,有20个蛋白与SKP1相互作用,有18个蛋白与ASK2相互作用。此外,At1g80110编码蛋白PP2-B11与ABI5相互作用,At5g49980编码蛋白AFB5与IAA相关蛋白互作。对蛋白互作因子的探索可以为今后研究这些基因的功能提供思路。
| 表 3 候选F-box蛋白的蛋白互作因子 Table 3 Interactors of selected F-box protein |
为进一步了解候选F-box基因在植物特定部位的功能作用。我们利用Genevestigator对38个候选F-box基因,在拟南芥不同组织器官中的表达进行了分析。从图 3中可看出,38个候选基因在保卫细胞中都有表达,其中At1g12710、At1g68050、At2g24540和At5g52120基因的表达量很高,可能与气孔的开闭相关。部分基因如At1g23780、At1g26930、At2g02870、At2g36090和At4g10930基因在种皮中的表达量较高,包括外种皮、普通种皮、合点端种皮。部分基因如At1g13200、At1g53320、At2g17310、At2g44030、At3g53000和At5g42360在花粉和精子中高表达。所有38个候选基因在衰老的叶片中均有表达,其中At1g15670、At2g02360、At2g25490和At4g14090表现为高表达。这表明了部分候选基因可能参与ABA信号通路影响植物的生长发育过程。
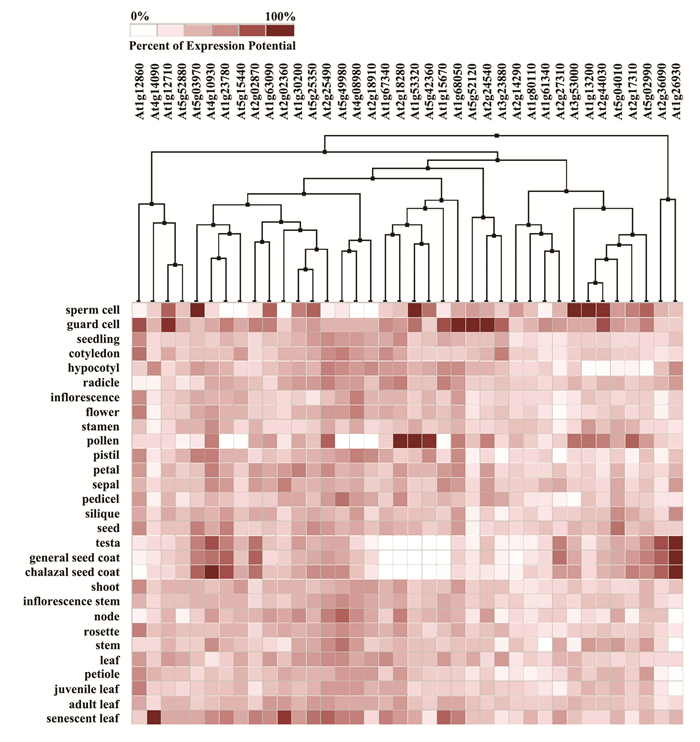
|
图 3 候选F-box基因在拟南芥不同组织中的表达谱 Figure 3 Expression profile of candidate F-box genes in different tissues of Arabidopsis |
激素之间通常可以形成复杂的调控网络,同时环境信号的作用,如温度、胁迫、光等信号同样与激素信号作用密不可分[21]。此外,ABA在促进种子休眠的建立和维持、防止早熟种子萌发、增强干燥种子的耐受性等方面发挥作用[22]。因此,利用Genevestigator分析了38个候选F-box基因响应激素(ABA、IAA)、胁迫(冷、热、干旱和盐)和光照,以及种子萌发过程中的表达谱。
从图 4可看出,大部分F-box基因对分析的激素及环境因子都有响应。在ABA处理的多组实验中At1g26930、At1g80110和At2g24540基因表达明显上调,At1g15670、At2g18910和At3g23880基因表达则明显下调。候选基因对IAA的响应并不明显。大多数基因对温度、干旱和盐胁迫有不同程度的响应。除个别基因外,候选基因对光照的响应并不强烈,且对于蓝光、红光和远红光的响应并无差别。但日照的长度对部分基因似乎有明显的影响。在种子萌发过程中,有更多的基因表现出上调或下调,且表现出下调的基因居多。在种子萌发过程中,如果以干燥种子中的表达量作为基准,则基因表达上调和下调明显;如果以吸胀处理后种子中的表达量为基准,则基因的表达量变化很小。这说明大多数基因在种子的吸胀过程中,表达量发生巨大的改变。综上分析结果,说明大部分候选基因可能通过ABA信号通路,参与植物种子萌发以及对环境胁迫的应答。
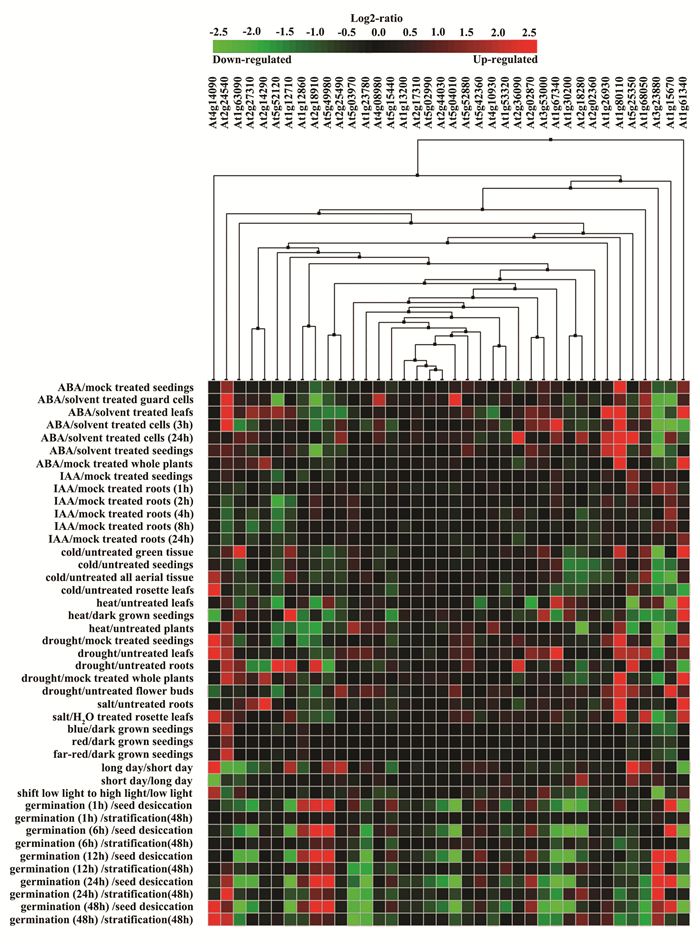
|
图 4 候选F-box基因在不同激素或环境因子处理下的表达谱 Figure 4 Expression profile of candidate F-box genes under different hormones or environment factors |
本研究利用GEO基因芯片数据,筛选受ABA调节的拟南芥F-box基因,初步筛选到38个差异表达的F-box基因。通过分析38个F-box基因的上游启动子序列,发现候选基因主要存在ABA、干旱、胁迫、分生组织表达和光照响应相关顺式作用元件。其中,13个基因具有不同数量的ABA响应元件(ABRE),24个基因存在干旱响应元件,19个基因存在胁迫响应元件,表明这些基因可能参与植物响应ABA及胁迫信号。
从38个候选基因中,筛选表达量改变倍数最大的6个基因,进行实时荧光定量PCR分析。结果显示,在ABA处理条件下,At1g26930、At1g80110和At5g04010表现出明显上调趋势,At2g18910和At3g23880表现出明显下调趋势,与基因芯片中的上调和下调结果相一致。此外,At4g14090在基因芯片分析结果中,对ABA的响应表现为上调,而在本实验中则表现为先下调后上调。对此,我们将通过缩短ABA处理的时间间隔,进一步探索该候选基因受ABA调节的模式。
本研究选择的6个候选基因的启动子中,至少含有ABA、干旱和胁迫相关响应元件中的一种。在拟南芥不同激素或环境因子的基因表达谱中,除At4g14090外,其他5个基因的表达均受ABA明显调节;除At1g26930外,其他5个基因的表达量在种子萌发过程中均显著改变;并且这6个基因对干旱均有一定程度的响应(见图 4)。说明这6个基因可能通过ABA信号通路参与植物种子萌发和干旱胁迫响应过程。
4 结论通过生物信息学方法,筛选到了受ABA调节的拟南芥F-box基因,并证明了其可靠性。这些基因可能通过ABA信号通路在植物种子萌发、生长发育、衰老以及抗逆境过程中起到重要作用。研究结果对探索ABA调控植物生长发育和抗逆的分子机制提供了重要的参考。
| [1] |
ZHANG X R, GARRETON V, CHUA N H. The AIP2 E3 ligase acts as a novel negative regulator of ABA signaling by promoting ABI3 degradation[J]. Genes & Development, 2005, 19(13): 1532-1543. DOI:10.1101/gad.1318705 (  0) 0) |
| [2] |
许智宏, 薛红卫. 植物激素作用的分子机理[M]. 上海: 科学技术出版社, 2012. XU Zhihong, XUE Hongwei. Plant hormones: Function and molecular mechanism[M]. Shanghai: Science and Technology Press, 2012. (  0) 0) |
| [3] |
LEUNG J, GIRAUDAT J. Abscisic acid signal transduction[J]. Annual Review of Plant Physiology & Plant Molecular Biology, 1998, 49(1): 199-222. DOI:10.1146/annurev.arplant.49.1.199 (  0) 0) |
| [4] |
AXEL H, THOMAS H, MARTIN L, et al. Homeodomain protein ATHB6 is a target of the protein phosphatase ABI1 and regulates hormone responses in Arabidopsis[J]. The EMBO Journal, 2002, 21(12): 3029-3038. DOI:10.1093/emboj/cdf316 (  0) 0) |
| [5] |
BUSK P K, PAGES M. Regulation of abscisic acid-induced transcription[J]. Plant Molecular Biology, 1998, 37(3): 425-435. DOI:10.1023/A:1006058700720 (  0) 0) |
| [6] |
JUDY C. The ubiquitination machinery of the ubiquitin system[J]. The Arabidopsis Book, 2014, 12(e0174): e0174. DOI:10.1199/tab.0174 (  0) 0) |
| [7] |
CLAUS S, LUZIRINA C V. Cullin-containing E3 ubiquitin ligases in plant development[J]. Current Opinion in Plant Biology, 2004, 7(6): 677-686. DOI:10.1016/j.pbi.2004.09.009 (  0) 0) |
| [8] |
EDWARD T K, MICHELE P. The F-box protein family[J]. Genome Biology, 2000, 1(5): 1-7. DOI:10.1186/gb-2000-1-5-reviews3002 (  0) 0) |
| [9] |
李丽, 刘德荣, 李新梅, 等. 受赤霉素调节的拟南芥F-box基因筛选分析[J]. 生物信息学, 2015, 13(3): 150-157. LI Li, LIU Derong, LI Xinmei, et al. Screening and analysis of gibberellin-regulated F-box genes in Arabidopsis[J]. Chinese Journal of Bioinformatics, 2015, 13(3): 150-157. DOI:10.3969/j.issn.1672-5565.2015.03.02 (  0) 0) |
| [10] |
WENDY J L, SOPHIA L S. Abiotic stress tolerance mediated by protein ubiquitination[J]. Journal of Experimental Botany, 2012, 63(2): 599-616. DOI:10.1093/jxb/err310 (  0) 0) |
| [11] |
吴丹, 唐冬英, 李新梅, 等. F-box蛋白在植物生长发育中的功能研究进展[J]. 生命科学研究, 2015, 19(4): 362-367. WU Dan, TANG Dongying, LI Xinmei, et al. Progress of F-box protein function in plant growth and development[J]. Life Science Research, 2015, 19(4): 362-367. DOI:10.16605/j.cnki.1007-7847.2015.04.012 (  0) 0) |
| [12] |
LAUREN E G, KRISTEN K, KAREN X C, et al. Transcriptome analysis uncovers Arabidopsis F-box STRESS INDUCED 1 as a regulator of jasmonic acid and abscisic acid stress gene expression[J]. BMC Genomics, 2017, 18(533): 1-15. DOI:10.1186/s12864-017-3864-6 (  0) 0) |
| [13] |
PETRA K, STEPHAN P, MICHAEL L, et al. EDL3 is an F-box protein involved in the regulation of abscisic acid signaling in Arabidopsis thaliana[J]. Journal of Experimental Botany, 2011, 62(15): 5547-5560. DOI:10.1093/jxb/err236 (  0) 0) |
| [14] |
HE Reqing, YU Dashi, LI Xinmei, et al. F-box gene FOA2 regulates GA-and ABA-mediated seed germination in Arabidopsis[J]. Science China Life Sciences, 2016, 59(11): 1-3. DOI:10.1007/s11427-016-0098-3 (  0) 0) |
| [15] |
GAGNE J M, DOWNES B P, SHIU S H, et al. The F-box subunit of the SCF E3 complex is encoded by a diverse superfamily of genes in Arabidopsis[J]. Proceedings of the National Academy of Sciences, 2002, 99(17): 11519-11524. DOI:10.1073/pnas.162339999 (  0) 0) |
| [16] |
EDGAR R, DOMRACHEV M, LASH A E. Gene expression omnibus: NCBI gene expression and hybridization array data repository[J]. Nucleic Acids Research, 2002, 30(1): 207-2l. DOI:10.1093/nar/30.1.207 (  0) 0) |
| [17] |
LESCOT M, DÉHAIS P, THIJS G, et al. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research, 2002, 30(1): 325-327. DOI:10.1093/nar/30.1.325 (  0) 0) |
| [18] |
HRUZ T, LAULE O, SZABO G, et al. Genevestigator v3: A reference expression database for the meta-analysis of tanscriptomes[J]. Advances in Bioinformatics, 2008, 2008(420747): 1-5. DOI:10.1155/2008/420747 (  0) 0) |
| [19] |
STARK C, BREITKREUTZ B J, REGULY T, et al. BioGRID: A general repository for interaction datasets[J]. Nucleic Acids Research, 2006, 34(Database issue): D535-D539. DOI:10.1093/nar/gkj109 (  0) 0) |
| [20] |
JAEHOON L, HYEJIN Y, WILLIAM T, et al. DWA1 and DWA2, two Arabidopsis DWD protein components of CUL4-Based E3 ligases act together as negative regulators in ABA signal transduction[J]. The Plant Cell, 2010, 22(6): 1716-1732. DOI:10.1105/tpc.109.073783 (  0) 0) |
| [21] |
KONG Lingyao, CHENG Jinkui, ZHU Yujuan, et al. Degradation of the ABA co-receptor ABI1 by PUB12/13 U-box E3 ligases[J]. Nature Communication: Nature Research, 2015, 6(8630): 1-19. DOI:10.1038/ncomms9630 (  0) 0) |
| [22] |
KENJI M, JIYOUNG L, JING B J, et al. Sumoylation of ABI5 by the Arabidopsis SUMO E3 ligase SIZ1 negatively regulates abscisic acid signaling[J]. PNAS, 2009, 106(13): 5418-5423. DOI:10.1073/pnas.0811088106 (  0) 0) |
 2018, Vol. 16
2018, Vol. 16


