通过物种学名、分类号(Taxon)、任意一段核酸或蛋白质的序列,判定其属于什么物种及其详细分类的信息如何,是生物信息分析的最为基础且重要的环节。生物分类信息的获取包含了生物分类的阶层系统和生物检索表的编制方法,是了解生物多样性、丰度、进化、遗传与变异的基础。
目前,一些大型生物学数据库包含了众多的生物学资源,可以通过多种方式提供物种信息的鉴定,最常用的是美国国立卫生研究院建立的美国国家生物技术信息中心(NCBI,Na-tional Center for Biotechnology Information)[1-2],其汇集了基因序列及蛋白质序列的大量数据,可提供全面和权威的生物序列信息检索(https://www.ncbi.nlm.nih.gov/)。例如当我们想要鉴定一条未知序列时,通常的方法是这样的:输入待测序列到blast[3]中(https://blast.ncbi.nlm.nih.gov/Blast.cgi),根据比对分数的排名手动选择最佳比对序列,接着获取比对序列的GI后,进入NCBI,在Search后的下拉框中选择Nucleotide,把GI号输入GO前面的文本框中,点“GO”,即可以检索到所需序列[4]。把该序列中taxon继续到Taxonomy数据库中(https://www.ncbi.nlm.nih.gov/guide/taxonomy/)查找,即可获取该序列的分类信息。
在查询或比对后,科研人员在获取网站的分类信息(门、纲、目、科、属、种)时,一般是通过手动方式复制、拷贝的方式到本地的文档,这种机械式的获取的方式费时费力,经过笔者测试,手动下载10个比对结果的时间为1 086 s; 同时,如果想获取大批量信息也极容易出错。在查询或比对的页面出现结果后,如果通过编程在NCBI网站上自动或批量获取物种信息,将会使枯燥的操作省时省力且准确; 但令人遗憾的是,目前尚无解决这个自动下载环节的报道或软件。
基于上述问题,结合Perl语言易学、简洁、正则表达式强大、适用于生物信息数据处理、高效便捷字符串的处理能力等特点[5-6],针对NCBI网页源代码遵循HTML语言规范,内容的分布具有一定规律的特点以及研究者们在分类鉴定中最常使用的操作[7],拟用Perl语言编写能在NCBI网站自动或批量获取物种信息的实用性脚本[8],旨在解决查询或比对后传统手动获取分类信息的问题。
1 材料方法 1.1 计算机配置及软件运行环境Perl是“practical extraction and report language”的缩写[9],它是由Larry Wall设计编写的,并由他不断更新和维护,用于在UNIX环境下编程。Perl可以跨系统平台运行,可用于UNIX、Linux、MAC和Windows等系统环境下编程和运行。
序列分析中字符串处理占了大部分的内容,如数据库搜索,序列比对等,所以在选择计算语言时必须考虑能很方便地进行字符串处理[5]。Perl具有独特的正则表达式(Regular expression)是一种基于文本处理的语言。特别适用于对HTML页面的解析。Perl可以直接进行模式匹配,Perl语言的复杂性也就是体现在高效使用正则表达式进行的模式查找上。在生物信息学领域可以很方便的用Perl的正则表达式进行序列的解析。BioPerl[10]模块可谓是人尽皆知,而它对各种BLAST、FASTA结果的解析也使用了大量的正则表达式。
在脚本中使用了LWP (Library for Web Access in Perl)标准编程模块。LWP是用于访问Web网站的Perl模块包。使用其中的User Agent、Cookies、Response等类, 可以很方便地模拟浏览器的行为[11]。
使用Perl的LWP模块包之前,应在本地计算机中安装这个模块包[12]。以ActivePerl为例说明安装方法:使用“win+R”调用调出“运行”,并输入“cmd”键入回车打开命令行界面,接着输入“cpan LWP”键入回车确认便会自动下载LWP模块。
在本次编写软件的过程中,考虑到Max OS以及Linux预装了Perl环境,因此采用了版本较低的Win XP系统来搭建软件的运行环境。同时也在各种主流系统,如Windows 7、Windows10、Ubuntu等都进行了逐一测试,均运行良好。
1.2 编程的思路 1.2.1 通过物种分类号或物种学名获取NCBI分类信息的编程在NCBI的Taxonomy网站页面(https://www.ncbi.nlm.nih.gov/guide/taxonomy/),如何通过taxon来获取对应的“门、纲、目、科、属、种”信息。编程思路如图 1.PROGRESS:调用LWP模块访问浏览器,通过传入的taxon来获取查询结果页面的源代码,通过物种信息在源代码的特征筛选出“门纲目科属种”对应信息并保存。
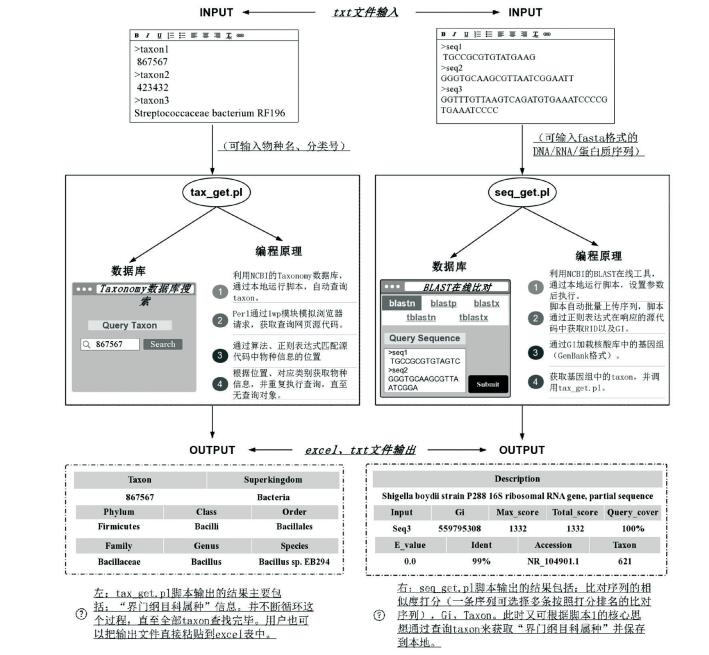
|
图 1 脚本tax_get.pl和seq_get.pl的编程原理 Figure 1 Principles of script tax_get.pl and seq_get.pl |
在NCBI的在线BLAST页面上,如何通过输入任意一段待测序列,获取比对后的物种信息。编程思路如图 1.PROGRESS:调用LWP模块访问浏览器,将待测序列上传到NCBI在线比对网站,选择对应的数据库以及BLAST方式后提交。当请求成功提交到NCBI服务器时,NBCI服务器会自动生成一个用于表示当前比对操作的Request ID(RID)。
当的脚本接收到NCBI服务器发回的响应后,首先脚本会根据用户设定的参数自动的从结果集中获取目标信息,目标信息包含了比对序列的描述、比对序列的打分、比对序列的GI号等。其次脚本会根据比对序列的GI号分批式加载位于核酸基因库(https://www.ncbi.nlm.nih.gov/nuccore)的基因组(genebank格式),直至检测到基因组片段包含taxon后结束加载操作。最后再执行1.21操作。
1.3 输入和输出tax_get.pl脚本中,输入的文件需要以.taxon后缀结尾,文件内容需要以”>taxon”作为首行,要查找的taxon依次放入其他行,键入回车进行换行操作。如果用户想要通过物种学名进行操作,在第二行开始依次放入物种学名放入即可,不需要变更其他信息。待查找结束,程序会自动生成名为“ncbi.reg.out“的输出文件。在输出文件的内容中,首行为:taxon superkingdom phylum class order family genus species,行内中间以tab分隔,以换行符结尾。而从第二行开始,则是保存查找后的结果,它们会填充到与首行相对应的类别中。
seq_get.pl脚本中,输入的文件为fasta格式,提供可以自定义比对条数的Rank参数。Rank表示序列比对后的结果序列中要保存的序列条数,其余参数与在线blast的参数保持一致。而输出的文件中,主要保留了三部分信息,第一部分是序列名以及该条比对序列的打分详情,这部分信息来自BLAST响应的比对序列:Seq_name Description Max_score Gi Total_score Query_cover E_value。第二部分是taxon,它是来自上述的GI对应的基因组文件。第三部分则是根据taxon查找的具体分类,这与上述脚本描述一致,便不再赘述。
1.4 Perl脚本的运行将上述编写好的脚本文件放入“C:\Perl\bin”目录,然后点击任务上的“开始-运行”,在运行框中输入“cmd”命令即可打开“cmd命令提示符”,在cmd窗口键入“cd C:\Perl\bin”后键入回车,便可进入Perl命令执行程序的目录下。
在“C:\Perl\bin>”的提示符下键入“perl tax_get.pl”,待出现“请输入查询的文件后”,输入当前目录下所要查找文件,该文件包含了所要查找的taxon。接着键入回车即可运行, 获取结束后会自动将结果保存到本地。
在“C:\Perl\bin>”的提示符下键入“perl seq_get.pl”回车运行后首先本脚本会在dos窗口以文字的形式展示首页菜单,用于介绍基本操作流程,以及提示用户键入输入的fasta格式序列。键入回车后,下一步程序会展示5种基本的Web-Blast方式供用户选择,这时用户只需根据自己的实际需求键入对应选项的标号。然后程序会根据用户输入的Web-Blast方式展示对应的Blast-Database供用户选择。选择完毕后,用户再根据提示键入输出的文件名以及要获取的比对序列数目即可。键入回车后,程序会自动将需要比对的序列上传给服务器并根据用户设定的参数发送相应的比对请求。而dos窗口则会同步展示服务器端响应的部分数据。待比对完毕,程序会自动将所有筛选后的信息保存到输出文件,并自动退出。
2 结果 2.1 核心代码(1) 通过taxon查找物种分类的编程(脚本1:tax_get.pl)
$tax_url="https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi";
$res=LWP::UserAgent->new->post($tax_url, ['name'=>$taxon]); #发送post请求
@web = split(/\r?\n/, $res->content);
@bio_info =qw(superkingdom phylum class order family genus species); #定义物种类名集合
$target_line; #定义目的行
foreach my $line(@web){#查找单元:“行“
my $i = 0;#定义物种类名在查找单元中出现的次数
foreach(@bio_info){#比对单元:“物种类名“
if ($line =~ /($_)/){
$i++;
if ($i >=2){ #当查找单元符合最小值时,结束查找。
$target_line = $line;
last; }}}}
#获取物种类名对应的物种对象foreach(0...6){
if($target_line =~ /(\=\"$bio_info[$_]\">)([^ < >]*)(< \/)/){
$organism.= "\t$2";
}else{
$organism.= "\t";
}
}
(2) 通过任意一段序列自动、批量在线blast的编程(脚本2:seq_get.pl)
a.通过post请求递交序列文件,并获取RID,
$url = "https://blast.ncbi.nlm.nih.gov/Blast.cgi?";
$submit_url=$url."PROGRAM=$program & PAGE_TYPE=BlastSearch & LINK_LOC=blasthome";
$res=$ua->post($submit_url, ['QUERYFILE'=>$file_name], 'DATABASE'=>$database, 'CMD'=>'Put', ], 'Content-Type'=>"form-data");
($rid) = $res->content =~ /^ RID = (.*$)/m;
b.通过get请求不断检测服务器响应的数据
my $res_web = $url."CMD=Get & RID=$rid";
while ("true"){
my $res= $ua->get($res_web); #获取目的页面
if($res->content =~ /Status=WAITING/){
sleep 10;
}elsif($res->content =~ /Status=READY/){
return $rid;
}else{exit;
}}
c.刷新完毕后则获取页面源代码,并对其结果进行初步的筛选。
my $url = "https://blast.ncbi.nlm.nih.gov/Blast.cgi?CMD=Get & RID=$rid & QUERY_INDEX=$query_index";
my $web_content = $ua->get($url)->content;
if ($web_content =~ /No significant similarity found/i){next; }#如果当前序列BLAST鉴定结果为空,则进行下一条序列的鉴定
d.通过HTML表单的构成规则,拆分blast结果页面
my ($tbody) = $web_content=~/(\ < tbody\>.*\ < \/tbody\>)/i;
my @trs = ();
my $ind = 0;
#获取tr标签包裹的比对序列, 总共100条
while($tbody =~ /(\ < tr>.*?\ < \/tr\>)/ig){
#遍历td标签,总共8个,包括打分、Gi、序列描述
my $td=$1;
my @tds = ();
#存放tds,如ind=0时,@tds存放8个该序列的第一部分信息
$trs[$ind++] = \@tds;
while($td =~ /\ < td[\s|\>](.*?)\ < \/td\>/ig){
push @tds, $1;
}
e.通过gi预览基因组,并获取taxon
$res = $ua->get("https://www.ncbi.nlm.nih.gov/sviewer/viewer.cgi?id=$gi",
‘:content_cb' => sub{
last if(($taxon)=$_[0] =~/\/db_xref\=\"taxon:(.*?)\"/i); },
‘:read_size_hint' => 16 * 1024, );#设置匿名子程序中预览的字节大小
2.2 原理描述(1) 通过观察Taxonomy查询页面的源代码,可知客户端是通过post请求[13]将单个查询对象$name发送到服务器端的,因此通过程序循环向服务器发送请求便可达到批量查询的目的。
接着手动使用不同的taxon多次查询,发现其分类结果均在一行展现,并且在该行中,物种类名与物种对象可以很好的匹配。因此获取的第一步是通过程序将源代码拆分为行与行的形式,通过固定的物种类名集合去获取目的行。经过不同种类的taxon验证,发现当筛选条件为物种类名至少出现两次时,便只有目的行符合。
(2) BLAST将比对的序列以及参数发送到服务器时,也是使用post请求的方式,由bioperl相关模块源码以及手动输入不同序列比对[2, 14]的多次验证,确定了不同blast时服务器端的地址变化规律以及post参数。如用户选择的Web-Blast方式为megablast,则服务器url地址中需要带上参数" & MEGABLAST=on",如果选择了blastp则需要带上参数"blastp & plain=on"
当将比对请求发送到服务器时,会获得唯一标识当前操作的常量RID,并且进入一个等待页面。根据RID可以不断获取服务器端的响应,当服务器端的响应信息包含“Status=WAITING”,程序会等待10 s再继续获取响应。而当响应的信息包含“Status=READY”时,标识着序列比对完成。发现比对结果页面与上传序列在文件的次序基本保持一致,如果上传序列是文件中的第一条(即索引为0),那么该条序列比对结果页面的url参数需要加入QUERY_INDEX=0。基于此,可以实现批量比对的操作,并且可操作的数量,也与ncbi设定的标准一致。
对于一条序列,Blast服务器按照打分的从高到低排序,展示100条比对结果,而这些比对结果,存在于HTML源码的 < tbody>标签中,根据HTML的标签规则以及设定的Rank值,利用正则表达式,即可获取比对序列的打分、GI等。
接着,为了获取序列对应的taxon,利用第一步获取的GI,到核酸库中加载相应的基因组(GeneBank格式)。由于基因组的大小参次不齐,因此我们采用分批式加载的策略,即边加载边检测是否包含taxon,如果有,则进入脚本一,通过taxon获取物种分类名。
2.3 测试结果测试了数千条DNA或蛋白质,序列类型包括细菌的rRNA和功能基因序列、动物和植物的功能基因等; 测试的方法包括megablast、blastn、blastx、tblastx、blastp、tblastn等; 测试的方数据库包括nr、swissprot、pdb等(见表 1);运行结果包括简单的BLAST结果和获取的详细的分类信息等(见图 1.OUTPUT)。
| 表 1 测试程序及对象 Table 1 Tested programs and objectives |
在编写代码时,考虑到研究者们通常使用序列文件进行比对,对上传的比对文件进行了设置,如测试中的“test.fasta”,序列名以“>”开头,换行结束。此外,充分利用NCBI对于多序列、不同序列的在线比对,只获取结果,不参与比对,保证了比对结果的可靠性。同时,编写的脚本尽量全面的获取待测序列的分类信息,包含了“门、纲、目、科、属、种”等。最后,对于结果的保存,我们使用了最常见的记事本,以tab进行间隔,便于研究者使用于其他地方。
3.2 测试结果的全面性本次研究所设计的脚本是以NCBI网站为基础的,因此输入的参数与NCBI比对时所要输入的参数保持一致。对脚本进行了全方位的测试,待测参数涵盖了:物种学名、分类号、任意一段核酸或蛋白质的序列,待测序列包含了各500条细菌、真菌、动物、植物; 涉及的序列类型包括DNA和蛋白质序列,功能基因和rRNA等; 在线比对使用的算法包括:megablast、blastn、blastp、blastx、blastp、tblastx等,比对查询数据库包括:16srRNA、nr、swissport等,均进行了逐一测试,均成功。
3.3 和常见程序以及手动获取的比较脚本基于在线blast,封装并实现了研究者最常用的操作,简化了众多繁琐步骤,用户只需结合实际情况设定几个参数,脚本便会自动的执行批量鉴定、查询、筛选、保存等一体化操作,非常适用于批量进行物种鉴定。Yin YH等使用Perl编写了GenScalpel[15],能够从NCBI大规模的序列集中批量的检索与提取特定序列。而物种的鉴定,其中第二步就得通过GI号去加载序列并获取taxon,脚本使用分批式加载的策略,获取到taxon即停止加载序列,节省了下载完整序列的时间,大大提高了检索的效率。
张成岗[16]等构建了局域网下的本地Blast的Web界面, 但发现此方法虽然简化了操作过程, 最后的分析结果以网页形式返回并且包含众多干扰信息,这不利于后期的分析。
周猛[17]、范彦辉[18]均使用Perl实现了本地blast自动化,在输入和输出上也做了较多的优化,但是想要进一步操作(如获取比对序列分类号),只得借助其他手工方式才能进行下去。
Guy L等编写了phyloSkeleton,它的优点在于能够借助taxonomy,通过手动设置筛选规则,便捷的选择一些具有代表性的物种[19]。脚本能保证NCBI的各种原用方法以及参数设置,Perl脚本只提供数据下载。
Lawrence TJ等利用Perl建立了一套FAST工具集利用快速命令对数据工作流进行组合编码[20]。基于此思路,将编写两个脚本(tax_get.pl和seq_get.pl)可以根据taxon、任意一段序列自动、批量自动获取物种信息,避免了手动获取方式的费时费力易出错等缺陷,同时获取50个序列比对结果时的运行时间(见图 2),显示了我们编写脚本时间上的优势。

|
图 2 分类鉴定50个对象所耗时间(s) Figure 2 Classification and identification of the time consumed by 50 objects(s) |
本脚本适用于细菌、真菌、动物、植物等物种学名、分类号、核酸或蛋白质的任意序列,可以为同行生物数据分析提供参考,获取的速度也与当前的系统以及网络带宽密切相关。本研究依然存在众多可改进之处:
(1) 尝试从更多的网站、或者以本地数据库的建立来进行分类鉴定。
(2) 将程序封装并进行可视化处理,即不通过命令行也可完成相应输入输出的操作。
(3) 增加断点续传的功能,即使查询的中途断电或者断网,下次执行还能继续上次的进度。
(4) 使用其他编程语言实现,如Python、Java的尝试等。
(5) 改进匹配鉴定下载的步骤,如使用双重法[21]增强程序运行的效率。
(6) 增加接口,能对结果数据进行自动采集、分析、挖掘[22]。
(7) 构建在线平台[7],依托数据库分析用户的实际需求,如用户可以根据实际需求动态组装脚本,以提升脚本交互性[23]。
| [1] |
徐晓婷, 王志恒, DimitarDimitrov, 等. 批量下载GenBank基因序列数据的新工具——NCBIminer[J]. 生物多样性, 2015, 23(04): 550-555. XU Xiaoting, WANG Zhiheng, DIMITAR D, et al. Using NCBIminer to search and download nucleotide sequences from GenBank[J]. Biodiversity Science, 2015, 23(04): 550-555. DOI:10.17520/biods.2015120 (  0) 0) |
| [2] |
王哲, 黄高升. NCBI的数据库资源及其应用[J]. 生命科学, 2002(01): 59-62. WANG Zhe, HUANG Gaosheng. Database resources of the national center for biotechnology information and its application[J]. Chinese Bulletin of Life Sciences, 2002(01): 59-62. DOI:10.3969/j.issn.1004-0374.2002.01.017 (  0) 0) |
| [3] |
吕军, 张颖, 冯立芹, 等. 生物信息学工具BLAST的使用简介[J]. 内蒙古大学学报(自然科学版), 2002(02): 179-187. LÜ Jun, ZHANG Ying, FENG Liqin, et al. A brief introduction of the bioinformatics tool blast[J]. Acta Scientiarum Naturalium Universitatis Neimongol, 2002(01): 59-62. DOI:10.3969/j.issn.1004-0374.2002.01.017 (  0) 0) |
| [4] |
陈源源, 沈微, 樊游, 等. GenBank数据库中微生物rDNA序列准确性研究[J]. 食品与发酵工业, 2012, 38(03): 28-31. CHEN Yuanyaun, SHEN Wei, FAN You, et al. Study on the accuracy of microbial rDNA sequence in GenBank database[J]. Food and Fermentation Industries, 2012, 38(03): 28-31. DOI:10.13995/j.cnki.11-1802/ts.2012.03.001 (  0) 0) |
| [5] |
郭文久. Perl语言环境下生物信息学的数据库技术[J]. 安康学院学报, 2007(05): 74-78. GUO Wenjiu. Database technology for bioinformatics research in the environment of perl language[J]. Journal of Ankang University, 2007(05): 74-78. DOI:10.3969/j.issn.1674-0092.2007.05.023 (  0) 0) |
| [6] |
刘强, 祈生胜, 罗中良. 用Perl编程进行因特网数据整理的方法(英文)[J]. 郑州轻工业学院学报, 2004(01): 66-69. LIU Qiang, QI Shengsheng, LUO Zhongliang. Method of data cleaning by Perl on the data of Internet(English)[J]. Journal of Zhengzhou University of Light Industry:Natural Science, 2004(01): 66-69. DOI:10.3969/j.issn.1004-1478.2004.01.020 (  0) 0) |
| [7] |
陈晨, 彭珂, 王海印, 等. 基于16S rDNA数据库的细菌在线分类鉴定平台的构建[J]. 疾病监测, 2013(03): 236-240. CHEN Chen, PENG Ke, WANG Haiyin, et al. An online platform SSUDB: database of bacteria identification and classification with 16S rDNA[J]. Disease Surveillance, 2013(03): 236-240. DOI:10.3784/j.issn.1003-9961.2013.3.019 (  0) 0) |
| [8] |
HERRERA-GALEANO J E, FREY K G, CER R Z, et al. A PERL module to plot next generation sequencing NCBI-BLAST results[J]. Source Code for Biology and Medicine, 2014, 9(1): 7. DOI:10.1186/1751-0473-9-7 (  0) 0) |
| [9] |
郑武, 老马. Linux下的Perl编程[M]. 北京: 人民邮电出版社, 2001. ZHENG Wu, LAO Ma. Perl programming under Linux[M]. Beijing: Posts & Telecom Press, 2001. (  0) 0) |
| [10] |
STAJICH J E, Block D, Boulez K, et al. The Bioperl toolkit: Perl modules for the life sciences[J]. Genome Research, 2002, 12(10): 1611-1618. DOI:10.1101/gr.361602 (  0) 0) |
| [11] |
郭文彬. 使用Perl脚本自动提取保存网页中的数据[J]. 中国管理信息化, 2015(20): 180. GUO Wenbin. Automatically extract and save data in web pages by using Perl script[J]. China Management Informationization, 2015(20): 180. DOI:10.3969/j.issn.1673-0194.2015.20.140 (  0) 0) |
| [12] |
李旭凯, 彭良才, 王令强. pep_pattern.pl, 搜索蛋白质序列模体的Perl脚本[J]. 华中农业大学学报, 2014, 33(04): 1-6. LI Xukai, PENG Liangcai, WANG Lingqiang. pep_pattern.pl, a perl script for searching motifs in a group of related DNA/protein sequences[J]. Journal of Huazhong Agricultural University, 2014, 33(04): 1-6. DOI:10.13300/j.cnki.hnlkxb.2014.04.001 (  0) 0) |
| [13] |
AMIRK, EITANR, 韩锴. Perl在生物研究中的应用[J]. 程序员, 2009(01): 104-106. AMIR K, EITAN R, HAN Kai. Application of Perl in biological research[J]. Porgrammer, 2009(01): 104-106. (  0) 0) |
| [14] |
刘文强, 贾玉萍, 赵宏坤. 16S rRNA在细菌分类鉴定研究中的应用[J]. 动物医学进展, 2006, 27(11): 15-18. LIU Wenqiang, JIA Yuping, ZHAO Hongkun. Progress on identification and classification of bacteria by means based on 16 S rRNA[J]. Progress in Veterinary Medicine, 2006, 27(11): 15-18. DOI:10.16437/j.cnki.1007-5038.2006.11.005 (  0) 0) |
| [15] |
YIN Y H, DU L M, YUE B S. GenScalpel: an application for sequence retrieval and extraction from the GenBank flatfile[J]. Journal of Heredity, 2012, 103(6): 908-911. DOI:10.1093/jhered/ess052 (  0) 0) |
| [16] |
张成岗, 贺福初, 欧阳曙光, 等. 序列同源性分析软件Blast的WEB界面构建及其应用[J]. 生物化学与生物物理进展, 2001, 28(6): 916918. ZHANG Chenggang, HE Fuchu, OUYANG Shuguang, et al. Construction and application of the WEB interface of blast package[J]. Progress in Biochemistry and Biophysics, 2001, 28(6): 916918. DOI:10.3321/j.issn:1000-3282.2001.06.034 (  0) 0) |
| [17] |
周猛, 童春发, 施季森. 基于Perl语言的序列同源性分析过程自动化的实现[J]. 生物技术, 2007, 17(01): 60-63. ZHOU Meng, TONG Chunfa, SHI Jishen. Realization of Perl-based automatic sequence homogeneous analysis[J]. Biotechnology, 2007, 17(01): 60-63. DOI:10.3969/j.issn.1672-5565.2008.01.015 (  0) 0) |
| [18] |
范彦辉, 陶士珩. 用Perl实现在Windows下本地化运行BLAST[J]. 生物信息学, 2008, 6(4): 178-179. FAN Yanhui, TAO Shiheng. Run stand-alone BLAST on Windows using Perl script[J]. China Journal of Bioinformatics, 2008, 6(4): 178-179. DOI:10.3969/j.issn.1672-5565.2008.04.011 (  0) 0) |
| [19] |
GUY L. phyloSkeleton: taxon selection, data retrieval and marker identification for phylogenomics[J]. Bioinformatics, 2017, 33(8): 1230-1232. (  0) 0) |
| [20] |
LAWRENCE T J, KAUFFMAN K T, AMRINE K C, et al. FAST: FAST analysis of sequences toolbox[J]. Frontiers in Genetics, 2015, 6(172): 172. DOI:10.3389/fgene.2015.00172.ecollection2015 (  0) 0) |
| [21] |
BERMAN J J. Nomenclature-based data retrieval without prior annotation: facilitating biomedical data integration with fast doublet matching[J]. Silico Biology, 2005, 5(5): 313-322. (  0) 0) |
| [22] |
SCHUBERT F, TAUSCH B, JOOS S, et al. CGH-Profiler: data mining based on genomic aberration profiles[J]. BMC Bioinformatics, 2005, 6(1): 188. DOI:10.1186/1471-2105-6-188 (  0) 0) |
| [23] |
BRESELL A, SERVENIUS B, PERSSON B. Ontology annotation treebrowser: an interactive tool where the complementarity of medical subject headings and gene ontology improves the interpretation of gene lists[J]. Applied Bioinformatics, 2005, 5(4): 225-236. DOI:10.2165/0082294-200605040-00005 (  0) 0) |
 2018, Vol. 16
2018, Vol. 16


