2. 生物电子学国家重点实验室,东南大学 生物科学与医学工程学院,南京 210096;
3. 生物医学工程国家级实验教学示范中心(东南大学),南京 210096
2. State Key Laboratory of Bio-electronics, School of Biological Sciences and Medical Engineering, Southeast University, Nanjing 210096, China;
3. National Demonstration Center for Experimental Biomedical Engineering Education (Southeast University), Nanjing 210096, China
环状RNA是区别于传统线性RNA的一类RNA,具有闭合环状结构,不受RNA外切酶影响,不易降解,表达更稳定[1-2]。由于检测方法的限制,环状RNA自1979年在Hela细胞的细胞质中被首次发现以来的近三十年时间内并没有得到研究者的关注。然而,随着近年来高通量测序技术的广泛应用,成千上万的环状RNA在不同物种的细胞和组织中被广泛发现[1-2]。研究发现,人类细胞中存在大量的环状RNA。通过与基因注释信息比对,85%的环状RNA能匹配到已知基因上,其中的84%与编码外显子重合[3]。进一步的研究发现,环状RNA通过多种途径在细胞中发挥着重要的生物学功能。一些环状RNA作为竞争性内源RNA,可以调控基因表达[3];环状RNA也可以发挥海绵作用,吸附miRNA,从而阻断后者对靶基因的抑制作用[4-5];环状RNA也可以通过与蛋白质结合,募集蛋白质复合体的组分,或调控蛋白质的活性[3, 6];有些环状RNA甚至能够被翻译成蛋白质[7]。
目前,常见的环状RNA的识别工具(如find_circ[3],CIRI[8],circRNAfinder[9],MapSplice[10],CIRCexplorer[11]等)都是从高通量测序数据中识别反向剪接位点(Back-splicing site)来实现环状RNA的识别。相关研究表明,上述5种环状RNA工具共同预测到的环状RNA比例很小,这些工具普遍存在较高的假阳性率和低灵敏度[12]。因此,如何充分利用序列本身的特征来区分环状RNA与其他线性RNA(特别是编码蛋白的信使RNA),是生物信息学研究中亟待解决的重要问题。近年来,序列特征与机器学习相结合的方法已成功地用于基因调控位点的预测、剪切位点的预测等生物学问题[13]。值得注意的是,前期研究已经表明基于序列特征的机器学习方法也能有效鉴别环状RNA与长链非编码RNA[14]。
本文首先提取一些影响RNA成环过程的序列特征,并筛选了一些在环状RNA与线性RNA之间存在显著差异的序列特征,采用机器学习的分类算法,构建用于仅仅基于基因组序列预测外显子环状RNA的预测工具。测试结果表明,我们的方法能很好地区分外显子环状RNA与编码蛋白的mRNA。
1 材料与方法 1.1 数据来源为了训练和测试模型,我们共选取了3组数据集及1组独立测试集。每组数据集由正负样本集两部分组成,正样本集代表环状RNA,负样本集代表线性蛋白质编码RNA(见表 1)。
| 表 1 组数据集中正负样本集所对应RNA的数目 Table 1 Correrponding number of RNAs in positive and negative sample sets in the dataset |
3组数据集中正样本集构成如下:组1:5种常见的环状RNA预测工具对相同的RNA测序数据集进行环状RNA预测,选择同时被5种工具检出的848条环状RNA[12]作为正样本。组2:同样来自该项工作,选择2种工具及以上检出的2 463条环状RNA。组3:来源于circBase数据库的人类环状RNA[15]。该数据库收集了不同研究小组的环状RNA识别结果,共包含了超过9万条环状RNA序列及其基因组注释信息。去除长度小于200 nt的短序列并且只保留同一基因的最长可变剪接转录本,最后得到11 472条环状RNA。对于每一个正样本集,我们建立对应的负样本集。负样本集来源于UCSC人类基因组数据hg19版本[16]。首先,选取人类基因组中约80 000条编码蛋白的序列。然后,剔除与正样本集中的序列及circBase和circNet[17]中预测出的circRNA相重叠的序列,剩下共9 976条序列。最后,随机抽取与每组正样本集数量相近的序列作为该组对应的负样本集。
另外,由于经过实验验证的环状RNA数目太少(文献[18]中共收集到282个经过PCR验证的环状RNA),不适合作为模型训练的数据集。因此,我们将实验验证的环状RNA作为独立数据集,仅用于模型预测能力的验证(见表 1)。
1.2 特征筛选现有研究结果表明,真核生物中RNA序列是否成环与其上下游序列有关。在内含子配对驱动RNA环化的过程中,5’端剪切位点存在GU重复序列,而对应的3’剪切位点存在AG重复序列,作为上下游剪切信号[19]。
A-to-I RNA编辑是在哺乳动物中普遍存在的RNA编辑方式,通过ADAR酶将某些特定位点的腺嘌呤(A)脱氨形成次黄嘌呤(I),在碱基配对中被识别作鸟嘌呤(G), 影响碱基配对及位点识别[20]。人类基因组中常伴随双链RNA上Alu反向互补重复序列[21-22],而Alu重复序列的配对结合能在空间上拉近两端剪切位点,从而促进RNA环化[2]。对比环状RNA与线性mRNA上下游内含子中存在的A-to-I编辑位点及反向互补重复序列,结果显示在环状RNA的上下游内含子中的分布密度显著高于线性mRNA,尤其体现在剪切位点附近[23]。同时,环状RNA上下游的内含子通常较长,且具有较多的Alu重复序列。这可能是由于内含子长度越长,在空间上更容易弯曲靠近,使得上下游Alu序列更容易结合,也就越容易发生反向剪接促进成环[2]。
另外,RNA结合蛋白(RBP)与RNA成环也存在着密切的联系。上下游内含子与RNA结合蛋白形成共聚体,容易在空间上拉近上下游剪切位点,促进反向剪接[3]。例如,QKI结合蛋白能调控RNA剪切过程,促进反向剪接发生。环状RNA与具有相似侧翼内含子长度的线性mRNA的QKI结合位点分布存在着显著差异[24]。
基于这些先验知识,我们选取序列组分特征(包括内含子长度、G/C含量、GT、AG剪切信号分布密度),剪接位点上下游内含子上A-to-I RNA编辑分布密度、Alu反向互补重复序列数量以及人类94种不同RBP[25]结合位点数共100种特征,分别计算不同数据集中所有特征的值,构建分类模型的特征值向量。
1.3 随机森林与支持向量机随机森林算法(RF)是一种用于数据分类和回归的机器学习算法,具有优越的分类性能,在生物信息学领域有着广泛应用[26]。支持向量机(SVM)是由Vapnik等提出的基于VC维度理论和结构风险最小化原理的机器学习方法,在求解小样本、非线性和高维模式识别方面具有许多特殊的优势[27-28]。
在本工作中,我们还利用RF来分析不同特征值对分类预测的重要性。另外,我们采用5折交叉验证对RF和SVM的分类能力进行了评估。
1.4 建立分类模型及评价利用R语言中的randomForest、ipred、predict、e1071、ROCR、caret等软件包来实现分类模型。分类特征向量为预先提取的100个特征组成的一维向量。首先,我们从每组待测数据集的正负数据集中随机抽取2/3作为训练集,剩余1/3作测试集,利用RF计算多维向量特征值重要性。其次,我们采用5折交叉验证,对3组待测数据分别进行RF和SVM算法分类,对预测结果进行评价。分类模型的基本流程见图 1。
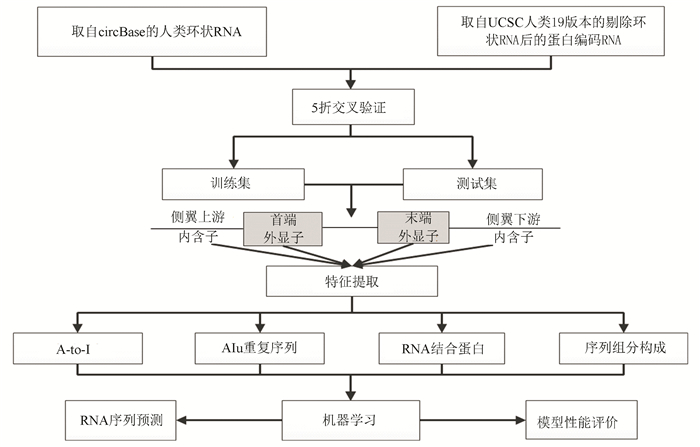
|
图 1 模型搭建流程图 Figure 1 Flowchart of the model building |
我们采用了下列的指标来进行模型评估:灵敏度(Sn)、特异性(Sp)、准确度(Acc)、和马修斯相关系数(MCC),分别定义如下:
| $ {\rm{Sn = }}\frac{{{\rm{TP}}}}{{{\rm{TP + FN}}}} $ | (1) |
| $ {\rm{Sp = }}\frac{{{\rm{TN}}}}{{{\rm{TN + FP}}}} $ | (2) |
| $ {\rm{Acc = }}\frac{{{\rm{TP + TN}}}}{{{\rm{TP + TN + FP + FN}}}} $ | (3) |
| $ {\rm{MCC = }}\frac{{{\rm{TP*TN - FP*FN}}}}{{\sqrt {\left( {{\rm{TP + FP}}} \right)\left( {{\rm{TP + FN}}} \right)\left( {{\rm{TN + FP}}} \right)\left( {{\rm{TN + FN}}} \right)} }} $ | (4) |
为了研究不同的特征在RNA成环分类预测中的重要性, 我们应用随机森林特征选择对其重要性进行排序。每个特征值的重要性分数是由森林多次置换前后错误的差异平均值决定的。分值越高表示特征值的重要性越高。图 2显示了对环状RNA分类贡献大的前40个特征。可以看出,排在前面的特征有Alu反向互补重复序列、A-to-I位点、多种剪切相关的RBP、内含子长度和GT剪切信号等。这一结果说明,这些特征在区分外显子环状RNA及线性mRNA中起关键性作用,而排序结果也基本符合我们的预期。
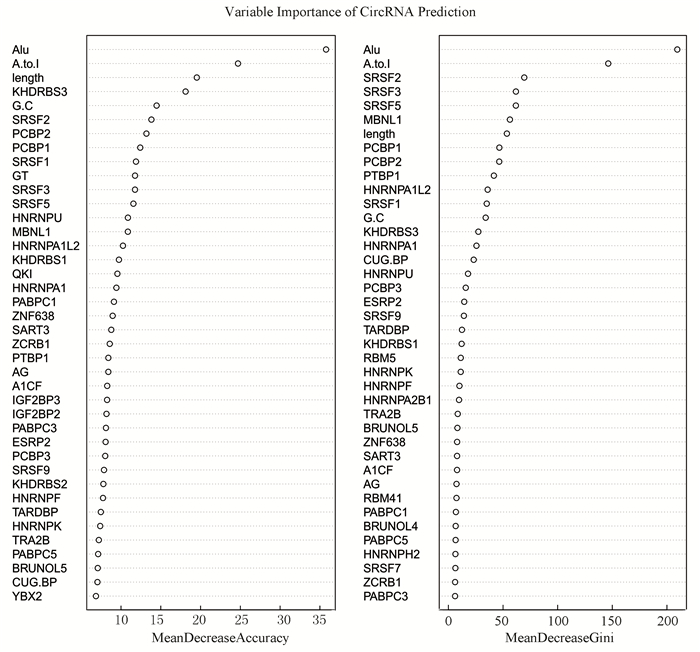
|
图 2 影响环状RNA预测的特征值排序前40 Figure 2 Top 40 features from Random Forest importance ranking that influence the prediction of circRNAs |
我们采用5折交叉检验来评估RF和SVM在特征向量数据集上的分类情况,结果见表 2。可以看出,对于第1、2组数据集,RF和SVM均能有效区别环状RNA与线性RNA,分类性能表现良好,RF分类表现略优于SVM。整体来说,这两类算法都能较好的整合不同的特征类型,兼容特征值的异质性,同时也体现出我们提取的特征较好的反映了环状RNA与线性RNA的特异性。第3组数据分类表现不如前两组优秀,但考虑到该组数据集主要来源于circBase数据库,该数据库集合了多个高通量测序数据的find_circ预测结果。虽然我们对数据进行了序列长度的过滤筛选,但仍有较高的噪声。但由于该数据集样本量大,模型在泛化性和抗过拟合上会有较好的表现。因此,在后续的独立样本预测中,我们选取该组数据训练的模型作为分类器。
| 表 2 模型训练分类性能打分 Table 2 Performance in model training |
为进一步验证模型的分类特性,使用基于第3组数据训练得到的模型对独立数据集中的RNA进行分类,分类结果见表 3。结果说明,RF模型和SVM模型均具有较高的环状RNA识别正确率。除了识别敏感性(Sn)外,RF均要优于SVM。从分类ROC曲线(见图 3)可以看出,RF模型的总体分类水平(AUC=0.947)要略优于SVM(AUC=0.937)。以上结果表明,不管采用RF和SVM,我们提取的100个序列特征均能很好地区分外显子区间的环状RNA和mRNA。结果证实,仅仅通过序列本身进行环状RNA预测是完全可行的。
| 表 3 独立样本测试集表现打分 Table 3 Performance of independent testing dataset |
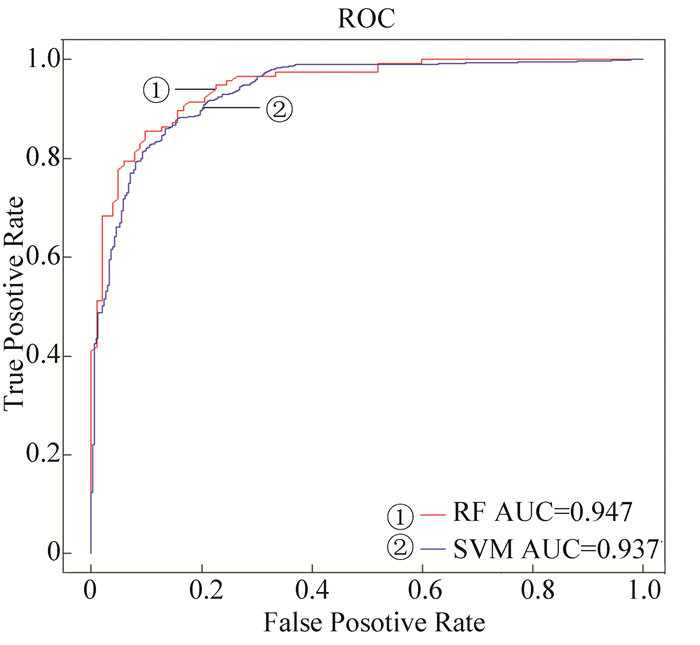
|
图 3 独立样本测试集分类结果ROC曲线 Figure 3 The ROC curves of prediction result of independent testing dataset |
另外,2016年发表的PredicircRNATool是基于侧翼内含子上双核苷酸构象特征和机器学习算法来识别环状RNA和组成型外显子[29]。和我们的模型不同,PredicircRNATool选取了完全不同的一组特征来实现环状RNA的识别。他们基于RNA构象及热力学特征,利用DiProDB数据库进行特征筛选,共得到了125个特征[29]。为了和该方法进行比较,我们计算了表 1三组数据集中PredictRNATool所提取的125个特征值,同样利用5折交叉验证和RF以及SVM机器学习方法进行分类测试,结果如表 4。比较表 2和表 4,我们可以看出尽管基于这两组不同特征所建立的模型的分类表现中,基于本研究的特征建立的模型具有更好的识别能力。对第3组数据的分类表现相似,但对于环状RNA的识别,我们的模型在RF分类方法下具有更好的灵敏度。另外,从特征提取的复杂性来看,我们的特征提取过程不依赖于特定的构象数据库,不需要实验数据的支持,提取过程更简单。同时,我们提取的每一类特征都具有明确的生物学解释,能较好地描述影响RNA成环的因素。
| 表 4 构象及热力学特征模型表现打分 Table 4 Performance of model based on conformational and thermodynamic features |
本文主要考虑RNA内在序列特征对RNA能否成环的影响,包括对成环有显著促进作用的Alu重复序列、A-to-I编辑位点、内含子上多种调控RNA剪切的RNA蛋白结合位点以及序列组分构成特征(包括内含子长度、G/C含量、GT、AG剪切信号分布密度)等,这些特征的选取综合了近年来环状RNA领域的诸多工作,有较好的广泛性和代表性。
模型的测试和验证结果表明:
1) 本文构建的基于序列特征的机器学习分类模型能有效地鉴别环状RNA与线性mRNA,准确度高达85%以上。
2) 对于机器学习分类方法,相比于SVM方法,RF分类的效果更好。
3) 不同特征对分类结果贡献度不同,其中以Alu重复序列及A-to-I RNA编辑位点对分类结果影响最显著。
4) 与已发表的相关工作比较也体现出本文特征选取方法能够更好地提升环状RNA的识别效率。
针对现有基于测序数据的环状RNA识别工具的低一致性、高假阳性率和高假阴性率问题,本论文提出的基于序列特征的机器学习模型能在现有工具基础上,进一步有效地区分环状RNA和线性mRNA,为环状RNA的后续功能研究提供更可靠的数据。
| [1] |
SALZMAN J, GAWAD C, WANG P L, et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J]. Plos One, 2012, 7(2): e30733. DOI:10.1371/journal.pone.0030733 (  0) 0) |
| [2] |
JECK W R, SORRENTINO J A, WANG K, et al. Circular RNAs are abundant, conserved, and associated with ALU repeats[J]. Rna-a Publication of the Rna Society, 2013, 19(2): 141-157. DOI:10.1261/rna.035667.112 (  0) 0) |
| [3] |
MEMCZAK S, JENS M, ELEFSINIOTI A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333-338. DOI:10.1038/nature11928 (  0) 0) |
| [4] |
HANSEN T B, JENSEN T I, CLAUSEN B H, et al. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495(7441): 384-388. DOI:10.1038/nature11993 (  0) 0) |
| [5] |
VALDMANIS P N, KAY M A. The expanding repertoire of circular RNAs[J]. Molecular Therapy, 2013, 21(6): 1112-1114. DOI:10.1038/mt.2013.101 (  0) 0) |
| [6] |
HENTZE M W, PREISS T. CIRCULAR RNAs: splicing's enigma variations[J]. Embo Journal, 2013, 32(7): 923-925. DOI:10.1038/emboj.2013.53 (  0) 0) |
| [7] |
WANG Y, WANG Z. Efficient backsplicing produces translatable circular mRNAs[J]. Rna-a Publication of the Rna Society, 2015, 21(2): 172-179. DOI:10.1261/rna.048272.114 (  0) 0) |
| [8] |
GAO Y, WANG J, ZHAO F. CIRI: an efficient and unbiased algorithm for de novo circular RNA identification[J]. Genome Biology, 2015, 16(1): 4. DOI:10.1186/s13059-014-0571-3 (  0) 0) |
| [9] |
FU X, LIU R. CircRNAFinder: a tool for identifying circular RNAs using RNA-Seq data[C]. Proceedings of the 6th International Conference on Bioinformatics and Computational Biology, BICOB. 2014. https://www.researchgate.net/publication/280068964_CircRNAFinder_a_Tool_for_Identifying_Circular_RNAs_Using_RNA-Seq_Data
(  0) 0) |
| [10] |
WANG K, SINGH D, ZENG Z, et al. MapSplice: accurate mapping of RNA-seq reads for splice junction discovery[J]. Nucleic Acids Research, 2010, 38(18): e178. DOI:10.1093/nar/gkq622 (  0) 0) |
| [11] |
ZHANG X O, WANG H B, ZHANG Y, et al. Complementary sequence-mediated exon circularization[J]. Cell, 2014, 159(1): 134-147. DOI:10.1016/j.cell.2014.09.001 (  0) 0) |
| [12] |
HANSEN T B, VENØ M T, DAMGAARD C K, et al. Comparison of circular RNA prediction tools[J]. Nucleic Acids Research, 2016, 44(6): e58. DOI:10.1093/nar/gkv1458 (  0) 0) |
| [13] |
XIONG H Y, ALIPANAHI B, LEE L J, et al. RNA splicing. The human splicing code reveals new insights into the genetic determinants of disease[J]. Science, 2015, 347(5218): 124-125. DOI:10.1126/science.1254806 (  0) 0) |
| [14] |
PAN X, XIONG K. PredcircRNA: computational classification of circular RNA from other long non-coding RNA using hybrid features[J]. Molecular BioSystems, 2015, 11(8): 2219-2226. DOI:10.1039/c5mb00214a (  0) 0) |
| [15] |
GLAŽAR P, PAPAVASILEIOU P, RAJEWSKY N. CircBase: a database for circular RNAs[J]. Rna, 2014, 20(11): 1666-1670. DOI:10.1261/rna.043687.113 (  0) 0) |
| [16] |
KENT W J, SUGNET C W, FUREY T S, et al. The Human Genome Browser at UCSC[J]. Genome Research, 2002, 12(6): 996-1006. DOI:10.1101/gr.229102.ArticlepublishedonlinebeforeprintinMay2002 (  0) 0) |
| [17] |
LIU Y C, LI J R, SUN C H, et al. CircNet: a database of circular RNAs derived from transcriptome sequencing data[J]. Nucleic Acids Research, 2016, 44(D1): D209-D215. DOI:10.1093/nar/gkv940 (  0) 0) |
| [18] |
ZENG X, LIN W, GUO M, et al. A comprehensive overview and evaluation of circular RNA detection tools[J]. Plos Computational Biology, 2017, 13(6): e1005420. DOI:10.1371/journal.pcbi.1005420 (  0) 0) |
| [19] |
GUO J U, AGARWAL V, GUO H, et al. Expanded identification and characterization of mammalian circular RNAs[J]. Genome Biology, 2014, 15(7): 409. DOI:10.1371/journal.pcbi.1005420 (  0) 0) |
| [20] |
WASHBURN M C, HUNDLEY H A. Controlling the editor: the many roles of RNA-binding proteins in regulating A-to-I RNA editing[M]//RNA Processing. Springer International Publishing, 2016: 189-213.DOI: 10.1007/978-3-319-29073-7_8.
(  0) 0) |
| [21] |
NISHIKURA K. Functions and regulation of RNA editing by ADAR deaminases[J]. Annual Review of Biochemistry, 2010, 79(1): 321-349. DOI:10.1146/annurev-biochem-060208-105251 (  0) 0) |
| [22] |
RAMASWAMI G, WEI L, PISKOL R, et al. Accurate identification of human Alu and non-Alu RNA editing sites[J]. Nature Methods, 2012, 9(6): 579-581. DOI:10.1038/nmeth.1982 (  0) 0) |
| [23] |
IVANOV A, MEMCZAK S, WYLER E, et al. Analysis of intron sequences reveals hallmarks of circular rna biogenesis in animals[J]. Cell Reports, 2015, 10(2): 170-177. DOI:10.1016/j.celrep.2014.12.019 (  0) 0) |
| [24] |
CONN S J, PILLMAN K A, TOUBIA J, et al. The RNA binding protein quaking regulates formation of circRNAs[J]. Cell, 2015, 160(6): 1125-1134. DOI:10.1016/j.cell.2015.02.014 (  0) 0) |
| [25] |
PAZ I, KOSTI I, ARES J R M, et al. RBPmap: a web server for mapping binding sites of RNA-binding proteins[J]. Nucleic Acids Research, 2014, 42(W1): 361-367. DOI:10.1093/nar/gku406 (  0) 0) |
| [26] |
BREIMAN L. Random forests[J]. Machine Learning, 2001, 45(1): 5-32. DOI:10.4236/abb.2010.14040 (  0) 0) |
| [27] |
VAPNIK V. The nature of statistical learning theory[J]. Conference on Artificial Intelligence, 1995, 10(5): 988-999. (  0) 0) |
| [28] |
WANG Y, LI Y, WANG Q, et al. Computational identification of human long intergenic non-coding RNAs using a GA-SVM algorithm[J]. Gene, 2014, 533(1): 94-99. DOI:10.1016/j.gene.2013.09.118 (  0) 0) |
| [29] |
LIU Z, HAN J, LV H, et al. Computational identification of circular RNAs based on conformational and thermodynamic properties in the flanking introns[J]. Computational Biology and Chemistry, 2016, 61: 221-225. DOI:10.1016/j.compbiolchem.2016.02.003 (  0) 0) |
 2018, Vol. 16
2018, Vol. 16


