生物信息学是由生物学、计算机科学、应用数学等学科交叉而成的新兴学科。随着人类基因组计划(HGP)的不断推进,生物信息学已经成为当今生命科学中最具活力的前沿领域之一。2002年,我国一些高等院校和研究机构开始向教育部申请设立生物信息学本科专业[1],生物信息学课程的教学实施也随之受到了越来越多的关注。目前,中国科技大学、山东大学、哈尔滨医科大学等高校已经开展了生物信息学课程。然而,中国目前的生物信息学教育尚未形成较成熟的教育体系,与欧美国家相比,还存在较大的差距[2]。因此,如何在高校内实施生物信息学课程教学,目前仍然处于探索和实践的阶段。
1 开设生物信息学课程的必要性 1.1 生物信息学的兴起20世纪后期,生物科学技术的迅猛发展,使得生物科学的数据资源无论是从数量上还是质量上都得到了极大的提升。生物科学领域相关数据的迅猛增长,就使得发展一种强有力的方法去存储、处理、分析这些生物数据成为迫切需求。直到20世纪80~90年代,计算机科学技术领域取得了巨大的进步,而以数据分析、处理为主的计算机科学技术也慢慢渗透到了生物科学的各个领域,并形成了良好的结合体。于是,一门崭新的、极具发展潜力的生物信息学就此产生了。
1995年,人类基因组计划[3]的总结报告中给了生物信息学一个较为完整的定义:生物信息学是一门由数学、计算机科学和生物学综合而成的交叉学科,它包含了生物信息的获取、加工、存储、分配、分析、解释等在内的所有方面,它综合运用数学、计算机科学和生物学的各种工具来阐明和理解大量数据包括的生物学意义。
1.2 在现代生命科学领域中的地位及意义“21世纪是生命科学的世纪,也是信息科学的世纪”[4]。21世纪以来,随着高通量测序技术的不断成熟,分子生物数据呈现爆炸式增长。建立数据库是存储生物医学相关数据的重要步骤,目前,生物信息学相关的数据库总数已经超过了1 078个[5],这些公共的数据库当中已经存储了上千个不同物种的序列数据。而这些庞大的数据背后蕴藏的是什么,这给生物信息科学家带来了巨大的挑战,所以利用计算机技术和信息论方法对蛋白质及核酸等多种生物信息进行解读就成为生物信息科学家的当务之急。生物信息学的产生在这种背景下应运而生,大大的推进了相关研究的开展,被誉为“解读生命天书的慧眼”[6]。
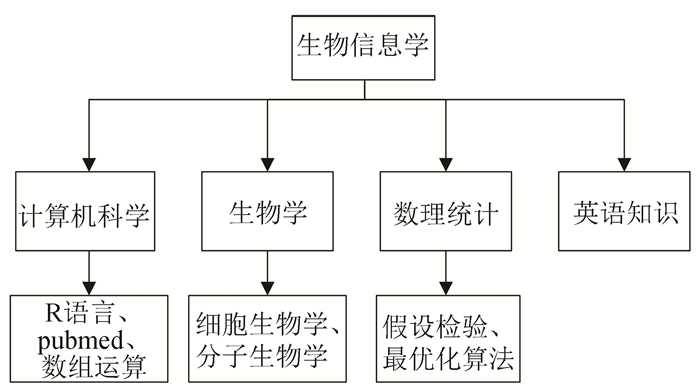
2 生物信息学课程教学模式构建 2.1 制定具有专业特色的生物信息学课程计划首先,考虑到生物信息学是一门由生物学、计算机科学、应用数学等多门学科交叉融合而产成的新兴学科。因此,在进行生物信息学课程的教学实施中,足够的生物学知识,计算机学知识,数学知识等基础知识课程对于生物信息学来说是必备的。这有助于增强学生对于生物信息学的全面了解,对后续的生物信息学课程学习来说起着尤为重要的基石作用。生物信息学课程对于计算机科学、互联网、各类应用软件的依赖性极强,在传授过程中要重点对相关知识进行讲解,例如R语言、pubmed等。R语言是一个专门用于统计分析和统计绘图的优秀工具。它是一款自由、免费、源代码开放的软件,内建多种统计学及数字分析功能,能够实现的功能有:数据存储和处理;数组运算(尤其是向量运算、矩阵运算);完整连贯的统计分析;优秀的统计制图等。简单而强大的特性使得它成为生物信息学家的新宠,R语言能够有效处理各种生物信息学数据,对于学生的实践应用过程来说是一个得心应手的工具。Pubmed医学文献检索系统提供生物医学方面的论文搜寻及摘要,是美国国家图书馆所属的国家生物技术信息中心(NCBI)于2000年开发的生物医学信息检索系统。Pubmed数据库内的数据类型主要是期刊文献、综述以及与其他数据库链接,是一个较为全面的生物医学资源,这对于生物信息学研究者来说是非常有效的资源。学会pubmed数据库的检索和使用,有助于学生了解生物信息领域内的专业内容和发展情况,并且在了解的过程中产生自己的思路,从而迅速进入生物信息学领域, 课程设计流程图见图 1。
|
图 1 专业特色的生物信息学课程计划流程 Figure 1 The process of specialized bioinformatics course plan |
而如何将这些涉及范围广、涉及内容深的生物信息学基础课程传授给学生,这对于老师们来说,是一个极其艰巨的任务。生物信息学人才是需要集生物学、计算机学、数理统计等知识于一身的高级复合型人才,然而在传授过程中,针对如此复杂多样的课程内容,我们要有侧重点,不能将其同计算机学等专业生对待,有侧重的讲解其中对于生物信息学有重要应用的内容,这样才不会导致学生课程压力过大,能够在有限的时间内最大量化的学习生物信息学相关课程。但同时必须注重扩宽学生的知识面,让学生了解到更多的专业内容,有助于学生对于专业的整体把握和理解。
英语知识学习一直都是生物信息课程教学必不可少的一部分。生物信息学是由国外科学家率先兴起的,这就导致领域内的相关知识、网站和文献等都是英文的,这是大多数学生都想要回避的,学生看到全英文的界面时,通常无从下手。如何引发学生学习英语的兴趣,提高学生学习学英语的积极性是一个重要课题。我们采用引导式、互动式教学模式,在教授过程中,充分重视举一反三,重视引导,重视互动。例如在数据库资源讲解过程中,我们可以例举几个典型数据库,GEO[7]、HGNC[8]、UCSC[9]等,对其应用进行充分讲解,让学生了解数据库相关资源的共通之处,这有利于学生对于相关资源的理解,使得原本无从下手的全英文界面变得有迹可循。课后还可以给学生布置一些小任务,自行根据课堂知识去了解GENCODE、Ensembl等重要数据库,及时进行反馈[10]。这样,能够引发学生学习英语、专业英语的积极性。学好英语无疑是生物信息学课程的好帮手[11]。
2.2 理论概念与实践应用相结合以往的教学经验发现,低年级学生在接受宽泛的基础知识教学时,会对自己所学知识产生一种迷茫的感觉,大都不清楚所学知识的应用之处,完全是被动的接受,这很不利于教学过程的开展以及学生对于课程知识的理解。针对这样的现状,我们采取理论与时间相结合的教学模式。在课程安排中夹杂着许多上机实验课,通常是两节理论课之后,就要进行一次实验课,在实验课上,设计一个小的课题,使学生能够将所学的知识及时的应用到实践当中,这有助于引发学生学习的积极性,同时强化学生对于课程知识的理解,使得理论和实践得到充分的结合[12]。
另外,我们还充分发展了教学科研相结合的教育模式。在课程教授过程的同时,引导学生进入科室,参与老师的科研课题。科研工作是一项严谨的工作,参与科研的过程能够激发学生们对于科研工作的兴趣,能够培养学生的责任感以及认真、严谨的科研思维和态度。科研工作中存在着一个个需要不断攻克的难题,引导学生参与科研工作,能够提高学生的动手能力,培养学生独立解决问题的能力[13]。科研工作是生物信息学课程的实际应用过程,让学生参与科研工作能够使学生充分理解生物信息学知识,切实把握课程概况,做到心中有数。
3 讨论生物信息学是一门新兴的、快速发展的、不断更新的学科,我们不能采用传统老套的教学模式,这样无法达到充分学习的目的。我们要在探索中行进,充分应用引导式、互动式、教学科研相结合的教学模式等,在教学过程中,发挥教师的主导作用的同时,突出学生的主体作用,充分调动学生的学习积极性,不断培养学生的严谨负责的态度,强调学生的动手能力和创新能力。此外,教师应该紧跟学科发展方向,了解领域的前沿动态,不断寻找最合适的教学模式,完善课程体系[1]。我们相信,随着生物信息学教学的不断发展,生物信息学课程教学一定会越来越完善,并且逐渐形成一套独特的教学体系。
| [1] |
戴凌燕, 姜述君, 高亚梅, 等. 《生物信息学》课程教学方法探索与实践[J]. 生物信息学, 2009, 7(4): 311-313. DAI Lingyan, JIANG Shujun, GAO Yamei, et al. Exploration and practice of teaching methods of bioinformatics course[J]. China Journal of Bioinformatics, 2009, 7(4): 311-313. DOI:10.3969/j.issn.1672-5565.2009.04.018 (  0) 0) |
| [2] |
虢毅, 胡德华, 邓昊, 等. 生物信息学课程"开放式、研究性"教学模式的探讨[J]. 生物信息学, 2009, 7(3): 227-228. GUO Yi, HU Dehua, DENG Hao, et al. Discussion on "open and research-based" teaching mode in bioinformatics course[J]. China Journal of Bioinformatics, 2009, 7(3): 227-228. (  0) 0) |
| [3] |
GREEN E, WATSON J, COLLINS F. Human Genome Project: Twenty-five years of big biology[J]. Nature, 2015, 526(7571): 29-31. DOI:10.1038/526029a (  0) 0) |
| [4] |
谭文斌. 迎接21世纪生命科学的新时代:———基因组与蛋白组计划时代[J]. 医学与哲学, 1998, 19(9): 449-452. TAN Wenbin. Greeting the new era of life science in twenty-first Century:--age of genome and proteome project[J]. Medicine and Philosophy, 1998, 19(9): 449-452. (  0) 0) |
| [5] |
KRISHNAMURTHY L, NADEAU J, OZSOYOGLU G, et al. Pathways database system: an integrated system for biological pathways[J]. Bioinformatics, 2003, 19(8): 930-937. DOI:10.1093/bioinformatics/btg113 (  0) 0) |
| [6] |
陈润生. 生物信息学[J]. 生物物理学报, 1999, 15(1): 5-12. CHEN Runsheng. Bioinformatics[J]. Biophysics journal, 1999, 15(1): 5-12. DOI:10.3760/cma.j.issn.1674-1927.2003.05.044 (  0) 0) |
| [7] |
BARRETT T, WILHITE S E, LEDOUX P, et al. NCBI GEO: archive for functional genomics data sets--update[J]. Nucleic Acids Research, 2013, 39(Database issue): 1005-10. DOI:10.1093/nar/gks1193 (  0) 0) |
| [8] |
GRAY K A, YATES B, SEAL R L, et al. Genenames.org: the HGNC resources in 2015[J]. Nucleic Acids Research, 2015, 43(Database issue): 1079-85. (  0) 0) |
| [9] |
KENT W J, SUGNET C W, FUREY T S, et al. The human genome browser at UCSC[J]. Genome Research, 2002, 12(6): 996-1006. DOI:10.1101/gr.229102 (  0) 0) |
| [10] |
李传星, 郝大鹏, 李霞, 等. 浅谈在生物信息学教学中的学科统一性[J]. 价值工程, 2010, 221(221): 184-184. LI Chuanxing, HAO Dapeng, LI Xia, et al. On discipline unity in bioinformatics teaching[J]. Value Engineering, 2010, 221(221): 184-184. DOI:10.3969/j.issn.1006-4311.2010.33.171 (  0) 0) |
| [11] |
张幸果, 丁俊强, 朱伟, 等. 关于如何提高生物信息学教学质量的探讨[J]. 江西农业学报, 2010, 22(3): 194-195. ZHANG Xing Guo, DING Junqiang, ZHU Wei, et al. Discussion about how to improve the quality of bioinformatics teaching[J]. Jiangxi Agricultural Journal, 2010, 22(3): 194-195. DOI:10.3969/j.issn.1001-8581.2010.03.063 (  0) 0) |
| [12] |
高亚梅, 王伟东, 戴凌燕, 等. 信息技术与生物信息学实验教学整合的探索与实践[J]. 生物信息学, 2011, 9(3): 247-249. GAO Yamei, WANG Weidong, DAI Lingyan, et al. The exploration and practice of the integration of the experimental teaching of information technology and bioinformatics[J]. China Journal of Bioinformatics, 2011, 9(3): 247-249. DOI:10.3969/j.issn.1672-5565.2011.03.017 (  0) 0) |
| [13] |
冉景盛, 姚启伦. 生物信息学创新教学模式初探[J]. 生物学通报, 2009, 44(1): 46-48. RAN Jingsheng, YAO Qilun. Innovative teaching mode of bioinformatics[J]. Biological Bulletin, 2009, 44(1): 46-48. DOI:10.3969/j.issn.0006-3193.2009.01.018 (  0) 0) |
 2018, Vol. 16
2018, Vol. 16

