2. 齐鲁工业大学(山东省科学院)山东省计算中心(国家超级计算济南中心),济南 250101
2. Shandong Computer Science Center(National Supercomputer Center in Jinan) Qilu University of Technology (Shandong Academy of Sciences), Jinan 250101, China
蛋白质二级结构是由蛋白质初级结构链折叠形成的一些通用结构,蛋白质初级结构是指构成蛋白质的氨基酸序列,蛋白质的组成包含20种天然的氨基酸,为了定义氨基酸序列折叠形成的结构,一种流行的算法是DSSP[1](Define secondary structure of proteins)算法,DSSP定义了八种蛋白质二级结构(β桥,β折叠,310螺旋,α螺旋,π螺旋,转角,β转角,其他的结构如螺旋),并将这八种状态结构合并成三种,分别为螺旋(用H表示),链(用E表示)和卷曲(用C表示)。
在生物信息学中,为了预测蛋白质二级结构,蛋白质的氨基酸序列携带的蛋白质结构信息是重要的样本信息来源[2]。PSSM(置特异性打分矩阵)作为一种蛋白质序列的编码方法,PSSM矩阵可以对蛋白质数据库中蛋白质序列进行比对和打分,将蛋白质原始序列编码后可以包含蛋白质残基序列的相对完整的信息,将PSSM应用于蛋白质二级结构预测开始于文献[3]。而许多深度学习应用于蛋白质二级结构预测的文献也都采用PSSM来表示蛋白质残基,文献[3, 6]采用基于深度学习的模型应用于蛋白质二级结构预测,原残基序列样本被编码成PSSM当作输入特征矩阵。
本文中对PSSM预测蛋白质二级结构做了研究和试验,在研究过程中,本文采用CNN(卷积神经网络)作为预测模型,考虑到卷积神经网络的预测结果不稳定,所以文中最终采用ensemble(集成方法)对实验结果进行整合,并且为了提高预测效果,本文对原本设计的卷积神经网络进行了改进,将LSTM(长短时记忆神经网络)与CNN结合,设计了两类CNN神经网络,经过试验,预测结果虽然没有显著提高,稳定性得到了提升。
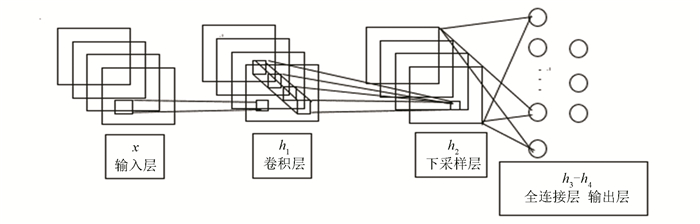
1 模型原理与方案提出 1.1 卷积神经网络简单结构根据文献[7],CNN基本包含卷积层和池化层(在文献[7]称作下采样层)单元,根据文献[7]这些卷积层和池化层单元的级联构成了卷积神经网络隐层,在文献[7]表明卷积神经网络的分类层之前要有全连接层。
如图 1所示的CNN中,只包含一个卷积层一个下采样层和一个全连接层,最后是输出层,假设输入层与卷基层设置α个卷积核,则卷积层的输出的第α个特征面h1, α=f(x*W1, α+b1, α),其中W1, α表示x样本的第α个卷积核,b1, α表示对应卷积核偏置。完成下采样后的特征面为h2, α=g(β2downλ2, τ2(h1, α)+γ2),那么全连接层可以表示为(hα1…hαn),如果有多个卷积层,假设第k层的下采样面是hk, αi(i∈{1, 2…n}),第k+1层表示卷积层,卷积核个数设置为w,则该卷积层构造的第w个卷积面
|
图 1 卷积神经网络简单结构图 Figure 1 Simple architecture of convolutional neural network |
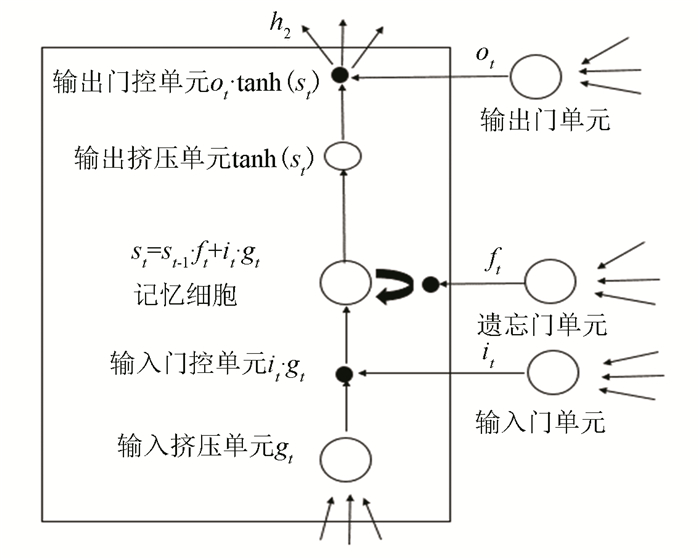
LSTM神经网络是一种循环神经网络,是在文献[9]中,为了解决其他循环网络消耗时间过长,梯度消失等不足而提出的,后来文献[10]在文献[9]基础上为LSTM加入了记忆扩展结构,使隐层记忆状态计算依赖于遗忘门中的权值矩阵,发展成了现代LSTM网络。
如图 2所示,在标准的LSTM模型中,t时刻样本通过输入门与t-1时刻LSTM模型的输出共同计算出输入it→it=σ(Wxi xt+Whi ht-1+bi),同时通过输入挤压单元与t-1时刻LSTM的输出计算出新的记忆gt→gt=tanh (Wxg xt+Whg ht-1+bg),再次通过遗忘门单元与t-1时刻LSTM的输出计算出决定记忆的状态因子ft→ft=σ(Wxf xt+Whf ht-1+bf),最后新的记忆gt与t时刻的输入it的点乘结果结合t-1时刻的记忆状态St-1与t时刻记忆状态因子ft的点乘结果共同更新了t时刻的最终记忆状态St→St=St-1∘ft+it∘gt,LSTM的输出是t时刻的样本输入与LSTM模型t-1时刻的输出通过输出门单元计算出的t时刻输出因子ot→ot=σ(Wxo xt+Who ht-1+bo),之后与t时刻LSTM模型的最终记忆状态的激活函数值点乘计算出的结果ht→ht=ot∘tanh (St)。
|
图 2 LSTM简单原理图 Figure 2 Simple schematic of LSTM |
LSTM的模型的输入包括样本大小维度,是指LSTM输入的样本个数,特别的包含一个时间维度,此维度代表时间片的大小,体现在图 1中就是t的大小,而最后的数据维度代表当前时间片的数据内容。
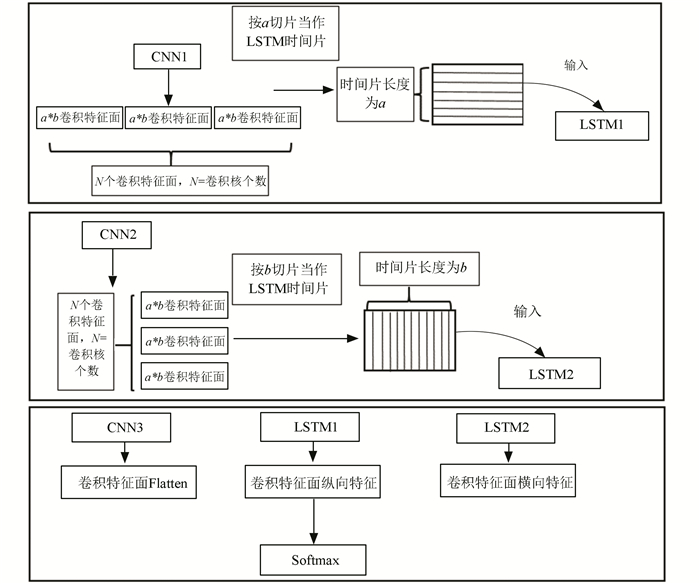
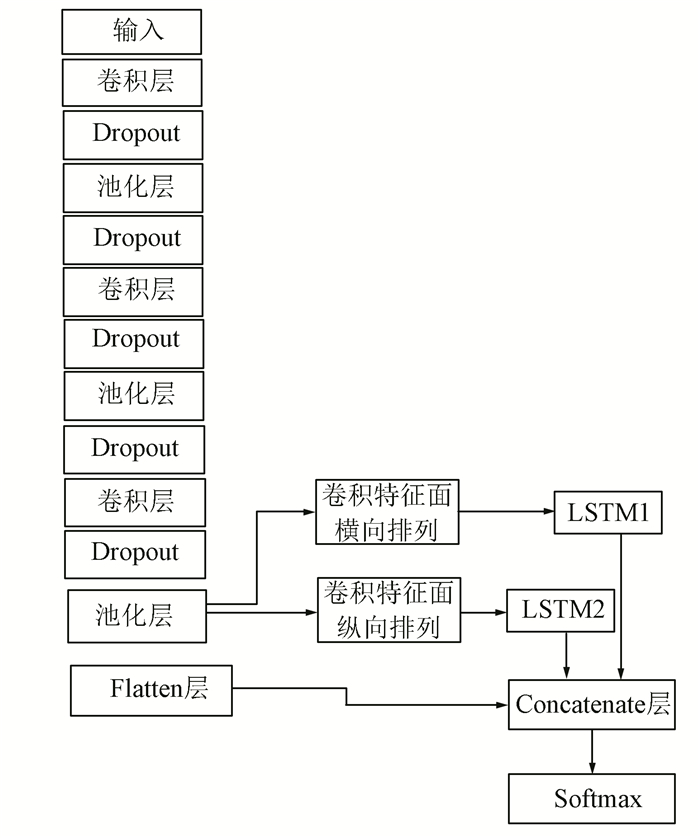
1.3 将LSTM与CNN组合在本文提出的方案中,在CNN的Flatten层之前(在图 1中为全连接层h3),假设h3是a×b×n的卷积特征面输出,其中a和b分别为卷积特征面的长度和宽度,n为卷积特征面的个数,为了训练两个LSTM模型,分别将卷积特征面纵向排列将卷积面的宽度作为时间维度输入LSTM神经网络,再将卷积特征面横向排列将卷积特征面的长度作为时间维度输入一个LSTM神经网络,最后将两个LSTM的输出连同卷积特征面共同经过Concatenate操作经过softmax完成分类(如图 3)。
|
图 3 LSTM与CNN组合原理图 Figure 3 Schematics of combination of LSTM and CNN |
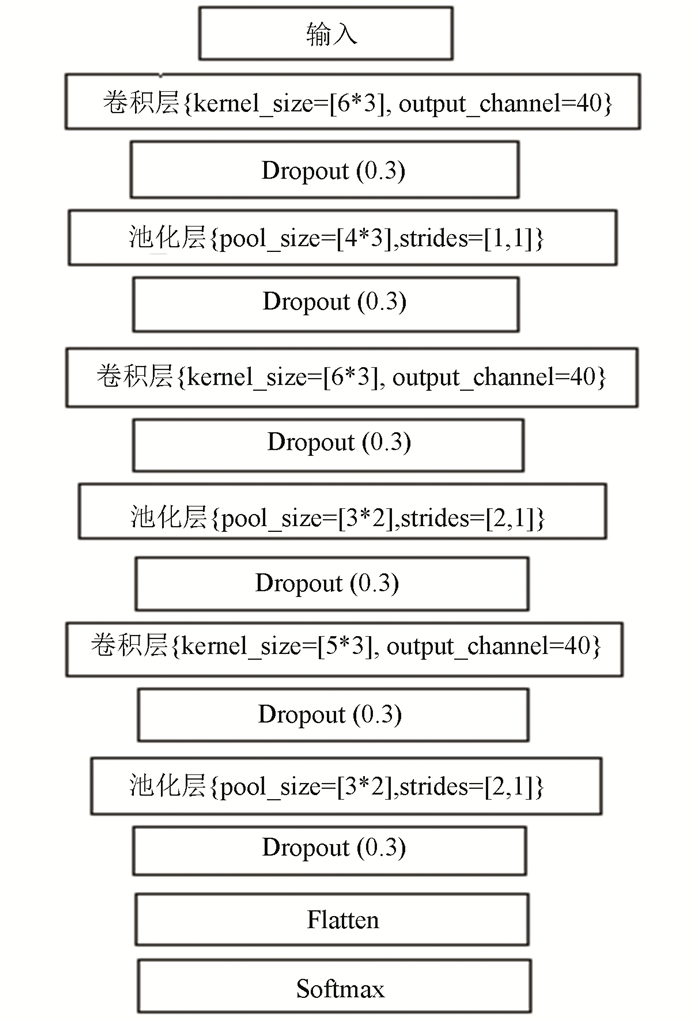
首先本文中CNN的设计原则是CNN的隐层越多越好,按照文献[11]中的描述,卷积神经网络的层数越多,每层的计算任务就越简单,因为每层的输入和输出相差就不会很大,简单的层模型有利于整个模型构建。在文献[12]中,作者说明在分类前的卷积特征面要足够小,同时卷积核要设置的足够大有利于模型的构建。由于PSSM组成的二维数据平面的大小的限制,本文CNN设计成了三层,每层设置40个卷积核,为了避免CNN训练过程中的过拟合,本文在CNN的设计中采用了Dropout[13]技术。本文CNN的结构图如图 4。
|
图 4 CNN结构图 Figure 4 Structural design of CNN |
在上述CNN设计完成的基础上,我们将CNN的最后计算的卷积特征面经过适当处理,分别作为两个LSTM模型的输入,模型设计结构如图 5。
|
图 5 CNN+LSTM结构图 Figure 5 Structural design of CNN+LSTM |
LSTM中也加入了dropout技术,同时设置LSTM的输出为整个时间片对应的序列,LSTM隐层个数设置为20个。
2.3 Ensemble方法的设计由于两类CNN网络产生的预测结果会不稳定,模型之间预测结果会存在差集,根据文献[14]本文采用了ensemble方法来解决这个问题,在多个分类器存在预测结果差异的情况下,ensemble方法会将多个分类器的结果结合在一起会取得更好的结果[14]。
本文ensemble方法中包含两类模型,一种是CNN模型,另一种模型是将LSTM与CNN组合的模型,这两类模型分别训练三个,一共六个模型,最后ensemble方法的输出为六个模型每类均值最大值。
关于Ensemble方法的实现,假设一共训练好N个预测模型,将预先训练好的N个模型分别预测数据样本,将预测结果进行整合,假设Modeln在第i类二级结构的预测结Outputni(i∈{1, 2, 3},n表示模型下标),Ensemble模型的输出为
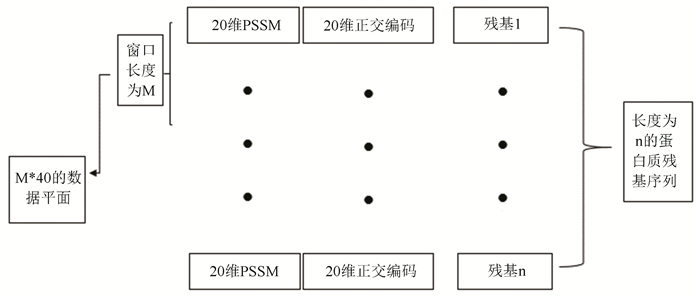
本文中将蛋白质序列取窗口大小为m来表示单个残基,蛋白质每个残基表示成20维的PSSM信息和20维的正交编码,每个残基被编码成了40维的信息,取窗口后,每个残基被编码成m*40的2维数据平面作为每个氨基酸残基的模型输入,如图 6所示。本文中采用CB513[15]蛋白质序列样本作为数据样本,CB513中包含513个蛋白质氨基酸序列, 同源相似性小于25%。PSSM用BLAST工具生成, 二级结构标签的定义采用DSSP的三类定义螺旋(H),折叠(E),卷曲(C),数据集中513个蛋白质样本一共84 119个氨基酸残基经PSSM编码后作为训练样本,螺旋(H)有29 097个, 折叠(E)有17 897个,卷曲(C)有37 125个。
|
图 6 蛋白质样本的编码示意图 Figure 6 Encoding diagram of protein sample |
在试验中,采用Q3作为预测结果的评估方法:
根据文献[16],利用PSI-BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)工具调用3次迭代,检测进化矩阵设置为BLOSUM62矩阵,E-value设置为0.00 l搜索CB513数据集中每个蛋白质样本生成相应的PSSM矩阵,在搜索蛋白质数据库是选择非冗余蛋白质序列库(nr),PSI-BLAST有本地化工具只需将nr(ftp://ftp.ncbi.nih.gov/blast/db/FASTA/nr.gz)序列库下载到本地即可。
4 结果与讨论在本文提出的方案的实现过程中,以keras[17]平台框架作为工具, 以Python3.6作为开发环境。在实验过程中我们采用三折交叉验证方法对试验结果进行了统计,将数据样本分为三份,2/3作为训练集1/3作为测试集。
因为ensemble需要三个模型,本文对CNN的设计一共训练了三个CNN模型。
对于CNN与LSTM组合的模型,对于Ensemble方法设计需要三个。
| 表 1 CNN模型交叉验证实验结果 Table 1 Cross validation test results for CNN |
| 表 2 (CNN+LSTM试验交叉验证结果) Table 2 Cross validation test results for CNN+LSTM |
图 7为前两个模型三次试验结果的对比折线图。

|
图 7 CNN与CNN+LSTM的对比结果 Figure 7 Comparison between CNN and CNN+LSTM |
在图10中,横向观察,不论是只用CNN还是LSTM与CNN的组合,蛋白质二级结构的预测结果都不很稳定,有一定的起伏;纵向观察,可以明显的观察出CNN模型中QH的结果要比LSTM与CNN的组合的QH结果高,而LSTM与CNN的组合的QC结果要高,所以两类模型有差集。文中最后采用了ensemble方法将每类这三个模型整合在一起取均值后作为ensemble模型的预测输出。
Ensemble方法的实验结果如表 3。
| 表 3 Ensemble方法的实验结果 Table 3 Test result of Ensemble method |
通过对比可以发现,两类模型成员经过集成方法,原本的预测结果并没有缩减,证明ensemble是可以增加模型的稳定性,并且集成方法的两类模型预测结果的差集通过ensemble可以很好的得到整合起到提高预测结果的作用。
其他方法与本文中方法的对比(CB513的实验结果)。
| 表 4 CB513数据集预测结果对比 Table 4 Comparison of prediction results for CB513 dataset by different methods |
由上表可以看出,Ensemble方法卷曲(C)的预测结果最高,Ensemble(CNN+LSTM)模型的卷曲预测结果达到了86.1%, 并且Ensemble(CNN only)的螺旋(H)的预测结果77.1%与最高结果79.8%相比,差距很小。
5 结论本文通过将CNN(卷积神经网络)应用到蛋白质二级结构预测,并且将LSTM与CNN组合之后,将两类CNN模型通过ensemble方法整合在一起,从实验结果来看ensemble方法的应用能够综合多个模型的输出结果并使结果有所提高,另一方面将LSTM应用到CNN之后,CNN模型预测结果会与原CNN预测结果产生差集,这使得将两类CNN模型整合在一起使结果更加稳定和有提高的空间。
| [1] |
KABSCH W, SANDER C. Dictionary of protein secondary structure: pattern recognition of hydrogen-bonded and geometrical features[J]. Biopolymers, 1983, 22(12): 2577-2637. DOI:10.1002/(ISSN)1097-0282 (  0) 0) |
| [2] |
DING Y S, ZHANG T L, CHOU K C. Prediction of protein structure classes with pseudo amino acid composition and fuzzy support vector machine network[J]. Protein & Peptide Letters, 2007, 14(8): 811-815. (  0) 0) |
| [3] |
JONES D T. Protein secondary structure prediction based on position-specific scoring matrices[J]. Journal of Molecular Biology, 1999, 292(2): 195-202. DOI:10.1006/jmbi.1999.3091 (  0) 0) |
| [4] |
LI Z, YU Y. Protein secondary structure prediction using cascaded convolutional and recurrent neural networks[A]. Proceedings of the twenty-fifth international joint conference on artificial intelligence[C]. AAAI Press, 2016: 2560-2567.
(  0) 0) |
| [5] |
SPENCER M, EICKHOLT J, CHENG J. A deep learning network approach to ab initio protein secondary structure prediction[J]. Computational Biology & Bioinformatics IEEE/ACM Transactions, 2015, 12(1): 103-112. (  0) 0) |
| [6] |
WANG S, PENG J, MA J, et al. Protein secondary structure prediction using deep convolutional neural fields[J]. Scientific Reports, 2016, 6: 18962. DOI:10.1038/srep18962 (  0) 0) |
| [7] |
DUMOULIN V, VISIN F. A guide to convolution arithmetic for deep learning[EB/OL]. https://pdfs.semanticscholar.org/7918/2aab186f0b68a8432540d8695e1646338479.pdf, 2016.
(  0) 0) |
| [8] |
李彦冬, 郝宗波, 雷航. 卷积神经网络研究综述[J]. 计算机应用, 2016, 36(9): 2508-2515. LI Yandong, HAO Zongbo, LEI Hang. Convolutional neural network research summary[J]. Journal of Computer Applications, 2016, 36(9): 2508-2515. DOI:10.11772/j.issn.1001-9081.2016.09.2508 (  0) 0) |
| [9] |
HOCHREITER S, SCHMIDHUBER J. Long short-term memory[J]. Neural Computation, 1997, 9(8): 1735-1780. DOI:10.1162/neco.1997.9.8.1735 (  0) 0) |
| [10] |
GERS F A, SCHMIDHUBER J, CUMMINS F. Learning to forget: continual prediction with LSTM[J]. Neural Computation, 2000, 12(10): 2451-2471. DOI:10.1162/089976600300015015 (  0) 0) |
| [11] |
SMITH L N, TOPIN N. Deep convolutional neural network design patterns[EB/OL]. https://arxiv.org/abs/1611.00847,2017.
(  0) 0) |
| [12] |
CAO X. A practical theory for designing very deep convolutional neural networks[EB/OL]. https://pdfs.semanticscholar.org/7922/2fad9f671be142bd7e42cd785a2cb06a1d30.pdf, 2015.
(  0) 0) |
| [13] |
SRIVASTAVA N, HINTON G, KRIZHEVSKY A, et al. Dropout: a simple way to prevent neural networks from overfitting[J]. Journal of Machine Learning Research, 2014, 15(1): 1929-1958. (  0) 0) |
| [14] |
MACLIN R, OPITZ D. Popular ensemble methods: an empirical study[J]. Journal of Artificial Intelligence Research, 2011, 11: 169-198. DOI:10.1613/jair.614 (  0) 0) |
| [15] |
CUFF J A, BARTON G J. Evaluation and improvement of multiple sequence methods for protein secondary structure prediction[J]. Proteins-structure Function & Bioinformatics, 1999, 34(4): 508. (  0) 0) |
| [16] |
CUFF J A, BARTON G J. Application of multiple sequence alignment profiles to improve protein secondary structure prediction[J]. Proteins-structure Function & Bioinformatics, 2000, 40(3): 502-511. (  0) 0) |
| [17] |
CHOLLET. Keras[EB/OL]. https://github.com/fchollet/keras,2015.
(  0) 0) |
| [18] |
WANG G, ZHAO Y, WANG D. A protein secondary structure prediction framework based on the Extreme Learning Machine[J]. Neurocomputing, 2008, 72(1-3): 262-268. DOI:10.1016/j.neucom.2008.01.016 (  0) 0) |
| [19] |
HUA S, SUN Z. A novel method of protein secondary structure prediction with high segment overlap measure: support vector machine approach[J]. Journal of Molecular Biology, 2001, 308(2): 397-407. DOI:10.1006/jmbi.2001.4580 (  0) 0) |
| [20] |
冯永娥. 基于打分函数的蛋白质二级结构的识别[J]. 生物数学学报, 2016(4): 455-460. FENG Yonge. Identification of protein secondary structure based on scoring function[J]. Journal of Biomathematics, 2016(4): 455-460. (  0) 0) |
 2018, Vol. 16
2018, Vol. 16


