多不饱和脂肪酸(Polyunsaturated fatty acid,PUFA),通常是指双键个数在两个或两个以上的直链脂肪酸,研究表明多不饱和脂肪酸是人体不可缺少的营养物质,具有多种生物学功能[1]。其中,ω-3多不饱和脂肪酸对心脑血管疾病以及癌症的防治等方面都有着极其重要的作用,并且是脑组织和一些神经细胞的重要组成成分,如果人体长期摄入不足,易导致心脏和大脑等重要器官的部分功能不能正常执行[2-3]。最近ω-3PUFAs还被确认具有抗癌性,也有证据表明当给药ω-3 PUFAs时,可有效地提高常规化疗的耐受性[4]。
目前人们依然是从深海鱼体内获取这种多不饱和脂肪酸,但由于鱼类自身合成十分有限,且近年来海洋污染加剧,很难满足人们对多不饱和脂肪酸的需求[5]。参考前人研究,可通过生物工程的方法将去饱和酶基因转入高等哺乳动物体内让动物体内自发产生这种脂肪酸,这种方法不仅可以降低成本同时可以减少对海洋生物的捕捞,还能满足人们的大量需求[6]。
但在生物体中存在着密码子使用的偏爱性[7],即不同生物对编码同一种氨基酸的同义密码子的使用情况具有很大的差异[8],这种偏爱性很大程度上与生物体细胞中tRNA丰度有联系[9],tRNA分子在翻译过程中通过与mRNA配对将氨基酸运载到核糖体,且相同物种tRNA池的数量及种类是一定的,而不同物种或不同器官之间的tRNA丰度会有很大的差异[10]。如果某种tRNA被大量使用,就会造成该种tRNA池缩小,反之tRNA池会有盈余[11-12]。当tRNA减少时,不能及时到达核糖体的胺酰部位,会导致核糖体翻译停止,产生报警信号,造成蛋白产量减少。所以当转入外源基因时,如果含有大量宿主的稀有密码子就有可能导致表达水平的降低,故应先对目的基因进行优化改造。
密码子优化是提高外源基因在宿主体内表达量使用最普遍的重要工具之一[13],为了满足需求,网上涌现大量用于优化基因的在线软件,本研究将对在线优化和手动优化的结果进行比较,以期得出最适于提高表达水平的优化方法,为后期生产转基因动物做准备。本文采用了大豆的ω-3和ω-6脂肪酸脱氢酶作为试验材料,大豆富含PUFAs及合成相关的酶类,且ω-3脂肪酸脱氢酶是合成α亚麻酸的关键酶,其上游基因ω-6脂肪酸脱氢酶为反应过程的限速酶[14-17],所以同时以这两个基因为基础根据人类基因组密码子的使用偏好对其优化,并通过分析一系列适应性参数来初步预测优化结果的表达水平,比较得出更优的序列来用于后期进一步的实验验证。
1 材料与方法 1.1 基因通过分析质体和微体在植物与动物体内的分布情况,质体只在植物细胞中出现,而微体在动植物细胞内均有分布,所以决定选用大豆微体中的ω-3和ω-6脂肪酸脱氢酶基因进行本次研究,ω-3脂肪酸脱氢酶基因编码序列引自GenBank的Glycine max cultivar Jack microsomal omega-3 fatty acid desaturase B (FAD3B) mRNA,登录号为EF632325,CDs序列长度为1 143 bp。ω-6脂肪酸脱氢酶基因引自GenBank的Glycine max FAD2-1 microsomal omega-6 desaturase mRNA,登录序列号为L43920,CDs长度为1 164 bp。
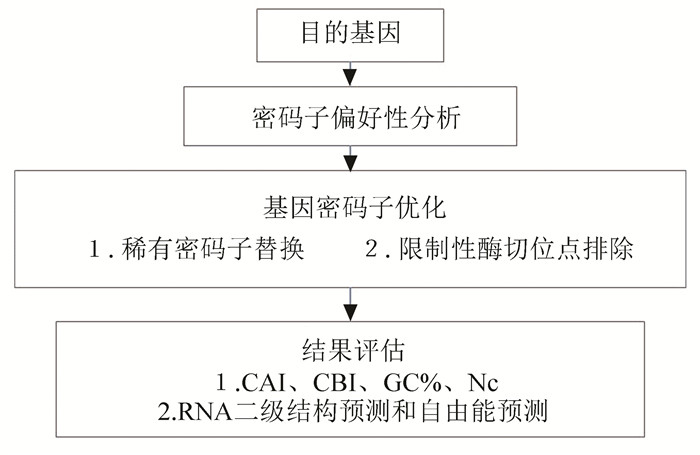
1.2 密码子的优化本研究优化的基本思路为:首先分析人类基因组和本次实验外源基因的密码子使用情况,然后比较并找出外源基因中的限速密码子,用人类基因组最优密码子替代。优化流程如图所示(见图 1)。人类基因组密码子使用情况的统计可由在线软件Codon Usage Database(http://www.kazusa.or.jp/codon/)得到。大豆ω-3和ω-6脂肪酸脱氢酶基因编码序列RNA的密码子使用偏好性通过CodonW软件分析得到。将大豆ω-3和ω-6脂肪酸脱氢酶基因与人类的密码子偏好性进行统计比较(见表 1),整理出可能的限速密码子,然后用人类基因组的最优密码子代替。另外使用网上已有的在线优化软件Jcat(http://www.jcat.de/)直接对该基因进行优化,JCat (Java Codon Adaptation Tool)是一种新的优化方法,可以根据大多数已测序的原核生物和真核宿主的密码子偏好性对目标基因序列进行适应性调整。在现有的软件中,JCat可直接输出优化后的高表达基因,是非常快速容易的一种方法[18]。该软件在网上免费开放(http://www.prodoric.de/JCat),可通过直接在网络提交序列进行分析,而且有多个物种可以进行选择。
|
图 1 优化流程图 Figure 1 Optimization progress |
| 表 1 目的基因与人类基因组密码子使用情况比较 Table 1 The codon usage comparison in target genes and human |
将优化后的序列用BioEdit软件翻译并进行序列对比,比较优化前后蛋白质序列,确认氨基酸序列没有改变。由于翻译起始的效率同RNA二级结构的稳定性有很大关系,包括与核糖体的结合,且对于基因的表达和蛋白质的合成有着重要的作用[19-20],所以二级结构的预测可以初步推断基因的表达水平,本研究使用在线软件ViennaRNA Services(http://rna.tbi.univie.ac.at/)中的RNAfold WebServer获得优化前后基因序列的RNA二级结构及最小自由能。在确定最佳优化后的基因序列之后,用BioEdit软件识别其中的常见酶切位点,并通过替换相关密码子,在不改变氨基酸序列的前提下排除一些常见的酶切位点。最后应用软件CodonW分别计算两种方式优化前后的基因序列CDS的CBI值、CG%、GC3s、Nc值和CAI值。
2 结果与讨论 2.1 密码子使用频率分析及改造根据表 1的统计结果对比原始序列和人类的偏好性,并进行一些适当调整。在登录号为EF632325的RNA中,最优密码子使用不一致的氨基酸有Gly、Leu、Asn、Pro、Gln和Thr等11个,除此之外Arg、Ser等的次优密码子频率也有较大差异。优化时在考虑替换最优密码子的同时,也要考虑到目的基因中一些次优密码子在人类基因组中可能会是使用频率极小或不使用的稀有密码子,有时这些密码子也会占有很大的比重。此时也要将此类密码子替换为人类最优密码子。L43920的改造也按照同样的方法,包括最优密码子不同的Ala、Asp、Gly、Leu、Gln等11个和次优密码子不同的Val等。另外用Jcat在线软件优化后,原序列中的密码子都替换为人类基因组的最优密码子。
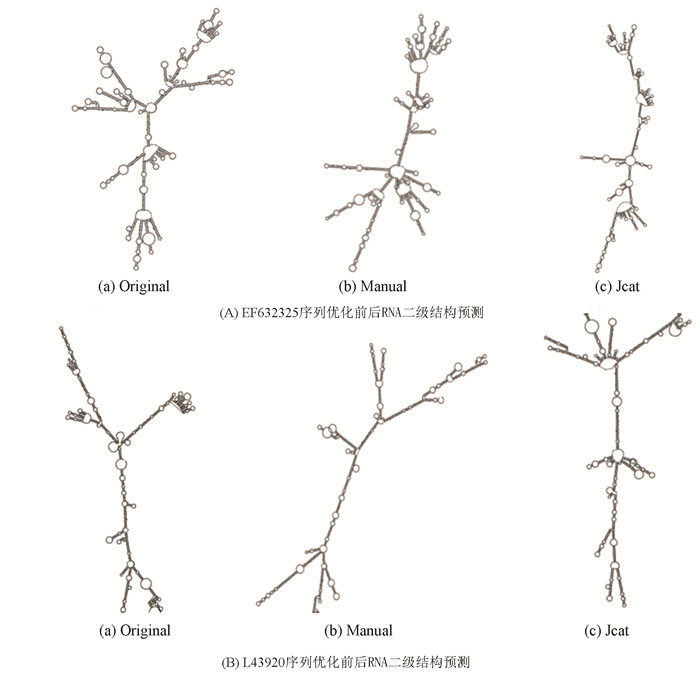
2.2 RNA二级结构预测根据研究表明RNA二级结构对于表达水平具有重要意义[19],用在线软件RNAfold Web Server预测得到的二级结构折叠情况见图 2,以及每个二级结构的最小自由能(见表 2)。结果显示,两个序列在优化后的结构分支和拥挤都减少了,最小自由能都较原来的小很多,可以推测两种优化后的RNA都较原来的结构更优更稳定,相应的表达水平也更高。手动优化和JCAT优化的结果都比较相似,但其中EF632325序列通过Jcat优化后的基因结构较手动同义密码子替换优化结果更加稳定,且没有过度拥挤,而L43920序列的结果中手动优化更优,两者计算得出的最小自由能也非常接近,初步预测结果为都可以表达,优化后的序列表达水平更高。
|
图 2 RNA二级结构预测 Figure 2 The RNA secondary structure prediction |
| 表 2 RNA二级结构最小自由能 Table 2 The minimum free energy of the RNA secondary structure |
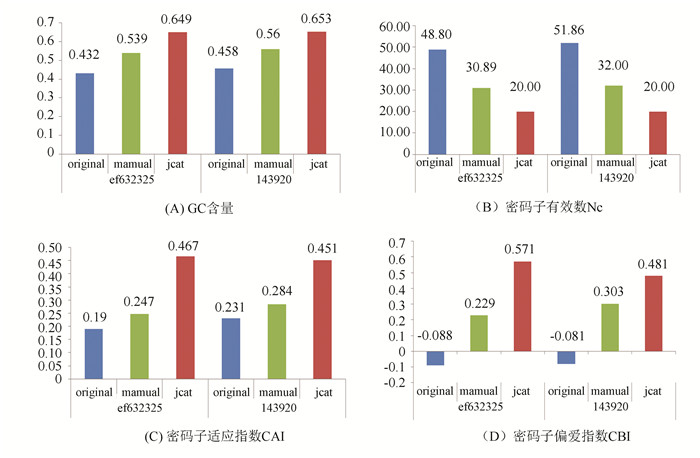
密码子有效数(Effective number of codons, Nc)是一个基因的密码子使用频率与同义密码子平均使用频率偏差的量化值,改制的范围为20~61,越接近20偏好性越强。此值可以反映同义密码子非均衡使用的偏好程度,提供一个客观的评价标准。GC含量表示密码子第三位碱基中G+C出现的频率,有研究表明,人类基因显示对以C或G,尤其C结尾的同义密码子的偏好。由于以C/G结尾的密码子比以A/U结尾的同义密码子具有更强的密码子-反密码子的结合能力,可以推断偏好C/G结尾的密码子有利于提高翻译准确度[21]。密码子适应指数(Codon Adaptation Index,CAI)常用于基因表达水平的测量。此值范围为0~1, 越接近1表示基因的表达水平越高。密码子偏爱指数(Codon Bias Index,CBI)表示的是在一个特定的基因中高表达的偏好密码子占总体的比重,可以非常明确地反映外源基因在其体内可能的表达情况。由数据可以推断两种优化方法得到的序列的偏好性和表达水平都有了明显的提升(见图 3)。
|
图 3 密码子适应性分析 Figure 3 The codon adaption analysis |
1) 本研究针对人类基因组密码子的使用特性对大豆ω-3和ω-6脂肪酸脱氢酶基因的编码序列进行改造,然后通过在线提供的软件分析预测了优化后结果的表达水平,从而得出最优的优化方法。
2) 优化过程中同时使用两种方法对目的基因进行了优化。一种是根据两物种密码子使用情况适当替换同义密码子,同时考虑最优密码子和次优密码子等密码子的使用,综合优化;另一种方法采用网上开放的软件Jcat对目标序列进行人源化,该方法直接用人类基因的最优密码子全部替换其他同义密码子。
3) 通过对两种优化方法优化后序列的适应性的分析,两种方法都可以明显的提高基因的表达量。其中Jcat优化后的基因序列的GC%、CAI、CBI都显著提高,并远远超过原序列和手动优化的结果,且Nc值为20,RNA二级结构也更稳定,但考虑到原基因的一些序列结构和特性,且如果全部使用最优密码子会造成该种密码子对应的tRNA被大量使用,远远超出其宿主体内该种tRNA的含量,而tRNA的匮乏会造成蛋白受阻,不能达到预期的结果。所以在考虑提高表达水平又能保持tRNA含量均衡的基础上,手动的优化结果更加适合进一步的转基因实验。
为了后期可以做到更精准的预测,接下来需要考虑更多影响外源基因在宿主体内表达的因素并加入到优化的方案中,比如启动子、UTR、密码子偏好性、内含子、DNA修饰水平等。另外,由于本研究只通过网上在线得到的参数对表达水平进行了预测,不能更直接地证明所获得的序列是否可以真的在哺乳动物体内表达,且本研究是基于人类基因组优化的,所以下一步将根据具体某种实验动物的密码子使用偏好进行优化,并进行实际实验操作来验证优化后的基因序列在宿主体内的表达水平是否有得到提升。
| [1] |
SCHMITZ G, ECKER J. The opposing effects of n-3 and n-6 fatty acids[J]. Progress in Lipid Research, 2008, 47(2): 147-155. DOI:10.1016/j.plipres.2007.12.004 (  0) 0) |
| [2] |
SARAVANAN P, DAVIDSON N C. Cardiovascular effects of marine omega-3 fatty acids[J]. Lancet, 2010, 376(9740):540-550. DOI:http://dx.doi.org/10.1016/S0140-6736(10)60445-X.
(  0) 0) |
| [3] |
DAS U N, FAMS M D. Long-chain polyunsaturated fatty acids in the growth and development of the brain and memory[J]. Nutrition, 2003, 19(1): 62-65. DOI:10.1016/S0899-9007(02)00852-3 (  0) 0) |
| [4] |
COCKBAIN A J, TOOGOOD G J, HULL M A. Omega-3 polyunsaturated fatty acids for the treatment and prevention of colorectal cancer[J]. Gut, 2012, 61: 135-149. DOI:10.1136/gut.2010.233718 (  0) 0) |
| [5] |
WILLIAMS C M, BURDGE G. Long-chain n-3 PUFA: plant v. marine sources[J]. Proceedings of the Nutrition Society, 2006, 65(1): 42-50. DOI:10.1079/PNS2005473 (  0) 0) |
| [6] |
李文宗, 王磊. 长链多不饱和脂肪酸EPA、DHA的基因工程研究进展[J]. 生物技术通报, 2016, 32(8): 1-7. LI Wenzong, Wang Lei. Research progress on genetic engineering for long-chain polyunsaturated fatty acids EPA and DHA[J]. Biotechnology Bulletin, 2016, 32(8): 1-7. DOI:10.13560/j.cnki.biotech.bull.1985.2016.08.001 (  0) 0) |
| [7] |
武志娟, 钟金城. 密码子偏性及其应用[J]. 生物学通报, 2012, 47(4): 9-11. WU Zhijuan, ZHONG Jincheng. Codon bias and its application[J]. Biology Bulletin, 2012, 47(4): 9-11. DOI:10.3969/j.issn.0006-3193.2012.04.004 (  0) 0) |
| [8] |
HIROSHI A. Gene expression and molecular evolution[J]. Current Opinion Genetics & Development, 2001, 11(6): 660-666. DOI:10.1016/S0959-437X(00)00250-1 (  0) 0) |
| [9] |
WEISS R B. Molecular model of ribosome frameshifting[J]. Proceeding of the National Academy of Sciences of the United States of America, 1984, 81(18): 5797-5801. DOI:10.1073/pnas.81.18.5797 (  0) 0) |
| [10] |
冯德江, 刘翔, 李旭刚, 等. tRNA丰度与基因表达的关系[J]. 中国生物工程杂志, 2002, 22(6): 4-8. FENG Dejiang, LIU Xiang, LI Xugang, et al. The relationship between tRNA abundance and gene expression[J]. Journal of Chinese Biotechnology, 2002, 22(6): 4-8. DOI:10.13523/j.cb.20020602 (  0) 0) |
| [11] |
IKEMUM T. Codon usage and tRNA content in unicellular and multicellular organisms[J]. Molecular Biology & Evolution, 1985, 2(1): 13-34. DOI:10.1093/oxfordjournals.molbev.a040335 (  0) 0) |
| [12] |
ANTEZANA M A, Kreitman M. The nonrandom location of synonymous codons suggests that reading frame-independent forces have patterned codon preferences[J]. Journal of Molecular Evolution, 1999, 49(1): 36-43. DOI:10.1007/PL00006532 (  0) 0) |
| [13] |
GUSTAFSSON C, GOVINDARAJAN S, MINSHULL J. Codon bias and heterologous protein expression[J]. Trends in Biotechnology, 2003, 22(7): 346-53. DOI:10.1016/j.tibtech.2004.04.006 (  0) 0) |
| [14] |
曹福亮, 王欢利, 郁万文, 等. 高等植物脂肪酸去饱和酶及编码基因研究进展[J]. 南京林业大学学报(自然科学版), 2012, 36(2): 125-132. CAO Fuliang, WANG Huanli, YU Wanwen, et al. Progress of research on fatty acid desaturase and their coding genes in higher plant[J]. Journal of Nanjing Forestry University, 2012, 36(2): 125-132. DOI:10.3969/j.issn.1000-2006.2012.02.026 (  0) 0) |
| [15] |
ALONSO D L, MAROTO F G, RUIZ R G, et al. Evolution of the membrane-bound fatty acid desaturases[J]. Biochemical Systematics and Ecology, 2003, 31(10): 1111-1124. DOI:10.1016/S0305-1978(03)00041-3 (  0) 0) |
| [16] |
MUSTARDY L, LOS D A, GOMBOS Z, et al. Immunocytochemical localization of acyl-lipid desaturases in cyanobacterial cells: Evidence that both thylakoid membranes and cytoplasmic membranes are sites of lipid desaturation[J]. Proceeding of the National Academy of Sciences of the United States of America, 1996, 93(19): 10524-10527. DOI:10.1073/pnas.93.19.10524 (  0) 0) |
| [17] |
刘永红, 张丽静, 张洪荣, 等. △12-脂肪酸脱氢酶及其编码基因研究进展[J]. 草业学报, 2011, 20(3): 256-267. LIU Yonghong, ZHANG Lijing, ZHANG Hongro, et al. Progress of research on △12 fatty acid desaturases and their coding genes[J]. Acta Prataculae Sinica, 2011, 20(3): 256-267. DOI:10.11686/cyxb20110332 (  0) 0) |
| [18] |
GROTE A, HILLER K, SCHEER M. JCat: a novel tool to adapt codon usage of a target gene to its potential expression host[J]. Nucleic Acids Research, 2005, 33(Web Server issue): W526-531. DOI:10.1093/nar/gki376 (  0) 0) |
| [19] |
KOZAK M. Influences of mRNA secondary structure on initiation by eukaryotic ribosomes[J]. Proceedings of the National Academy of Science, 1986, 93(9): 2850-2954. DOI:10.1073/pnas.83.9.2850 (  0) 0) |
| [20] |
HALL M N, GABAY J, DÉBARBOUILLÉM. A role for mRNA secondary structure in the control of translation initiation[J]. Nature, 1982, 295(5850): 616-618. DOI:10.1038/295616a0 (  0) 0) |
| [21] |
石秀凡, 黄京飞, 柳树群, 等. 人类基因同义密码子偏好的特征以及与基因GC含量的关系[J]. 生物化学与生物物理进展, 2002, 29(3): 411-414. SHI Xiufan, HUANG Jingfei, LIU Shuqun, et al. The features of synonymous codon bias and GC-content relationship in human genes[J]. Progress in Biochemistry and Biophysics, 2002, 29(3): 411-414. DOI:10.3321/j.issn:1000-3282.2002.03.017 (  0) 0) |
 2018, Vol. 16
2018, Vol. 16


