转录因子对植物的生长、发育和响应外界环境的变化起着重要的作用[1],生长素响应因子(ARF)就是其中的一类,生长素与植物种子的萌发,器官的形成和向性反映等诸多生理过程有密切联系[2]。生长素响应因子能够与生长素响应原件TGTCTC序列特异性结合并调控生长素响应基因的表达[3],这些响应基因包括AUX/IAA家族、CH3家族和受生长素调控的RNA调控的SAUR基因家族[4]。ARF家族成员在N-端有一个能与DNA特异性结合的B3结构域(DBD);在中间有一个与转录活性有关的结构域(MR),MR区域的氨基酸构成和序列长度决定了其分子量和转录活性[5];一般认为,中间区域含丝氨酸(S)、苏氨酸(T)和脯氨酸(P)是抑制子(RD),而富含谷氨酰胺(Q)的多为激活子(AD)[6]。在拟南芥中,AtARF1、2、3、4、9是转录抑制子;ATARF5、6、7、8、9是转录激活子。大多数ARF基因还含有2个C端的结构域(功能域Ⅲ和Ⅳ),能够与ARF蛋白本身或AUX/IAA结合形成同源或异源二聚体[7]。生长素响响应因子蛋白是由一个大的基因家族编码的[7],目前,拟南芥[8]、玉米[6]、雷蒙德氏棉[9]等物种的ARF基因家族均被鉴定出来,且关于模式植物ARF基因家族的功能也有很多研究。亚洲棉与雷蒙德氏棉同为二倍体棉花,有关ARF基因家族的报道还较少。
亚洲棉(Gossypium arboretum L.)是一种原产于亚洲的棉花种类的合称,是人类最早种植的农作物之一,同时也是目前世界最重要的经济棉种陆地棉的祖先。2014年4月,亚洲棉基因组(A基因组)测序的完成[10],为研究ARF基因家族成员的生物学功能提供了丰富的资源。本研究利用生物信息学的方法,鉴定了亚洲棉ARF基因家族,命名为GaARF从基因水平上分析了ARF基因在亚洲棉中的数目、序列特征、进化关系,旨在为进一步研究生长素相关基因提供参考。
1 材料和方法 1.1 GaARF基因家族的筛选及氨基酸序列属性分析以锦葵科亚洲棉为研究对象,亚洲棉全基因组数据来源于中国农业科学院棉花研究所CGP数据库(http://cgp.genomics.org.cn)。使用“BioEdit”软件将获得的亚洲棉全基因组氨基酸序列建立一个本地数据库,然后以拟南芥ARF蛋白序列作为Query序列与建立的亚洲棉氨基酸序列进行BlastP(E-value=0.001)序列比对,将所得结果在Pafm[11]数据库(http://pfam.xfam.org/)和CDD[12](https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进行检测,筛选出含ARF基因特征结构域(B3、AUX/IAA)的序列。将所得到的氨基酸序列使用ExPAsy[13](http//www.expasy.org/)进行在线分析,以确定氨基酸的等电点(PI)、蛋白质的分子量(MW)和不稳定系数。
1.2 GaARF基因家族进化树的构建与分类利用MEGA7.0[14]软件里提供的ClustalW工具对亚洲棉ARF基因氨基酸序列进行多序列比对,然后通过邻接法(NJ,Nwighbor-Joining),校验参数(Bootstrap)设置为1 000,构建系统进化树。不同物种ARF蛋白进化树的绘制:采用拟南芥、玉米共计83个ARF蛋白序列进行综合分析,拟南芥、玉米ARF蛋白序列均来自NCBI数据库(https://www.ncbi.nlm.nih.gov/)。用上述方法进行综合分析,用上述方法绘制ARF蛋白家族的复合进化树。
1.3 染色体定位、基因结构及序列保守性分析亚洲棉染色体长度及起始位点信息从棉花基因数据库(https://www.cottongen.org)中得到,使用MapInspect软件将得到的GaARF基因在染色体上的物理位置绘制成图。按照GaARF基因在染色体上的位置进行命名。采用GSDS[15](http://gsds.Cbi.pku.edu.cn/)在线工具获得亚洲棉ARF家族外显子-内含子结构图。用已得到的蛋白序列,使用MEME[16]在线分析工具(http://meme.sdsc.edu/)分析得到亚洲棉ARF家族蛋白的motif模式。
1.4 信号肽检测及亚细胞定位利用TargetP[17]信号肽预测在线网站(http://www.cbs.dtu.dk/services/TargetP/)信号肽预测程序,对29条亚洲棉ARF蛋白进行信号肽预测。
1.5 组织表达模式分析在棉花转录组数据库(http://www.plexdb.org/plex.php?database=Cotton)中将亚洲棉基因基因数据通过Blast比对,得到GaARF基因在根和叶中的表达情况。
2 结果与分析 2.1 GaARF基因家族成员鉴定利用生物信息学方法,从亚洲棉中共获得29个ARF家族成员(见表 1),同时通过EsPAsy在线网站查询所有的GaARF基因的基本信息,并对蛋白质的长度、分子量、等电点、不稳定系数等理化性质进行分析。结果显示,亚洲棉中29个蛋白序列差异很大,编码ARF蛋白的氨基酸长度范围从576 aa(GaARF11)到1 141 aa(GaARF14), 分子量范围从63.44 kD(GaARF11)到127.54 kD(GaARF14), 等电点范围从5.21(GaARF21)到8.93(GaARF24)。从蛋白的基本特征可以看出该基因家族无论从基因长度到蛋白特性变化都很大,表明该基因家族具有不同的特性,发挥着不同的生物学作用。一般认为,蛋白的不稳定系数大于40时,则蛋白属于不稳定蛋白,本研究中亚洲棉蛋白的不稳定系数都大于43,因此均属于不稳定蛋白。值得注意的是,亚洲棉大部分蛋白的等电点都小于7,表明多数GaARF编码的蛋白呈弱酸性,可能在酸性的亚细胞环境中发挥相应的作用。
| 表 1 GaARF基因家族的鉴定及特性 Table 1 Characteristics of identified GaARF gene family |
系统进化分析是比较同源序列和研究分子进化的方法,利用本研究中的GaARF蛋白构建进化树(见图 1),参照SUN[9]等人的分类方式,将GaARF基因家族分为:Ⅰ、Ⅱ、Ⅲ(Ⅲa、Ⅲb、Ⅲc)、Ⅳ四大类,以此来更好的了解GaARF基因系统的进化关系。由图 1可以看出,GaARF基因中存在11个同源对,其中8对步长值(Bootstrap values)为100,说明这八对ARF基因的亲缘关系非常近;同一亚家族的基因序列和保守序列很相似,表明它们的功能可能很相似,体现了GaARF基因在进化过程的保守性;不同亚家族中的GARF基因在结构上有较大的差异,如第Ⅳ基因家族只有2~4个外显子,而其他三类的外显子数量明显多于第Ⅳ基因家族。所有的GaARF均含motif1、2、3、4,而不同亚家族间的保守区域不同,如亚家族Ⅳ中均含有motif18,不含有motif13、16,表明这些motif可能存在某些特定的功能。基因结构和保守基序的相似性和差异性从侧面反映了GaARF基因家族在漫长的进化过程中的相对保守性和为适应环境而产生的多样性。
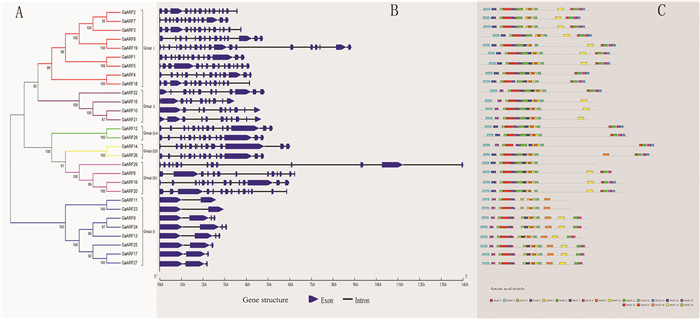
|
图 1 GaARF基因家族的系统发育、基因结构及保守区域 Figure 1 Phylogenetic tree, gene structure and conserved region of GaARF gene family |
为了更好的明确亚洲棉与单、双子叶物种间ARF基因亲缘关系的远近,将已公布的序列信息的单子叶植物玉米(31个);双子叶植物亚洲棉(29个)、拟南芥(23个)共计83个ARF蛋白构建了系统进化树(见图 2)。从图中可以看出,83个ARF家族成员被明显的分成四大类,由进化关系可以发现,亚洲棉和拟南芥较多的聚类到一起,由此可以看出它们之间的进化关系较近。从亚洲棉与拟南芥的聚类关系可以看出,ClassⅠ、Ⅱ中的ARF蛋白是转录抑制子,可能在转录过程中抑制转录的发生;ClassⅢ中的ARF蛋白是转录激活子,可能在转录过程中促进转录的进行。
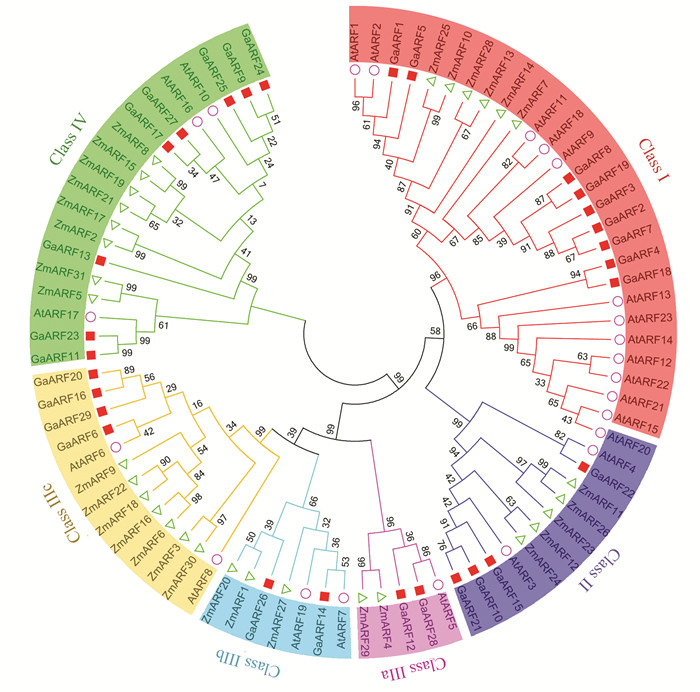
|
图 2 ARF蛋白多物种系统进化树 Figure 2 Phylogenetic tree of ARF protein in multi-species |
物种间基因数量的增加被认为是促进物种进化的方式,基因家族数量增加的主要方式是基因复制,基因复制又分为基因内复制和基因间复制[18]。亚洲棉基因组在复制的过程中经历了几次的基因数量加倍(Gene duplication)事件,这些基因的复制品通常可幸免于选择压力[10]。这不仅为亚洲棉的进化提供了保障,也丰富了亚洲棉基因家族的多样性。为了进一步研究基因分化对GaARF基因家族的影响,我们利用Mapinspect软件将29个GARF基因定位在9个染色体上(见图 3),其中2、3、5、11号染色体不含ARF基因。由图中可以看出,亚洲棉29个ARF基因是随机分布的,其中10号染色体分布最多,有9个ARF基因。一般认为200 kb核苷酸有3个以上基因的基因群,就认为是一个基因簇,亚洲棉中在10号染色体上存在一个基因簇,它们可能编码催化同一个新陈代谢途径中不同步骤的酶的结构基因。根据基因串联复制的定义,GaARF2、3、7;GaARF6、16、20;GaARF17、27;还存在基因间的串联复制。
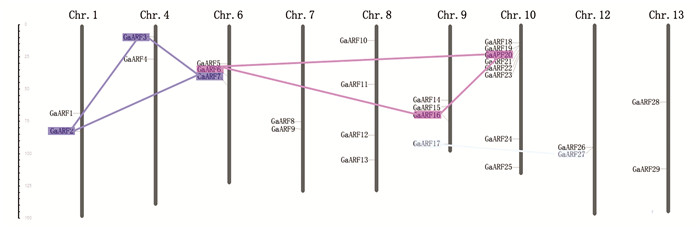
|
图 3 GaARF基因染色体定位图 Figure 3 Chromosomal locations of GaARF gene family |
利用TargetP软件检测,发现有7条(GaARF8、10、15、20、21、22、23)定位到叶绿体上,GaARF11蛋白定位到线粒体上,其余21条蛋白未检测到信号肽信息。
2.6 亚洲棉ARF基因家族表达模式分析为了进一步了解ARF基因的功能和特性,对基因的组织特异性表达和诱导表达模式进行分析(见图 4)。分析结果表明:GaARF1、2、3、7、12、26、28在根中表达较高,推测它们可能参与根的形成;GaARF1、15、17、22、26在叶中的表达较高,表明它们可能在叶的发育过程起到重要的作用。淹水处理后,GaARF11、13、24在根和叶都存在差异表达,暗示着它们可能是参与响应涝害的关键基因,但ARF基因在不同部位的表达差异及作用还有待于进一步研究。
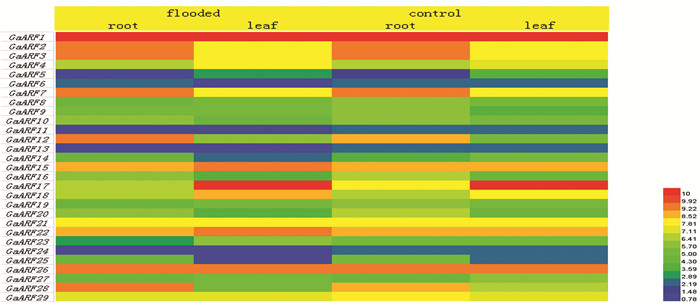
|
图 4 GaARF基因家族表达热图 Figure 4 Heat map of GaARF gene expression |
ARF基因家族是参与生长素信号调节的重要转录因子,关于生长素的功能及作用机制的研究正在持续进行。Ulmasov等于1997年利用酵母单技术以人工合成的AuxREP3(4x)作为分子诱饵,筛选拟南芥cDNA表达文库,鉴定了第一个ARF基因,即AtARF1[3]。到目前为止,在拟南芥中共发现23个ARF基因[8],番茄中发现21个ARF基因[19],玉米中发现31个ARF基因[6],水稻中共发现25个ARF基因[20],苹果中共发现29个ARF基因[21],雷蒙德氏棉共发现35个[9],目前在亚洲棉中还没有ARF基因家族的系统分析和研究,基于此,本文进行了亚洲棉ARF家族的生物信息学分析。
本研究从基因组水平上初步筛选并分析了29亚洲棉ARF基因,并将其分为四大类,系统的阐述了其进化关系。前人研究发现,AtARF1和AtARF2基因参与花器官衰老和脱落的过程[22],通过系统进化树分析发现,GaARF1和GaARF5与AtARF1、2同源性最高,可以推测亚洲棉中GaARF1和GaARF5也参与花器官衰老和脱落过程;AtARF3控制KANAD蛋白合成,参与叶的极性反应,可以推测亚洲棉中同源性最高的GaARF15有着相似的作用;AtARF7和AtARF19作为转录激活子,介导拟南芥侧根形成[8],暗示着GaARF14可能有着相似的作用;AtARF6和AtARF8基因参与调控逆境相关的茉莉酸合成[23],可以推测亚洲棉中GaARF16、20、29可能参与逆境调控相关激素的合成;AtARF10、AtARF16基因的单突变体没有明显的表型,而双突变型根冠和根系向地性生长发生异常[24],亚洲棉中有5个基因和这两个基因属于一个类别,但具体作用还有待考究。
最近的研究还表明ARF基因的表达受到microRNA的调控,microRNA160调控拟南芥AtARF10和AtARF16基因的表达[24];玉米中,microRNA167高表达的突变株能够导致ZmARF3、9、16、18、22、30转录本的降解,暗示ZmARF基因起到转录后的调控作用[6]。亚洲棉由于研究较少,目前还没有ARF基因诱导表达的报道。
前人研究表明,基因家族数量的增加主要是基因复制,基因复制又分为基因内和基因间复制[25],从进化树中可以看出,亚洲棉中存在基因复制的现象。亚洲棉基因组(A)和雷蒙德氏棉基因组(D)在距今约5百万年(2~13百万年)之前从同一祖先分化而来,二者的基因数目和基因序列都极为相近,染色体水平上也保留了高度的共线性,但由于A基因组发生过多次大规模的反转座子插入事件,导致其基因组膨胀至超过D基因组的两倍。但亚洲棉中ARF基因家族数量多于雷蒙德氏棉,可能是因为亚洲棉含有的41 330个编码基因中,68.5%的基因组序列是由重复序列组成的造成的[10]。
ARF基因家族调节植物的激素代谢,在植物的生长发育中起到至关重要的作用。本研究以亚洲棉全基因组序列为研究背景,为研究ARF基因的功能奠定了基础。亚洲棉中ARF基因家族是如何调控生长素发挥作用还需要后人进一步研究。
| [1] |
HORI S, NOMURA T, SAKAGUCHI S. Control of regulatory t cell development by the transcription factor foxp3[J]. Science, 2003, 299(5609): 1057-1061. DOI:10.1126/science.1079490 (  0) 0) |
| [2] |
吴蓓, 吴建勇, 蔡刘体, 等. 生长素反应因子[J]. 植物生理学通讯, 2005, 41(3): 273-278. WU Bei, WU Jianyong, CAI Liuti, et al. Auxin response factor[J]. Plant Physiology Communications, 2005, 41(3): 273-278. DOI:10.13592/j.cnki.ppj.2005.03.001 (  0) 0) |
| [3] |
ULMASOV T, HAGEN G, GUILFOYLE T J. Arf1, a transcription factor that binds to auxin response elements[J]. Science, 1997, 276(5320): 1865-1868. DOI:10.1126/science.276.5320.1865 (  0) 0) |
| [4] |
ABEL S, THEOLOGIS A. Early genes and auxin action[J]. Plant Physiology, 1996, 111(1): 9-17. DOI:10.1104/pp.111.1.9 (  0) 0) |
| [5] |
RAMOS J A, ZENSER N, LEYSER O, et al. Rapid degradation of auxin/indoleacetic acid proteins requires conserved amino acids of domain ii and is proteasome dependent[J]. Plant Cell, 2001, 13(10): 2349-2360. DOI:10.1105/tpc.13.10.2349 (  0) 0) |
| [6] |
XING H, PUDAKE R N, GUO G, et al. Genome-wide identification and expression profiling of auxin response factor (arf) gene family in maize[J]. BMC Genomics, 2011, 12(1): 178. DOI:10.1186/1471-2164-12-178 (  0) 0) |
| [7] |
TIWARI S B. The roles of auxin response factor domains in auxin-responsive transcription[J]. Plant Cell, 2003, 15(2): 533-543. DOI:10.1105/tpc.008417 (  0) 0) |
| [8] |
OKUSHIMA Y, OVERVOORDE P J, ARIMA K, et al. Functional genomic analysis of the auxin response factor gene family members in arabidopsis thaliana: Unique and overlapping functions of arf7 and arf19[J]. Plant Cell, 2005, 17(2): 444-463. DOI:10.1105/tpc.104.028316 (  0) 0) |
| [9] |
SUN R, WANG K, GUO T, et al. Genome-wide identification of auxin response factor (arf) genes and its tissue-specific prominent expression in gossypium raimondii[J]. Functional & Integrative Genomics, 2015, 15(4): 1-13. DOI:10.1007/s10142-015-0437-0 (  0) 0) |
| [10] |
LI F, FAN G, WANG K, et al. Genome sequence of the cultivated cotton gossypium arboreum[J]. Nature Genetics, 2014, 46(6): 567-572. DOI:10.1038/ng.2987 (  0) 0) |
| [11] |
PUNTA M, COGGILL P C, Eberhardt R Y, et al. The pfam protein families database[J]. Nucleic Acids Research, 2008, 36(Database issue): 263-266. DOI:10.1002/047001153x.g306303 (  0) 0) |
| [12] |
MARCHLERBAUER A, LU S, ANDERSON J B, et al. Cdd: A conserved domain database for the functional annotation of proteins[J]. Nucleic Acids Research, 2011, 39(Database issue): D225-229. DOI:10.1093/database/bar058 (  0) 0) |
| [13] |
(  0) 0) |
| [14] |
KUMAR S, STECHER G, TAMURA K. Mega7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology & Evolution, 2016, 33(7): 1870. DOI:10.1093/molbev/msw054 (  0) 0) |
| [15] |
GUO A Y, ZHU Q H, CHEN X, et al. [gsds: A gene structure display server[J]. Hereditas, 2007, 29(8): 1023. DOI:10.1360/yc-007-1023 (  0) 0) |
| [16] |
BAILEY T L, BODEN M, BUSKE F A, et al. Meme suite: Tools for motif discovery and searching[J]. Nucleic Acids Research, 2009, 37(Web Server issue): W202-W208. DOI:10.1093/nar/gkp335 (  0) 0) |
| [17] |
EMANUELSSON O, BRUNAK S, VON H G, et al. Locating proteins in the cell using targetp, signalp and related tools[J]. Nature Protocol, 2007, 2(4): 953-971. DOI:10.1038/nprot.2007.131 (  0) 0) |
| [18] |
SANKOFF D. Gene and genome duplication[J]. Current Opinion in Genetics & Development, 2001, 11(6): 681. DOI:10.1002/9780470619902.ch15 (  0) 0) |
| [19] |
WU J, WANG F, CHENG L, et al. Identification, isolation and expression analysis of auxin response factor (arf) genes in solanum lycopersicum[J]. Plant Cell Reports, 2011, 30(11): 2059-2073. DOI:10.1242/dev.067918 (  0) 0) |
| [20] |
WANG D, PEI K, FU Y, et al. Genome-wide analysis of the auxin response factors (arf) gene family in rice (oryza sativa)[J]. Gene, 2007, 394(1-2): 13-24. DOI:10.1016/j.gene.2007.01.006 (  0) 0) |
| [21] |
李慧峰, 冉昆, 何平, 等. 苹果生长素响应因子(ARF)基因家族全基因组鉴定及表达分析[J]. 植物生理学报, 2015, 51(7): 1045-1054. LI Huifeng, RAN Kun, HE Ping, et al. Genome-wide identification and expression analysis of auxin response factor(arf)gene family in apple[J]. Plant Physiology Journal, 2015, 51(7): 1045-1054. DOI:10.13592/j.cnki.ppj.2015.0173 (  0) 0) |
| [22] |
REMINGTON D L, VISION T J, GUILFOYLE T J, et al. Contrasting modes of diversification in the aux/iaa and arf gene families[J]. Plant Physiology, 2004, 135(3): 1738-1752. DOI:10.1104/pp.104.039669 (  0) 0) |
| [23] |
NAGPAL P, ELLIS C M, WEBER H, et al. Auxin response factors arf6 and arf8 promote jasmonic acid production and flower maturation[J]. Development, 2005, 132(18): 4107-4118. DOI:10.1242/dev.01955 (  0) 0) |
| [24] |
WANG J W, WANG L J, MAO Y B, et al. Control of root cap formation by microrna-targeted auxin response factors in arabidopsis[J]. Plant Cell, 2005, 17(8): 2204-2216. DOI:10.1105/tpc.105.033076 (  0) 0) |
| [25] |
GUAN X, XU T, GAO S, et al. Temporal and spatial distribution of auxin response factor genes during tomato flower abscission[J]. Journal of Plant Growth Regulation, 2014, 33(2): 317-327. DOI:10.1007/s00344-013-9377-x (  0) 0) |
 2018, Vol. 16
2018, Vol. 16


