2. University of Michigan Medical School, Ann Arbor, MI, 48109 USA;
3. 贵州大学医学院,贵州省人民医院,贵阳 550002;
4. 国家人口与健康科学数据共享服务平台,北京 100700;
5. 中国中医科学院 中医药信息研究所,北京 100700;
6. 哈尔滨医科大学大庆校区,黑龙江, 大庆 163319;
7. 广州中医药大学,广州 510000;
8. 重庆邮电大学,重庆 400065;
9. University of PennsylvaniaPerelman School of Medicine, PA, 19104 USA;
10. 国家蛋白质科学中心北京凤凰中心,北京 102206
2. University of Michigan Medical School, Ann Arbor, MI 48109, USA;
3. Guizhou University Medical College, Guizhou Province People's Hospital, Guiyang 550002, China;
4. National Scientific Data Sharing Platform for Population and Health, Beijing 100700, China;
5. Institute of Information on Traditional Chinese Medicine, China Academy of Chinese Medical Sciences, Beijing 100700, China;
6. Daqing Branch of Harbin Medical University, Daqing 163319, China;
7. Guangzhou University of Chinese Medicine, Guangzhou, 510000, China;
8. Chongqing University of Posts and Telecommunications, Chongqing 400065, China;
9. University of Pennsylvania Perelman School of Medicine, PA 19104, USA;
10. The National Center for Protein Sciences, Beijing 102206, China
本体是事物本质概念化的详细说明,其核心作用在于用人和计算机都可以理解的术语(Terms)及关系(Relations)描述某一领域内的实体(Entities)以及实体之间的相互关系,从而提供一个对此领域事物本质的统一认识。这一系列概念犹如建造大厦的基石,在其支持下,知识搜索、积累和共享的效率将大大提高,真正意义上的知识重复利用和共享也成为可能。在过去十多年中,本体广泛应用于生物医学数据整合、共享、检索、分析和新知识的挖掘。通过本体中的标准化术语,不同数据集合的元数据可以被注释并进行术语的统一,进而消除异质性,实现数据的整合。本体作为生物医学数据标准化的一种重要手段,在大数据整合与复用的过程中发挥着日益重要的作用。在最近提出的精准医学的理念中,本体更是发挥着不可替代的作用。
1 本体的概念与研发进展作为一个古老的哲学分支,本体论(Ontology)是用来研究事物的本源和存在(“Onto”)。古希腊米利都哲学学派最早探索组成万物的最基本元素——“本原”(Arche)。米利都学派哲学家巴门尼德(Parmenides)对being(是,存在)的探讨成为本体论的先声。巴门尼德提出现实存在(Reality)是唯一不变的本原, 并且存在不能从经验之中感知获取。亚里士多德(Aristotle)认为哲学研究的主要对象是实体,而实体或本体的问题是关于本质、共相和个体事物。所以,亚里士多德认为本体学是第一哲学。在中国古代哲学中,本体论叫做“本根论”,它是探究天地万物产生、存在、发展变化根本原因和根本依据的学说。
随着现代科技特别是计算机学的快速发展,本体论已经开始被大量用于知识和数据的表达、数据分析和人工智能。在现代,作为一种知识组织方式,本体可以定义为,人和计算机可识别的,用来表达某个领域的实体(Entity)、概念、特性及其相互关系的词汇术语体系。本体的目标通常是用于表达相关领域的知识,提供对该领域知识的共同理解,并对这些知识和理解给出词汇和词汇间相互关系的逻辑性的定义。本体一般是采用本体语言来编制。作为当今常用的本体编写语言,OWL(Web Ontology Language)是World Wide Web Consortium (W3C)推荐的语义互联网中本体描述语言的标准[1]。
按照本体的概念涵盖范围可以分为上层本体(Upper level ontology)、参考本体(Reference ontology)和应用本体(Application ontology)。上层本体,如基本形式化本体(Basic Formal Ontology,BFO)[2], 提供一种高于特定领域的上层框架结构以指导特定领域本体的构建。参考本体,如人类表型本体(Human Phenotype Ontology, HPO)[3],表达可被重复使用的某一学科上的基本知识[4]。应用本体针对的是某一特定使用场景或应用[5]。
目前国外本体研究与应用发展很快。比如,The Open Biological/Biomedical Ontologies (OBO),即开放性生物信息本体体系[6],OBO Foundry (可直译为开放性生物与生物医学本体铸造工厂)是一个由众多生物与生物医学本体开发者发起的基于自愿原则的国际联盟(http://obofoundry.org/)[6]。OBO Foundry的使命是开发一族具有可互操作性、逻辑性、科学性和准确性的本体。为了保障完成OBO Foundry的使命, OBO Foundry的参与者自发地共同开发一系列原则,如开放(Open)、合作(Commitment To Collaboration)和通用格式(Common Format),等等[6]。OBO本体库目前已有160多个本体,如BFO[2]、HPO[3]、Gene Ontology (GO)基因本体[7]、Relations Ontology (RO)关系本体[8]、Ontology for Biomedical Investigations(OBI)生物医学调查本体等[9]。
按本体的作用,我们可以把本体分为三类:1)作为增强版控制术语集的本体;2)作为知识库的本体; 3)元数据本体(见图 1)。在当今生物医学大数据与精准医学研究时代,这些类型的本体对数据、元数据、与知识的标准化表达、注释、整合、传递与分析有着重要的应用(见图 1)。以下我们就对这些方面进行阐述与举例说明,并对未来发展进行展望。
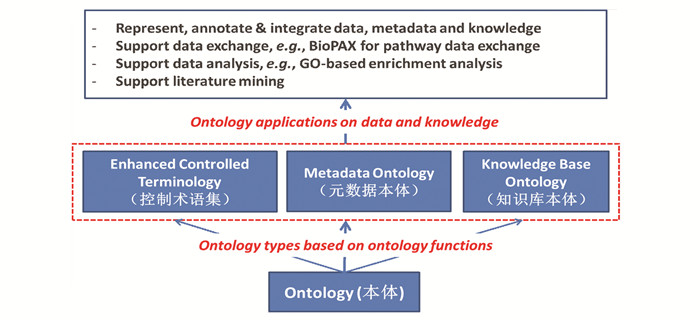
|
图 1 本体按作用的分类及其应用 Figure 1 Function-based ontology classification and applications |
控制术语集(Controlled terminology, 又称为受控词表,控制词表)是一种对知识术语(Term)加以组织整理,权威认定,赋予特定编码,以利于特定语境下的标记与检索。比较而言,自然语言词典(Dictionary)或叙词表(Thesaurus)没有权威认定与赋予编码的限制。控制术语集在医学界广泛应用。比如,MedDRA (Medical Dictionary for Regulatory Activities,国际医学用语词典)术语集[10]在临床药物安全检验中发挥了重要的术语标准化的作用。SNOMED-CT(Systematized Nomenclature of Medicine-Clinical Terms)医学系统命名法—临床术语标准涵盖大多数方面的临床信息,如疾病、所见、操作、微生物、药物等[11]。MeSH(Medical Subject Headings,医学主题词表[12]是一部庞大的控制术语集,广泛应用于医学信息检索,标引期刊文献和书籍。
本体继承和发展了传统的控制术语系统。传统控制术语集大致等于本体中用“is a”(是一个)定义的从属关系层次结构(Hierarchical structure), 但他们的“is a”从属关系经常定义不够准确。除了“is a”关系,本体也提供了各种其他的关系。如在OAE(Ontology of Adverse Events, 不良反应本体)[13]里,我们可以用“preceded by”(在……前)关系来表述一个医学干预(Medical intervention)与不良反应(Adverse event)之间时间上的先后关系(见图 2A),用“occurs in” (在……内发生)关系来表述一个不良反应与其所发生身体部位的关系(见图 2B)。实体间关系的逻辑性表述在本体内被称“公理”(Axiom)(见图 2B)。公理可以提供用于推导的必要(Necessary)和/或充分(Sufficient)条件(Condition)。因此,本体表述实体之间的逻辑关系更为严格,用处更广泛。
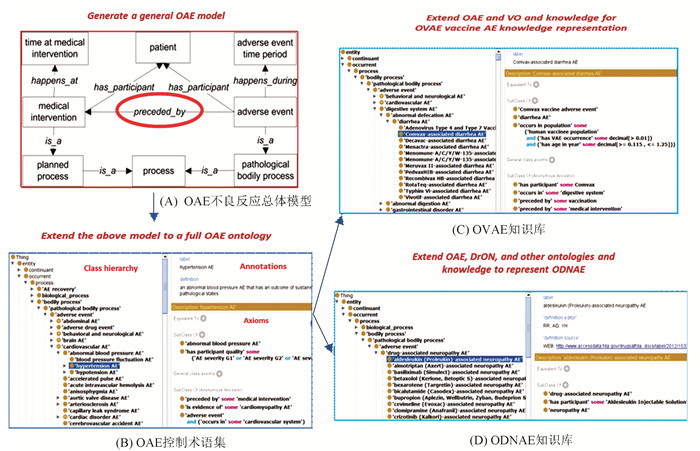
|
图 2 本体研发与分类的实例 Figure 2 Illustration of ontology development and classification |
知识可以用不同的方式表述和储存。传统的纸质文档应用广泛,但纸质文档表达的知识是非结构化的,我们无法用计算机查询。结构化的关系数据库(Relational database)可以储存大量的数据。但是关系数据库储存的数据和知识只能被特定服务器理解和查询,不能被其他计算机理解和共享。本体可以作为知识库去表述一个领域已知的知识,并解决上述的问题。比如,OVAE(Ontology of Vaccine Adverse Events, 疫苗不良反应本体)被开发成一种知识库来表达在美国上市疫苗的不良反应[14]。OVAE重复使用了很多OAE和VO(Vaccine Ontology, 疫苗本体)[15, 16]里的术语并用它们来定义新的OVAE术语[14](见图 2C)。通过OVAE我们可以很快地查询与疫苗相关的不良反应的发生率与严重程度。本体的查询可以用SPARQL(SPARQL Protocol and RDF Query Language)语言,比如,简短的SPARQL本体查询语句就可以查到诸如哪个不良反应在所有疫苗中出现得最频繁,哪个疫苗有最多的不良反应[14]。类似的方法也被用来构建ODNAE(Ontology of Drug Neuropathy Adverse Events, 药物神经不良反应本体)本体知识库[17]。ODNAE用到了OAE和DrON(Drug Ontology, 药物本体)[18]和已知的药物导致的神经不良反应的知识(见图 2D)。
2.3 元数据本体对元数据的标准化本体可以被用来标准化各种数据。例如,人类表型本体(HPO)[3]里的术语可以用以直接描述某个病人的各种病变的表型,UBERON[19]本体可以用来表述疾病发生的解剖部位,细胞本体(Cell Ontology, CL)[20]可以表述发生形态或功能发生异常变化的细胞,疾病本体(Disease Ontology, DOID)[21]用以描述经诊断的疾病,等等。
元数据(metadata)是“关于数据的数据”(Data about data),主要描述数据内涵或属性的信息,用来支持不同的功能。一个共同认可的元数据系统是很有必要的。如“都柏林核心(Dublin Core)元数据”,其核心包含十五个元数据元素(如题名、日期、创建者、主题等),用以描述资源对象的语义信息,已成为许多国家和机构的标准。
元数据本体可以支持对元数据的更进一步标准化。传统元数据之间的关系缺乏有效的表达,计算机不能理解。而本体可以有效地标准化元数据,给出元数据之间关系的逻辑定义,并增强不同元数据系统之间的互操作性(Interoperability)。如美国NIH资助与组织构建了一套人病原基因组序列的标准化元数据体系,并对这些元数据进行了基于OBI(Ontology for Biomedical Investigations, 生物医学调查本体)的本体化[9]。这种元数据的本体化提供了一个基于本体的丰富的语义框架(Semantic framework),实现了对现有本体的再利用和数据表述的最优化,并可以扩展以支持更多项目的数据标准化与整合分析[9]。
3 基于本体的生物医学大数据(Big data)的整合与分析随着人类基因组计划的完成和高通量测序等新技术的快速发展,生命科学研究产生了大量的生物学数据,包括基因组、蛋白质组等生物组学数据和临床医疗大数据。这些生物医学大数据具有数据量大(Volume)、数据多样化(Variety)、高速(Velocity)等特点[22]。生物医学大数据针对的是不同领域不同来源的数据。随着生物医学基础研究、转化医学研究和医院临床数据的海量收集与处理,各方面的大数据缺乏融合贯通,已成为制约生物医学进一步研究发展的瓶颈。如何实现不同数据源的整合,方便研究者检索利用这些大数据,挖掘数据里的知识,已经成为当前研究热点之一。
本体可以用于生物医学大数据的整合与分析。本体理论为数据与知识的表达、管理、整合、索引和高水平的分析与展示提供了强大的表达和推理能力,可以用于解决数据异构性问题,并促进数据的大整合与研究。一个被广泛应用于数据整合的本体是OBI(Ontology for Biomedical Investigations, 生物医学调查本体)[23]。尽管不同生物医学领域的调查研究往往有很多共性,但是生物医学各领域之间缺乏一个统一的标准,不同的研究数据难以共享。有鉴于此,超过20个社区的科学家共同研发出了OBI,来提供一个生命科学与临床调查研究的统一的标准,弥补了生物医学标准不统一,信息不能共享的空白,更好的实现了数据信息资源的优化利用。一个最典型的本体支持应用的例子是GO基因本体[7]的应用。作为一种适用于各种物种的本体,GO描述基因产物的分子功能(Molecular Function)、参与的生物过程(Biological Process)和在细胞组件(Cellular Component)的存在部位。GO被广泛应用于基因组学数据分析,判定与特定表型(如疾病与衰老)有关的基因产物的表达变化(增强或减弱),生物功能与生物学途径。此外,GO也被用于文献检索等。几乎所有与基因和蛋白有关的大数据分析都要用到GO,自2000年发表以来,GO已经被数万篇文献引用。
4 本体是精准医学的基础精准医学(Precision Medicine),又称精准医疗,是以个体化医疗为宗旨的新型医学概念与医疗模式[24]。精准医学的本质是通过生物组学和其他医学前沿技术,对于大样本人群与特定疾病类型进行从基因型到表型各个方面的数据进行整合分析与标准化处理,建立不同数据之间的关联性和差异性,对病理发生发展的过程建立定量模型,提出假设与验证,从而寻找到特定人群甚至个体疾病的精确原因和治疗靶点,最终实现对于疾病和特定患者进行个性化精准预防和治疗的目的。精准医学的基础是大量生物科学新技术(如基因组测序)产生的生物医学大数据。对生物医疗大数据快速有效的生物信息整合与分析是精准医学的关键(见图 3)。
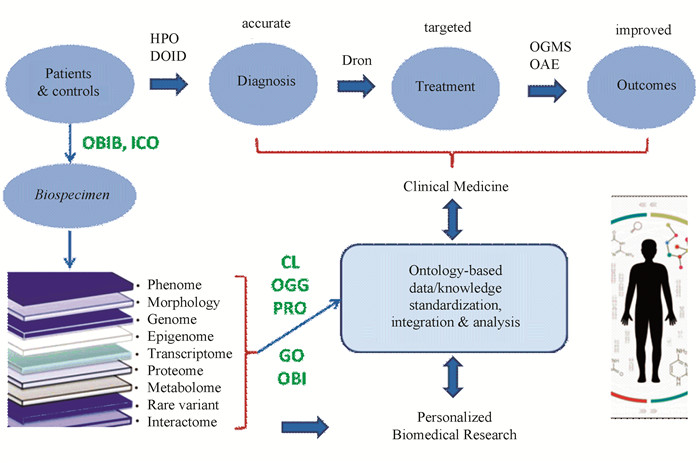
|
图 3 本体在精准医学中的应用 Figure 3 Ontology applications in precision medicine |
不同个体的生物样本经采集后可以进行各种临床与实验室测试,数据经标准化处理、整合与分析,可以帮助对个体病人的精确诊断、针对性治疗、和改善预后。本体(如GO, OBI)在各个阶段都起到重要的作用。本体对精准医学的发展起到基础支撑的作用(见图 3)。
首先,作为生物医学数据标准化的一种重要手段,本体可以对个体从生物样本的收集使用,到基因型(如基因突变)到表型(如疾病症状)的各个方面进行精确的实体定义和层次分类。比如,我们可以用OBIB(Ontology for BioBanking,生物银行本体)[25]和ICO(Informed Consent Ontology, 知情同意本体)[26]来表达经过知情同意后对研究对象生物样本的采集、储存、与使用。多种本体(如CL, GO, 和OBI)可以用于临床与实验室测试对象、过程、数据与结果的标准化。基因与蛋白可以分别用OGG(Ontology of Genes and Genomes,基因与基因组本体)[27]和PRO(Protein Ontology,蛋白本体)[28]来表示。同时,疾病症状和诊断可以用HPO和DOID标注,治疗用药物可以用DrON,药物效果可以用OGMS(Ontology for General Medical Science, 一般医学本体)[29]和OAE等本体表示(见图 3)。现在常用的临床控制术语集,如SNOMED和LOINCS[30]等,可以和本体映射并一起使用。
本体可以精确定义不同生物过程的模式和个体差异对生物过程变化的作用。比如,OAE可以逻辑化地定义医疗干预与不良反应之间的关系及个体差异对不良反应后果的影响[13]。大部分药物(或疫苗)使用者对药物(或疫苗)耐受良好,但是少数个体会出现不良反应,甚至严重的不良反应。这些严重不良反应的发生与个体差异(如基因型、年龄、性别、身体状态等)有一定关系。OAE及其相关本体可以对这些个体差异及其与不良反应的关系进行精确分类与描述,有助于对个体不良反应的预测、预防与及时治疗。
第二,利用上述本体化的精确定义与分类,我们可以进一步构建精准医学知识库本体来准确表述基因-RNA-蛋白质-代谢产物等相关内环境的信息和关系,以及个体表型和行为差异对疾病的影响。
第三,我们也可以利用本体标准化数据与元数据,并据此更好地分析复杂的精准医学大数据。基于本体的标准化可以消除异质性,实现数据的整合、注释与分析。在精准医学大数据应用中,80%以上的时间与精力是花在数据整合上。集约化的本体数据标准化可以节省大量时间,对精准医学目标的最终实现起着不可替代的重要作用。
5 前景与展望本体对现代生物医学大数据与精准医疗的研究应用很重要,然而本体这个外来词对很多生物医学研究者与临床医疗工作者还很陌生。了解生物医学本体概念和研究方法有助于加速计算机对于现实世界的理解,支撑大数据、人工智能技术在医疗健康场景落地。因此,本文介绍了本体的基本概念与主要作用,并进而阐述本体在生物医学大数据和精准医疗发展中的关键应用。
生物医学本体的快速发展得益于本体工具的开发与使用。例如Protégé-OWL editor[31]是公认使用最为广泛的的本体编辑工具。BioPortal[32]和Ontobee[33]被用于本体的储存与查询, Ontofox[34]可以方便地提取已知本体的术语及关系并在新建本体中再利用。各种GO基因本体也在基因表达富集分析上广泛应用[35]。最近一套XOD (eXtensible Ontology Development) “可扩展的本体开发”原则被提出来并用来指导可互操作性的本体开发[36]。在本体中,每个术语都有其独一无二的Internationalized Resource Identifier (IRI,国际化资源标识符),因此本体IRI可以被用来解决不同语言上的交流阻碍,例如我们可以用本体IRI来注释中文数据。
目前健康医疗大数据与精准医疗的兴起正推动全世界本体研究的快速推进。数据的规范和标准化是大数据与精准医疗的基础。而作为数据标准化的主要手段,当代本体学刚刚起步,各个方面的本体研究还有着大量的研究与应用机会。例如随着中医研究与应用的日益兴起,中医领域亟需一系列的本体支持。最近我们开发了一个抗风湿中药本体(Ontology of Chinese Medicine for Rheumatism, OCMR),该本体对26种抗风湿中药的生物来源、化学成分、药理作用及其副作用进行了中药知识与数据的整合与挖掘。我们基于OCMR本体的分析更便捷与直观地发现了中药化学成分与其抗风湿功能和副作用之间的关系,并对以后抗风湿中药的机制与安全性研究提供新的假设与方向[37]。这个研究显示了本体在中医知识与数据的标准化与整合分析上的应用前景。
相较而言,国内本体研究相对滞后。国内在生物医学研究与临床数据结构化与标准化方面与世界先进水平还存在着巨大差距,导致临床科研大数据整合的能力不足,影响精准医疗数据分析的准确度。各个单位的本体构建研究水平参差不齐,本体构建中存在本体工作的重合,需要加强沟通避免重复。直接通过翻译引进现有本体也存在一些问题,英文经常无法完全表述中文领域的学科知识,中文本体、中文化本体和英文本体的构建选择比较盲目。同时中国的本体建设还缺乏一个顶层设计来进行通用性本体的构建。
有鉴于此,最近中国生物医学本体联合工作组(China Biomedical Ontology Consortium,OntoChina)由国家人口与健康科学数据共享服务平台组织成立,宗旨是通过生物医学领域的广泛协作,引入先进本体建设理念和模式,建设为国内生物医学信息系统和相关领域科学研究服务的本体资源,促进生物医学本体在信息化建设和科学研究中的使用。
致谢 我们感谢北京大学罗静初教授和中国医学科学院王恒教授的讨论、建议与修改。何勇群2017年6月底到10月初在中国医学科学院基础医学研究所的访学与研究由中国医学科学院基础医学研究所,国家人口与健康科学数据共享服务平台基础医学科学数据中心,以及University of Michigan Medical School Global Reach Fund课题资助。
| [1] |
BAO J, KENDALL E F, MCGUINNESS D L, et al. OWL 2 web ontology language quick reference guide (Second edition)[EB/OL]. http://www.w3.org/TR/owl2-quick-reference/. 2012-12-11.
(  0) 0) |
| [2] |
ARP R, SMITH B. Spear AD: Building ontologies using basic formal ontology[M]. Cambridge, MA, USA: MIT Press, 2015.
(  0) 0) |
| [3] |
GROZA T, KOHLER S, MOLDENHAUER D, et al. The human phenotype ontology: semantic unification of common and rare disease[J]. American journal of human genetics, 2015, 97(1): 111-124. DOI:10.1016/j.ajhg.2015.05.020 (  0) 0) |
| [4] |
BRINKLEY J F, SUCIU D, DETWILER L T, et al. Rosse C: A framework for using reference ontologies as a foundation for the semantic web[J]. AMIA Annual Symposium Proceedings Archive, 2006, 96-100. (  0) 0) |
| [5] |
MALONE J, PARKINSON H. Reference and application ontologies[J]. Motricidad European Journal of Human Movement, 2010, 2(1): 113-127. (  0) 0) |
| [6] |
SMITH B, ASHBURNER M, ROSSE C, et al. The OBO Foundry: coordinated evolution of ontologies to support biomedical data integration[J]. Nature Biotechnology, 2007, 25(11): 1251-1255. DOI:10.1038/nbt1346 (  0) 0) |
| [7] |
ASHBURNER M, BALL C A, BLAKE J A, et al. The Gene Ontology Consortium[J]. Nature Genetics, 2000, 25(1): 25-29. DOI:10.1038/75556 (  0) 0) |
| [8] |
SMITH B, CEUSTERS W, KLAGGES B, et al. Relations in biomedical ontologies[J]. Genome Biology, 2005, 6(5): R46. DOI:10.1186/gb-2005-6-5-r46 (  0) 0) |
| [9] |
DUGAN V G, EMRICH S J, GIRALDO-CALDERON G I, et al. Standardized metadata for human pathogen/vector genomic sequences[J]. PloS One, 2014, 9(6): e99979. DOI:10.1371/journal.pone.0099979 (  0) 0) |
| [10] |
BROWN E G. Methods and pitfalls in searching drug safety databases utilising The medical dictionary for regulatory activities (MedDRA)[J]. Drug Safety, 2003, 26(3): 145-158. DOI:10.2165/00002018-200326030-00002 (  0) 0) |
| [11] |
BROWN S H, ELKIN P L, BAUER B A, et al. Rosenbloom ST: SNOMED CT: utility for a general medical evaluation template[J]. AMIA Annual Symposium Proceedings Archive, 2006, 101-105. (  0) 0) |
| [12] |
LIPSCOMB C E. Medical subject headings (MeSH)[J]. Bulletin of the Medical Library Association, 2000, 88(3): 265. (  0) 0) |
| [13] |
HE Y, SARNTIVIJAI S, LIN Y, et al. OAE: The ontology of Adverse events[J]. Journal of Biomedical Semantics, 2014, 5: 29. DOI:10.1186/2041-1480-5-29 (  0) 0) |
| [14] |
MARCOS E, ZHAO B, HE Y. The ontology of vaccine adverse events (OVAE) and its usage in representing and analyzing adverse events associated with US-licensed human vaccines[J]. Journal of Biomedical Semantics, 2013, 4(1): 40. DOI:10.1186/2041-1480-4-40 (  0) 0) |
| [15] |
HE Y, COWELL L, DIEHL A D, et al. VO: Vaccine Ontology. The 1st international conference on biomedical Ontology (ICBO 2009)[C]. UK: Nature Precedings, 2009. DOI: 10.1038/npre.2009.3552.1.
(  0) 0) |
| [16] |
OZGUR A, XIANG Z, RADEV DR, et al. Mining of vaccine-associated IFN-gamma gene interaction networks using the vaccine ontology[J]. Journal of Biomedical Semantics, 2011, 2(Suppl 2): S8. DOI:10.1186/2041-1480-2-S2-S8 (  0) 0) |
| [17] |
GUO A, RACZ R, HUR J, et al. Ontology-based collection, representation and analysis of drug-associated neuropathy adverse events[J]. Journal of Biomedical Semantics, 2016, 7(1): 29. DOI:10.1186/s13326-016-0069-x (  0) 0) |
| [18] |
HANNA J, JOSEPH E, BROCHHAUSEN M, et al. Building a drug ontology based on RxNorm and other sources[J]. Journal of Biomedical Semantics, 2013, 4(1): 44. DOI:10.1186/2041-1480-4-44 (  0) 0) |
| [19] |
MUNGALL C J, TORNIAI C, GKOUTOS G V, et al. Uberon, an integrative multi-species anatomy ontology[J]. Genome Biology, 2012, 13(1): R5. DOI:10.1186/gb-2012-13-1-r5 (  0) 0) |
| [20] |
DIEHL A D, MEEHAN T F, BRADFORD Y M, et al. The cell ontology 2016: enhanced content, modularization, and ontology interoperability[J]. Journal of Biomedical Semantics, 2016, 7(1): 44. DOI:10.1186/s13326-016-0088-7 (  0) 0) |
| [21] |
SCHRIML L M, ARZE C, NADENDLA S, et al. Disease ontology: a backbone for disease semantic integration[J]. Nucleic Acids Research, 2012, 40(Database issue): D940-946. DOI:10.1093/nar/gkr972 (  0) 0) |
| [22] |
KESSEL K A, COMBS S E. Review of developments in electronic, clinical data collection, and documentation systems over the last decade-are we ready for big data in routine health care?[J]. Frontiers in Oncology, 2016, 6(1): 75. DOI:10.3389/fonc.2016.00075 (  0) 0) |
| [23] |
BANDROWSKI A, BRINKMAN R, BROCHHAUSEN M, et al. The ontology for biomedical investigations[J]. PloS One, 2016, 11(4): e0154556. DOI:10.1371/journal.pone.0154556 (  0) 0) |
| [24] |
ASHLEY E A. The precision medicine initiative: a new national effort[J]. JAMA, 2015, 313(21): 2119-2120. DOI:10.1001/jama.2015.3595 (  0) 0) |
| [25] |
BROCHHAUSEN M, ZHENG J, BIRTWELL D, et al. OBIB-a novel ontology for biobanking[J]. Journal of Biomedical Semantics, 2016, 7(1): 23. DOI:10.1186/s13326-016-0068-y (  0) 0) |
| [26] |
LIN Y, HARRIS M R, MANION F J, et al. Development of a BFO-based informed consent ontology (ICO). Proceedings of the 5th international conference on biomedical ontologies (ICBO)[C]. Web: CEUR Workshop Proceedings, 2014. https://jbiomedsem.biomedcentral.com/articles/10.1186/s13326-016-0062-4
(  0) 0) |
| [27] |
HE Y, LIU Y, ZHAO B. OGG: A biological ontology for representing genes and genomes in specific organisms. Proceedings of the 5th international conference on biomedical ontologies (ICBO)[C]. Web: CEUR Workshop Proceedings, 2014. https://jbiomedsem.biomedcentral.com/articles/10.1186/2041-1480-6-2
(  0) 0) |
| [28] |
NATALE D A, ARIGHI C N, BLAKE J A, et al. Protein Ontology: a controlled structured network of protein entities[J]. Nucleic Acids Research, 2014, 42(Database issue): D415-421. DOI:10.1093/nar/gkt1173 (  0) 0) |
| [29] |
The ontology for general medical science (OGMS)[EB/OL]. https://github.com/OGMS/ogms. 2017-10-14. https://bmcsystbiol.biomedcentral.com/articles/10.1186/s12918-017-0510-5
(  0) 0) |
| [30] |
VREEMAN D J, MCDONALD C J, HUFF S M. LOINC(R) -A universal catalog of individual clinical observations and uniform representation of enumerated collections[J]. International Journal of Functional Informatics and Personalised Medicine, 2010, 3(4): 273-291. DOI:10.1504/IJFIPM.2010.040211 (  0) 0) |
| [31] |
RUBIN D L, NOY N F, MUSEN M A. Protege: a tool for managing and using terminology in radiology applications[J]. Journal of Digital Imaging, 2007, 20(Suppl 1): 34-46. DOI:10.1007/s10278-007-9065-0 (  0) 0) |
| [32] |
WHETZEL P L, NOY N F, SHAH N H, et al. BioPortal: enhanced functionality via new Web services from the National Center for Biomedical Ontology to access and use ontologies in software applications[J]. Nucleic Acids Research, 2011, 39(Web Server issue): W541-545. DOI:10.1093/nar/gkr469 (  0) 0) |
| [33] |
ONG E, XIANG Z, ZHAO B, et al. Ontobee: A linked ontology data server to support ontology term dereferencing, linkage, query and integration[J]. Nucleic Acids Research, 2017, 45(D1): D347-D352. DOI:10.1093/nar/gkw918 (  0) 0) |
| [34] |
XIANG Z, COURTOT M, BRINKMAN R R, et al. OntoFox: web-based support for ontology reuse[J]. BMC Research Notes, 2010, 3(1): 175:1-12. DOI:10.1186/1756-0500-3-175 (  0) 0) |
| [35] |
HUANG D A W, SHERMAN B T, LEMPICKI R A. Bioinformatics enrichment tools: paths toward the comprehensive functional analysis of large gene lists[J]. Nucleic Acids Research, 2009, 37(1): 1-13. DOI:10.1093/nar/gkn923 (  0) 0) |
| [36] |
HE Y, XIANG Z, ZHENG J, et al. The eXtensible ontology development (XOD) principles and tool implementation to support ontology interoperability[J]. Journal of Biomedical Semantics, 2018, 9(1): 3. DOI:10.1186/s13326-017-0169-2 (  0) 0) |
| [37] |
LIU Q, WANG J, ZHU Y, et al. Ontology-based systematic representation and analysis of traditional Chinese drugs against rheumatism[J]. BMC systems biology, 2017, 11(Suppl 7): 130. DOI:10.1186/s12918-017-0510-5 (  0) 0) |
 2018, Vol. 16
2018, Vol. 16

