2. 长治医学院附属和济医院 医务科, 山西 长治 046000
2. Medical Department, Heji Hospital Affiliated to Changzhi Medical College, Changzhi 046000, Shanxi, China
肝癌(Hepatocellular carcinoma,HCC)是世界第3大致死癌症,也是中国乃至亚洲最广泛的一种癌症。目前对于肝癌细胞癌变机制的研究尚无系统工作。临床上通常采用肝移植、手术切除、肝动脉化疗栓塞等方法治疗肝癌,但术后复发率和转移率偏高。有研究人员[1]发现,PPM1D的mRNA表达水平与肝癌患者的肿瘤体积、肿瘤分级和总体生存时间密切相关。
Mg2+依赖性蛋白磷酸酶1δ(PPM1D),亦被称为野生型p53诱导的蛋白磷酸酶1(Wip1,wild-type p53-induced phosphatase 1)。在乳腺肿瘤、卵巢透明细胞癌、胰腺癌、神经母细胞瘤和髓母细胞瘤等固体肿瘤中,发现PPM1D常过量表达,且与癌症患者生存状况不佳呈正相关[2]。研究人员[1, 3]发现,对于中分化或低分化肝癌患者,肿瘤抑制基因p53通常发生突变或表达受到抑制。PPM1D能够抑制p53信号途径和p38 MAPK信号通路,推测其为致癌基因[4-5]。但PPM1D的致癌机制和预后价值仍未完全阐明,针对该分子的基因治疗手段也尚未见报道。
越来越多的证据表明,在多种固体肿瘤中,PPM1D是一个潜在的肿瘤相关分子,也是一个有价值的预后标志物。PPM1D在接近59 %的肝癌患者体内过量表达,且高表达PPM1D的患者特点是:AFP水平偏高、肿瘤体积偏大、TNM分期偏高、肿瘤复发率偏高、存在肝癌家族史,总体存活时间偏短[1]。文献[2]使用siRNA下调PPM1D表达,发现能够抑制肿瘤异种移植小鼠模型体内肝癌细胞的增殖和迁移能力,降低其致瘤性。因此,PPM1D作为肝癌的一个潜在预后标志物和治疗靶点,它的功能及其参与的信号通路值得深入研究。本文使用生物信息学分析方法,研究PPM1D蛋白的理化性质、结构与功能,可为进一步研究PPM1D的作用机制及其作为肝癌治疗靶点提供依据。
1 材料与方法 1.1 材料以“PPM1D+物种名”为关键词,在美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)的GenBank数据库(https://www.ncbi.nlm.nih.gov/),搜索获得人PPM1D基因转录本序列和PPM1D蛋白的氨基酸序列信息。
1.2 方法使用Clustal2.1(http://www.clustal.org/download/current/)进行PPM1D同源蛋白间的多重序列比对。使用MEGA6软件,Neighbor-joining方法,Boot-strap分析重复数设置为1 000,构建系统进化树,并计算进化距离[6]。使用NCBI的UniGene数据库中EST结果分析PPM1D的组织表达特异性,PSORTII(https://psort.hgc.jp/)进行亚细胞定位预测。使用ExPASy数据库中的ProtParam在线工具(http://web.expasy.org/protparam/)对PPM1D进行理化性质分析。使用SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)和TMHMM 2.0工具(http://www.cbs.dtu.dk/services/TMHMM/)预测PPM1D的信号肽和跨膜区域。使用NetPhosK3.1(http://www.cbs.dtu.dk/services/NetPhos/)和PhosphoSitePlus工具(http://www.phosphosite.org/homeAction.action)分析PPM1D的翻译后修饰情况。使用SOPMA工具(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)分析PPM1D的二级结构,NCBI的Conserved Domain数据库分析结构域,SWISS-MODEL建模服务器(https://swissmodel.expasy.org/)预测三维结构。使用STRING数据库(http://string-db.org/),构建与PPM1D相互作用的蛋白网络。
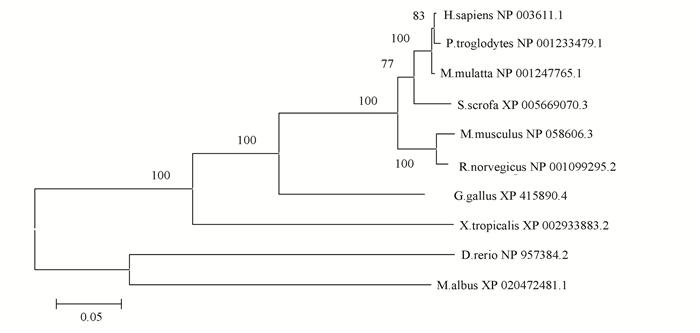
2 结果与分析 2.1 PPM1D蛋白多序列比对和进化关系分析在NCBI数据库中搜索到人PPM1D蛋白在哺乳动物、两栖类和鱼类中的同源序列。人PPM1D蛋白的氨基酸序列与黑猩猩、猕猴、野猪、小鼠、大鼠、鸡、非洲爪蟾、黄鳝和斑马鱼的相似性分别为99.34 %、98.51 %、94.05 %、89.13 %、78.45 %、73.38 %、61.02 %、54.75 %和50.94 %。图 1表明,人PPM1D蛋白与其他几种哺乳动物序列相似性较大,组成一支,而与两栖类和鱼类几种动物的序列相似性较小。人与黑猩猩的同源性最高,进化距离为0.004,与野猪、小鼠和鸡的同源蛋白进化距离分别为0.027、0.044、0.111,进化距离最远的是斑马鱼和黄鳝,分别为0.323、0.305。由此推测,PPM1D蛋白的同源性与物种间亲缘关系呈正相关,亲缘关系越近,蛋白的同源性越高,提示该蛋白在物种进化过程中扮演一定角色。
|
图 1 人PPM1D蛋白与其同源序列比对的系统进化树 Figure 1 The sequence alignment of human PPM1D protein and its homologous sequences |
根据NCBI的UniGene数据库中EST结果显示,PPM1D在以下正常组织均有表达,拷贝数分别是:脑34、睾丸26、肺10、肾8、肠8、子宫8、胰腺5、肝1。由此推测,PPM1D的组织表达特异性不强,在多数组织中均有表达。PSORTII预测,PPM1D定位于细胞核的可能性最大(60.9 %),其次可能定位于细胞质(13.0 %)、质膜(8.7 %)、细胞骨架(8.7 %)、分泌系统囊泡(4.3 %)和高尔基体(4.3 %)。由此推测,PPM1D主要在细胞核中发挥生理功能,而在其他亚细胞结构中也可能动态存在。
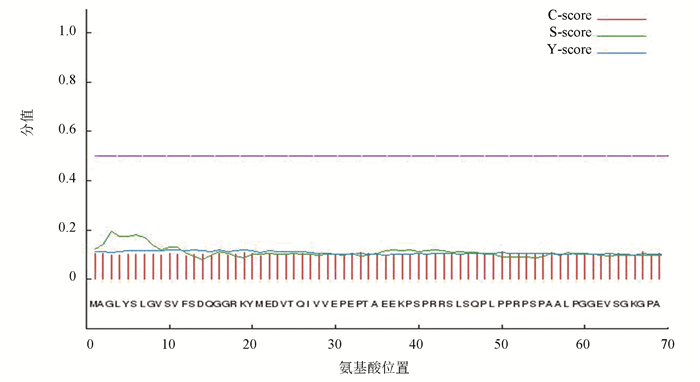
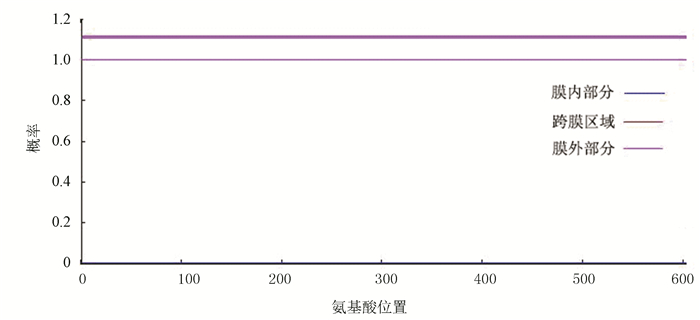
SignalP 4.0预测PPM1D蛋白不含切割位点,无信号肽序列如图 2所示,说明该蛋白不是分泌蛋白。TMHMM 2.0预测PPM1D无跨膜结构域如图 3所示。图 3中,红色细线、蓝色细线和粉色细线分别表示跨膜区域、膜内部分和膜外部分。图中PPM1D蛋白位于膜外的概率几乎为100 %,跨膜区域和位于膜内的概率几乎为0。蓝色细线与纵坐标为0的基线重叠。粉色粗线用来标识多肽链中跨膜区域所在位置,因PPM1D蛋白没有跨膜区域,所以在粗线上没有相应标记。
|
图 2 PPM1D蛋白的信号肽分析结果 Figure 2 The analysis result of PPM1D signal peptide |
|
图 3 PPM1D蛋白跨膜结构分析 Figure 3 Trans-membrane domain analysis of PPM1D |
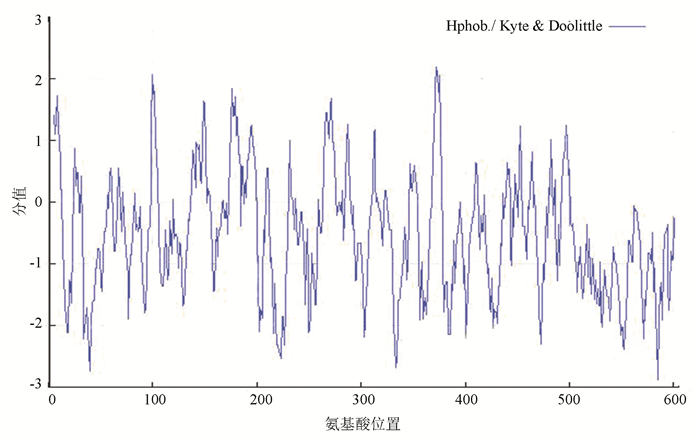
人PPM1D基因(NM003620.3)位于17号染色体上(17q23.2),编码产物NP_003611.1为该基因的蛋白质共识编码序列。PPM1D蛋白含有605个氨基酸,分子式为C2905H4669N857O880S31,分子量为66 675.1 Da,理论等电点预测为9.14,属于碱性蛋白质。在哺乳动物网织红细胞内半衰期为30 h,不稳定系数为56.41,属于不稳定蛋白质。脂肪系数为72.99,总的平均亲水性为-0.517。ExPASy数据库中的ProtScale在线工具预测,PPM1D亲水性最强的位点是第585位的精氨酸,分值为-2.878;疏水性最强的位点是第372位的缬氨酸,分值为2.189。由图 4可知,PPM1D蛋白的亲水区域多于疏水区域,属于亲水蛋白质。
|
图 4 ProtScale分析PPM1D蛋白的亲疏水性 Figure 4 Hydrophobicity profile of the PPM1D protein analyzed by ProtScale |
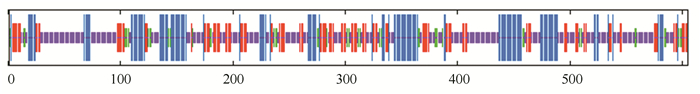
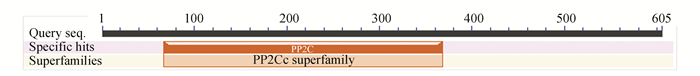
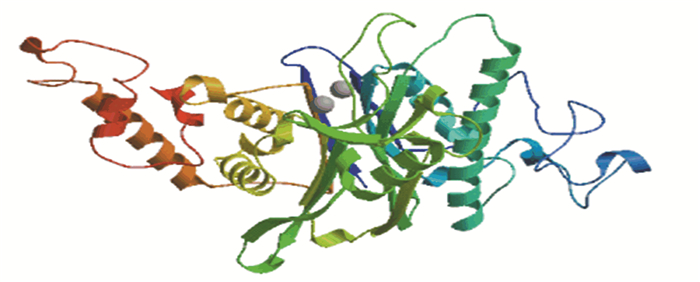
由图 5知,在PPM1D蛋白质的二级结构中,随机卷曲(图中橘色)占47.6 %,α-螺旋(图中蓝色)占23.8 %,延伸链(图中红色)占21.16 %,β-转角(图中绿色)占7.44 %。NCBI的Conserved Domain数据库预测PPM1D蛋白属于蛋白磷酸酶2C(Protein phosphatase 2C, PP2C)超家族的一员(如图 6所示),PP2C是一类丝氨酸/苏氨酸蛋白磷酸酶。PPM1D含有一个PP2Cc(Complementary DNA encoding the isoform of protein phosphatase 2C)结构域,PP2Cc是约42 kDa的单体酶,含有约390个氨基酸,与PP2C1异构体相似性达76%, 其底物特异性不强,活性依赖于二价阳离子(主要是锰和镁),确切的生理作用尚不清楚[7]。使用SWISS-MODEL建模服务器预测PPM1D蛋白三维结构(如图 7所示),序列与模板相似度为30.09 %,三维结构预测表明其符合PP2C结构域模型特点。
|
图 5 SOPMA预测PPM1D蛋白二级结构 Figure 5 Predicted secondary structure of PPM1D protein by SOPMA |
|
图 6 PPM1D蛋白保守结构域 Figure 6 Conserved domain of PPM1D protein |
|
图 7 PPM1D三维结构预测 Figure 7 Three dimensional structure prediction of PPM1D |
使用NetPhosK 3.1和PhosphoSitePlus分析PPM1D翻译后修饰情况,发现丝氨酸、苏氨酸和酪氨酸位点能够被磷酸化。根据PPM1D能够使其互作蛋白发生去磷酸化,探索其磷酸化程度及动态变化能够加深对该蛋白功能的理解。此外,赖氨酸会发生乙酰化、甲基化和泛素化修饰,针对该蛋白如何发生泛素化及降解,可以进一步探索。
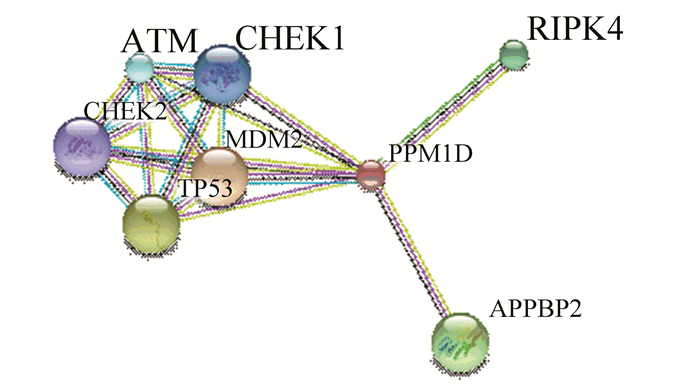
2.5 PPM1D蛋白质相互作用分析利用STRING数据库搜索与PPM1D相互作用的蛋白质信息,设置为高置信度0.7,数量限制在10个以内,构建PPM1D蛋白相互作用网络如图 8所示。与PPM1D相互作用的蛋白主要有TP53、ATM(ataxia telangiectasia mutated)、MDM2(murine double minute 2)、CHEK1(checkpoint kinase 1)、CHEK2(checkpoint kinase 1)、RIPK4(receptor interacting protein kinase 4)和APPBP2(Amyloid beta precursor protein (cytoplasmic tail) binding protein 2)。值得关注的是,互作蛋白中包括MDM2、RIPK4和APPBP2。
|
图 8 STRING预测PPM1D蛋白相互作用网络 Figure 8 Protein-protein interaction network for PPM1D predicted by STRING |
对于肿瘤患者,肿瘤抑制基因p53通常发生突变或表达受到抑制,研究者曾一度尝试通过逆转p53的肿瘤抑制活性进行癌症治疗。PPM1D能够由p53激活表达,其产物作为一种丝氨酸/苏氨酸磷酸酶,是细胞生长和应激反应的重要调控分子,能够使关键的细胞周期检查点蛋白和细胞损伤修复相关蛋白(如:p53、ATM、p38MAPK等)去磷酸化,从而抑制相关蛋白表达,在肿瘤发生发展过程中发挥作用。PPM1D能够以p53依赖的方式,在电离辐射和紫外线等胁迫环境下表达[8]。通过使p38 MAPK第180位苏氨酸残基去磷酸化,PPM1D能够调节p38 MAPK信号通路,抑制p38 MAPK的激酶活性和p53的肿瘤抑制活性[9-10]。另外,PPM1D能够抑制p16INK4A和p19ARF等肿瘤抑制因子的活性[5],也能够与RAS、MYC和ERBB2等致癌基因协同作用[11-12]。由此可知,PPM1D能够负反馈调节p53,PPM1D受p53激活后,其产物又能使p53去磷酸化,发挥拮抗作用[9]。在肿瘤组织中,PPM1D通常过量表达。但是,若PPMD1的3'端编码区发生截短突变,其负调控活性会受到影响[13-14]。
本文通过STRING预测知PPM1D与MDM2存在相互作用。除PPM1D以外,MDM2也能由p53激活表达,作为一种负调控因子的MDM2是一种E3泛素连接酶,能够导致p53泛素化,蛋白酶体发生降解[15-16]。在多种恶性肿瘤中都发现MDM2过量表达。Sriraman等[17]认为MDM2和PPM1D拥有不同的拮抗机制,单独靶向其一都不足以完全激活p53。研究发现,同时使用这两种分子各自的靶向药物(Nutlin-3a和GSK2830371)处理肿瘤细胞,能够显著增强p53活性,抑制或阻滞肿瘤进程。目前,联合用药已成为肿瘤药物治疗的研究热点。若两种或药物协同作用,使用联合治疗法能够增强疗效,降低每种药物的副作用。针对PPM1D与MDM2联合用药的显著疗效,有望为下一步开发联合用药策略提供思路。
STRING预测知APPBP2能够与PPM1D相互作用。Hirasawa等[18]发现APPBP2和PPM1D在卵巢透明细胞癌中都过量表达,可作为潜在靶点和预后标志物。因此,PPM1D与相互作用蛋白之间的关系及参与的具体生理生化过程具有一定的研究价值,有助于阐明PPM1D的作用机制和生理功能。
4 结论1) PPM1D的理论分子量为66.6751 kDa,理论等电点为9.14,预测为碱性蛋白质。不稳定系数为56.41,预测为不稳定蛋白质。脂肪系数为72.99,总平均亲水性为-0.517,预测为亲水蛋白质。
2) PPM1D的二级结构主要为随机卷曲,属于PP2C超家族,含有一个PP2Cc结构域。无信号肽和跨膜结构域,含有磷酸化、乙酰化、甲基化和泛素化修饰位点。该蛋白表达的组织特异性不强,定位于细胞核的可能性较大。系统进化树显示,人PPM1D蛋白与哺乳动物(如:黑猩猩、猕猴)序列相似性较大,组成一支,而与两栖类和鱼类(如:非洲爪蟾、斑马鱼)的序列相似性较小,该蛋白的同源性与物种间亲缘关系呈正相关。与PPM1D相互作用的蛋白主要是细胞周期检查点蛋白(如:CHEK1、CHEK2)和细胞损伤修复相关蛋白(如:p53、ATM、p38MAPK)。
3) 本文通过生物信息学方法,构建PPM1D的系统进化树,分析PPM1D蛋白的理化性质、组织特异性、亚细胞定位、结构域及相互作用蛋白质,为全面认识PPM1D,研究其功能及参与的具体信号通路提供一定的参考。
| [1] |
LI Guangbing, ZHANG Xueli, YUAN Li, et al. Protein phosphatase magnesium-dependent 1d (PPM1D) mRNA expression is a prognosis marker for hepatocellular carcinoma[J]. PLoS One, 2013, 8(3): e60775. DOI:10.1371/journal.pone.0060775 (  0) 0) |
| [2] |
XU Zhi, CAO Chunxiang, XIA Haiyan, et al. Protein phosphatase magnesium-dependent 1δ is a novel tumor marker and target in hepatocellular carcinoma[J]. Frontiers of Medicine, 2016, 10(1): 52-60. DOI:10.1007/s11684-016-0433-3 (  0) 0) |
| [3] |
BIEGING K T, MELLO S S, ATTARDI L D. Unravelling mechanisms of p53-mediated tumor suppression[J]. Nature Reviews Cancer, 2014, 14(5): 359-370. DOI:10.1038/nrc3711 (  0) 0) |
| [4] |
KOOM W S, PARK S Y, KIM W, et al. Combination of radiotherapy and adenovirus-mediated p53 gene therapy for MDM2-overexpressing hepatocellular carcinoma[J]. Journal of Radiation Research, 2012, 53(2): 202-210. DOI:10.1269/jrr.11110 (  0) 0) |
| [5] |
BULAVIN D V, PHILLIPS C, NANNENGA B, et al. Inactivation of the Wip1 phosphatase inhibits mammary tumorigenesis through p38 MAPK-mediated activation of the p16(Ink4a)-p19(Arf) pathway[J]. Nature Genetics, 2004, 36(4): 343-350. DOI:10.1038/ng1317 (  0) 0) |
| [6] |
TAMURA K, DUDLEY J, NEI M, et al. MEGA4: molecular evolutionary genetics analysis(MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24(8): 1596-1599. DOI:10.1093/molbev/msm092 (  0) 0) |
| [7] |
WENK J, TROMPETER H I, PETTRICH K G, et al. Molecular cloning and primary structure of a protein phosphatase 2C isoform[J]. FEBS Letters, 1992, 297(1/2): 135-138. DOI:10.1016/0014-5793(92)80344-G (  0) 0) |
| [8] |
FUKAMI S, RIEMENSCHNEIDER M J, KOHNO M, et al. Expression and gene doses changes of the p53-regulator PPM1D in meningiomas: a role in meningioma progression?[J]. Brain Tumor Pathology, 2016, 33(3): 191-199. DOI:10.1007/s10014-016-0252-x (  0) 0) |
| [9] |
TAKEKAWA M, ADACHI M, NAKAHATA A, et al. p53-inducible wip1 phosphatase mediates a negative feedback regulation of p38 MAPK-p53 signaling in response to UV radiation[J]. EMBO Journal, 2000, 19(23): 6517-6526. DOI:10.1093/emboj/19.23.6517 (  0) 0) |
| [10] |
BULAVIN D V, DEMIDOV O N, SAITO S, et al. Amplification of PPM1D in human tumors abrogates p53 tumor-suppressor activity[J]. Nature Genetics, 2002, 31(2): 210-215. DOI:10.1038/ng894 (  0) 0) |
| [11] |
DUDGEON C, SHREERAM S, TANOUE K, et al. Genetic variants and mutations of PPM1D control the response to DNA damage[J]. Cell Cycle, 2013, 12(16): 2656-2664. DOI:10.4161/cc.25694 (  0) 0) |
| [12] |
MA Dan, ZHANG Chaojun, CHEN Zulin, et al. Prognostic value of PPM1D in 800 gastric cancer patients[J]. Molecular Medicine Reports, 2014, 10(1): 191-194. DOI:10.3892/mmr.2014.2165 (  0) 0) |
| [13] |
KLEIBLOVA P, SHALTIEL I A, BENADA J, et al. Gain-of-function mutations of PPM1D/Wip1 impair the p53-dependent G1 checkpoint[J]. The Journal of Cell Biology, 2013, 201(4): 511-521. DOI:10.1083/jcb.201210031 (  0) 0) |
| [14] |
RUARK E, SNAPE K, HUMBURG P, et al. Mosaic PPM1D mutations are associated with predisposition to breast and ovarian cancer[J]. Nature, 2013, 493(7432): 406-410. DOI:10.1038/nature11725 (  0) 0) |
| [15] |
WADE M, LI Yaocheng, WAHL G M. MDM2, MDMX and p53 in oncogenesis and cancer therapy[J]. Nature Reviews Cancer, 2013, 13(2): 83-96. DOI:10.1038/nrc3430 (  0) 0) |
| [16] |
KHOO K H, VERMA C S, LANE D P. Drugging the p53 pathway: understanding the route to clinical efficacy[J]. Nature Reviews Drug Discovery, 2014, 13(3): 217-236. DOI:10.1038/nrd4236 (  0) 0) |
| [17] |
SRIRAMAN A, RADOVANOVIC M, WIENKEN M, et al. Cooperation of Nutlin-3a and a Wip1 inhibitor to induce p53 activity[J]. Oncotarget, 2016, 7(22): 31623-31638. DOI:10.18632/oncotarget.9302 (  0) 0) |
| [18] |
HIRASAWA A, SAITO-OHAR F, INOUE J, et al. Association of 17q21-q24 gain in ovarian clear cell adenocarcinomas with poor prognosis and identification of PPM1D and APPBP2 as likely amplification targets[J]. Clinical Cancer Research, 2003, 9(6): 1995-2004. (  0) 0) |
 2017, Vol. 15
2017, Vol. 15


