2. 发酵工程教育部重点实验室(湖北工业大学),武汉 430068
2. Key Laboratory of Fermentation Engineering, Ministry of Education(Hubei University of Technology), Wuhan 430068, China
依据中国国家疾病控制中心数据,手足口病(hand foot mouth disease,HFMD)年感染率位居丙类传染病首位,肠道病毒71型(enterovirus 71,EV71)是引起手足口病的常见病原体,是继脊髓灰质炎病毒之后严重危害幼儿身体健康的重要病原体之一[1]。目前国内已有EV71灭活疫苗上市,对于病毒预防已有保障,但无抗EV71特效药物,急需深入研究病毒感染过程中的宿主调控机制,发现新颖的抗病毒治疗靶标,助力抗EV71特效药开发。
EV71和宿主细胞因子作用复杂,现有报道显示细胞转录因子Zif268由EGR1基因表达,发现可以直接和EV71的RNA结合促进病毒的复制[2]。另外发现病毒的2A蛋白酶1裂解宿主的eIF4GI或是eIF4GⅡ,转换宿主依赖帽子的翻译过程为不依赖帽子的IRES翻译过程[3]。此外有研究发现病毒非结构蛋白与宿主有较为复杂的相互作用,其中2A、3C蛋白是病毒的前体蛋白水解酶,具有阻断宿主自身抗病毒反应的作用。2A蛋白既可抑制Ⅰ型干扰素的产生又可阻断细胞中少量β干扰素激活的Ⅰ型干扰素信号通路,在EV71免疫逃逸过程中发挥重要作用。2A蛋白具有蛋白酶活性和转录激活因子功能,并切割Nup62而抑制其核转运过程[4]。2C蛋白通过结合RelA(p65)蛋白抑制NF-kB的激活,并且重排宿主膜蛋白[5]。3C蛋白切割干扰素调节因子7(interferon regulatory factor 7,IRF7)抑制细胞应答[6],并通过抑制维甲酸诱导基因-Ⅰ(retinoid acid-inducible gene-Ⅰ,RIG-Ⅰ)介导的干扰素调控因子3(interferon regulatory factor 3,IRF3)的活性和切割接头蛋白TRIF起到抑制干扰素产生的作用[7],因此干扰素不能用于治疗EV71引起的手足口病。3D蛋白充当病毒基因组复制酶,具有RNA依赖的RNA聚合酶功能,Yu等[8]发现EV71可以通过3D蛋白使细胞周期停滞于S期,并可以结合NLRP3增强炎症小体的组装[9]。Wong等[10]研究发现EV71感染Vero细胞0.5~6.0 h期间能够活化PI3K/AKT通路并且通过抑制GSK-3过活性抑制细胞凋亡的发生。对于病毒感染研究发现“细胞因子风暴”可能与疾病的进程密切相关。所以仅仅从局部出发关注某几个基因的表达差异不足以描述这种高度复杂的调控关系。RD细胞是研究EV71与宿主相互作用的常用感染细胞系[11]。本文使用WGCNA方法构建了与感染时间相关差异基因共表达网络,通过差异基因间的关联度识别与EV71感染相关的关键基因,为抗EV71药物研究提供新视角。
1 材料与方法 1.1 数据与材料本研究9个样本(0、4、8 h各3个)表达谱数据集来源于BEI数据库中的数据集GEOD-15323,对应的NCBI数据集是GSE15323(https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE15323)。q-PCR引物参考相关文献或是自行设计,引物合成由昆泰瑞公司完成。RD细胞(rhabdomyosarcoma cells)和EV71病毒来源于武汉大学中国典型培养物保藏中心。培养细胞用含10%胎牛血清(FBS)的DMEM培养基(血清和培养基购自Biological Industries ©)
1.2 加权基因共表达网络构建 1.2.1 构建差异表达谱矩阵数据芯片数据中实际每一行对应一个探针序列号,多个探针对应同一个基因,通过数据控制将探针检测值的差异系数小于50%的探针剔除,并采用SAM方法对保留的探针组进行筛选,设定SAM参数为FDR≤0.05。通过3个时间点之间的两两比较,对筛选的探针组取合集,构建基因表达谱矩阵。
1.2.2 离群样本的检测离群样本定义为在聚类树图中某个样本高度分离于集体样本或是分类样本。离群样本对网络模块的分析结果具有较大的影响,进行网络构建前需要检查离群样本的存在性,去除离群值。本研究将9个样本的基因表达值矩阵根据其马氏距离绘制树形图,剔除离群样本。
1.2.3 基因模块分析WGCNA方法在生物信息学中属于系统生物学分析工具,使用该方法依赖的前提假设是生物网络中节点的连接度分布分析服从幂律分布。WGCNA方法使用一种巧妙的方法将基因表达数据进行幂律分布拟合。具体方法为对基因表达值的皮尔森相关系数取n次幂指数加权,转换基因表达皮尔生相关系数矩阵Sij为邻接系数矩阵Aij。相关系数值的分布在β次幂作用下逐渐趋于无尺度分布。其中算法定义了无尺度分布的拟合系数应满足方程的R2>0.8,表达数据可转换为调控关系数据。动态剪切数聚类方法对拟合后的无尺度分布数据进行聚类分析获取共表达模块,模块内的基因代表着一类表达模式类似的基因簇,使用颜色命名模块。如图 1所示显示WGCNA算法的基本步骤。

|
图 1 WGCNA算法基本步骤 Figure 1 WGCNA basic steps |
利用生物信息学在线软件DAVID进行基因富集分析和通路富集分析[12],使用统计学方法Fisher Exact检验计算p值,注释模块所具有的生物学功能。
1.4 枢纽基因筛选枢纽基因的筛选可以直接使用阈值法,选择连接度排在网络模块前3位的基因,或是使用TOM(拓扑矩阵)导出网络图到VisANT软件对网络模块进行可视化,设定权重值筛选枢纽基因。
1.5 qPCR验证筛选基因的转录水平为了验证枢纽基因在病毒感染中的表达差异,本文使用EV71感染8 h后的细胞进行提取总RNA、反转录和qPCR检测[11]。
选择基因表达时间序列数据中持续性上调3个基因,合并上述8个枢纽基因进行转录本表达情况验证。本文涉及到的基因引物见表 1,引物由昆泰锐(武汉)公司合成。qPCR试验按照Takara公司的PrimeScript RT reagent kit反转录试剂盒和SYBR Premix Ex Taq Ⅱ试剂盒说明书操作。
| 表 1 引物列表 Table 1 Primers list |
GSE15323全基因组表达数据包含有9个样本的病毒感染细胞数据,这些数据均经过了背景校正和标准化处理。由于数据中包含的探针数量太大,含有56 475个探针组,质量低且差异性不强的数据太多,会造成数据分析时背景过大的情况,为了减少噪音和计算机分析的运行负荷,先筛选差异基因保留显著性较高的探针组。使用R语言中的siggene包实现差异基因筛选,滤除不显著的探针组后得到6 642个探针组(q<0.05)纳入本文的重点研究。
2.2 样本数据初筛通过聚类得到病毒感染细胞的样本聚类图,发现9个样本可明显分为3类,这和试验设计一致,因为设计的感染时间是分为3个时间点,0、4 h感染时间点内的类中距离相对较近说明组内的样本一致性较强,8 h感染时间点内的类中距离较远,说明该组内样本因为病毒感染持续到后期有大量基因出现表达变化导致样本一致性较弱。图 2聚类树图结果显示9个样本不存在离群样本,将所有样本纳入后续网络建模分析。

|
图 2 样本聚类树 Figure 2 Samples clustering |
选择合适的邻接矩阵权重参数β进行基因表示数值无尺度拟合。参数的拟合区间为1~30,对某节点连接度的对数log(i)与该节点出现概率的对数log(p(i))建立线性模型,参数β拟合情况通过线性回归系数R2反映,选择R2首次接近0.9的β作为构建网络模块的目标β值。针对本文芯片数据选择β为9。β拟合情况如图 3所示。其中, 图 3的横轴代表权重参数β, 图 3(a)的纵轴表示对应网络中log(i)和log(p(i))的线性相关系数R2,该系数越高(至少要达到0.8,完美的无尺度网络模型适用指数是1),表明该网络越趋近于无尺度网络;图 3(b)的纵轴代表模型中所有基因接邻系数的均值,反映网络的平均连接水平。
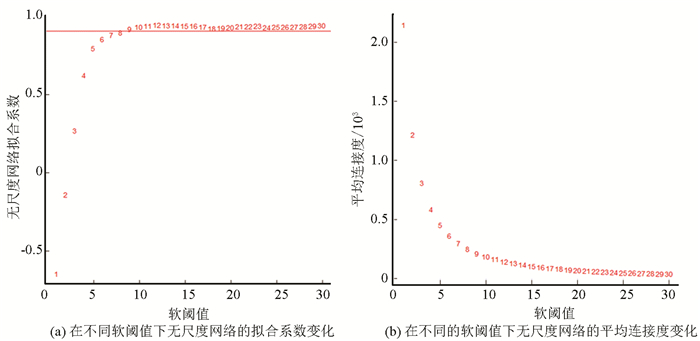
|
图 3 参数β拟合情况 Figure 3 Parameter β fitting |
选择β=9计算网络拓扑重叠TOM,利用层次聚类法得到基因系统聚类树。根据动态分层剪切数算法挖掘出基因模块,设定每一个基因模块的最小基因数为100,基因聚类的高度上限为0.995。基因模块构建完成后,计算每个基因模块的特征向量基因(module eigengene, ME)。
如图 4所示,通过动态层次聚类本文得到了18个模块,其中grey模块表示不属于任何模块的聚类。
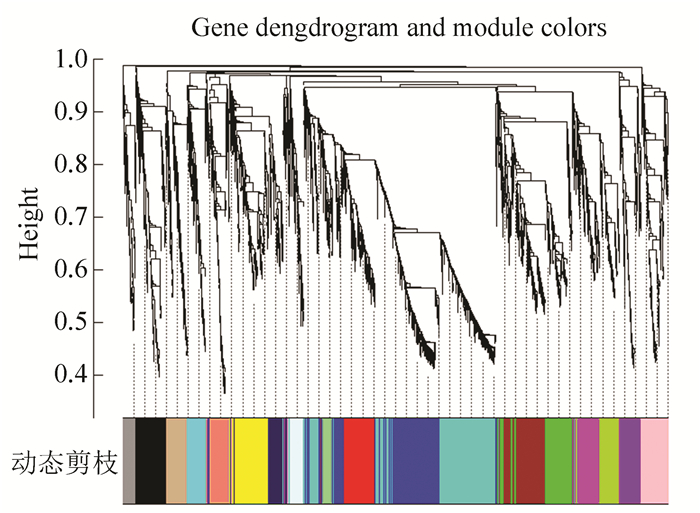
|
图 4 动态分层聚类 Figure 4 Dynamic hierarchical cluster |
对于这18个模块和感染时间的关联性,本文使用模块的第一主成分和感染时间的相关系数进行定量化。分析得出pink模块和darkgreen模块和感染时间的关联性最高, 如图 5所示。
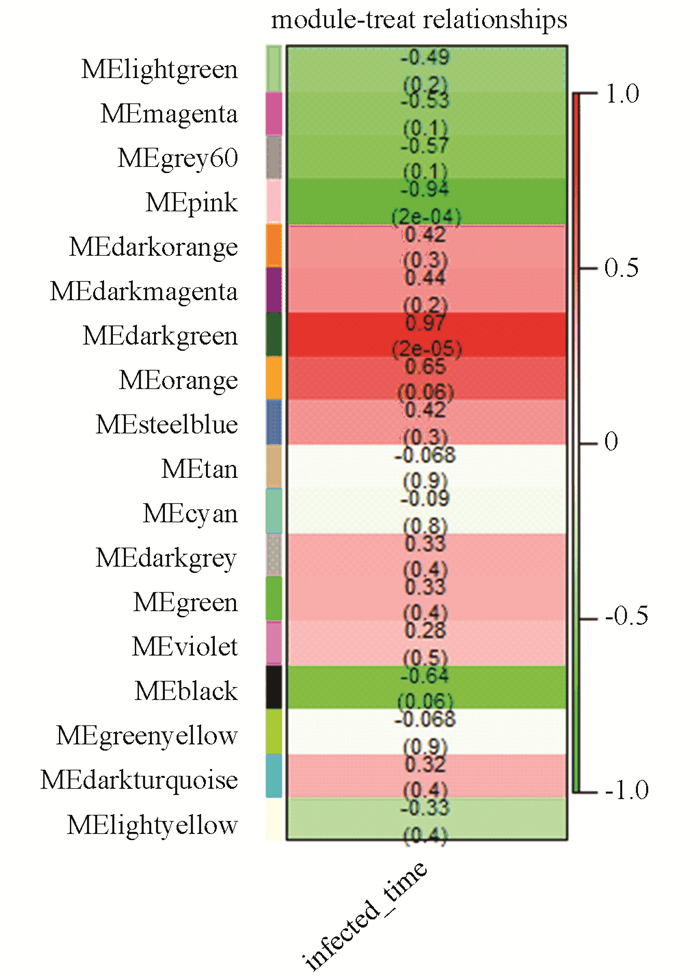
|
图 5 模块与感染时间的关联性 Figure 5 Relationship between modules and infected time |
从图 5中可以看出pink模块和感染时间呈负关联性,相关系数为-0.94,darkgreen模块和感染时间呈正关联性,相关系数为0.97。还有相关性较强的其他模块比如orange模块和black模块,本文对关联性最强的pink模块和darkgreen模块进行生物功能注释。
呈现加权基因共表达网络的一种直观方式是热图,将所有基因加权共表达值转换得到的拓扑重叠矩阵绘制于一副全像素图中,可以明显看到图 6中有颜色较深区域,展示的即是加权共表达模块。
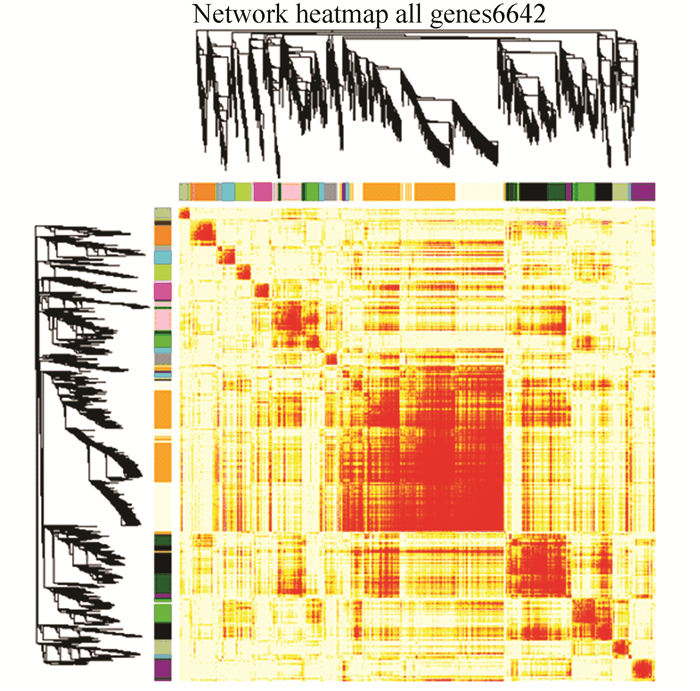
|
图 6 基于TOM矩阵的加权基因共表达网络热图 Figure 6 Heatmap of network based on TOM matrix 注:图中基于所有基因的TOM矩阵绘制,热图中每行及每列均对应一个基因,颜色越深,提示基因间的拓扑重叠度越高,基因间的调控关系越强。图中的左侧和上侧为基因聚类树图中的基因模块。 |
通过GO富集分析和通路富集分析可以得到目标模块在基因本位角度和代谢通路角度的信息。其中p值小于0.05的结果见表 2。GO富集分析结果显示目标模块主要参与前剪切体的反式组装、cAMP的应答、U5 snRNP、翻译起始、膜结构、多聚腺苷酸尾的结合,可能与手足口病的发生有一定的关联。通路富集分析结果显示目标模块高度富集于核糖体通路。
| 表 2 GO功能富集分析和KEGG代谢通路富集分析结果 Table 2 Result of enrichment analysis of GO and KEGG pathway |
结合模块基因富集分析和通路富集分析的结果,将目标模块pink模块和darkgreen模块中的基因调控关系进行可视化展示。如图 7、8所示(同一模块中拓扑重叠值相同),其中pink模块的权重值区间设置为0.40~1.00,选择连接度大于或等于7视为枢纽基因,设置darkgreen模块的权重值区间为0.38~1.00,选择连接度大于或等于8视为枢纽基因。从pink模块的共表达网络可以看出3个基因ABLIM1、CTSS和SUPT7L形成一定的核心,其余基因围绕在其周围,这种状态呈现出小世界性。
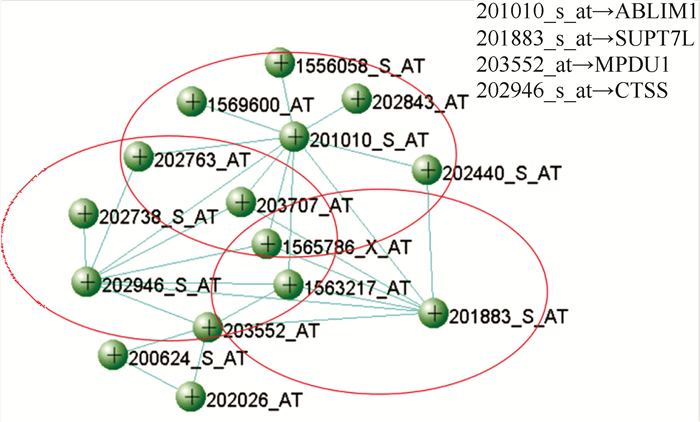
|
图 7 pink模块共表达网络 Figure 7 Network of pink module |

|
图 8 darkgreen模块共表达网络 Figure 8 Network of darkgreen module |
darkgreen模块形成的共表达网络没有明显的小世界性或是成簇性,只有4个基因CXCL9、RPS4X、CD55和HSPA13的连接度较高。表 3显示目标模块中的关键节点基因。
| 表 3 目标模块中的关键节点基因 Table 3 Hub genes of targeted modules |
为了比较目标基因的检测可靠性,将3个持续性上调的差异基因TXNIP、FOS和EGR1纳入验证试验。q-PCR的结果如图 9所示。纵坐标是相对表达值log 2处理后的数据,柱形图高度为1表示感染24 h该基因的表达强度为原来2倍。比较芯片数据和q-PCR数据发现9个基因的变化方向是一致的,有2个基因上、下调情况不明显,其中3个持续性上调基因在原始芯片和q-PCR试验中表达趋势相同。
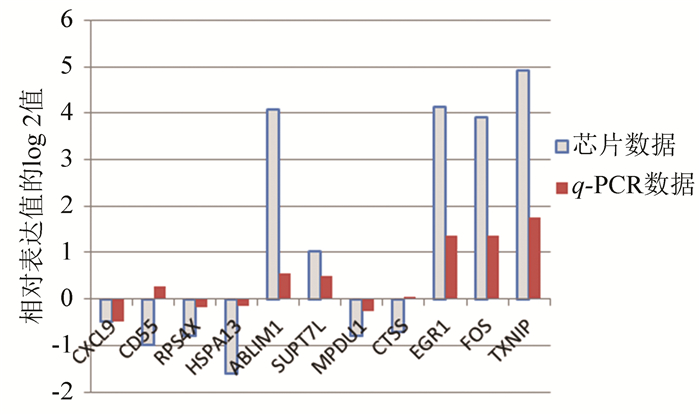
|
图 9 11个基因的相对表达 Figure 9 Relatively expression of 11 genes |
本文首先从NCBI的GEO数据库下载EV71感染相关的表达谱数据集GSE15323(含有9个样本),使用WGCNA方法构建了18个模块,并重点关注感染时间高关联性的pink模块和darkgreen模块,对目标模块进行GO功能注释、KEGG通路富集分析和共表达网络可视化展示,从中筛选了8个Hub基因。pink模块GO术语富集为cis assembly of pre-catalytic spliceosome、response to cAMP和U5 snRNP,darkgreen模块GO术语富集为translational initiation、membrane和poly(A) RNA binding;pink模块没有富集到显著的通路,darkgreen模块富集的通路为Ribosome。
WGCNA方法筛选的基因RPS4X、HSPA13、ABLIM1、MPDU1、CXCL9、CD55、SUPT7L和CTSS在EV71感染中可能具有重要作用。Wang等[13]调研细胞因子和病毒感染关系发现,CXCL9在脑干脑炎患者血浆中含量比正常人有显著升高,比较CXCL9在感染EV71的肺水肿患者血浆中含量比无症状脑干脑炎患者和正常组有显著升高,并且随着病情加重CXCL9表达值逐渐上升。CD55分子被确定为肠道病毒属的Echovirus 1病毒的细胞受体,并且CD55会诱导骨肉瘤细胞的c-FOS基因表达[14]。其余的6个基因RPS4X、HSPA13、ABLIM1、MPDU1、SUPT7L和CTSS没有发现和EV71有间接或是直接的关系。TXNIP、EGR1和c-FOS这3个基因中的TXNIP和EGR1在病毒感染细胞过程中可能发挥重要作用。硫氧蛋白相互作用蛋白(thioredoxin interacting protein, TXNIP), 该蛋白是细胞炎症反应中的重要因子,维持细胞氧化还原压力的平衡,而EV71感染细胞后通过增加NOX2介导的ROS产物上调氧化还原压力[15]。EGR1(early growth response 1),是一种转录调控因子,又称为锌指蛋白268(Zif268,zinc finger protein 268)或神经生长因子诱导蛋白A(NGFI-A,nerve growth factor-induced protein A),其靶向激活细胞分化和有丝分裂的必需基因,被认为是一种肿瘤抑制基因[16],报道显示EGR1可以直接和EV71的RNA结合促进病毒的复制,并且其促进方式不依赖于病毒的IRES结构。有文献报道[17]EV71感染RD细胞8 h后c-FOS基因表达上调3.34倍和本文的q-PCR结果较为接近。
4 结语本文成功构建了与EV71感染时间相关的差异基因共表达网络,挖掘出8个关键基因RPS4X、HSPA13、ABLIM1、MPDU1、CXCL9、CD55、SUPT7L和CTSS,经q-PCR实验验证3个持续上调的基因TXNIP、EGR1、c-FOS和8个枢纽基因的转录水平与芯片数据一致,其中TXNIP、EGR1、c-FOS表达上调超过2.5倍,CXCL9下调4.5倍。这些关键基因的发现可能为EV71感染机制研究和抗病毒药物靶标发现提供一定参考。
| [1] |
杨芳, 于石成, 张菊英, 等. 2008—2011年我国大陆地区重症手足口病流行特征分析[J]. 疾病监测, 2013, 28(11): 888-893. YANG Fang, YU Shicheng, ZHANG Juying, et al. Epidemiology of severe hand foot and mouth disease in the mainland of China, 2008-2011[J]. Disease Surveillance, 2013, 28(11): 888-893. DOI:10.3784/j.issn.1003-9961.2013.11.006 (  0) 0) |
| [2] |
SONG Yu, CHENG Xin, YANG Xiaoxia, et al. Early growth response-1 facilitates enterovirus 71 replication by direct binding to the viral genome RNA[J]. The International Journal of Biochemistry & Cell Biology, 2015, 62: 36-46. DOI:10.1016/j.biocel.2015.02.012 (  0) 0) |
| [3] |
HSU Y Y, LIU Y N, LU W W, et al. Visualizing and quantifying the differential cleavages of the eukaryotic translation initiation factors eIF4GI and eIF4GⅡ in the enterovirus-infected cell[J]. Biotechnol Bioeng, 2009, 104(6): 1142-1152. DOI:10.1002/bit.22495 (  0) 0) |
| [4] |
张亚洲, 甘星, 宋娟, 等. EV71通过2A蛋白酶切割Nup62而抑制核转运[J]. 病毒学报, 2013, 29(4): 421-425. ZHANG Yazhou, GAN Xing, SONG Juan, et al. The 2A protease of enterovirus 71 cleaves nup62 to inhibit nuclear transport[J]. Chinese Journal of Virology, 2013, 29(4): 421-425. DOI:10.13242/j.cnki.bingduxuebao.002412 (  0) 0) |
| [5] |
DU Haiwei, YIN Peiqi, YANG Xiaojie, et al. Enterovirus 71 2C Protein Inhibits NF-kappaB Activation by Binding to RelA(p65)[J]. Sci Rep, 2015, 5: 14302. DOI:10.1038/srep14302 (  0) 0) |
| [6] |
LEI Xiaobo, XIAO Xia, XUE Qinghua, et al. Cleavage of interferon regulatory factor 7 by enterovirus 71 3C suppresses cellular responses[J]. Journal of Virology, 2013, 87(3): 1690-1698. DOI:10.1128/JVI.01855-12 (  0) 0) |
| [7] |
LEI Xiaobo, LIU Xinlei, MA Yijie, et al. The 3C protein of enterovirus 71 inhibits retinoid acid-inducible gene Ⅰ-mediated interferon regulatory factor 3 activation and type Ⅰ interferon responses[J]. Journal of Virology, 2010, 84(16): 8051-8061. DOI:10.1128/JVI.02491-09 (  0) 0) |
| [8] |
YU Jinghua, ZHANG Liying, REN Penyou, et al. Enterovirus 71 mediates cell cycle arrest in S phase through non-structural protein 3D[J]. Cell Cycle, 2015, 14(3): 425-436. DOI:10.4161/15384101.2014.980631 (  0) 0) |
| [9] |
WANG Hongbin, LEI Xiaobo, XIAO Xia, et al. Reciprocal Regulation between Enterovirus 71 and the NLRP3 Inflammasome[J]. Cell Reports, 2015, 12(1): 42-48. DOI:10.1016/j.celrep.2015.05.047 (  0) 0) |
| [10] |
WONG W R, CHEN Y Y, YANG S M, et al. Phosphorylation of PI3K/Akt and MAPK/ERK in an early entry step of enterovirus 71[J]. Life Sciences, 2005, 78(1): 82-90. DOI:10.1016/j.lfs.2005.04.076 (  0) 0) |
| [11] |
CHANG Y L, HO B C, SHER S, et al. miR-146a and miR-370 coordinate enterovirus 71-induced cell apoptosis through targeting SOS1 and GADD45 β[J]. Cell Microbiol, 2015, 17(6): 802-818. DOI:10.1111/cmi.12401 (  0) 0) |
| [12] |
HUANG D W, SHERMAN B T, LEMPICKI R A. Bioinformatics enrichment tools: paths toward the comprehensive functional analysis of large gene lists[J]. Nucleic Acids Research, 2009, 37(1): 1-13. DOI:10.1093/nar/gkn923 (  0) 0) |
| [13] |
WANG S M, LEI H Y, YU C K, et al. Acute chemokine response in the blood and cerebrospinal fluid of children with enterovirus 71-associated brainstem encephalitis[J]. Journal of Infectious Diseases, 2008, 198(7): 1002-1006. DOI:10.1086/591462 (  0) 0) |
| [14] |
WARD T, PIPKIN P A, CLARKSON N A, et al. Decay-accelerating factor CD55 is identified as the receptor for echovirus 7 using CELICS, a rapid immuno-focal cloning method[J]. EMBO Journal, 1994, 13(21): 5070-5074. (  0) 0) |
| [15] |
PAIVA C N, BOZZA M T. Are reactive oxygen species always detrimental to pathogens?[J]. Antioxid Redox Signal, 2014, 20(6): 1000-1037. DOI:10.1089/ars.2013.5447 (  0) 0) |
| [16] |
KNAPSKA E, KACZMAREK L. A gene for neuronal plasticity in the mammalian brain: Zif268/Egr-1/NGFI-A/Krox-24/TIS8/ZENK?[J]. Progress in Neurobiology, 2004, 74(4): 183-211. DOI:10.1016/j.pneurobio.2004.05.007 (  0) 0) |
| [17] |
SHI Weifeng, HOU Xueling, LI Xiang, et al. Differential gene expressions of the MAPK signaling pathway in enterovirus 71-infected rhabdomyosarcoma cells[J]. The Brazilian Journal of Infectious Diseases, 2013, 17(4): 410-417. DOI:10.1016/j.bjid.2012.11.009 (  0) 0) |
 2017, Vol. 15
2017, Vol. 15


