2. 大理州人民医院 病理科,云南 大理 671000;
3. 云南大学 生命科学学院,昆明 530061
2. Department of Pathology, Dali Bai Autonous Prefecture People's Hospital, Dali 671000, Yunnan, China;
3. College of Life Sciences, Yunnan University, Kunming 530061, China
胃癌是源自胃黏膜上皮的恶性肿瘤,占消化道恶性肿瘤的首位[1]。中国是胃癌高发国家,每年约有17万人死于胃癌,是一种严重威胁人类身体健康和生命的疾病[2]。其发生、发展机制颇为复杂,涉及一系列的遗传学改变[3]。从分子水平研究胃癌发生、发展的机制有利于开发新的治疗手段,对减轻病人的痛苦和延长生命尤为重要。
人线粒体转录终止因子1(human mitochondrial transcription termination factor 1, hMTERF1)基因是人类MTERF超基因家族中最早被发现的第1个成员[4]。目前,已确定hMTERF1基因位于7q21.2,基因全长7 845 bp,其含有4个外显子和3个内含子[5]。hMTERF1蛋白由399个氨基酸残基构成,具有3个亮氨酸拉链结构和2个独立的DNA结合区[6]。该蛋白与线粒体DNA(mitochondrial DNA, mtDNA)上编码16S rRNA基因与tRNALEU(UUR)基因分界处的一段28 bp的序列特异结合,从而终止mtDNA重链的转录[7]。有研究发现,与hMTERF1蛋白结合的线粒体tRNALEU(UUR)基因A3243G位点的碱基突变,将引起老年性糖尿病、线粒体脑肌病伴高乳酸血症和卒中样发作综合症、进行性眼外肌麻痹等线粒体疾病。mtDNA靶序列A3243G点突变引起hMTERF1与DNA亲合力下降,但并不改变线粒体转录产物的比例,但线粒体蛋白质合成能力和细胞的呼吸活性下降将导致线粒体疾病[8]。但目前为止,关于hMTERF1基因与胃癌的关系尚未见相关报道。本文基于BioGPS数据库、Oncomine数据库以及Kaplan-Meier Plotter数据库的数据挖掘分析hMTERF1基因在胃癌中的表达及意义,为阐明hMTERF1基因在胃癌发生发展中的作用奠定基础。
1 材料与方法 1.1 Bio GPS数据库分析hMTERF1在正常组织中的表达利用BioGPS数据库[9](http://biogps.org/)分析hMTERF1基因(204871_at)在人体正常组织中的表达。
1.2 Oncomine数据库提取数据Oncomine数据库[10]是一个基于基因芯片的数据库和整合数据挖掘平台,在此数据库中可根据自己的需要设定筛选条件。本文设定的筛选条件为:1)“Gene: MTERF1”;2)“Analysis Type: Cancer vs. Normal Analysis”;3)“Cancer Type: Gastric Cancer”;4)“Date Type: mRNA and DNA copy number”;5)“Sample Type: Clinical Specimen”;6)临界值设定条件(P value < 0.000 1, fold change 2, gene rank=top 10%)。
1.3 Kaplan-Meier Plotter进行患者生存周期分析利用Kaplan-Meier Plotter数据库[11](http://kmplot.com/analysis/)的胃癌数据集进行在线生存分析。筛选条件如下:1)“Cancer: Gastric Cancer”; 2)“Gene: MTERF1”; 3)“Survival: OS”; 4)“Lauren classification: all/intestinal/diffuse/mixed”。
1.4 hMTERF1在不同分期胃癌患者中的预后作用设置截尾数据为OS,然后探针选择为JetSet,所需的有效Affymetrix ID: 204871_at (MTERF1),在Stage选项下面分别选择1、2、3、4期,Lauren classification选项下面分别选择all/intestinal/diffuse/mixed,在线分析hMTERF1在不同临床分期的胃癌患者中的预后作用。
1.5 统计学分析胃癌组织与正常胃组织中hMTERF1基因表达差异采用t检验。hMTERF1基因表达与患者预后的关系采用Kaplan-Meier模型进行生存分析,两组间生存率的比较采用Log-Rank检验,检验水准α=0.05。
2 结果分析 2.1 hMTERF1蛋白在正常组织中的表达BioGPS数据库显示,hMTERF1蛋白在人体各个正常组织中均有表达,其中在心肌组织、子宫体和骨骼肌组织中含量较高,如图 1所示。由此可知,hMTERF1蛋白在机体新陈代谢旺盛的组织中呈现高表达,所以hMTERF1是一种典型的线粒体蛋白质。
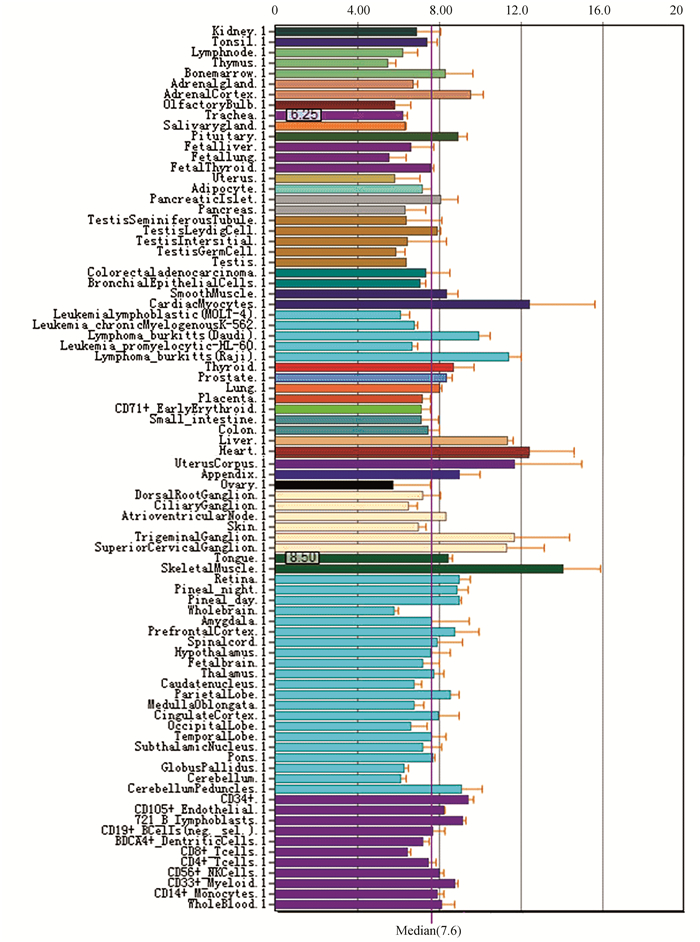
|
图 1 BioGPS数据库显示hMTERF1在人体各种组织中的表达 Figure 1 BioGPS microarray expression data for hMTERF1 in different human tissues |
Oncomine数据库中共收集了946个不同类型的基因芯片研究结果,其中关于hMTERF1表达有统计学差异的研究结果共152个,hMTERF1表达增高的研究有59个,hMTERF1表达降低的研究有93个(如图 2所示)。在人类消化道系统常见的恶性肿瘤中,食管癌中高表达的研究有2个,低表达的研究有2个;胃癌中高表达的研究有4个,低表达的研究有5个;肝癌中高表达的研究有2个;结直肠癌中高表达的研究有2个,低表达的研究有8个。
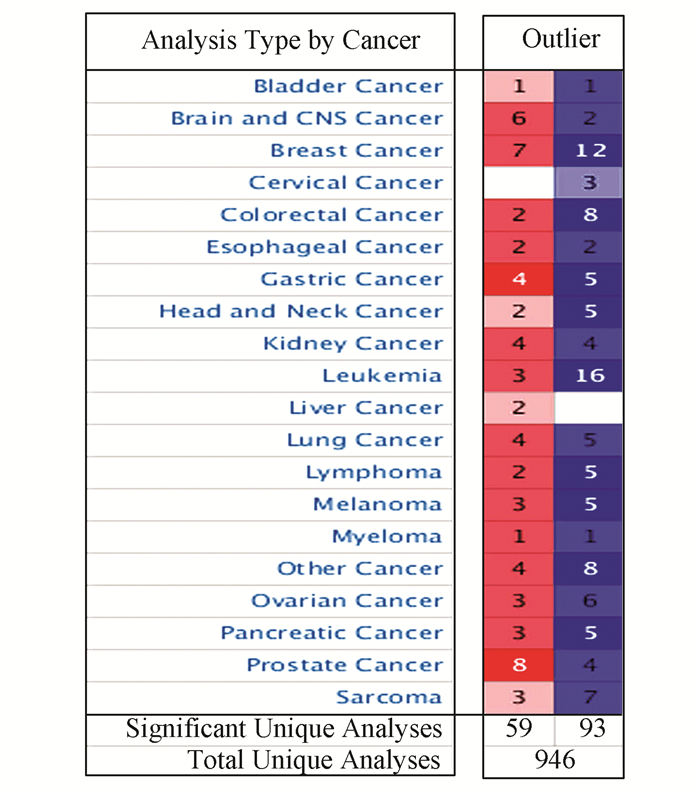
|
图 2 hMTERF1在Oncomine数据库所有肿瘤相关研究中的表达 Figure 2 All the related research of tumor the hMTERF1 in Oncomine database |
Oncomine数据库中检索发现,从2003年至今,共有20项研究涉及hMTERF1在胃癌组织和正常胃组织中的表达,共计798个样本(如图 3所示)。相关的文献分别发表在Mol Biol Cell[12]、Clin Cancer Research[13]、Nucleic Acids Research[14]、Gut[15]、Med Oncol[16]期刊。详细分析20项研究结果发现,与正常胃组织相比,hMTERF1在胃癌组织中呈现高表达,中位数值为4 728.5,P=0.003,表明其高表达具有显著性差异。

|
图 3 hMTERF1在Oncomine数据库胃癌研究中的表达 Figure 3 Expression of hMTERF1 in gastric cancer in the studies identified in the Oncomine database 注:1-20分别表示20项研究结果,红色越深表示hMTERF1基因在该芯片中表达越高。 |
对Oncomine数据库的数据挖掘显示,相比正常胃组织,hMTERF1基因在弥漫型胃癌组织、肠型胃癌组织和混合型胃癌组织中均呈高表达,且差异具有统计学意义(P<0.050),如图 4所示。
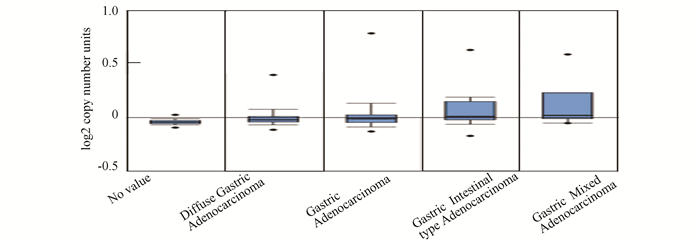
|
图 4 Oncomine芯片数据库中hMTERF1在不同胃癌组织中的表达情况 Figure 4 Expression of hMTERF1 mRNA evaluated in gastric cancers by using Oncomine database analysis |
为进一步明确hMTERF1表达水平与胃癌患者预后之间的关系,本文在Oncomine数据库中提取hMTERF1基因表达水平与胃癌患者预后相关的数据,通过Kaplan-Meier Plotter数据库分析发现:与hMTERF1高表达组相比,hMTERF1低表达组胃癌患者的累积生存时间增高(HR=1.19, P=0.045),说明hMTERF1表达是胃癌患者预后的风险因素。在肠型胃癌患者中,hMTERF1高表达胃癌患者的生存时间增高(HR=0.69, P=0.028),说明hMTERF1表达是肠型胃癌患者预后的保护因素。在弥漫型胃癌患者中,hMTERF1低表达胃癌患者的生存时间增高(HR=1.48, P=0.024),说明hMTERF1表达是弥漫型胃癌患者预后的风险因素。在混合型胃癌患者中,hMTERF1高表达患者与低表达患者生存时间的差异无统计学意义(HR=0.57, P=0.310),如图 5所示。
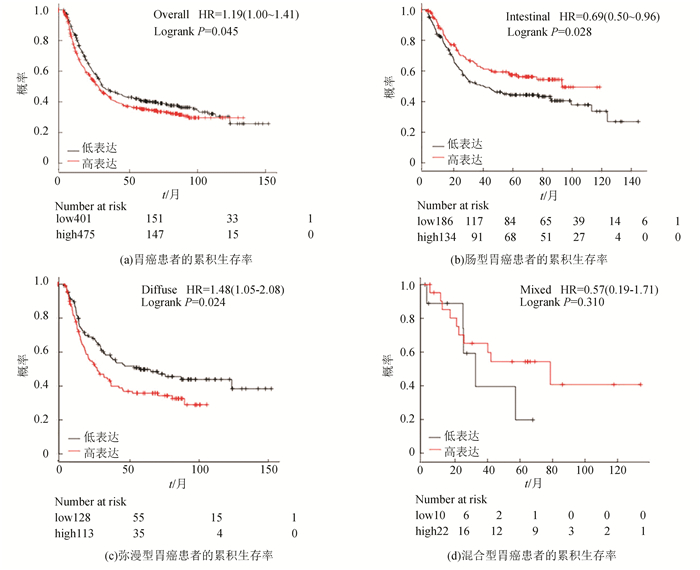
|
图 5 Kaplan-Meier Plotter数据库分析hMTERF1对胃癌患者预后的影响 Figure 5 Relationship between the expression of hMTERF1 and the prognosis of gastric cancer Unicariate Kaplan-Meier survival analysis performed using datasets compiled at Kaplan-Meier Plotter |
在所有不同类型的胃癌中,早期胃癌患者(2期)hMTERF1表达水平越高,患者预后越差(HR>1),说明hMTERF1表达对胃癌患者的总体生存率(OS)为风险因素。在肠型胃癌中,早期、中期和晚期hMTERF1表达水平越高,患者预后越好(HR<1),说明hMTERF1表达对不同分期的肠型胃癌患者均为保护因素。在弥漫型胃癌中,胃癌患者(2期+3期)hMTERF1表达水平越高,患者预后越差(HR>1)。在混合型胃癌中,由于数据库中的样本量有限,未能获得相关数据,见表 1。
| 表 1 各个分期胃癌患者的预后与hMTERF1 mRNA水平的关系 Table 1 The relationship of hMTERF1 mRNA levels and the prognosis of different stage of gastric cancer patients |
胃癌是常见的消化道恶性肿瘤,是严重危害人类健康和生命的恶性肿瘤之一,其发病率和死亡率较高,以中老年居多[17-18]。在中国,胃癌的死亡率位居所有恶性肿瘤的第3位[19]。胃癌的发生是一个复杂的过程,是多基因、多因素及其相互作用的结果[20]。近年来国内外学者对mtDNA与恶性肿瘤发生发展之间的关联提出一些假说,已有的研究资料显示肿瘤的发生不仅取决于核内遗传物质,而且与核外的mtDNA有一定的联系。mtDNA是环境致癌物作用的重要靶标,其损伤程度和突变率显著高于核内DNA。
MTERF1蛋白是线粒体转录终止因子(MTERF)蛋白家族的第1个成员,也是最早被发现的MTERF蛋白[21]。MTERF1蛋白广泛存在于后生动物与植物中。蛋白质相互作用分析表明,MTERF1蛋白与线粒体转录起始因子A(mitochondrial transcription factor A, TFAM)、线粒体RNA聚合酶(mitochondrial RNA polymerase, PLORMT)和线粒体转录延伸因子(mitochondrial transcription elongation factor, TEFM)等蛋白存在相互作用,共同参与线粒体基因复制、转录、氧化磷酸化活性和细胞周期等的调控[22]。Haväinen等[23-24]发现,在人胚肾293T细胞内过表达MTERF1,将导致mtDNA复制的终止和mtDNA拷贝数的下降,而且MTERF1蛋白在协调mtDNA的复制和转录过程中发挥重要的作用。Chen等[25]对人类宫颈癌HeLa细胞株的体外研究发现,hMTERF1蛋白可调控肿瘤细胞的线粒体基因表达和氧化磷酸化水平,从而促进肿瘤细胞的增殖,这为探讨hMTERF1在肿瘤细胞的发生发展提供了新的视角。目前,hMTERF1在胃癌中的表达及其预后意义还未见相关报道。
BioGPS数据库是一个在线的基因注释数据库,包含了公共资源的信息以及诺华研究部门所产生的数据。基于BioGPS数据库挖掘的结果显示,hMTERF1蛋白在人体正常组织中均有表达,尤其在心肌组织、子宫体和骨骼肌组织等新陈代谢旺盛的组织中表达较高,是一个典型的线粒体蛋白质。Oncomine数据库是目前世界上最大的基因芯片数据库和整合数据挖掘平台,其中的所有数据都来自基因芯片,研究方法一致。本文利用该数据平台避免了因为样本量的问题导致的误差,增加了结论的可信度[26]。本文分析结果显示,相较于正常胃组织,hMTERF1在不同类型的胃癌组织中均表达增高,且差异具有统计学意义(P<0.050)。
1965年Lauren根据胃癌的组织结构和生物学行为,将胃癌分为肠型和弥漫型。肠型胃癌起源于肠化生黏膜,一般具有明显的腺管结构,瘤细胞呈柱状或立方形,可见刷状缘,瘤细胞分泌酸性黏液物质,类似于肠癌的结构;常伴有萎缩性胃炎和肠化生,多见于老年男性,病程较长,发病率较高,预后较好。弥漫型胃癌起源于胃固有黏膜,癌细胞分化较差,呈弥漫性生长,缺乏细胞连接,一般不形成腺管,许多低分化腺癌和印戒细胞癌属于此型;多见于年轻女性,易出现淋巴结转移和远处转移,预后较差。Lauren分型简明有效,常在病理研究中应用。但有10%~20%的病例兼有肠型和弥漫型的特征,难以归入其中任何一种,从而称为混合型。关于hMTERF1蛋白表达水平对胃癌患者预后的影响,目前为止一直缺乏强有力的证据和实验研究。本文通过数据挖掘发现hMTERF1在不同Lauren分型的胃癌患者中的预后有所差异。与hMTERF1高表达组相比,hMTERF1低表达组胃癌患者的累积生存时间增高,差异有统计学意义(P<0.050),说明hMTERF1表达是胃癌发生的风险因素(HR=1.19)。在肠型胃癌患者中,hMTERF1高表达胃癌患者的生存时间增高,差异有统计学意义(P<0.050),说明hMTERF1表达是肠型胃癌的保护因素(HR=0.69)。而在弥漫型胃癌患者中,hMTERF1低表达胃癌患者的生存时间增高,差异有统计学意义(P<0.050),说明hMTERF1表达是肠型胃癌的风险因素(HR=1.48)。在混合型胃癌患者中,hMTERF1高表达患者与低表达患者生存时间的差异无统计学意义(P>0.050),既不是预后的保护因素也不是风险因素。目前,hMTERF1在胃癌发生发展中具体的作用及其分子机制仍不清楚。上述结果表明,hMTERF1在不同类型的胃癌组织中可能具有不同的病理作用,其表达水平与不同类型胃癌患者的预后关系存在差异,亟待进一步通过免疫组织化学和Western blot技术在蛋白质表达水平上分析验证hMTERF1在不同类型胃癌组织中的表达水平。hMTERF1蛋白将有望成为判别不同类型胃癌的一个潜在的生物标记物。
综上所述,本文通过对BioGPS数据库、Oncomine基因芯片数据库和Kaplan-Meier Plotter数据库中肿瘤相关基因信息的深入挖掘,提出hMTERF1在胃癌组织中高表达,且与胃癌的预后相关。通过对数据库进行大样本量的数据挖掘和分析,可以避免单一研究样本量过小导致的误差,为胃癌的临床治疗提供重要的线索和思路。
4 结论1) 利用BioGPS数据库分析hMTERF1在正常组织中的表达,发现hMTERF1在人体正常组织中均有表达,其中在心肌组织、子宫体和骨骼肌组织中含量较高。
2) 利用Oncomine数据库对hMTERF1基因在胃癌中的表达进行汇总分析,发现与正常胃组织相比,hMTERF1在胃癌组织中呈现高表达,且差异具有统计学意义(P=0.003)。
3) 利用Kaplan-Meier Plotter数据库分析hMTERF1表达对不同Lauren分型胃癌患者生存时间的影响,结果显示,与hMTERF1高表达组相比,hMTERF1低表达组胃癌患者的累积生存时间增高(P=0.045)。在肠型胃癌患者中,hMTERF1高表达胃癌患者的生存时间增高(P=0.028)。而在弥漫型胃癌患者中,hMTERF1低表达胃癌患者的生存时间增高(P=0.024)。在混合型胃癌患者中,hMTERF1高表达患者与低表达患者生存时间的差异无统计学意义(P=0.310)。
| [1] |
FERLAY J, SHIN H, BRAY F, et al. Estimates of worldwide burden of cancer in 2008: GLOBOCAN 2008[J]. International Journal of Cancer, 2010, 127(12): 2893-2917. DOI:10.1002/ijc.25516 (  0) 0) |
| [2] |
王晓瑜. 胃癌研究相关文献热点变化分析[J]. 临床军医杂志, 2015, 43(9): 955-959. WANG Xiaoyu. The study of gastric cancer was analyzed[J]. Clinical Journal of Medical Officers, 2015, 43(9): 955-959. DOI:10.16680/j.1671-3826.2015.09.24 (  0) 0) |
| [3] |
ZHOU Yong, LI Ni, ZHUANG Wen, et al. P53 codon 72 polymorphism and gastric cancer: a meta-analysis of the literature[J]. International Journal of Cancer, 2007, 121(7): 1481-1486. DOI:10.1002/ijc.22833 (  0) 0) |
| [4] |
TERZIOGLU M, RUZZENENTE B, HARMEL J, et al. MTERF1 binds mtDNA to prevent transcriptional interference at the light-strand promoter but is dispensable for Rrna gene transcription regulation[J]. Cell Metabolism, 2013, 17(4): 618-626. DOI:10.1016/j.cmet.2013.03.006 (  0) 0) |
| [5] |
GUSTAFSSON C M, LARSSON N G. MTERF1 gives mtDNA an unusual twist[J]. Cell Metabolism, 2010, 12(1): 3-4. DOI:10.1016/j.cmet.2010.06.006 (  0) 0) |
| [6] |
熊伟, 杨勇琴, 张海洋, 等. 人线粒体转录终止因子1 (hMTERF1)蛋白的生物信息学分析[J]. 生物信息学, 2015, 13(1): 23-30. XIONG Wei, YANG Yongqin, ZHANG Haiyang, et al. Bioinformatics analysis of human mitochondrial transcription termination factor 1 (hMTERF1)[J]. Chinese Journal of Bioinformatics, 2015, 13(1): 23-30. DOI:10.3969/j.issn.1672-5565.2015.01.05 (  0) 0) |
| [7] |
KRUSE B, NARASIMHAN N, ATTARDI G. Termination of transcription in human mitochondria: identification and purification of a DNA binding protein factor that promotes termination[J]. Cell, 1989, 58(2): 391-397. DOI:10.1016/0092-8674(89)90853-2 (  0) 0) |
| [8] |
阳娅玲, 肖红利, 管敏鑫. 人类线粒体tRNA生物合成与线粒体疾病[J]. 中国生物化学与分子生物学报, 2013, 29(10): 916-925. YANG Yaling, XIAO Hongli, GUAN Minxin. Human mitochondrial tRNA biosynthesis and mitochondrial disease[J]. China Journal of Biochemistry and Molecular Biology, 2013, 29(10): 916-925. DOI:10.3321/j.issn:1000-3282.2008.08.001 (  0) 0) |
| [9] |
WU Chunlei, OROZCO C, BOYER J, et al. BioGPS: an extensible and customizable portal for organizing and querying gene annotation resources[J]. Genome Biology, 2009, 10(11): R130. DOI:10.1186/gb-2009-10-11-r130 (  0) 0) |
| [10] |
王巍, 张志常, 宋晓雯, 等. 基于数据挖掘分析PDCD5表达对胃癌预后的影响[J]. 现代肿瘤医学, 2016, 24(24): 3957-3959. WANG Wei, ZHANG Zhichang, SONG Xiaowen, et al. The prognosis roles of PDCD5 in gastric cancer: Evidence from data mining[J]. Modern Oncology, 2016, 24(24): 3957-3959. DOI:10.3969/j.issn.1672-4992.2016.24.023 (  0) 0) |
| [11] |
SZASZ A M, LANCZKY A, NAGY A, et al. Cross-validation of survival associated biomarkers in gastric cancer using transcriptomic data of 1, 065 patients[J]. Oncotarget, 2016, 7(31): 49322-49333. DOI:10.18632/oncotarget.10337 (  0) 0) |
| [12] |
CHEN Xin, LEUNG S Y, YUEN S T, et al. Variation in gene expression patterns in human gastric cancers[J]. Mol Biol Cell, 2003, 14(8): 3208-3215. DOI:10.1091/mbc.E02-12-0833 (  0) 0) |
| [13] |
CHO J Y, LIM J Y, CHEONG J H, et al. Gene expression signature-based prognostic risk score in gastric cancer[J]. Clin Cancer Research, 2011, 17(7): 1850-1857. DOI:10.1158/1078-0432.CCR-10-2180 (  0) 0) |
| [14] |
CUI Juan, CHEN Yunbo, CHOU Wenchi, et al. An integrated transcriptomic and computational analysis for biomarker identification in gastric cancer[J]. Nucleic Acids Research, 2011, 39(4): 1197-1207. DOI:10.1093/nar/gkq960 (  0) 0) |
| [15] |
DENG Niantao, GOH L K, WANG H, et al. A comprehensive survey of genomic alterations in gastric cancer reveals systematic patterns of molecular exclusivity and co-occurrence among distinct therapeutic targets[J]. Gut, 2012, 61(5): 673-684. DOI:10.1136/gutjnl-2011-301839 (  0) 0) |
| [16] |
MA Yanlei, QIN Huanlong, ZHENG QI, et al. Neoadjuvant chemotherapy with a combination of docetaxel, cisplatin, fluorouracil, and leucovorin in nonresectable advanced gastric cancer[J]. Medical Oncology, 2010, 27(4): 1089-1095. DOI:10.1007/s12032-009-9340-7 (  0) 0) |
| [17] |
CHEN Wanqing, ZHENG Rongshou, ZHANG Siwei, et al. Report of cancer incidence and mortality in China, 2010[J]. Annals of Translational Medicine, 2014, 2(7): 61. DOI:10.3978/j.issn.2305-5839.2014.04.05 (  0) 0) |
| [18] |
王婕敏, 林三仁. 胃癌研究及诊治新进展[J]. 胃肠病学和肝病学杂志, 2012, 21(1): 3-5. WANG Jiemin, LIN Sanren. Progress in the clinical research of gastric cancer[J]. Chinese Journal of gastroenterology and Hepatology, 2012, 21(1): 3-5. DOI:10.3969/j.issn.1006-5709.2012.01.002 (  0) 0) |
| [19] |
CHEN Wanqing, ZHENG Rongshou, BAADE P D, et al. Cancer stastistics in China, 2015[J]. CA: A Cancer Journal for Clinicians, 2016, 66(2): 115-132. DOI:10.3322/caac.21338 (  0) 0) |
| [20] |
高燕, 崔艳欣, 姜相君. Dnmt1基因单核苷酸多态性与胃癌的关系[J]. 中国癌症防治杂志, 2015, 7(6): 394-397. GAO Yan, CUI Yanxin, JIANG Xiangjun. Association of Dnmtl single-nucleotide polymorphisms and risk of gastric cancer[J]. Chinese Journal of Oncology Prevention and Treatment, 2015, 7(6): 394-397. DOI:10.3969/j.issn.1674-5671.2015.06.02 (  0) 0) |
| [21] |
熊伟, 余敏, 左绍远. 线粒体转录终止因子蛋白家族在线粒体基因表达中的调节作用[J]. 中国生物化学与分子生物学报, 2015, 31(3): 223-231. XIONG Wei, YU Min, ZUO Shaoyuan. Regulatory roles of mitochondrial transciption termination factor (MTERF) family proteins in mitochondrial gene expression[J]. Chinese Journal of Biochemistry and Molecular Biology, 2015, 31(3): 223-231. DOI:10.13865/j.cnki.cjbmb.2015.03.01 (  0) 0) |
| [22] |
ROBERTI M, POLOSA P L, BRUNI F, et al. MTERF factors: a multifunction protein family[J]. Biomol Concepts, 2010, 1(2): 215-224. DOI:10.1515/bmc.2010.015 (  0) 0) |
| [23] |
HYVÄRINEN A K, POHJOISMAKI J L, REYES A, et al. The mitochondrial transcription termination factor mTERF modulates replication pausing in human mitochondrial DNA[J]. Nucleic Acids Research, 2007, 35(19): 6458-6474. DOI:10.1093/nar/gkm676 (  0) 0) |
| [24] |
HYVÄRINEN A K, KUMANTO M K, MARJAVAARA S K, et al. Effect on mitochondrial transcription of manpulating mTERF protein levels in cultured human HEK293 cells[J]. BMC Molecular Biology, 2010, 11(9): 72-78. DOI:10.1186/1471-2199-11-72 (  0) 0) |
| [25] |
CHEN Guiyuan, DAI Jie, TAN Shirui, et al. MTERF1 regulates the oxidative phosphorylation activity and cell proliferation in HeLa cells[J]. Acta Biochimica Et Biophysica Sinica, 2014, 46(6): 512-521. DOI:10.1093/abbs/gmu029 (  0) 0) |
| [26] |
王进峰, 卢晓明, 王礼平, 等. 基于Oncomine芯片数据库荟萃分析碳酸苷酶Ⅸ在肾细胞癌中的表达及意义[J]. 现代泌尿生殖肿瘤杂志, 2015, 7(4): 231-235. WANG Jinfeng, LU Xiaoming, WANG liping, et al. Application of data-mining from Oncomine database in the research of renal cell carcinoma[J]. Journal of Contemporary Urologic and Reproductive Oncology, 2015, 7(4): 231-235. DOI:10.3870/j.issn.1674-4624.2015.04.011 (  0) 0) |
 2017, Vol. 15
2017, Vol. 15


