2. 北京北科德源生物医药科技有限公司,北京 100094
2. Beijing Beike Deyuan Bio-Pharm Technology Co., Ltd., Beijing 100094, China
创新药物研发是一个耗资巨大、周期漫长、风险极高的产业。据统计,平均每10 000个新化学实体(NCEs)中只有1个最终可能成为药物,并且整个过程要花费10~12年时间和12~20亿美元的投入[1]。导致药物在临床前和临床研究阶段研发失败的原因是多方面的,主要包括药效不明显、毒性及副作用等安全性问题、药代动力学性质不理想以及商业因素等。药代动力学性质是指化合物在体内的行为,包括吸收(adsorption)、分布(distribution)和清除(excretion)过程,简称ADME,与毒性(toxicity)合称ADMET性质。ADMET性质是衡量化合物成药性最重要的参考指标[2-3]。上世纪90年代之前,导致药物研发失败的主要因素中,ADME性质不理想所占的比例达到39%,随着人们加大了对药代动力学研究的重视和投入,这一比例逐渐下降到了10%以下。毒性和副作用等安全性因素所占的比例则从21%上升到接近30%,成为药效因素之外,导致药物研发失败的最主要原因[4]。遵循“Fail early, fail cheaply”(失败越早,损失越小)的原则,很多药物化学家提出在药物开发的早期,甚至在先导化合物发现阶段就应该进行化合物的ADMET性质评价,选择ADMET性质理想的化合物进行实验筛选,可从一定程度上缓解实验筛选的经济压力;同时,将本来是在药物研发过程后期才考虑的毒性和代谢问题,提前到先导化合物发现的前期阶段来完成,可以有效提高候选药物后期开发的成功率。
面对目前组合化学蛋白质组学和计算机药物筛选的快速发展,常规的生物试验方法(包括体外试验和体内试验)显得比较昂贵和滞后[5-6]。如何快速、廉价地在药物研发早期进行化合物ADMET性质预测成为各大制药公司和研究机构十分关心和需要迫切解决的问题。近年来,利用计算方法预测药物的ADMET性质引起了国内外科学家的广泛关注,这一方法的基本思想是根据已知分子的ADMET性质数据,通过机器学习和模式识别方法建立计算预测模型,从而预测未知分子的ADMET性质。与实验方法相比,使用计算机方法对化合物进行ADMET性质预测具有明显的优势[7-9].首先,计算机方法可以快速地对大批量化合物进行处理和预测,而且花费极低;其次,只要化合物结构已知,即使化合物尚未合成出来,也可以通过计算机模型预测该化合物的ADMET性质。
本文针对人体小肠吸收、血脑屏障透过、人体表观分布体积、清除率、致癌性、急性毒性、发育毒性、肝毒性、生殖毒性等28个ADMET相关的性质,使用机器学习方法分别建立预测模型,构建了一个覆盖重要的吸收、分布、代谢、排泄及毒性性质评价的化合物ADMET性质预测平台,供国内外研究者使用。
1 材料与方法 1.1 数据收集与整理高质量的实验数据,是保证ADMET预测模型构建成功的关键。本文采用文本挖掘和手工核对方法从文献报道和专业数据库中收集整理已有的化合物ADMET性质实验数据。通过文献交叉参照等方式对收集到的数据进行汇总和去冗余、除错等处理。经过整理,得到超过11万条化合物ADMET相关的试验数据。保存的数据包括化合物分子的名称、ADMET相关的性质、实验测试方法和材料、分子结构信息、数据来源(原始文献来源)等。这里收录了28种ADMET相关的性质端点(endpoint),包括人体小肠吸收[10]、P-糖蛋白抑制剂和底物[11-12]、血脑屏障通透性[10, 13]、人体表观分布体积[14]、血浆蛋白结合率[15]、CYP450底物和抑制剂(CYP1A1、1A2、2C9、2C19、2D6和3A4)[16-18]、肾清除率[14]、大鼠口服毒性[19-20]、AMES致突变毒性[21]、慢性毒性[22]、发育毒性[23]、肝毒性[24-26]、眼毒性[27]、生殖毒性[23]、hERG通道抑制性[28-29]、致癌性[30]、鸟毒性[31]、鱼毒性[32]和昆虫毒性[33]等。
1.2 模型构建在化合物ADMET性质预测模型构建过程中,需要选择适当的分子结构表征方式和建模方法。分子结构的表征通常采用分子描述符和分子指纹方式[34-35]。分子描述符是对分子物理化学性质的数学度量,如分子量、脂水分配系数、原子数目等。分子指纹则是根据分子片段描述分子,其实现方法主要是对分子结构进行切分,获得一个个的片段作为分子结构的表征。收集到的针对不同ADMET性质端点建立预测模型的文献中,大部分都明确给出了建模所用的分子描述符和分子指纹类型。
目前化合物ADMET预测研究中,机器学习是占据主导地位的建模方法。机器学习方法从观测数据(样本)出发寻找规律,利用这些规律对未来数据或无法预测的数据进行预测。其中诸如支持向量机(support vector machine, SVM)[36]、决策树(decision tree, DT)[37]、朴素贝叶斯(Naïve Bayes, NB) [38]、k-最近邻算法(k-Nearest Neighborhoods, kNN) [39]、人工神经网络(artificial neuron network, ANN)[40]等算法大量成功的应用实例证明了这些机器学习算法的有效性和实用性。在本文中,将支持向量机算法作为首选的建模方法。支持向量机是Vapnik等[41]1995年提出的一种机器学习方法,它在解决小样本、非线性以及高维模式识别问题中表现出许多特有的优势,能够推广应用到函数拟合等其他机器学习问题中。对于非线性问题的处理,SVM也可以通过松弛变量(slack variables)和核函数(Kernel Functions)实现。因此,SVM已经成为目前应用最广泛的QSAR建模方法之一。不同的核函数都有各自的关键参数,如RBF核函数的惩罚因子C和核参数γ等。本文使用台湾大学林智仁教授[42]开发的libsvm工具箱进行模型的复原与构建。
本文把收集到的针对不同ADMET性质端点建立预测模型的文献分为3类:使用支持向量机算法并提供关键参数的、使用支持向量机算法但未提供关键参数的以及未使用支持向量机算法建模的。对于第1类,本文直接按照文献中提供的参数复原模型;对于第2类,本文通过邮件向文献原作者请求提供主要参数,然后实现模型复原;对于第3类以及无法获取原作者使用的分子描述符的文献,本文使用文献中的化合物ADMET性质数据,通过支持向量机算法结合分子指纹方式与分子描述符方式,构建模型并进行验证。
这里以hERG通道抑制性为例,介绍使用libsvm进行参数选择与模型训练的过程。
经过文献调研,本文选择文献[29]中的数据做为训练集,用于模型构建;使用文献[28]中的数据作为验证集进行模型验证。这里将hERG抑制活性IC50≥10 μM的化合物归类为hERG抑制剂,IC50<1 μM的化合物归类为非抑制剂。所有数据经过去重复、去除混合物、去除无机物等预处理,最终训练集剩余962个抑制剂和966个非抑制剂,测试集剩余抑制剂与非抑制剂数量分别为235个和363个。
本文采用一种常见的分子指纹MACCS keys(MACCS)表征分子结构。MACCS分子指纹包含了166个结构片段,这种分子指纹可以使用PaDEL-Descriptor软件计算得到[43]。模型的构建采用的是libsvm工具箱,核函数采用RBF核函数。如上所述,使用RBF核函数需要确定两个关键参数惩罚因子C和核参数γ。通过libsvm自带的网格搜索算法结合五折交叉验证方法对这两个关键参数进行了优化,得到最优参数组合为(3.030 0,0. 018 8)。使用这一参数组合训练得到的模型,对训练集进行五折交叉验证,得到整体预测准确率为85.94%,敏感率与特异性分别为85.14%、86.75%。使用验证集进行验证,得到的整体预测准确率、敏感率与特异性分别为82.61%、80.85%、83.75%,表现出很好的预测能力。
2 结果与讨论 2.1 ADMET性质预测模型本文复原或者构建了25个分类模型(其中11个根据原始文献提供的参数复原的模型和14个自建模型)和3个回归模型(其中2个根据原始文献提供的参数复原的模型和1个自建模型),用于预测化合物的28种ADMET相关的性质。经过外部数据集的验证,分类模型的整体预测准确率都在75%以上,其中大部分(19个)模型的整体预测准确率超过了80%;3个回归模型中,两个模型的决定系数R2超过了0.7(见表 1)。根据原始文献复原的模型,预测能力已经得到了认可,本文自建的模型也都表现出了较高的预测能力,这样就可以保证化合物ADMET性质预测结果的准确性。
| 表 1 化合物ADMET性质预测模型评价指标 Table 1 Summary of the models integrated in the ADMET prediction platform |
基于分布式虚拟筛选架构,结合高性能计算集群优势,本文搭建了化合物ADMET性质预测平台(http://www.vslead.com/?r=admet/index),为研究者提供在线的化合物ADMET性质预测服务。平台界面简洁友好,操作简单方便。图 1(a)展示了平台的任务提交界面。
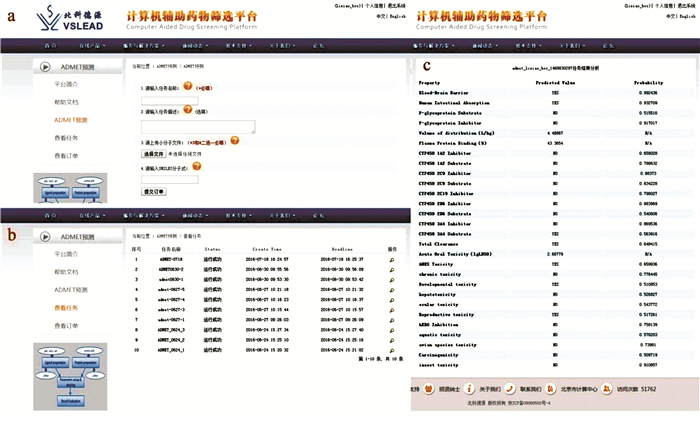
|
图 1 ADMET性质预测平台页面截图 Figure 1 A screenshot of the platform for estimation of chemical ADMET properties 注:(a)平台任务提交界面;(b)平台任务查看界面;(c)化合物ADMET性质预测结果展示页面。 |
用户进行化合物ADMET性质预测的主要步骤包括:1)输入任务名称,用户可任意拟定一个名称作为本次ADMET预测的任务名称;2)输入任务描述,简要描述项目研究内容,帮助用户备注筛选任务细节;3)上传小分子文件或者输入SMILES分子式,平台目前支持3种小分子格式文件, 即mol2、sdf和smi,也支持SMILES分子式输入,用户可以自由选择上传小分子文件或者输入SMILES分子式的方式提交化合物结构;4)任务提交,用户完成上述步骤后,可以点击下方的“提交订单”按钮,提交本次化合物ADMET性质预测任务。点击页面中的问号图标可以显示每一步骤的说明。用户提交任务并完成支付后,就可以在“查看任务”板块中看到自己提交的任务(如图 1(b)所示),点击任务名称可以显示任务详情,点击“操作”栏下的“查看分析结果”按钮,可以查看本次提交的小分子化合物ADMET性质预测结果(如图 1(c)所示)。在平台使用过程中,用户也可以通过点击左侧的“平台简介”和“帮助文档”查看关于平台的基本情况介绍和使用方法。
本文搭建的化合物ADMET性质预测平台整合了28个模型,涵盖了目前比较重要的吸收、分布、代谢、排泄及毒性性质。研究者可以快捷方便地同时预测28种ADMET性质,在药物研发早期对候选化合物进行成药性评价和风险评估,有助于降低药物研发的失败率,节省研发时间和经费的投入。平台中各个模型,在构建过程中都采用了较全面的目前已知的ADMET性质实验数据,涵盖了较大的化学空间,在最大程度上保证了模型的应用域;平台中部分模型直接采用高质量文献中的建模数据和参数,自建模型也采用了目前最先进的建模方法,可以保证各模型具有很好的预测能力。
为了更方便为用户提供服务,平台目前只对注册用户开放使用。平台的支付系统是为将来对商业用户开放使用进行准备。当前状态下,使用本平台,支付页面实际需要付款金额为0元,因而是供用户免费使用的。
使用平台预测化合物ADMET性质具有一些限制条件:首先,用于ADMET性质预测模型构建的化合物都是小分子化合物,因此平台只适用于小分子化合物的ADMET性质预测,不支持大分子化合物;其次,用户提交的小分子必须是单一的有机化合物,平台不支持无机物、混合物、水合物、有机盐等化合物的ADMET性质预测;第三,大多数模型构建过程中,只考虑了化合物的二维结构特征,因此对于二维结构相同但三维构象不同的化合物,平台的预测结果没有区分。
另外需要说明的是,鉴于目前机器学习算法与可用数据的局限性以及化合物ADMET性质的复杂性,这里的化合物ADMET性质预测平台与其他计算机辅助系统一样,预测结果可以提供有效的辅助与参考作用,但并不能完全替代生物实验结果。对于化合物的ADMET性质,还是需要用户结合平台预测结果与人工专家经验进行综合判断,以助于提高判断的准确率。
3 结论1) 本文借助文本挖掘、机器学习等技术,构建了28种重要的ADMET性质预测模型,经过检验,模型都表现出了较高的预测能力,这样就可以保证化合物ADMET性质预测结果的准确性。
2) 在构建的机器学习模型基础上,本文基于分布式虚拟筛选架构,结合高性能计算集群优势,搭建了化合物ADMET性质预测平台(http://www.vslead.com/?r=admet/index)。平台界面简洁友好,操作简单方便。
3) 这一平台可以帮助研究者在药物研发早期阶段,快速准确地判断候选化合物的成药性和安全性,进而降低药物研发的失败率,节省研发时间和经费的投入。
| [1] |
PAUL S M, MYTELKA D S, DUNWIDDIE C T, et al. How to improve R & D productivity: the pharmaceutical industry's grand challenge[J]. Nature Reviews Drug Discovery, 2010, 9(3): 203-214. DOI:10.1038/nrd3078 (  0) 0) |
| [2] |
郭宗儒. 药物分子设计的策略:药理活性与成药性[J]. 药学学报, 2010, 45(5): 539-547. GUO Zongru. Strategy of molecular drug design: activity and druggability[J]. Acta Pharmaceutica Sinica, 2010, 45(5): 539-547. DOI:10.16438/j.0513-4870.2010.05.016 (  0) 0) |
| [3] |
李晓, 孔德信. 化合物成药性的预测方法[J]. 计算机与应用化学, 2012, 29(8): 999-1003. LI Xiao, KONG Dexin. Predicting ligand druggability for drug discovery[J]. Computers and Applied Chemistry, 2012, 29(8): 999-1003. (  0) 0) |
| [4] |
KOLA I, LANDIS J. Can the pharmaceutical industry reduce attrition rates?[J]. Nature Reviews Drug Discovery, 2004, 3(8): 711-716. DOI:10.1038/nrd1470 (  0) 0) |
| [5] |
MODI S, HUGHES M, GARROW A, et al. The value of in silico chemistry in the safety assessment of chemicals in the consumer goods and pharmaceutical industries[J]. Drug Discovery Today, 2012, 17(3/4): 135-142. DOI:10.1016/j.drudis.2011.10.022 (  0) 0) |
| [6] |
NIGSCH F, MACALUSO N J M, MITCHELL J B O, et al. Computational toxicology: an overview of the sources of data and of modelling methods[J]. Expert Opinion on Drug Metabolism & Toxicology, 2009, 5(1): 1-14. DOI:10.1517/17425250802660467 (  0) 0) |
| [7] |
CHENG Feixiong, LI Weihua, LIU Guixia, et al. In silico ADMET prediction: recent advances, current challenges and future trends[J]. Current Topics in Medicinal Chemistry, 2013, 13(11): 1273-1289. DOI:10.2174/15680266113139990033 (  0) 0) |
| [8] |
GLEESON M P, MODI S, BENDER A, et al. The challenges involved in modeling toxicity data in silico: a review[J]. Current Pharmaceutical Design, 2012, 18(9): 1266-1291. DOI:10.2174/138161212799436359 (  0) 0) |
| [9] |
MERLOT C. Computational toxicology-a tool for early safety evaluation[J]. Drug Discovery Today, 2010, 15(1/2): 16-22. DOI:10.1016/j.drudis.2009.09.010 (  0) 0) |
| [10] |
SHEN Jie, CHENG Feixiong, XU You, et al. Estimation of ADME properties with substructure pattern recognition[J]. Journal of Chemical Information and Modeling, 2010, 50(6): 1034-1041. DOI:10.1021/ci100104j (  0) 0) |
| [11] |
BROCCATELLI F, CAROSATI E, NERI A, et al. A novel approach for predicting P-glycoprotein (ABCB1) inhibition using molecular interaction fields[J]. Journal of Medicinal Chemistry, 2011, 54(6): 1740-1751. DOI:10.1021/jm101421d (  0) 0) |
| [12] |
WANG Zhi, CHEN Yuanying, LIANG Hu, et al. P-glycoprotein substrate models using support vector machines based on a comprehensive data set[J]. Journal of Chemical Information and Modeling, 2011, 51(6): 1447-1456. DOI:10.1021/ci2001583 (  0) 0) |
| [13] |
HOU T J, XU X J. ADME evaluation in drug discovery[J]. Journal of Chemical Information and Computer Science, 2003, 43(6): 2137-2152. DOI:10.1021/ci034134i (  0) 0) |
| [14] |
GOMBAR V K, HALL S D. Quantitative structure-activity relationship models of clinical pharmacokinetics: clearance and volume of distribution[J]. Journal of Chemical Information and Modeling, 2013, 53(4): 948-957. DOI:10.1021/ci400001u (  0) 0) |
| [15] |
ZHU Xiangwei, SEDYKH A, ZHU Hao, et al. The use of pseudo-equilibrium constant affords improved QSAR models of human plasma protein binding[J]. Pharmaceutical Research, 2013, 30(7): 1790-1798. DOI:10.1007/s11095-013-1023-6 (  0) 0) |
| [16] |
HAMMANN F, GUTMANN H, BAUMANN U, et al. Classification of cytochrome p(450) activities using machine learning methods[J]. Molecular Pharmaceutics, 2009, 6(6): 1920-1926. DOI:10.1021/mp900217x (  0) 0) |
| [17] |
CARBON-MANGELS M, HUTTER M C. Selecting relevant descriptors for classification by bayesian estimates: a comparison with decision trees and support vector machines approaches for disparate data sets[J]. Molecular Informatics, 2011, 30(10): 885-895. DOI:10.1002/minf.201100069 (  0) 0) |
| [18] |
CHENG Feixiong, YU Yue, SHEN Jie, et al. Classification of cytochrome P450 inhibitors and noninhibitors using combined classifiers[J]. Journal of Chemical Information and Modeling, 2011, 51(5): 996-1011. DOI:10.1021/ci200028n (  0) 0) |
| [19] |
ZHU Hao, MARTIN T M, YE Lin, et al. Quantitative structure-activity relationship modeling of rat acute toxicity by oral exposure[J]. Chemical Research in Toxicology, 2009, 22(12): 1913-1921. DOI:10.1021/tx900189p (  0) 0) |
| [20] |
LI Xiao, CHEN Lei, CHENG Feixiong, et al. In silico prediction of chemical acute oral toxicity using multi-classification methods[J]. Journal of Chemical Information and Modeling, 2014, 54(4): 1061-1069. DOI:10.1021/ci5000467 (  0) 0) |
| [21] |
XU Congying, CHENG Feixiong, CHEN Lei, et al. In silico prediction of chemical Ames mutagenicity[J]. Journal of Chemical Information and Modeling, 2012, 52(11): 2840-2847. DOI:10.1021/ci300400a (  0) 0) |
| [22] |
MAZZATORTA P, ESTEVEZ M D, COULET M, et al. Modeling oral rat chronic toxicity[J]. Journal of Chemical Information and Modeling, 2008, 48(10): 1949-1954. DOI:10.1021/ci8001974 (  0) 0) |
| [23] |
PLUNKETT L M, KAPLAN A M, BECKER R A. Challenges in using the ToxRefDB as a resource for toxicity prediction modeling[J]. Regulatory Toxicology and Pharmacology, 2015, 72(3): 610-614. DOI:10.1016/j.yrtph.2015.05.013 (  0) 0) |
| [24] |
FOURCHES D, BARNES J C, DAY N C, et al. Cheminformatics analysis of assertions mined from literature that describe drug-induced liver injury in different species[J]. Chemical Research in Toxicology, 2010, 23(1): 171-183. DOI:10.1021/tx900326k (  0) 0) |
| [25] |
LIEW C Y, LIM Y C, YAP C W. Mixed learning algorithms and features ensemble in hepatotoxicity prediction[J]. Journal of Computer-Aided Molecular Design, 2011, 25(9): 855-871. DOI:10.1007/s10822-011-9468-3 (  0) 0) |
| [26] |
HUANG S H, TUNG C W, FULOP F, et al. Developing a QSAR model for hepatotoxicity screening of the active compounds in traditional Chinese medicines[J]. Food and Chemical Toxicology, 2015, 78: 71-77. DOI:10.1016/j.fct.2015.01.020 (  0) 0) |
| [27] |
SOLIMEO R, ZHANG Jun, KIM M, et al. Predicting chemical ocular toxicity using a combinatorial QSAR approach[J]. Chemical Research in Toxicology, 2012, 25(12): 2763-2769. DOI:10.1021/tx300393v (  0) 0) |
| [28] |
DODDAREDDY M R, KLAASSE E C, SHAGUFTA, et al. Prospective validation of a comprehensive in silico hERG model and its applications to commercial compound and drug databases[J]. ChemMedChem, 2010, 5(5): 716-729. DOI:10.1002/cmdc.201000024 (  0) 0) |
| [29] |
CZODROWSKI P. hERG me out[J]. Journal of Chemical Information and Modeling, 2013, 53(9): 2240-2251. DOI:10.1021/ci400308z (  0) 0) |
| [30] |
LI Xiao, DU Zheng, WANG Jie, et al. In silico estimation of chemical carcinogenicity with binary and ternary classification methods[J]. Molecular Informatics, 2015, 34(4): 228-235. DOI:10.1002/minf.201400127 (  0) 0) |
| [31] |
ZHANG Cheng, CHENG Feixiong, SUN Lu, et al. In silico prediction of chemical toxicity on avian species using chemical category approaches[J]. Chemosphere, 2015, 122: 280-287. DOI:10.1016/j.chemosphere.2014.12.001 (  0) 0) |
| [32] |
SUN Lu, ZHANG Chen, CHEN Yingjie, et al. In silico prediction of chemical aquatic toxicity with chemical category approaches and substructural alerts[J]. Toxicology Researc, 2015, 4(2): 452-463. DOI:10.1039/C4TX00174E (  0) 0) |
| [33] |
SINGH K P, GUPTA S, BASANT N, et al. QSTR modeling for qualitative and quantitative toxicity predictions of diverse chemical pesticides in honey bee for regulatory purposes[J]. Chemical Research in Toxicology, 2014, 27(9): 1504-1515. DOI:10.1021/tx500100m (  0) 0) |
| [34] |
DONG Jie, CAO Dongsheng, MIAO Hongyu, et al. ChemDes: an integrated web-based platform for molecular descriptor and fingerprint computation[J]. Journal of Cheminformatics, 2015, 7(1): 1-10. DOI:10.1186/s13321-015-0109-z (  0) 0) |
| [35] |
LI Xiao, CHEN Lei, CHENG Feixiong, et al. In silico prediction of chemical acute oral toxicity using multi-classification methods[J]. Journal of Chemical Information and Modeling, 2014, 54(4): 1061-1069. DOI:10.1021/ci5000467 (  0) 0) |
| [36] |
NOBLE W S. What is a support vector machine?[J]. Nature Biotechnology, 2006, 24(12): 1565-1567. DOI:10.1038/nbt1206-1565 (  0) 0) |
| [37] |
QUINLAN J R. Induction of decision trees[J]. Machine Learning, 1986, 1(1): 81-106. DOI:10.1023/A:1022643204877 (  0) 0) |
| [38] |
FRANK E, TRIGG L, HOLMES G, et al. Technical note: naive bayes for regression[J]. Machine Learning, 2000, 41(1): 5-25. DOI:10.1023/A:1007670802811 (  0) 0) |
| [39] |
LI Lili, ZHANG Yanxia, ZHAO Yongheng. k-Nearest Neighbors for automated classification of celestial objects[J]. Science in China Series G: Physics, Mechanics and Astronomy, 2008, 51(7): 916-922. DOI:10.1007/s11433-008-0088-4 (  0) 0) |
| [40] |
OLAWOYIN R. Application of backpropagation artificial neural network prediction model for the PAH bioremediation of polluted soil[J]. Chemosphere, 2016, 161: 145-150. DOI:10.1016/j.chemosphere.2016.07.003 (  0) 0) |
| [41] |
CORTES C, VAPNIK V. Support-vector networks[J]. Machine Learning, 1995, 20(3): 273-297. DOI:10.1007/BF00994018 (  0) 0) |
| [42] |
CHANG C C, LIN C J. LIBSVM: a library for support vector machines[J]. ACM Transactions on Intelligent Systems and Technology, 2011, 2(3): 1-27. DOI:10.1145/1961189.1961199 (  0) 0) |
| [43] |
YAP C W. PaDEL-descriptor: an open source software to calculate molecular descriptors and fingerprints[J]. Journal of Computational Chemistry, 2011, 32(7): 1466-1474. DOI:10.1002/jcc.21707 (  0) 0) |
 2017, Vol. 15
2017, Vol. 15


