真核生物染色质会通过核小体与DNA链的多重缠绕折叠形成紧密的三维高级结构[1-2],这种结构在基因表达中远程调节的物理实现上起到了关键作用[3-5]。胚胎干细胞(ES cells)是早期胚胎的原始细胞,具有分化为三胚层各种细胞[6]的潜在能力。在干细胞分化过程中,细胞核内染色质的三维结构会发生相应的构象变化[7]。在哺乳动物[8]中通过表达某一基因能够激活其他成体细胞特征性基因,致使细胞命运发生改变,而染色质三维结构变化可对这一过程进行调控。研究干细胞与其衍生细胞的染色质结构差异可以更深地了解这种调控关系。
利用Hi-C技术[9],研究者可以高通量地获得染色质高级结构中相互作用的DNA片段。通过检测这些DNA片段在染色质上的分布,可以绘制染色质相互作用图谱[10],此图谱展示了染色质上各位点间的相互作用情况。然而,除了存在单个位点仅与单个位点作用的情况外,还存在单个位点与多个位点作用以及多个位点与多个位点相互作用的情况(图 1),本文将这种位点称为染色质多重相互作用位点(chromosome multiple interaction sites,CMI sites)。这些位点可能涉及某些复杂的生物学过程,甚至是其中的关键节点。

|
图 1 CMI位点示意 Figure 1 An illustration of CMI site (red oval) 注:细胞中染色质(线条)通过DNA位点的接触来相互作用,包括发生在两个DNA片段间的作用(绿色椭圆),和发生在多个DNA片段间的作用(红色椭圆)。 |
如果将CMI位点所涵盖的范围称之为CMI区域,那么染色质上其他区域则可称为非CMI区域。比较不同细胞系中这两种区域内的表观遗传修饰富集情况、基因数目及其表达情况可以粗略的反映出CMI位点与细胞分化调控的关联性。目前,尚未见此类分析,本文通过分析Hi-C数据,系统分析了染色质多重相互作用的特征以及在细胞分化中的变化。
1 数据和方法 1.1 数据本文利用文献[11]提供的Hi-C Reads回帖后的数据,包括人类ES细胞系以及中内胚层细胞(ME cells)、间充质干细胞(MS cells)、神经祖细胞(NP cells)、滋养层细胞(TR cells)等4种ES衍生细胞系,获取存在多次相互作用的位点。同时,结合mRNA和人类参考基因组数据,以及组蛋白甲基化与乙酰化、CTCF、DNA甲基化(DNA Meth)、DNaseI高敏感位点(DHS)等表观遗传信息,本文进行了进一步的分析。所有数据的GEO登录号为GSE52457。
1.2 方法首先统计各细胞系中相同位置Hi-C Reads的出现频率,频率超过2就意味着此位点在Hi-C数据中重复出现,即视为CMI位点。然后进一步统计CMI位点数目与其作用频率,可得到CMI位点在所有Hi-C位点中的比例以及同一作用频率位点的数目。最后,通过汇总CMI位点在染色质中的位置信息,计算CMI区域所涵盖的范围(以bp为单位)。通过将CMI位点匹配到人类基因组上,计算与CMI区域有接触的基因数目。
结合RNA-Seq数据,进一步计算各基因的FPKM值,从而反映基因的表达水平。本文取整个细胞系中FPKM值的中位数为标准,来判定某个基因是否高表达,FPKM值高于中位数的基因被认为是高表达的基因,FPKM值低于中位数的基因则被认为是低表达的基因。
以多个分辨率(10~100 kbp,步长为10 kbp)计算CMI区域和所有相互作用区域上表观遗传修饰reads数目,即两区域中表观遗传修饰发生频率的总和,两者之间的比率作为CMI区域发生表观遗传修饰的相对概率。然后,计算各个CMI位点的Hi-C作用频率以及染色质各位点上组蛋白修饰的频率,对染色质上非CMI位点以及未发生组蛋白修饰位点的作用频率记为0,进而对两组数据进行相关(皮尔森相关系数),并在不同分辨率下求平均相关性系数,以此反映CMI频率与组蛋白修饰频率的相关性强弱。
对比ES细胞系与其4种衍生细胞系的CMI位点数目、CMI区域相关基因数目及表达量、CMI区域相关表观遗传修饰数目、CMI与组蛋白乙酰化与甲基化的相关性系数,从而比较细胞分化过程中人类ES细胞与其分化后细胞的差异。
2 结果与讨论 2.1 染色质多重相互作用(CMI)的数目及分布CMI位点的统计结果表明其占所有相互作用位点数目的近30% (图 2(a)),单点作用频率最高达500次,所有CMI位点遍布整个染色质。可以看出CMI位点在所有染色质相互作用中占据了一定的重要性。其中ES细胞系的所有相互作用位点总数以及CMI位点数均明显多于其他4种衍生细胞系(见表 1)。同时,不同作用频率的CMI位点的出现次数也会随着作用频率的增加而衰减,且ES细胞具有更多的复杂相互作用(图 2(b))。这说明CMI会随着细胞分化而变化,但总体分布的趋势相对保守。

|
图 2 CMI位点数目与频率 Figure 2 The frequency and count of CMI sites |
| 表 1 CMI位点数目统计 Table 1 Count of CMI sites |
通过统计所有CMI位点及Hi-C位点占据的染色质区域,本文发现CMI区域约占所有相互作用区域的15%,但是与人类基因的匹配结果显示其与近70%的基因有接触。这表明CMI可能参与了许多复杂的生物学过程。
值得注意的是,通过计算各个细胞系中不同区域的基因平均FPKM表达量,发现CMI区域中基因表达量明显高于非CMI区域(图 3(a))。进一步计算高表达与低表达基因数目在两区域中的比例,发现CMI区域的高表达基因明显多于低表达基因,而非CMI区域的高表达基因则明显少于低表达基因,并且,CMI区域高表达基因显著的多于非CMI区域(图 3(b))。可以推测,与CMI区域相关联的基因倾向于高表达,与非CMI区域相关联的基因倾向于低表达,但是这种倾向性随着细胞分化有减弱的趋势。上述结果说明,在干细胞中,有更多的CMI形成,且随分化而逐渐减弱,暗示CMI是干细胞的标识性结构。

|
图 3 CMI区域基因表达情况 Figure 3 Gene expression in CMI regions |
通过比较CMI区域与所有染色质相互作用区域表观遗传修饰发生的频率,发现仅占染色质相互作用区域15%的CMI区域发生表观遗传修饰的相对概率能达到30%~60%,表明表观遗传修饰在CMI区域存在富集现象。但是ES细胞系中CMI区域发生表观遗传修饰的相对概率明显大于其他4种衍生细胞系(图 4(a)),这表明干细胞分化可能会导致这种富集效应减弱。为了进一步研究CMI区域与表观遗传修饰之间的关系,将CMI位点的相互作用频率与表观遗传修饰的发生频率进行相关性分析,发现CMI的作用频率与某些表观遗传修饰发生频率具有较强相关性(相关性系数介于0.6~0.8之间),也有一些具有弱相关性(相关性系数介于0.4~0.6之间),不同细胞系之间的频率相关性(以下简称相关性)具有显著差异(图 4(b))。
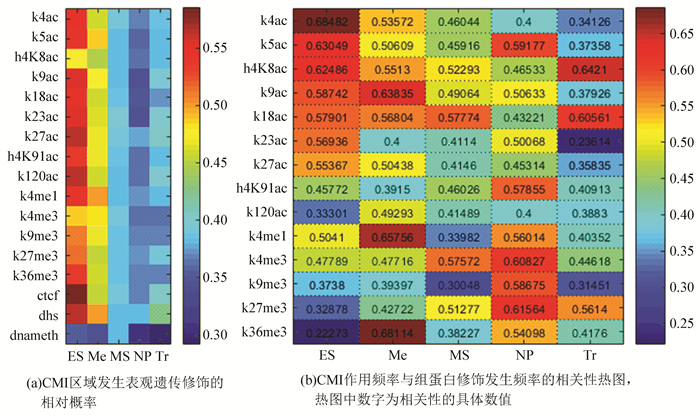
|
图 4 各细胞系中CMI区域的表观遗传修饰情况 Figure 4 Epigenetic modifications in CMI regions of cell lines |
ES细胞系中CMI整体与组蛋白乙酰化的相关性较强,而与组蛋白甲基化的相关性较弱,比如与H3K23ac、H3K4ac、H2BK120ac的相关性均大于0.6,而与组蛋白甲基化相关性最大仅0.5,与其他组蛋白甲基化更是几乎不相关(相关性系数低于0.4);Me细胞系与H3K4甲基化的相关性相比于ES细胞系显著增加,与其他乙酰化相关性均有下降,与组蛋白乙酰化相关性明显增强;MS细胞系与组蛋白修饰的相关性整体明显下降;NP细胞系与组蛋白甲基化的相关性显著上升,同时与组蛋白乙酰化相关性显著增强;TR细胞系与组蛋白乙酰化的相关性急剧减弱,整体变化比较复杂。如果H3K27ac是增强子的重要标志,那么该结果表明此类Enhancer在空间上可能和多个染色质的区域靠近并调控相关基因[12-13]。
总体来看,随着分化过程中CMI区域在所有相互作用区域中表观遗传修饰的占比减少,虽然4种ES衍生细胞系与组蛋白乙酰化的相关性相对于ES细胞系趋于弱化,但是与组蛋白甲基化的相关性趋于强化,其中强化最明显的是与H3K4me3的相关性。可以推测,干细胞分化过程中与组蛋白乙酰化相关的CMI位点趋于减少,而与组蛋白甲基化相关的CMI位点趋于增多,从而共同维护了染色质相互作用中CMI位点与非CMI位点以及区域范围大小的动态平衡。H3K4me3和H3K27me3的双重修饰是分化基因的重要标志[14-15],本文结果表明分化中这两种甲基化修饰在CMI区域中增强(图 4(b)),这说明分化过程中H3K4me3和H3K27me3的双重修饰可能凭借多重作用结构,一次调控多个区域的基因转录。
3 结论本文基于Hi-C数据,系统分析了一类特殊的染色质高级结构——染色质多重相互作用的特征。结果表明:染色质多重相互作用在所有染色质高级结构中占有较高的比例(约30%),而且此类结构关联的基因占所有基因的70%。染色质多重相互作用富集组蛋白修饰,具有高乙酰化的特征,位于此类区域的高表达基因比例较大。在干细胞分化期间,染色质多重相互作用数量和比例有变小的趋势,H3K27ac的修饰有减弱的趋势,而甲基化却有增加的趋势(H3K4me3和H3K27me)。推测这些修饰可能凭借多重作用结构,一次调控多个区域的基因转录。总之,染色质多重相互作用在干细胞分化过程中动态变化,具有重要的基因转录调节作用。
| [1] |
HU Ming, DENG Ke, QIN Zhaohui, et al. Understanding spatial organizations of chromosomes via statistical analysis of Hi-C data[J]. Quantitative Biology, 2013, 1(2): 156-174. DOI:10.1007/s40484-013-0016-0 (  0) 0) |
| [2] |
MIELE A, DEKKER J. Long-range chromosomal interactions and gene regulation[J]. Molecular BioSystems, 2008, 4(11): 1046-1057. DOI:10.1039/b803580f (  0) 0) |
| [3] |
GORKIN D U, LEUNG D, REN Bing. The 3D genome in transcriptional regulation and pluripotency[J]. Cell Stem Cell, 2014, 14(6): 762-775. DOI:10.1016/j.stem.2014.05.017 (  0) 0) |
| [4] |
LI Guoliang, RUAN Xiaoan, AUERBACH R K, et al. Extensive promoter-centered chromatin interactions provide a topological basis for transcription regulation[J]. Cell, 2012, 148(1/2): 84-98. DOI:10.1016/j.cell.2011.12.014 (  0) 0) |
| [5] |
HANDOKO L, XU Han, LI Guoliang, et al. CTCF-mediated functional chromatin interactome in pluripotent cells[J]. Nature Genetics, 2011, 43(7): 630-638. DOI:10.1038/ng.857 (  0) 0) |
| [6] |
THOMSON J A, ITSKOVITZ-ELDOR J, SHAPIRO S S, et al. Embryonic stem cell lines derived from human blastocysts[J]. Science, 1998, 282(5391): 1145-1147. DOI:10.1126/science.282.5391.1145 (  0) 0) |
| [7] |
JOFFE B, LEONHARDT H, SOLOVEI I. Differentiation and large scale spatial organization of the genome[J]. Current Opinion in Genetics & Development, 2010, 20(5): 562-569. DOI:10.1016/j.gde.2010.05.009 (  0) 0) |
| [8] |
DAVIS R L, WEINTRAUB H, LASSAR A B. Expression of a single transfected cDNA converts fibroblasts to myoblasts[J]. Cell, 1987, 51(6): 987-1000. DOI:10.1016/0092-8674(87)90585-X (  0) 0) |
| [9] |
LIEBERMAN-AIDEN E, van BERKUM N L, WILLIAMS L, et al. Comprehensive mapping of long-range interactions reveals folding principles of the Human genome[J]. Science, 2009, 326(5950): 289-293. DOI:10.1126/science.1181369 (  0) 0) |
| [10] |
DEKKER J, MARTI-RENOM M A, MIRNY L A. Exploring the three-dimensional organization of genomes: interpreting chromatin interaction data[J]. Nature Reviews Genetics, 2013, 14(6): 390-403. DOI:10.1038/nrg3454 (  0) 0) |
| [11] |
DIXON J R, JUNG I, SELVARAJ S, et al. Chromatin architecture reorganization during stem cell differentiation[J]. Nature, 2015, 518(7539): 331-336. DOI:10.1038/nature14222 (  0) 0) |
| [12] |
ZHU Yun, SUN Lin, CHEN Zhao, et al. Predicting enhancer transcription and activity from chromatin modifications[J]. Nucleic Acids Research, 2013, 41(22): 10032-10043. DOI:10.1093/nar/gkt826 (  0) 0) |
| [13] |
CREYGHTON M P, CHENG A W, WELSTEAD G G, et al. Histone H3K27ac separates active from poised enhancers and predicts developmental state[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(50): 21931-21936. DOI:10.1073/pnas.1016071107 (  0) 0) |
| [14] |
BOGLIOTTI Y S, ROSS P J. Mechanisms of histone H3 lysine 27 trimethylation remodeling during early mammalian development[J]. Epigenetics, 2012, 7(9): 976-981. DOI:10.4161/epi.21615 (  0) 0) |
| [15] |
LIU Xiaoyu, WANG Chenfei, LIU Wenqiang, et al. Distinct features of H3K4me3 and H3K27me3 chromatin domains in pre-implantation embryos[J]. Nature, 2016, 537(7621): 558-562. DOI:10.1038/nature19362 (  0) 0) |
 2017, Vol. 15
2017, Vol. 15


