2. 辽宁大学 生命学院, 沈阳 110036;
3. 辽宁大学 轻型产业学院, 沈阳 110036
2. School of Life Science, Liaoning University, Shenyang 110036, China;
3. College of Light Industry, Liaoning University, Shenyang 110036, China
Hsp40在原核细胞和真核细胞中广泛存在,它作为Hsp70的辅助伴侣可以与Hsp70共同参与多种重要的生命活动,如非天然多肽的折叠、多聚体的组装、细胞器蛋白的转运及错误折叠蛋白的降解等[1-2]。Ydj1p是Hsp40分子伴侣家族成员之一,通过调节Hsp70的ATP酶活性结合展开多肽促进蛋白正确折叠。Ydj1p也可单独发挥其生物学功能,能够识别并结合错误折叠的蛋白使其重新展开,起到抑制蛋白聚集的作用,Hsp40重新展开的多肽片段传递给Hsp70折叠成有生物活性的蛋白质[3-4]。
锌指结构是由若干保守的氨基酸残基同锌离子结合, 形成相对独立的“指”状四面体结构, 这一结构最早由Miller等[5]1985年在非洲爪蟾转录因子TFⅢA中发现。锌指结构域含有两个锌离子结合位点(ZBDⅠ和ZBDⅡ)与半胱氨酸形成配位键维持结构稳定。生化实验表明,Ydj1p锌指结构有助于Hsp70捕获多肽底物,锌指Ⅰ和锌指Ⅱ突变不改变Ydj1p结合化学变性荧光素酶的能力。因此蛋白折叠的缺陷可能是在Ydj1p结合多肽之后,ZFLR的功能可能是帮助多肽从Ydj1p到Hsp70的传递[6],这个发现与之前报道一致。蛋白折叠依赖于锌指Ⅰ和锌指Ⅱ的原因之一是Hsp70需要它们来转移非本地多肽。
锌指ⅠC143S和锌指ⅡC162S突变,失去刺激Hsp70与底物结合的能力。锌指Ⅱ突变呈现温度敏感型生长缺陷,但是锌指Ⅰ突变体没有这种现象,热激条件下C143S有功能与Hsp70结合抑制荧光素酶聚集而且与野生型相似,C143S有部分生物学功能可以保护高温下的细胞[7]。相反C162S不能抑制荧光素酶聚集,C162S在高温下不能生长,原因是高温使蛋白变性。因此,锌指Ⅱ对于酵母高温下生存是至关重要的,与Hsp70合作抑制热压力导致的蛋白聚集[8]。这一现象反映了锌指结构Ⅰ和Ⅱ在作为分子伴侣功能上的差异,但是从分子水平无法解释产生这一现象的原因。所以本文通过分子动力学手段模拟Ydj1p以及各突变体,对模拟后的数据进行进一步分析找到不同突变体差异的原因[9-10]。
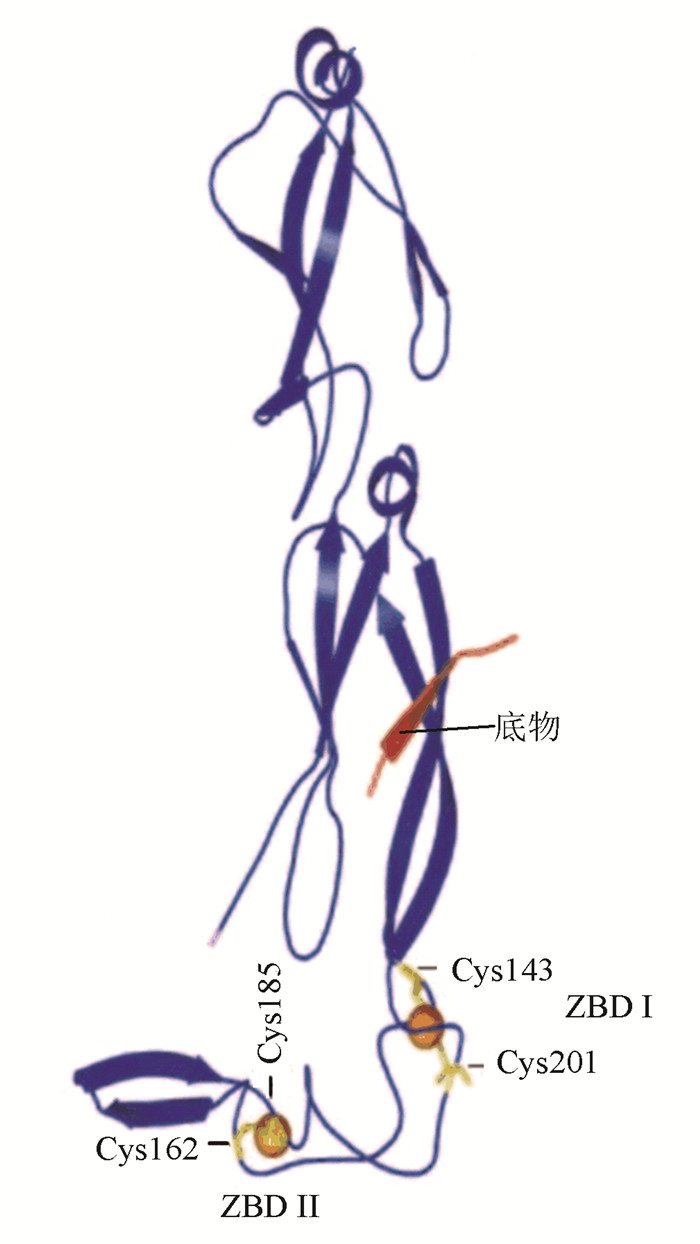
1 计算方法与条件 1.1 突变体模型构建采用RCSB蛋白数据库中酵母Ydj1p部分晶体结构(PDB:1NLT)为模型,通过Swiss-Pdb突变软件以上述模型为模板构建突变体C143S、C162S、C185S、C201S模型如图 1所示。
|
图 1 Ydj1p结构图及锌指一区域Cys-201, Cys-143和锌指二区域Cys-162, Cys-185局部图 Figure 1 The structure of Ydj1p and the side chains for Cys-201 and Cys-143 in zinc-binding domain (ZBD) Ⅰ and Cys-162 and Cys-185 in ZBDⅡ are depicted |
利用分子动力学模拟软件Gromacs 4.5.5和三维结构分析软件PyMOL进行。各模型的模拟步骤及参数设定均相同, 具体步骤及参数如下.
1) 力场选择。根据不同力场偏向于不同的结构状态模拟,该模拟各体系均采用NPT系综,计算力场选用GROMOS96 43a1[11]。
2) 溶解蛋白。将结构模型放进一个立方体的周期性边界盒子中并置于该盒子的中心,设定模型中任一原子距盒子边缘的距离大于1.0 nm, 以保证其不会和另一个盒子中的镜像相互作用。水溶液环境采用SPC模型水分子(约含25 169个水分子)。
3) 平衡电荷。正常的生理条件下,体系的pH应接近中性[12-13],总电荷为0。向各溶液体系中加入不同数量的Na+或Cl-,保持体系pH=7,总电荷为0。
4) 能量最小化。为了消除可能的原子碰撞,根据蛋白质折叠的热力学,先将各模型在真空中以最陡下降法(Steep)优化2 000步的能量使其达到结构稳定状态[14]。
5) 限制性模拟。根据固定主链,优化侧链的原则,为了使侧链找到一个较稳定的状态避免出现溶剂分子分布不均的问题,进行了80×10-12s的限制性模拟。
6) 最终动力学模拟。达到上述平衡之后,各体系在恒定的温度和压力下进行10×10-9 s的分子动力学模拟。分子的运动轨迹每4×10-12 s收集一次,以便用于后续的数据分析[15-17]。
此外,为了保持整个模拟过程中体系温度稳定在300 K、压力稳定在1.0×105 Pa,分别采用“V-rescale”和“Parrinello-Rahman”[18]方法对体系温度和压力进行控制, 耦合常数分别为0.1×10-12 s和0.5×10-12 s。模拟的时间积分步长为2×10-15 s, 整个体系使用周期性边界条件,采用LINCS[18](linear constraint solver)算法对体系中所有键的键长进行约束,静电相互作用采用PME(Particle mesh Ewald)算法进行估算。
1.3 拉伸分子动力学模拟取模拟后的平衡结构作为拉伸动力学的初始结构,采用GROMACS恒速拉伸动力学算法进行计算。拉伸过程中采用的弹簧系数为300 kJ/(mol·nm),拉伸速度为0.002 nm/s,拉伸方向为Y轴方向。拉伸过程中底物七肽沿着结合口袋延伸的方向拉出。分子的运动轨迹每0.1×10-12 s保持一次,拉伸时间为2×10-9 s[19-20]。
1.4 模拟结果分析软件通过GROMACS软件包内置模块完成,所有的三维结构图像通过Pymol软件呈现。均方根偏差(root mean square deviations,RMSD)计算了蛋白质所有Cα在模拟过程中的偏移程度。氢键估算键长为0.35 nm,键角为300°。疏水相互作用的强弱使用Ligplot+软件计算[23]。
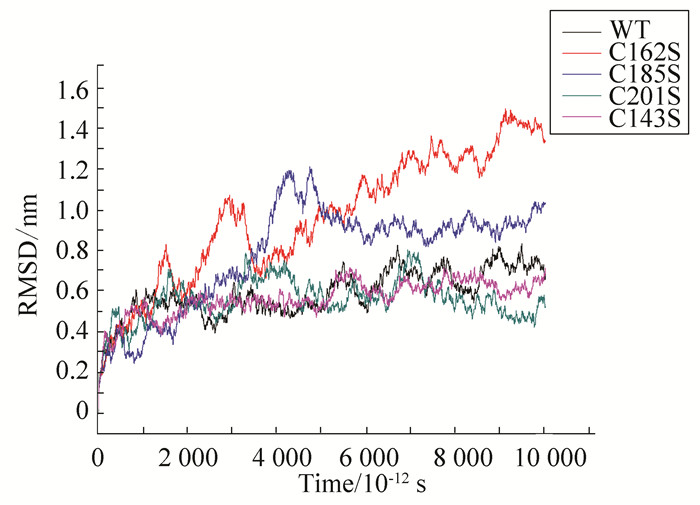
2 结果分析 2.1 突变体稳定性分析差异均方根(root mean square deviation,RMSD)值可用来测量进行比对的两个蛋白质相同位点的氨基酸残基的碳原子之间的距离,它是用来衡量模拟过程中蛋白是否达到稳定结构的重要参数。该值的大小直接反映了不同时刻蛋白构象与初始构象的差异程度[27-30]。当RMSD值趋于恒定时可以认为该蛋白构象稳定,相对值越低的RMSD对应的蛋白质结构越稳定。如图 2所示,ZBDⅠ突变体C143S、C201S的RMSD值与野生型相似,而ZBDⅡ突变体C162S、C185S的RMSD值与野生型相比略高,在模拟了7×10-9 s之后各突变蛋白基本达到稳定。由数据结果可知,C143S、C201S突变体在模拟过程中结构变化不大,而C162S、C185S结构变化较大,一方面,ZBDⅠ位于CTDI和ZFLR之间,ZBDⅠ单点突变有增加结构的灵活性的可能性,但可能不显著影响整体ZFLR结构。ZBDⅡ位于在ZFLR的中间,由氨基包围具有相对高的B-因子酸,根据数据推测ZBDⅠ结构可能比ZBDⅡ结构更稳定。
|
图 2 模拟过程中WT和突变体RMSD随时间的变化* Figure 2 Time dependence of RMSD values of WT and Mutants *彩图见电子版(http://swxxx.alljournals.cn/ch/index.aspx)(2017年第2期doi:10.3969/j.issn.1672-5565.20160715001)。 |
另一方面,生化试验表明通过对Ydj1p锌指区域突变体分析,其无法支持酵母在高温下生长,当温度升高到非酵母正常生长温度后,锌指结构突变使抑制荧光素酶聚集的能力缺失,ydj1p锌指Ⅱ突变体在37°时生长缓慢,到达43°时不能生存[21]。纯化的ydj1p锌指Ⅱ突变体不能与Hsp70合作抑止蛋白折叠,故本文中C162S不能支持酵母在非正常条件下生长,锌指Ⅱ功能的损坏,阻止Hsp40与Hsp70合作去抑制热激条件下的蛋白质损伤。相反,ZBDⅠ突变体C143S能部分行使功能与Hsp70合作抑制蛋白质聚集,在热激条件下保护蛋白。通过RMSD分析,模拟7×10-9 s后C162S最不稳定,其次是C185S,都位于ZBDⅡ,C143S和C201S平衡后波动较平稳。也证实了ZBDⅡ构象的改变对整个锌指区域构象造成了大的影响,因此,该ZBDⅡ的突变是有可能破坏ZFLR整体结构并使其不起作用的。Hsp40,Hsp70与底物共同存在,并且ATP提供能量时,底物能进行正确折叠。而当仅含有Hsp70或Hsp40时,底物错误聚集都在80%以上。Hsp40锌指区域进行突变后,ZBDⅠ突变体C143S使Hsp70与底物的结合率降低至35%,底物的正常折叠可达到75%,ZBDⅡ突变体C162S使Hsp70与底物的结合率降低至25%,底物80%发生错误聚集;上述数据说明:在一定热激或者化学条件改变下,当Hsp40锌指区域发生突变后,突变体严重影响底物与Hsp70的传递,使Hsp70与底物结合率降低,Hsp70不能帮助底物应对热激或者化学条件的改变,而进行错误的折叠组装。
2.2 RMSF分析均方根涨落(root mean square fluctuation,RMSF)是蛋白体系最终原子位置同参考位值的比较,它可以通过蛋白中各个氨基酸相对于平均结构的位置偏差,来计算蛋白质二级结构变化剧烈程度[26]。为了分析局部残基的构象变化,本文分析了WT和突变体所有氨基酸残基Cα原子的均方根位移(Root-mean square fluctuations,RMSF)。如表 1所示,与野生型相比,C162S、C185S的ZBDⅡ和ZBDⅠ区域均发生明显偏移,而C143S、C201S的ZBDⅡ和ZBDⅠ区域偏移较小,其他结构没有发生明显的偏移。分析可知各突变体锌指结构都有不同程度的变化,因此,影响蛋白结构稳定性的原因可能是蛋白突变后锌指结构的变化,为了进一步证实这一推测,对突变后的蛋白与野生型进行结构比对。
| 表 1 模拟过程中WT和突变体的RMSF平均值 Table 1 The average RMSF of WT and Mutants during MDs |
由表 1可以看出,锌指Ⅰ点突变体的平均RMSF比野生型降低,锌指Ⅱ点突变体的平均RMSF比野生型升高,并且所有模型中锌指Ⅱ部分的平均RMSF均比锌指Ⅰ部分的平均RMSF大,说明锌指Ⅱ部分结构更易受到影响,在其部分的突变可能对Ydj1功能有更大的影响。
2.3 Ydj1p及各突变体与多肽底物的静电能量和范德华能量分析分子动力学模拟的方法分析相互作用力时,通常以疏水和静电作用为主来判断结合能力的强弱[27-28],为了定量分析底物与Ydj1p之间相互作用能量的变化,本文对Ydj1p野生型和各突变体与多肽底物进行了拉伸动力学模拟。由表 2可知,Ydj1p突变体的氢键数量以及疏水数量略少于野生型,C143S,C162S较为明显,降低约10%,其C143S较C162S降低的更多,说明突变后与底物结合能力稍差,稍许影响结合。而C201S,C185S的疏水以及氢键数量均与野生型相似,底物结合能力不发生改变,这与之前生化报道的数据一致[21]。
| 表 2 WT和不同突变体对底物结合的影响 Table 2 The effect of WT and Mutants in substrates combination |
当与底物结合时,只含有Ydj1p野生型和各突变体的多肽底物,不含Hsp70时,底物发生错误聚集;当Hsp70,Ydj1p野生型与多肽底物结合后,底物进行正常折叠[29]。相比之下,当Hsp70,Ydj1p各突变体与多肽底物结合后,Ydj1p锌指Ⅰ突变体C143S与底物结合后,正确折叠相较ydj1p野生型与底物结合的正确折叠率略低,而ydj1p锌指Ⅱ区域突变体C162S与底物结合后,严重影响底物的正确折叠,结合上述结论,突变后并未大幅度影响与底物的结合能力,即可推测出Ydj1p C162S突变后影响底物与Hsp70之间的传递,从而影响底物的正确折叠。拉伸数据如图 3所示,能量分析与前面的疏水数量和氢键数量的分析结果相符,由表 2可知,从能量角度来看,C143S,C162S的静电能量以及范德华能量都略少野生型,C185S和C201S的静电能量与野生型相似,范德华能量略高于野生型。C185S,C201S突变体与底物之间的能量没有太大变化,对底物结合能力的影响也不大。而C143S,C162S能量降低,这与之前的文献报道一致[21]。进而推测出ZBDⅠ和ZBDⅡ的突变C143S和C162S是对底物与Hsp70之间的传递产生了一定的影响。
|
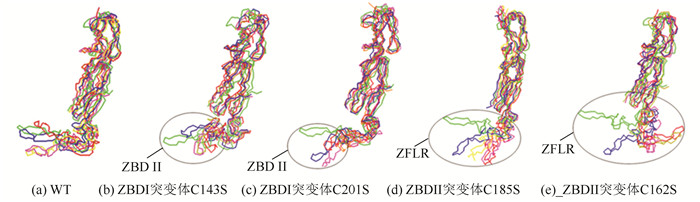
图 3 WT及各变异体锌指模拟不同时刻的结构变化图 Figure 3 The construction change of the zinc finger of WT and Mutants during different times of the MDs *彩图见电子版(http://swxxx.alljournals.cn/ch/index.aspx)(2017年第2期DOI:10.3969/j.issn.1672-5565.20160715001)。 |
通过Pymol可视化软件做了10×10-9 s时WT与所有突变体的结构比对,如图 3所示,其中图 3(a)为WT、图 3(b)、(c)为ZBDⅠ突变体(C143S、C201S)与野生型结构比对,图 3(d)、(e)为ZBDⅡ突变体(C185S、C162S)与野生型结构比对,绿色*为模拟0×10-9 s时的构象,红色*为模拟10×10-9s时的构象,模拟10×10-9 s过程中每隔500×10-12 s所有构象进行比对,由比对结果可知,ZBDⅠ突变体只是锌指结构末端,位于ZBDⅡ区域发生偏移,而ZBDⅡ突变导致整体锌指结构明显受到影响。那么突变体结构变化后是否会影响与Hsp70的合作的功能呢?本文通过ClusterPro 2.0 Server在线服务器对野生型及C143S、C162S突变体进行对接[30]。
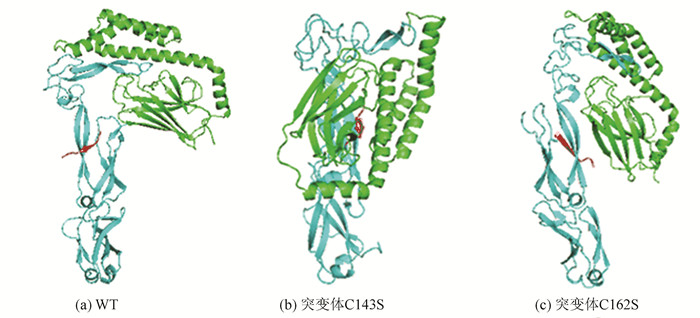
2.5 Ydj1p与Hsp70对接结果分析采用蛋白晶体结构数据库中的Ydj1(PDB code:1NLT)为对接模型, 以Ydj1为模型利用Swiss-Pdb Viewer进行同源建模构建C143S、C162S突变体模型,考虑到大肠杆菌的HscA与Ssa1的序列相似性较高,采用大肠杆菌的C末端为对接模型(PDB code:1U00),首先对WT、C143S、C162S进行分子动力学模拟使分子结构处于稳定,以此为初始模型使用ClusterPro 2.0 Server分别做了Ydj1p以及突变体与HscA的对接。Hsp40与Hsp70分子伴侣相互作用,分子伴侣Ydj1p暂时与未折叠的多肽迅速结合,Ydj1p的J结构域通过刺激Hsp70-ATP酶结构域促进ATP酶水解,使Hsp70的盖子结构打开,然后底物转移到Hsp70的底物结合口袋中,Ydj1p解离,核苷酸交换因子与Hsp70复合体结合,引起ADP释放盖子关闭。Hsp70进一步对底物进行折叠。Ydj1p与大肠杆菌中HscAC-末端相互作用模型的结果有很多种,依据相关文献报道选择了最优的对接模型,如图 4所示,图 4中红色*片段为多肽底物,通过Hsp40将其转移到Hsp70底物结合部位,通过对图 4分析,Ydj1单体都与Hsp70-SBD结构域结合的可能,但考虑到Ydj1在酵母细胞中是以二聚体形式存在的,C143S的这种对接形式由于空间位阻的影响会阻碍与Hsp70的结合,底物传递到Hsp70的效率会降低。但不会影响Ydj1p的生物学功能,所以抑制变性蛋白的聚集也不会受到影响。而突变体C162S模拟之后锌指结构偏移程度较大,在锌指结构与Hsp70碳末端结合时C162S的锌指结构反向与Hsp70结合,使Ydj1p底物结合口袋背离Hsp70,因此,底物的传递会受到影响。C162S与Hsp70这种对接方式排名靠后,能结合的对接模型数量较少,可能现实中并不存在,锌指Ⅱ突变阻碍了与Hsp70的进一步合作。
|
图 4 WT及突变体C143S,C162S与Hsp70对接,底物结合模式图(红色部分为底物*) Figure 4 The docking models among WT and C143S, C162S with Hsp70, substrate(the red part is substrate) *彩图见电子版(http://swxxx.alljournals.cn/ch/index.aspx)(2017年第2期DOI:10.3969/j.issn.1672-5565.20160715001)。 |
对WT和突变体的对接模型进行了分析,也只是从结果上证实确实结构的变化可能影响了底物的传递的原因,如果更进一步对Hsp40与Hsp70之间的相互作用进行研究,推测出Hsp40(Ydj1p)与Hsp70(1U00) 相互作用的第2位点,那对两者之间的研究将更进一步。
3 结论1) 突变体C143S,C162S与底物之间的氢键数量、疏水数量以及总能量略少于野生型,与多肽底物的结合能力降低约10%。而突变体C185S,C201S与底物之间的疏水、氢键数量以及总能量与野生型相似,对多肽底物的结合能力影响不大,总体来讲不影响底物结合。
2) ZBDⅠ单点突变有增加结构的灵活性的可能性,但可能不显著影响整体ZFLR结构。ZBDⅡ构象的改变对整个锌指区域构象造成了大的影响,因此,该ZBDⅡ的突变是有可能破坏ZFLR整体结构并使其不起作用的。
3) 野生型及C143S、C162S突变体都能与Hsp70作用,由于空间位阻以及构象变化的原因,Ydj1p与Hsp70的合作受到影响,底物传递效率降低。
| [1] |
LINKE K, WOLFRAM T, BUSSEME J, et al. The roles of the two Zinc binding sites in DnaJ[J]. The Journal of Biolo-gical Chemistry, 2003, 278(45): 44457–66.
DOI:10.1074/jbc.M307491200( 0) 0)
|
| [2] |
SCOTT W R P, HVNENBERGER P H, TIRONI I G, et al. The GROMOS biomolecular simulation program package[J]. Journal of Physical Chemistry A, 1999, 103(19): 3596–3607.
DOI:10.1021/jp984217f( 0) 0)
|
| [3] |
KARPLUS M, PETSKO G A. Molecular dynamics simulations in biology[J]. Nature, 1990, 347(6294): 631–639.
DOI:10.1038/347631a0( 0) 0)
|
| [4] |
VJESTICA A, ZHANG Dan, LIU Jianhua, et al. Hsp70-Hsp40 chaperone complex functions in controlling polarized growth by repressing Hsf1-Driven heat stress-associated transcription[J]. Plos Genetics, 2013, 9(10): e1003886.
DOI:10.1371/journal.pgen.1003886( 0) 0)
|
| [5] |
FAN Chunyang, REN Hongyu, LEE P, et al. The type Ⅰ Hsp40 zinc finger-like region is required for Hsp70 to capture non-native polypeptides from Ydj1*[J]. Journal of Biological Chemistry, 2005, 280(1): 695–702.
DOI:10.1074/jbc.M410645200( 0) 0)
|
| [6] |
FLORIN E L, MOY V T, GAUB H E. Adhesion forces between individual ligand-receptor pairs[J]. Science, 1994, 264(5157): 415–417.
DOI:10.1126/science.8153628( 0) 0)
|
| [7] |
XU Yechun, SHEN Jianhua, LUO Xiaomin, et al. Steered molecular dynamics simulations of protein-ligand interactions[J]. Science in China Series B: Chemistry, 2004, 47(5): 355–366.
DOI:10.1007/BF02990895( 0) 0)
|
| [8] |
CYR D M. Cooperation of the molecular chaperone Ydj1 with specific Hsp70 homologs to suppress protein aggregation[J]. FEBS Letters, 1995, 359(2/3): 129–132.
DOI:10.1016/0014-5793(95)00024-4( 0) 0)
|
| [9] |
SILVA J C, BORGES J C, CYR D M, et al. Central domain deletions affect the SAXS solution structure and function of Yeast Hsp40 proteins Sis1 and Ydj1[J]. BMC Structural Biology, 2011, 11(1): 3348–3353.
DOI:10.1186/1472-6807-11-40( 0) 0)
|
| [10] |
SUMMERS D W, DOUGLAS P M, CYR D M. Prion pro-pagation by Hsp40 molecular chaperones[J]. Prion, 2009, 3(2): 59–64.
DOI:10.4161/pri.3.2.9062( 0) 0)
|
| [11] |
CHAARI A, HOARAU-VÉCHOT J, LADJIMI M. Applying chaperones to protein-misfolding disorders: Molecular chaperones against α-synuclein in Parkinson's disease[J]. International Journal of Biological Macromolecules, 2013, 60: 196–205.
DOI:10.1016/j.ijbiomac.2013.05.032( 0) 0)
|
| [12] |
MASISON D C, KIRKLAND P A, SHARMA D. Influence of Hsp70s and their regulators on yeast prion propagation[J]. Prion, 2009, 3(2): 65–73.
DOI:10.4161/pri.3.2.9134( 0) 0)
|
| [13] |
KIRKLAND P A, REIDY M, MASISON D C. Functions of Yeast Hsp40 chaperone sis1p dispensable for prion propagation but important for prion curing and protection from prion toxicity[J]. Genetics, 2011, 188(3): 565–577.
DOI:10.1534/genetics.111.129460( 0) 0)
|
| [14] |
LANGER T, LU Chi, ECHOLS H, et al. Successive action of DnaK, DnaJ and GroEL along the pathway of chaperone-mediated protein folding[J]. Nature, 1992, 356(6371): 683–689.
DOI:10.1038/356683a0( 0) 0)
|
| [15] |
WU Yunkun, LI Jingzhi, JIN Zhongmin, et al. The crystal structure of the C-terminal fragment of Yeast Hsp40 Ydj1 reveals novel dimerization motif for Hsp40[J]. Journal of Molecular Biology, 2005, 346(4): 1005–1011.
DOI:10.1016/j.jmb.2004.12.040( 0) 0)
|
| [16] |
王浩, 徐利楠, 孙玉娜, 等. Ydj1p二聚体中β14~β15与domain-Ⅲ分离的拉伸分子动力学模拟研究[J]. 生物信息学, 2013, 11(3): 167–171.
WANG Hao, XU Li'nan, SUN Yuna, et al. Steered molecular dynamics simulation of the binding of β14-β15 and domain-Ⅲ in Ydj1p dimer[J]. Chinese Journal of Bioinformatics, 2013, 11(3): 167–171. DOI:10.3969/j.issn.1672-5565.2013.03.02(  0) 0)
|
| [17] |
ZHANG Jilong, ZHENG Qingchuan, ZHANG Hongxing. Unbinding of glucose from human pulmonary surfactant protein D studied by steered molecular dynamics simulations[J]. Chemical Physics Letters, 2010, 484(4/5/6): 338–343.
DOI:10.1016/j.cplett.2009.12.022( 0) 0)
|
| [18] |
SHEN Manli, GUAN Jing, XU Linan, et al. Steered molecular dynamics simulations on the binding of the appendant structure and helix-B2 in domain-swapped human cystatin C dimmer[J]. Journal of Biomolecular Structure & Dynamics, 2012, 30(6): 652–661.
DOI:10.1080/07391102.2012.689698( 0) 0)
|
| [19] |
HESS B, KUTZNER C, VAN DER SPOEL D, et al. GROMACS 4: Algorithms for highly efficient, load-balanced, and scalable molecular simulation[J]. Journal of Chemical Theory and Computation, 2008, 4(3): 435–447.
DOI:10.1021/ct700301q( 0) 0)
|
| [20] |
PRONK S, PÁLL S, SCHULZ R, et al. GROMACS 4.5: a high-throughput and highly parallel open source molecular simulation toolkit[J]. Bioinformatics, 2013, 29(7): 845–854.
DOI:10.1093/bioinformatics/btt055( 0) 0)
|
| [21] |
LINKE K, WOLFRAM T, BUSSEMER J, et al. The roles of the two zinc binding sites in DnaJ[J]. Journal of Biological Chemistry, 2003, 278(45): 44457–44466.
DOI:10.1074/jbc.M307491200( 0) 0)
|
| [22] |
HESS B, BEKKER H, BERENDSEN H J C, et al. LINCS: A linear constraint solver for molecular simulations[J]. Journal of Computational Chemistry, 1997, 18(12): 1463–1472.
DOI:10.1002/(SICI)1096-987X(199709)18:12<1463::AID-JCC4>3.0.CO;2-H( 0) 0)
|
| [23] |
DARDEN T, YORK D, PEDERSEN L. Particle mesh Ewald: An N·log(N) method for Ewald sums in large systems[J]. Journal of Chemical Physics, 1993, 98: 10089–10092.
DOI:10.1063/1.464397( 0) 0)
|
| [24] |
ESSMANN U, PERERA L, BERKOWITZ M L, et al. A smooth particle mesh Ewald potential[J]. Journal of Chemical Physics, 1995, 103(19): 8577–8593.
DOI:10.1063/1.470117( 0) 0)
|
| [25] |
LIN E S, YANG Y S, YANG J M. Modeling the binding and inhibition mechanism of nucleotides and sulfotransferase using molecular docking[J]. Journal of the Chinese Chemical Society, 2003, 50(3B): 655–663.
DOI:10.1002/jccs.200300095( 0) 0)
|
| [26] |
SHEN Lingling, SHEN Jianhua, LUO Xiaomin, et al. Molecular dynamics simulation on the binding of NNRTI to HIV-1 RT[J]. Biophysical Journal, 2003, 84(6): 3547–3563.
DOI:10.1016/S0006-3495(03)75088-7( 0) 0)
|
| [27] |
CUÉLLAR J, PERALES-CALVO J, MUGA A, et al. Structural insights into the chaperone activity of the 40-kDa heat Shock protein DnaJ binding and remodeling of a native substrate[J]. Journal of Biological Chemistry, 2013, 288(21): 15065–15074.
DOI:10.1074/jbc.M112.430595( 0) 0)
|
| [28] |
ZHURAVLEVA A, CLERICO E M, GIERASCH L M. An interdomain energetic Tug-of-War creates the allosterically active state in Hsp70 molecular chaperones[J]. Cell, 2012, 151(6): 1296–1307.
DOI:10.1016/j.cell.2012.11.002( 0) 0)
|
| [29] |
ZHURAVLEVA A, GIERASCH L M. Substrate-binding domain conformational dynamics mediate Hsp70 allostery[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(22): E2865–E2873.
DOI:10.1073/pnas.1506692112( 0) 0)
|
| [30] |
周雷, 徐利楠, 周小红, 等. Hsc70-Auxilin复合物的拉伸分子动力学模拟[J]. 生物信息学, 2014, 12(2): 110–116.
ZHOU Lei, XU Li'nan, ZHOU Xiaohong, et al. Steered molecular dynamics simulations of Hsc70-Auxilin interactions[J]. Chinese Journal of Bioinformatics, 2014, 12(2): 110–116. DOI:10.3969/j.issn.1672-5565.2014.02.06(  0) 0)
|
 2017, Vol. 15
2017, Vol. 15


